修回日期: 2021-06-16
接受日期: 2021-07-07
在线出版日期: 2021-08-28
乙型肝炎病毒(hepatitis B virus, HBV)体外感染模型是开展HBV生命周期、致病机理、药物筛选等研究的基础. 随着临床抗HBV治疗进入"功能性治愈""完全治愈"新时代的趋势, 对能稳定模拟共价闭合环状DNA分子(covalently closed circular DNA, cccDNA)转录机制, 乙型肝炎病毒X蛋白(hepatitis B virus X protein, HBx)致病机理的细胞模型的需求日益迫切. HBV1.3倍(1.3-fold HBV)全基因组包含了全部HBV生物信息, 能依赖自身启动子启动转录过程, 支持cccDNA形成和完整病毒复制, 最接近于HBV体内感染状态下的生命周期. 慢病毒转染是一种以慢病毒为载体, 将外源分子如DNA, RNA等导入真核细胞的技术, 可形成稳定转染.
采用慢病毒转染技术构建HBV 1.3倍基因组HepG2稳转细胞模型, 筛选并鉴定出能稳定、高效表达HBV生物标志物的优势单克隆株.
构建含1.3-fold HBV基因组信息的慢病毒质粒并包装慢病毒; 以最佳感染复数(MOI)值将1.3-fold HBV慢病毒液侵染靶细胞HepG2, 以抗生素杀稻瘟菌素(blasticidin, BSD)最佳筛选剂量筛选出稳定整合1.3-fold HBV基因的HepG2细胞系(1.3-fold HBV-HepG2), 继而采用PCR法鉴定该细胞模型中的HBV DNA序列. 将1.3-fold HBV-HepG2稳转细胞进行培养并挑选出9株候选阳性单克隆, 通过测序各单克隆细胞的侧翼序列, 以确定9株候选阳性单克隆相应的基因组在HepG2细胞基因组中的插入位置, 再根据各候选单克隆株表达HBsAg、HBeAg的水平, 筛选出最优势单克隆株. 将该优势单克隆株与HepG2.2.15细胞株分别持续培养20代, 比较这两种细胞株表达HBsAg、HBeAg、HBx、cccDNA、HBV DNA等标志物的水平及稳定性.
(1)将慢病毒pLenti-BSD-1.3-fold HBV以MOI = 30的参数侵染HepG2细胞, 72 h后加入BSD抗生素(终浓度1 μg/mL)进行筛选, 连续培养15-20 d后, 获得1.3-fold HBV-HepG2稳转细胞系, PCR鉴定出HBV DNA序列; (2)在挑选出的9株候选阳性单克隆中, 编号A14的细胞株(命名为HepGA14)的HBsAg、HBeAg表达水平最高, 分别为24.28 IU/mL、39.62 NCU/mL, 其插入HepG2基因组位置为1:166461695-166461715, 确定为优势单克隆株; (3)与HepG2.2.15细胞株比较, HepGA14优势单克隆株1-20代能稳定、高表达HBsAg、HBeAg、HBx、HBV DNA、cccDNA, 差异均具有统计学意义(P<0.05).
成功构建并筛选出1.3-fold HBV基因组HepG2稳转细胞模型优势单克隆株HepGA14, 这为后续开展HBV-宿主关系、发病机制及药物筛选等奠定良好的基础.
核心提要: 成功构建并筛选出乙型肝炎病毒(hepatitis B virus, HBV)1.3倍基因组HepG2稳转细胞模型优势单克隆株HepGA14, 其能稳定、高效地表达共价闭合环状DNA分子(covalently closed circular DNA, cccDNA)、乙型肝炎病毒X蛋白(hepatitis B virus X protein, HBx)、HBVDNA、HBsAg、HBeAg等生物标志物, 有望在研制乙肝"治愈"药物及探索HBV相关疾病发病机制等领域发挥积极作用.
引文著录: 邱华, 林栋毅, 李锦源. HBV 1.3倍基因组HepG2稳转细胞模型优势单克隆株的筛选及鉴定. 世界华人消化杂志 2021; 29(16): 934-944
Revised: June 16, 2021
Accepted: July 7, 2021
Published online: August 28, 2021
Hepatitis B virus (HBV) infection model in vitro is the basis for studying HBV life cycle and pathogenesis and for drug screening. With the clinical anti-HBV therapy entering the new trend of "functional cure" and "complete cure", there is an urgent need for cell models that can stably simulate the transcription mechanism of covalently closed circular DNA (cccDNA) and the role of hepatitis B virus X protein (HBx). The 1.3-fold HBV genome contains all the biological information of HBV. It can start the transcription process by its own promoter, support the formation of cccDNA, and complete viral replication, which is closest to the life cycle of HBV in vivo. Lentivirus transfection is a technology that takes lentivirus as vector and introduces foreign molecules such as DNA and RNA into eukaryotic cells, which can form stable transfection.
To construct a HepG2 cell model with 1.3-fold HBV genome by lentivirus transfection technology, and to screen and identify the dominant monoclonal strain that can stably and efficiently express HBV biomarkers.
A lentiviral plasmid containing 1.3-fold HBV genome information was constructed, and the recombinant lentivirus culture was used to infect HepG2 cells at the optimal multiplicity of infection (MOI). Blasticidin (BSD) was used to select HepG2 cell strains (1.3-fold HBV-HepG2) stably integrating the 1.3-fold HBV genome, and then the HBV in the cell model was identified by PCR. HepG2 cells stably carrying the 1.3-fold HBV genome were cultured and nine candidate positive monoclones were selected. The flanking sequences of each monoclonal cell were sequenced to determine the insertion position of the corresponding HBV genome in the genome of HepG2 cells. The most dominant monoclones were selected according to the expression levels of HBsAg and HBeAg. The expression levels and stability of HBsAg, HBeAg, HBx, cccDNA, and HBV DNA in HepG2 cells stably carrying the 1.3-fold HBV genome were compared.
The lentiviral plasmid plenti-bsd-1.3-fold HBV was used to infect HepG2 cells at an MOI of 30. After 72 h, BSD (final concentration 1 μg/mL) was added for screening. After 15-20 d of continuous culture, stable 1.3-fold HBV-HepG2 cell line was obtained. HBV DNA sequence was then identified by PCR. Among the nine selected candidate positive monoclones, A14, in which the 1.3-fold HBV genome was inserted into the HepG2 genome at 1:166461695-166461715 (named HepGA14), had the highest expression levels of HBsAg and HBeAg at 24.28 IU/mL and 39.62 NCU/mL, respectively. HepGA14 can stably and highly express HBV biomarkers. Compared with HepG2.2.15 cell line, the expression levels of HBx and cccDNA in Hepga14 dominant monoclonal line in 1-20 passages were significantly higher (P < 0.05).
We have successfully constructed and screened HepGA14, a dominant monoclonal HepG cell strain with HBV 1.3-fold genome, which lays a good foundation for further research of HBV-host relationship and pathogenesis as well as for drug screening.
- Citation: Qiu H, Lin DY, Li JY. Screening and identification of dominant monoclonal HepG2 cell strain with 1.3-fold HBV genome. Shijie Huaren Xiaohua Zazhi 2021; 29(16): 934-944
- URL: https://www.wjgnet.com/1009-3079/full/v29/i16/934.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v29.i16.934
乙型肝炎病毒(hepatitis B virus, HBV)感染仍是全球重要的公共卫生问题, 每年将近100万人死于HBV感染相关的肝硬化、肝癌及肝衰竭等疾病, 而中国作为世界上HBV感染者最多的国家, 防控压力尤为沉重. HBV体外感染与复制模型是研究HBV生活史、致病机理、传染途径的重要工具, 并在HBV相关肝硬化和肝癌的发病机制探索、治疗靶点发现和候选药物筛选等方面发挥关键性作用[1]. 因此, 构建一种高效、稳定、经济、可重复性的HBV体外细胞模型具有重要的意义. 而对于HBV体外复制模型来说, 目前主要是通过各种病毒载体递送系统, 将HBV基因组转染到肝癌细胞系的Huh7和HepG2细胞内构建而成, 如HepG2.2.15、HepAD38、HepDE19等. 本课题组前期采用重组腺病毒转染技术构建了HBV全基因组1.3倍体HepG2瞬染细胞模型, 该模型在感染第2天即可分别在上清液和细胞内中检测出HBV DNA、共价闭合环状DNA分子(covalently closed circular DNA, cccDNA)、HBsAg及HBeAg等标志物的表达, 在4-6 d达到峰值水平, 7 d之后逐渐下降[2]. 但由于该模型HBV DNA不是整合到HepG2细胞的基因组中, 因此其表达HBV生物标志物是不稳定的, 且随着时间延长其表达活性会自然衰减, 每次使用时都需重新构建. 有鉴于此, 本项目组拟采用重组慢病毒转染技术, 构建并筛选出HBV1.3倍(1.3-fold HBV)基因组HepG2稳转细胞模型的优势单克隆株, 以更好服务于HBV感染相关疾病的发病机制及治疗药物研究.
HepG2细胞、HepG2.2.15细胞、293A细胞、293T细胞购自中国科学院上海细胞生物研究所; DH5α感受态细胞购自北京全式金生物技术有限公司; 包装质粒Packaging Mix购自江苏百奥生物技术有限公司; 0.05% Trypsin购自上海玉博生物科技有限公司; DMEM培养基购自美国HyClone公司; 慢病毒骨架载体pLenti-BSD载体购自上海诺百生物科技有限公司; 胎牛血清购自法国Biowest公司; Opti-MEM 购自美国GIBCO公司; Trizol Reagent、荧光染料SYBR Green Ⅰ、oligo dT/Random primer/特异性引物、逆转录酶SuperScriptⅢReverse Transcriptase、Platinum Taq DNA Polymerase、100mM dNTPs、DEPC H2O购自美国invitrogen公司; Rnase Inhibitor购自加拿大Fermentas公司; HBsAg、HBeAg检测试剂盒购自郑州安图生物股份有限公司(批号: 20191103). CO2培养箱、生物安全柜、高速冷冻离心机购自上海力康生物医疗科技控股有限公司; 荧光显微镜购自日本Olympus公司; 荧光定量PCR仪购自美国Bio Rad公司. 基因序列、PCR引物合成、测序由上海诺百生物科技有限公司完成.
本研究严格遵循《中华人民共和国生物安全法》中"病原微生物实验室生物安全"的相关法律规定进行管理与操作.
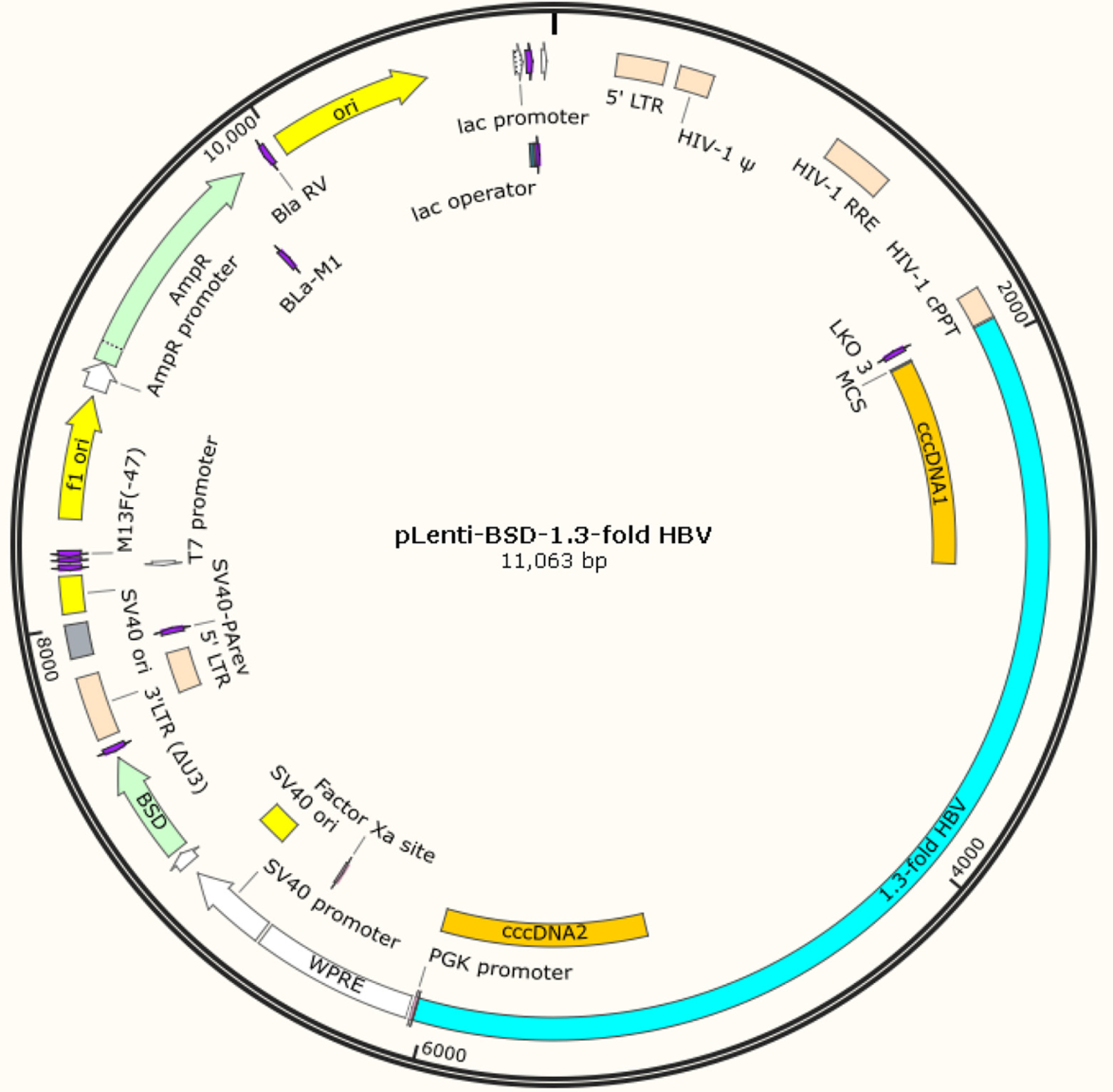
1.2.1 1.3-fold HBV基因组慢病毒质粒构建: (1)1.3-fold HBV基因组序列扩增: 通过NCBI检索, 查出HBV全基因组序列(NC_003977.2), 并确定1.3-fold HBV基因组序列(图1). 按照序列设计oligo, 全基因合成1.3-fold HBV的序列, 将1.3-fold HBV序列插入到慢病毒骨架载体pLenti-BSD的ClaI和MluI切点之间, 获得慢病毒载体pLenti-BSD-1.3-fold HBV(图2). 根据实验要求设计In-fusion引物, 引物5'端引物重组必需的序列, 上游引物(F): AATTCAAAATTTTATCGATcTCGAGttcaatctaagcaggct, 下游引物(R): TTACTAACCGGTACGCGTgcggccGCgatctcgtactgaagg. 扩增体系: 10×PCR Buffer 5 μL、dNTP 5 μL、Mg2+ 3 μL、Primer F/R 1.5 μL、模板1 μL、KOD 1 μL, 加水至50 μL. 扩增条件: 94 ℃ 15 s、58 ℃ 30 s, 28个循环. 扩增产物通过0.8%琼脂糖凝胶电泳鉴定, PCR产物记为 1.3-fold HBV(PCR); (2)1.3-fold HBV PCR产物回收以及In-fusion连接: pLenti-BSD质粒在37 ℃进行双酶切, 酶切条件: pLenti-BSD 2 μg、10×Buffer T 5 μL、SalI 2 μL、BglII 2 μL, 加水至50 μL. 酶切产物经0.8%琼脂糖凝胶电泳, 切胶回收, 回收产物记为pLenti-BSD(ClaI/MluI). pLenti-BSD(ClaI/MluI)和1.3-fold HBV DNA(PCR)通过In-fusion连接, 连接产物记为pLenti-BSD-1.3-fold HBV. 连接体系: 5×CE II Buffer 4 μL、pLenti-BSD(ClaI/MluI)50 ng、HBV DNA 1.3P(PCR)150 ng、Exnase® Ⅱ 2 μL, 加入至20 μL, 混匀. 连接条件: 37 ℃反应30 min后, 立即将反应管置于冰水浴中冷却; (3)pLenti-BSD-1.3-fold HBV重组质粒鉴定: pLenti-BSD-1.3-fold HBV连接产物转化DH5α感受态细胞, 37 ℃过夜培养, 挑取阳性克隆经PCR鉴定, 根据1.3-fold HBV序列设计特异性引物(F: CTCGTGGTGGACTTCTCTC; R: CAGCAGGATGAAGAGGAA)进行PCR扩增, 扩增产物大小为166bp; 同时设置阴性对照(dd H2O作为模板)和阳性对照(PCR扩增1.3-fold HBV); PCR反应体系和反应条件参照1.2.1.1; (4)pLenti-BSD-1.3-fold HBV慢病毒包装及滴度测定:将慢病毒载体pLenti-BSD-1.3-fold HBV和包装质粒(packaging mix)共转染293T细胞, 包装病毒, 收集病毒原液, 超速离心浓缩, 浓缩后的慢病毒侵染293A细胞, 48 h后qPCR测定慢病毒活性滴度[2]. qPCR反应体系: 10×PCR buffer 1 μL、Mg2+ 0.4 μL、dNTPs 0.2 μL、Primer F/R 0.5 μL、Probe(FAM)0.25 μL、Taq酶0.1 μL、Template 1 μL、Total volume 10 μL; qPCR反应条件: 95 ℃10 s、60 ℃30 s, 40个循环.
1.2.2 HBV 1.3倍基因组慢病毒质粒侵染HepG2细胞及筛选: (1)慢病毒载体pLenti-BSD-1.3-fold HBV侵染HepG2最佳MOI值: 培养靶细胞HepG2至状态良好, 处于对数生长期; 细胞用0.05%胰酶消化, 用含8 μg/mL polybrene的完全培养液重悬细胞, 细胞计数后, 按照每孔第2天细胞密度60%-70%的细胞数量进行接种于96孔板的每个孔中, 设置1个复孔(病毒滴度设1复孔); 培养过夜, 观察细胞生长状态. 进行GFP荧光慢病毒侵染实验; 取出已知滴度的阴性对照病毒液, 冰上融化, 根据设计的MOI测定范围, 以及细胞接种量, 计算每孔所需要病毒量, 按照不同感染复数(multiplication of infection, MOI), 慢病毒MOI = 0、2、5、10、30、60、120、240, 向每孔加入病毒液. 在37 ℃, 5%的CO2培养箱培养6 h后, 更换为完全培养液后继续培养至24 h-96 h; 荧光显微镜下观察细胞生长情况和荧光比率, 确定最佳MOI, 并拍摄每个MOI下的白光和荧光视野下的照片; (2)1.3-fold HBV DNA慢病毒侵染HepG2细胞及抗生素筛选:培养靶细胞HepG2至状态良好, 处于对数生长期; 细胞用0.05%胰酶消化, 用完全培养基悬浮成单细胞悬液, 细胞计数后, 按照5×105个细胞接种于12孔板的每个孔中; 感染培养基预先配制完全培养基+8 μg/mL ploybrene; 培养孔换上新配制的感染培养基, 添加1 mL培养基, 按照MOI测定值的病毒液侵染靶细胞, 混匀后置于5%的CO2的培养箱中培养4-6 h, 更换新鲜的完全培养基, 继续培养24 h-72 h, 根据细胞状态进行抗生素筛选(梯度分为0, 2 μg/mL, 4 μg/mL, 6 μg/mL, 8 μg/mL, 10 μg/mL); 完全培养基中加入合适量的抗生素进行筛选, 筛选期间每天观察细胞状态, 抗生素筛选周期视细胞状态而定, 筛选成功的稳转细胞按照常规培养并冻存; (3)1.3-fold HBV-HepG2稳转细胞的PCR鉴定: 抽提1.3-fold HBV-HepG2稳转细胞和HepG2细胞的基因组DNA, 根据1.3-fold HBV序列设计特异性引物(F: CTCGTGGTGGACTTCTCTC; R: CAGCAGGATGAAGAGGAA)进行PCR扩增, 扩增产物大小为166 bp; PCR反应体系和反应条件参照1.2.1.1.
1.2.3 1.3-fold HBV-HepG2稳转细胞优势单克隆株的筛选及鉴定: (1)1.3-fold HBV-HepG2稳转细胞候选阳性单克隆的筛选: ①铺板培养: 取1.3-fold HBV-HepG2稳转细胞株消化重悬计数, 以每孔1个细胞量调整细胞密度, 接种到96孔板中, 2周后显微镜下观察已长出的单克隆细胞团, 收集单克隆细胞上清并消化扩培到24孔板中, 24孔板长满再扩培到6孔板中, 依次扩大培养. 再取扩增后的稳转单克隆株分批次铺板培养并挑选, 第一批单克隆依次命名为A1、A2、A3等, 第二批单克隆依次命名为B1、B2、B3等, 依此类推. ②初筛: 所有单克隆株均收集细胞上清, 利用HBsAg和HBeAg检测试剂盒, 分别检测HBsAg、HBeAg表达水平, 初步筛选出阳性单克隆株, 进行后续实验. ③复筛: 源于初筛的阳性单克隆株以同一细胞数铺板, 培养3 d后收集细胞上清, 再根据HBsAg、HBeAg的表达水平, 筛选出9株候选阳性单克隆株, 进入后续靶基因插入位置测序及表达活性等实验; (2)检测9株候选阳性单克隆1.3-fold HBV基因组整合进HepG2基因组位置: 通过测序9株候选阳性单克隆细胞株的侧翼序列, 以确定其相应的基因组插入HepG2细胞基因组的位置. 培养候选的9株阳性单克隆株至1×106以上, 分别抽提基因组DNA; 用XspI对基因组DNA进行酶切, 将酶切后的产物与NdeI切开的pUC57-Amp载体进行连接反应, 针对连接产物, 进行巢式PCR扩增, 第一轮所用的引物F: gttccgcagtatggatcggc, R: cgatcgcccttcccaacagt; 第二轮所用的引物F: gaggagccgaaaaggttcca, R: cagcctgaatggcgaatgg. 针对巢式PCR产物, 进行Sanger测序(使用cagcctgaatggcgaatgg为测序引物)将测序获得的序列信息, 与人基因组数据库进行Blast比对, 可找出相应的基因组位置信息; (3)筛选出1.3-fold HBV-HepG2稳转优势单克隆株: 收集9株候选阳性单克隆株细胞上清, 分别检测上清中HBsAg、HBeAg水平, 筛选出表达量及稳定性最优的单克隆, 作为优势单克隆株, 培养备用; (4)检测优势单克隆株与HepG2.2.15各代HBsAg、HBeAg表达水平与稳定性: 取1.3-fold HBV-HepG2稳转优势单克隆株和HepG2.2.15细胞株, 各连续培养20代, 分别收集第1代、第2代、第3代、第4代、第5代、第10代、第15代、第20代的细胞上清, 检测细胞株上清中HBsAg、HBeAg水平; (5)检测优势单克隆株与HepG2.2.15各代乙型肝炎病毒X蛋白(hepatitis B virus X protein, HBx)、cccDNA、HBV DNA的表达水平及稳定性: 分别收集两种细胞株, 连续培养后, 收集第1代、第2代、第3代、第4代、第5代、第10代、第15代、第20代的细胞, 抽提RNA和DNA; 采用qPCR检测HBx mRNA、cccDNA、HBV DNA的表达水平. HBx的检测引物为F: tctgtgccttctcatctgc, R: tcggtcgttgacattgctg, cccDNA的检测引物为F: ctccccgtctgtgccttct, R: gccccaaagccacccaag, HBV DNA的检测引物为F: ctcgtggtggacttctctc, R: cagcaggatgaagaggaa, 以18S rDNA为内参, 检测引物为引物F: GAATTGACGGAAGGGCACCAC, R: AAGAACGGCCATGCACCACCA. 实验组与对照组中基因的倍数关系以2-ΔΔCt表示 . 参考前期反应体系[2]: 2×SYBR Green PCR Master Mix 10 μL、Primer F/R 1 μL、模板 1 μL、RNase-free H2O 5 μL; PCR反应条件: 95 ℃ 30 s、60 ℃ 30 s、72 ℃ 30 s, 40个循环. 检测过程中以HepG2.2.15细胞为阳性对照. 细胞按50%密度铺6孔板(每组细胞数一致, 约2E+6/孔), 培养3 d后收集细胞并检测.
统计学处理 应用软件SPSS 22.0进行统计分析, 所有实验均重复3次, 符合正态分布的计量资料以均数±标准差(mean±SD)表示, 两组间比较用独立样本t检验. 统计学显著性用aP<0.05或bP<0.01(P>0.05不注)表示.
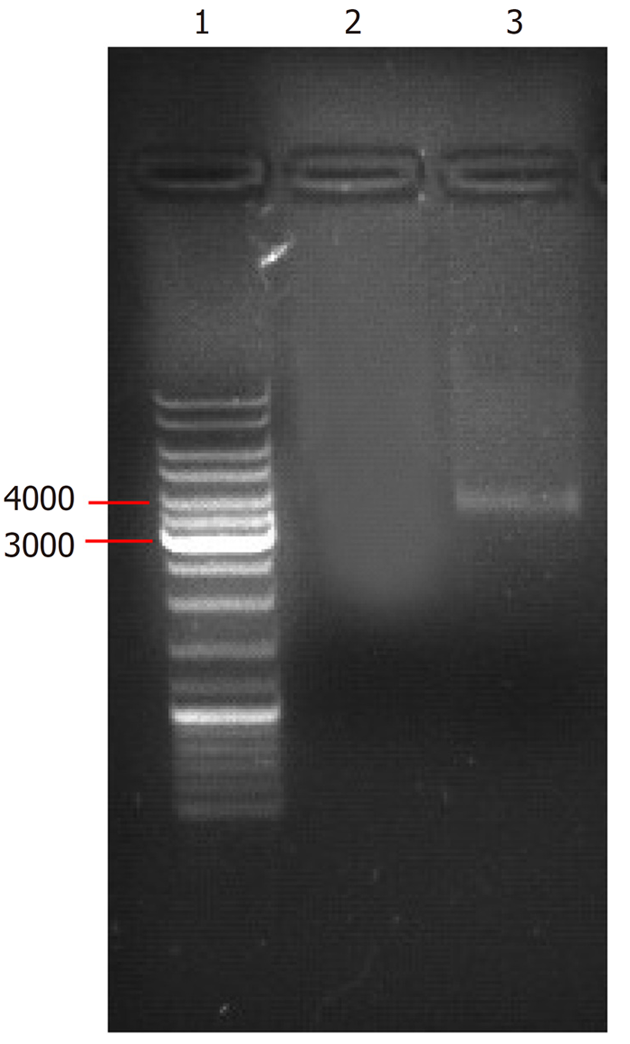
1.3-fold HBV基因组扩增产物经0.8%琼脂糖凝胶电泳, 在4100 bp处可见清晰的条带, 其大小与理论大小一致(图3).
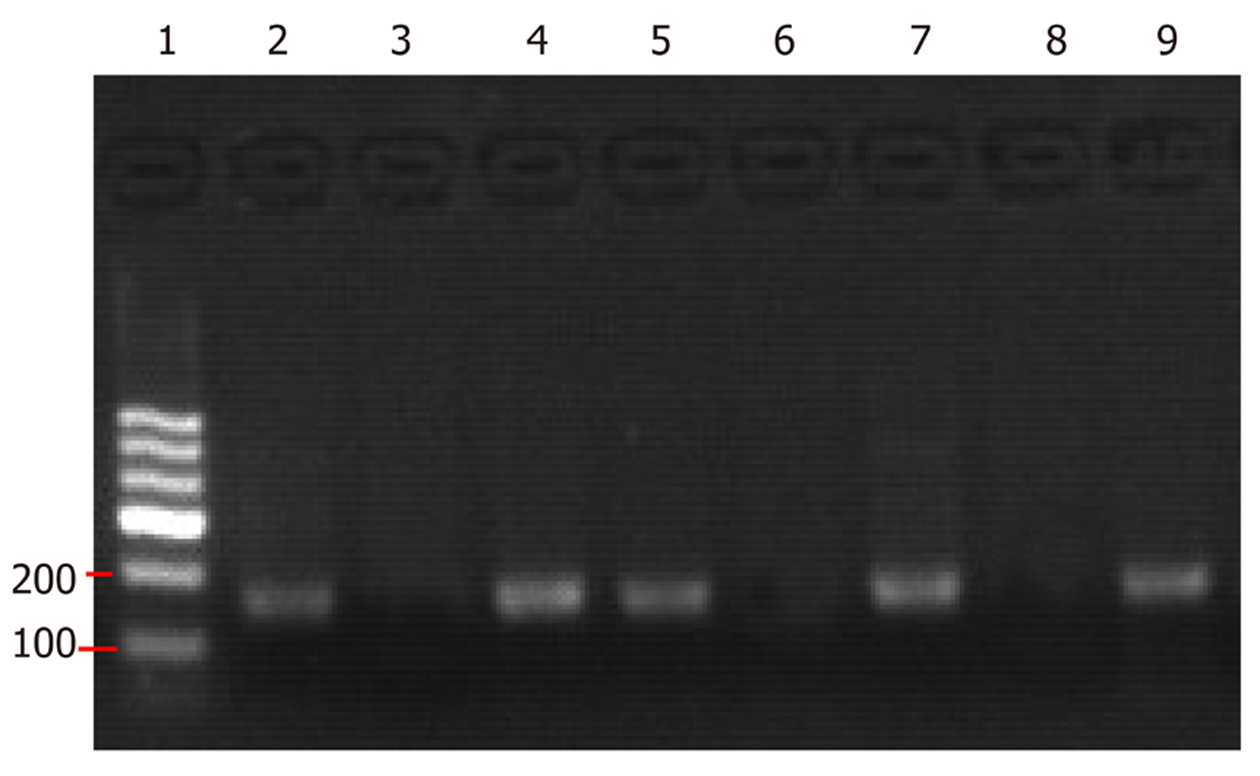
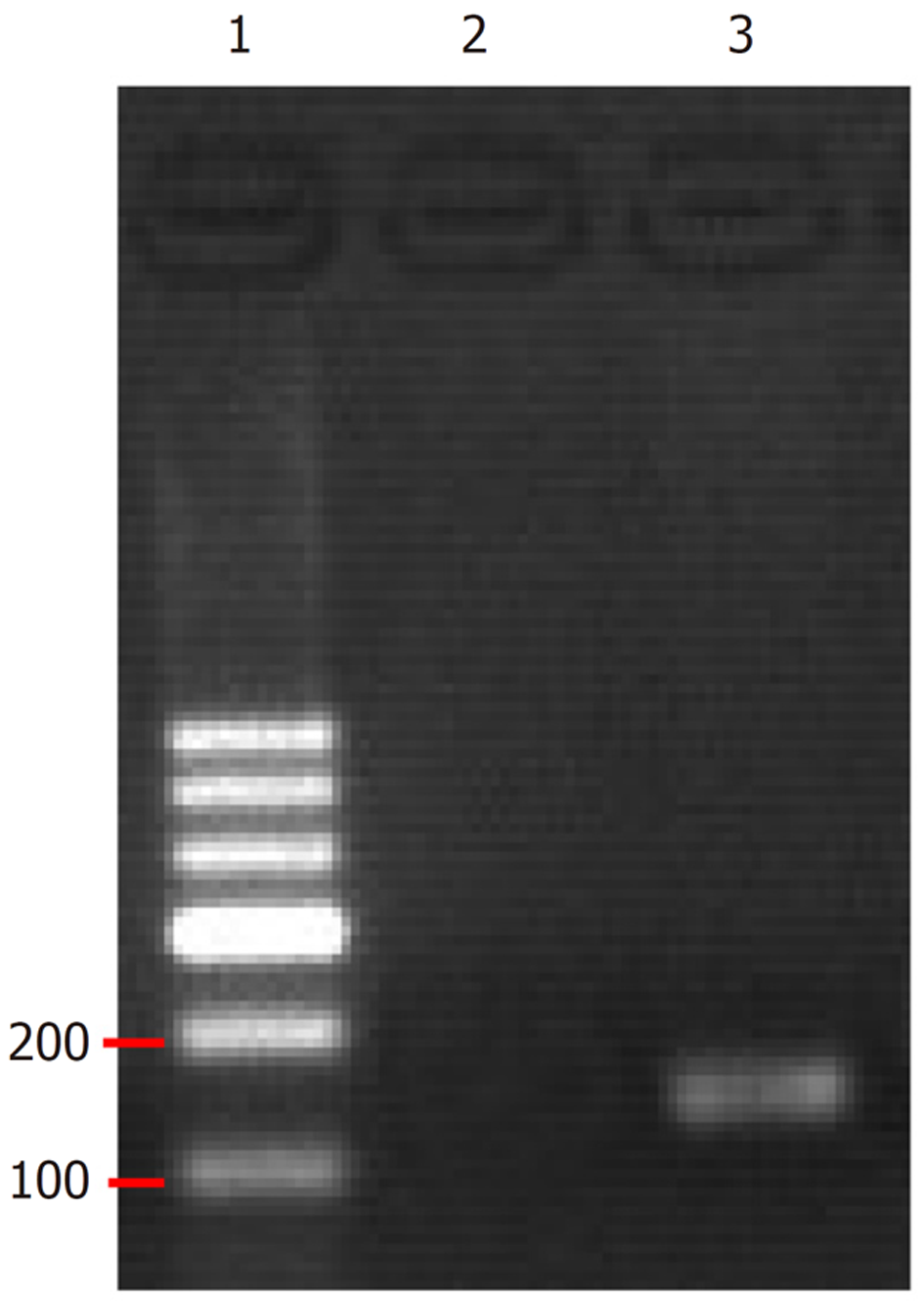
扩增产物经0.8%琼脂糖凝胶电泳鉴定, 鉴定条带约为166 bp, 符合预期条带大小(图4).
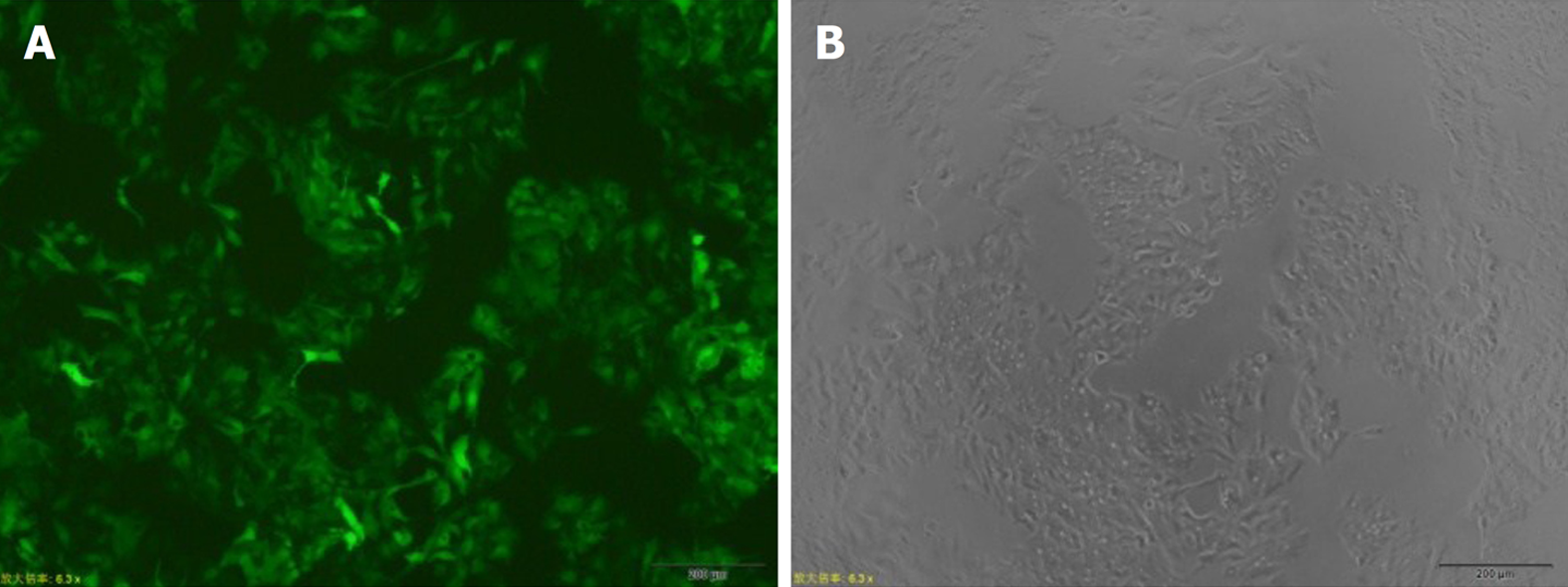
浓缩后的慢病毒侵染HepG2细胞, 48 h后抽提细胞基因组. 测定病毒滴度为1.28×108 TU/mL. MOI = 30时, 综合分析细胞阳性比例达到80%以上, 细胞状态较好, 满足后续实验要求(图5).
HepG2细胞抗生素最佳筛选剂量为1 μg/mL, 筛选时间为10 d. 将慢病毒载体pLenti-BSD-1.3-fold HBV以MOI = 30的参数侵染HepG2细胞, 72 h后加入BSD抗生素(终浓度1 μg/mL)进行筛选, 连续培养15-20 d后, 获得1.3-fold HBV -HepG2稳转细胞系(图6).
从1.3-fold HBV-HepG2稳转细胞中扩增出预期的HBV DNA序列(约166bp), 符合预期(图7).
表1.
| 单克隆编号 | 基因组定位 | 核心序列信息 |
| A4 | 6:950537-950557 | aaaaaaagcacgcaaatttttgtgctgggaaacattagaacttaagactttcttacacttcctctgtggtttagtctttgtggtagtttgggaagatggagtcatataattcttttcacttttgagggatgctgtaga |
| A14 | 1:166461695-166461715 | ttcataaaactgtagtaataaatattcctttctctttggtcttttgaaagagtttgagatgaattggctttaattcttcaaatgtttgatagaacttgccaaagaagtcatttggtcttgggcttttccatcttgggaggt |
| A23 | 13:36028711-36028732 | cttcctcgtttcctttgccttgcttcctcccgccctccaggctgtttcttggcctgggaaatgctcat |
| B4 | 18:65485740-65485760 | caaaggtgttattgattttaatagattttcatatattacaaagtcattgatgtagtctgtgaatttcattataatacatcaaacatgttacaggtatagtcttatactaagttggcaattcatagctaacac |
| B8 | 4:98253463-98253488 | accatctcttccaaagtcttgtgaaataggtatatgtgttttatgagtggtcttttaggcaaaaataaagtacaattgatccttgaacaacacaaggttgaatgtcatgggtccacatatacctggatgggtccacatatacctgagataattgactgcttatgttatcagtaatgtcaacaagtaagctat |
| B17 | 13:21513207-21513227 | tgaaacagtctcttttcgtagatggcattgtctctgtagaaactcccaggaaatctgcaaaaaaaaaatcacaaaacaaaacaaagcttctagaactaataattgattttaggaaggtgactaggtacaagaa |
| B22 | 12:1947817-1947837 | gccccattcctttcattcttcctcaaacatgtttccttcttttgtagtctcgctatgttgctcaggctggtcttgaactcctggcctcaagcaatgctcccaccttggcttcccaaagtgctgggattacaggcgtgagcagccactgtgcccggccaacatggaacctttttaaaaatgttgtactaattgagaccactgtctttttaagtaggataagacaaccgcagaagg |
| B23 | 13:99716126-99716148 | cttgagcccagcctgagcaacatagcgagaccttgtctctaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa |
| B27 | X:81891372-81891396 | aatagacaaatcaatttccaaagtgtctactttggtaagagcagagtatttctcctttcatagtgaaagtaagccaatgtaatccacctgccacgaggta |
由表2可知, 1.3-fold HBV DNA-HepG2稳转细胞单克隆A14的HBsAg、HBeAg表达水平最高, 分别为24.28 IU/mL、39.62 NCU/mL, 确定为优势单克隆株, 并命名为HepGA14, 可用于后续实验(表2).
| 序号 | 样品 | HBsAg浓度(IU/mL) | HBeAg浓度(NCU/mL) |
| 1 | A4 | 2.57 | 36.20 |
| 2 | A14 | 24.28 | 39.62 |
| 3 | A23 | 7.68 | 26.71 |
| 4 | B4 | 14.64 | 10.10 |
| 5 | B8 | 11.21 | 31.16 |
| 6 | B17 | 1.02 | 26.03 |
| 7 | B22 | 6.64 | 33.82 |
| 8 | B23 | 13.02 | 27.76 |
| 9 | B27 | 18.88 | 39.75 |
由表3可知, HepGA14优势克隆株培养的第1-20代HBsAg、HBeAg浓度均较稳定, 而HepG2.2.15株培养的第1-20代HBsAg、HBeAg浓度呈逐渐降低趋势(表3).
| 代数 | HBsAg浓度(IU/mL) | HBeAg浓度(NCU/mL) | ||
| HepGA14 | HepG2.2.15 | HepGA14 | HepG2.2.15 | |
| 第1代 | 24.98 | 29.87 | 36.30 | 26.50 |
| 第2代 | 24.70 | 29.61 | 37.53 | 26.58 |
| 第3代 | 25.01 | 28.83 | 35.47 | 26.65 |
| 第4代 | 24.02 | 29.26 | 36.38 | 26.20 |
| 第5代 | 24.38 | 29.98 | 36.92 | 27.69 |
| 第10代 | 25.49 | 27.61 | 37.93 | 25.02 |
| 第15代 | 26.20 | 24.38 | 38.89 | 22.68 |
| 第20代 | 26.89 | 21.51 | 39.26 | 19.27 |
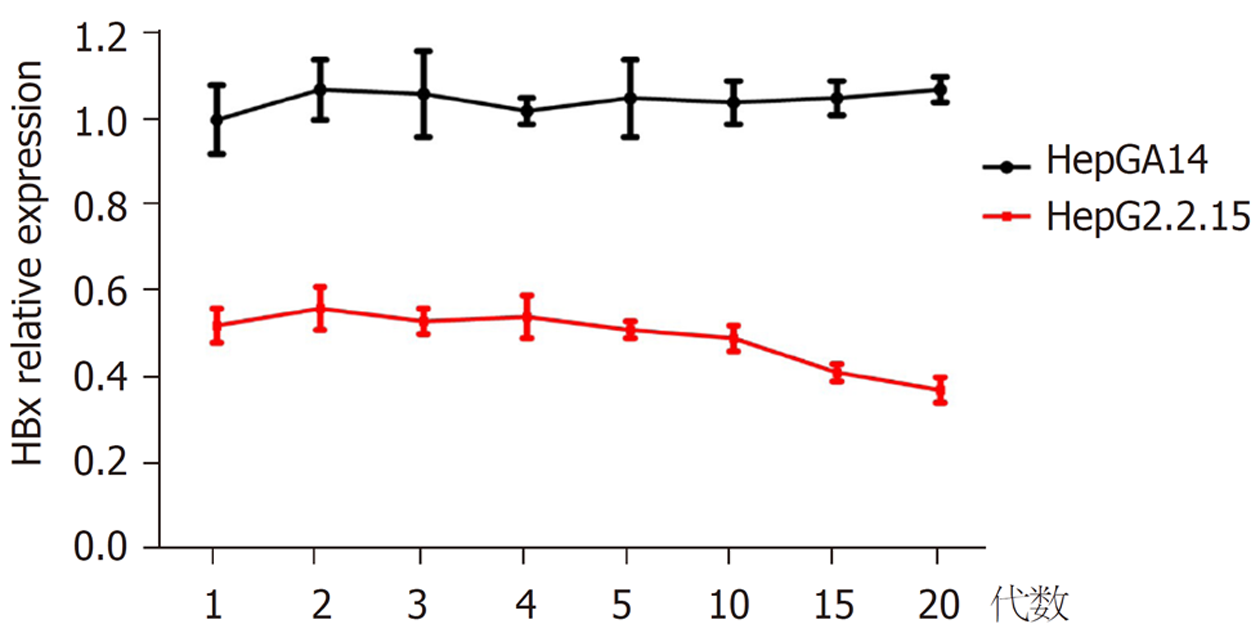
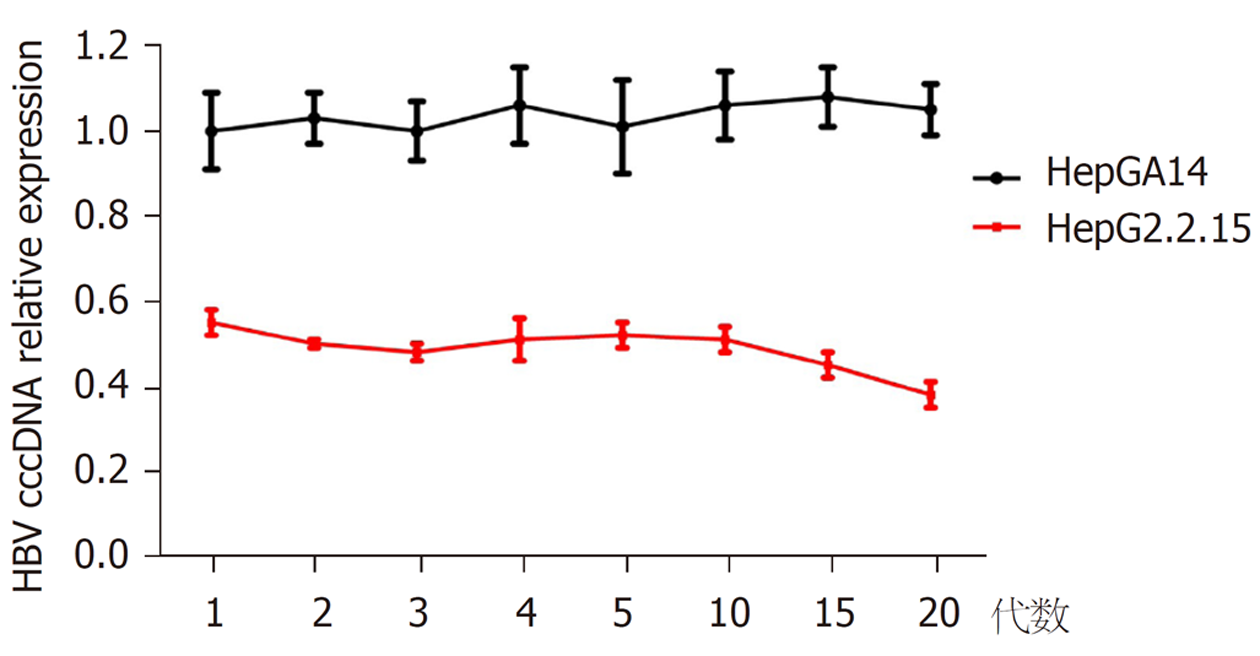
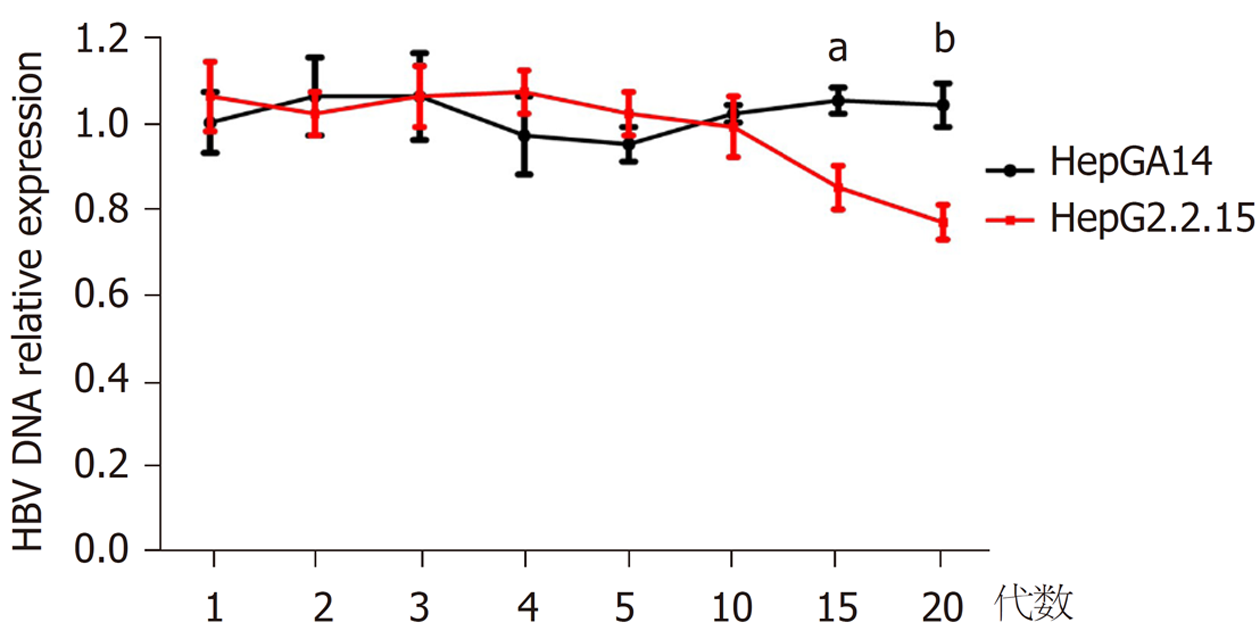
HepGA14优势单克隆株培养1-20代的HBx、HBV DNA、cccDNA相对表达水平均较稳定, 而HepG2.2.15株HBx、HBV DNA、cccDNA相对表达水平随着传代数的增加, 逐渐降低. HepGA14组各代HBx、cccDNA相对表达水平均高于HepG2.2.15组, 差异均具有统计学意义(P<0.05)(图8、图9). HepG2.2.15组的HBV DNA相对表达水平从第15、20代显著下降, 与同代HepGA14比较, 差异具有统计学意义(P<0.05)(图10).
HBV是一种是噬肝病毒属小包膜DNA病毒, 由于其具有严格的种属限制性及组织亲嗜性, 宿主范围狭窄, 只能感染人和少数灵长类动物(如黑猩猩)[3]. 因此支持HBV感染和复制的体外细胞模型就成了研究HBV生命周期、病毒-宿主相互作用、致病机理及新型抗病毒药物等的重要工具[4]. 根据研究目的、感染途径等不同又分为可直接感染HBV的细胞模型和依赖基因工程技术所建立的重组细胞系模型2类. 对于直接感染HBV的细胞模型, 由于人类原代肝细胞、胚胎肝细胞保留了肝细胞的重要细胞学特性, 自然条件下允许HBV穿入和完整复制, 能模拟最接近HBV体内感染的真实过程及状态, 相当长时间内占据着HBV感染模型的主要位置[5]. 但这类细胞模型都具有细胞来源有限, 体外培养技术要求较高且不能连续扩增, 生命周期短暂, HBV易感性迅速降低, 不同捐赠者之间个体差异较大等缺点, 因此只适合于对HBV感染早期阶段研究[6]. HepaRG细胞是唯一可直接感染HBV的肝癌细胞系, 增殖能力较强, 但是感染效率较低且操作复杂, 在诱导分化数周后才可被感染[7]. 近年来, 随着钠离子-牛磺胆酸共转运蛋白(sodium-taurocholate cotransporting polypeptide, NTCP)的发现, 使肝癌细胞系通过外源表达NTCP直接感染HBV成为了可能[8]. NTCP是多次跨膜的胆汁酸转运蛋白, 特异性地在肝细胞膜表面表达, 能与HBV病毒外膜蛋白pre-S1区域特异性结合. HepG2、HuH7等传代细胞系由于缺乏内源性NTCP表达, 故在正常培养条件下不能支持HBV感染, 但在其中外源性表达NTCP可恢复其HBV易感性. 以HepG2-NTCP、HuH7-NTCP为代表的新型HBV感染细胞模型, 为针对HBV生命周期早期阶段的研究提供了新选择[9]. 但该类模型与体内感染相比, 感染是短暂的, 所产生的子代病毒不能有效地在细胞间传播, 从而限制了其作为HBV病毒源的研究应用, 且只有少量cccDNA可被检测到.
同时, 为解决研究HBV生命周期中后阶段自然史、筛选抑制HBV转录与复制药物及获得稳定细胞培养源性HBV病毒颗粒等需要, 科学家们采用基因工程技术建立了系列整合HBV基因组, 能持续扩增, 高效、稳定表达HBV标志物的稳转细胞模型. 1987年, Sells等[10]通过将首尾相连的2倍HBV基因组整合进入HepG2细胞, 从而构建成能稳定表达乙肝病毒颗粒的HepG2.2.15细胞系. 该细胞系中HBV可持续复制, 并能检测到共价闭合环状DNA分子(cccDNA)和松弛环状 DNA(rcDNA)的产生; 能产生一定量的病毒感染颗粒, 将其注射到黑猩猩体内可以诱发显著的肝炎症状[11]. 该模型已被广泛用于HBV基础生物学问题研究, 为早期抗HBV药物的发展做出了重要贡献, 但该模型存在病毒复制活性弱、cccDNA表达水平较低等不足, 且随着培养代数增多, HBV主要抗原(HBsAg、HBeAg)表达量会逐渐降低, 更难以满足以cccDNA等为目标新型抗病毒药物研发的需要. 针对HepG2.2.15存在的问题, 学者们将1.1倍的HBV全基因组整合入HepG2细胞, 成功构建了HepAD38细胞模型. 该模型HBV的产生受四环素严格调控, 当培养基中含有四环素时由于抑制了前基因组RNA(pregenomic RNA, pgRNA)的合成, 而无法合成HBV, 去除培养基中四环素后, 细胞立即表达pgRNA、cccDNA和HBV DNA等标志物; 同时HepAD38产生HBV DNA、cccDNA等的量比HepG2.2.15大幅增加[12]. 但HepAD38对四环素敏感的巨细胞病毒(CMV)启动子, 与自然感染条件下, HBV通过内源启动子作用下转录、复制的机制是不同的[13]. 进一步, 针对HepAD38存在的问题, 科学家们提出了1.3-fold HBV基因组的解决方案: 在HBV1.1倍上游加入其自身启动子形成的, 使其依赖自身启动子启动转录, 这样可以充分反映病毒株的自身复制情况, 便于研究HBV复制和转录调节的全过程. 如HepAD79、HepG2-H1.3整合了1.3倍的HBV全基因组, 表达完全靠HBV自身启动子, 更接近于病毒自然状态, 支持cccDNA形成和完整病毒复制, 但上述模型的病毒产量较低, 不能满足以降解、清除cccDNA为靶点的新型抗病毒药物研发的需要[14].
本项目组在前人基础上, 采用慢病毒质粒技术将1.3倍的HBV全基因组整合入HepG2细胞基因组中, 构建HBV 1.3倍HepG2稳转细胞模型. 由于慢病毒将靶基因插入目标细胞基因组中的位置是随机的, 而不同的目标细胞基因插入位置, 由于自身转录活性的差异, 会对靶基因HBV的表达水平产生较大影响, 故需进行优势株的筛选. 本项目组以构建成功的HBV 1.3倍HepG2稳转细胞模型为基础, 按照以下研究步骤: 分批次挑选、扩增单克隆株→初筛出阳性单克隆株→复筛出9株候选阳性单克隆株→检测9株候选阳性单克隆株的基因组插入HepG2基因组的位置→检测9株候选阳性单克隆株表达HBsAg、HBeAg的活性及稳定性→选出最优势单克隆株→与HepG2.2.15比较该优势单克隆株表达HBVDNA、cccDNA、HBx、HBsAg、HBeAg等生物标志物的活性及稳定性, 从而进一步筛选并鉴定出1.3-fold HBV基因组HepG2稳转细胞模型优势单克隆株. 结果表明, 在挑选出的9株候选阳性单克隆中以A14插入HepG2基因组位置为1:166461695-166461715(命名为HepGA14)的HBsAg、HBeAg表达水平最高, 分别为24.28 IU/mL、39.62 NCU/mL, 初步确定为优势单克隆株. 以HepG2.2.15细胞株为对照, 进一步检测HepGA14优势单克隆株1-20代表达HBx、cccDNA、HBVDNA的水平, 结果表明HepGA14能高效、稳定地表达HBV生物标志物, 差异具有统计学意义(P<0.05). HepGA14有望为以HBx为功能蛋白的肝纤维化启动机制、肝细胞癌发病机制、HBx-免疫系统跨细胞竞争机制等前沿研究奠定基础; 为以cccDNA为靶点的新型HBV"治愈"药物的研发提供有力的工具, 具有积极的应用价值.
综上, 所筛选出的优势单克隆株HepGA14在表达cccDNA、HBx、HBVDNA、HBsAg、HBeAg等标志物的稳定性及生物活性上, 与常规的HepG2.2.15细胞株比较, 具有明显的优势, 其有望在新型HBV"治愈"药物及致病机制等领域发挥积极作用.
慢性乙型肝炎的抗病毒治疗已进入到要实现"临床性治愈""完全治愈"的新时代. 已知共价闭合环状DNA分子(covalently closed circularDNA, cccDNA)、乙型肝炎病毒X蛋白(hepatitis B virus X protein, HBx)是导致乙型肝炎病毒(hepatitis B virus, HBV)感染慢性化和难以治愈的关键物质基础. 但目前尚缺乏能稳定、高效表达上述标志物的体外细胞模型, 从而严重限制了以"治愈"为目标的药物筛选及机制探索. 本研究拟采用慢病毒转染技术, 以HepG2细胞为载体, 构建并筛选出能稳定、高效表达cccDNA、HBx等生物标志物的优势单克隆株, 以期为以cccDNA为靶点的新型HBV"治愈"药物研发, 以HBx为功能蛋白的肝纤维化启动机制、肝细胞癌发病机制、HBx-免疫系统跨细胞竞争机制等探索提供新的研究工具.
本研究的主题是筛选出HBV1.3倍(1.3-fold HBV)基因组HepG2稳转细胞模型优势单克隆株, 并鉴定出该优势单克隆株的HBV基因组整合入HepG2靶细胞的基因组位置, 及其在表达HBeAg、HBeAg、HBVDNA、cccDNA、HBx等标志物上的稳定性与活性.
本研究的主要目标是筛选并鉴定出能稳定、高效表达cccDNA、HBx等标志物的1.3-fold HBV全基因组HepG2稳转细胞模型优势单克隆株, 为"治愈"乙肝药物的研制及HBV相关疾病发病机制的探索奠定基础.
本研究以最佳MOI值将1.3-fold HBV慢病毒液侵染靶细胞HepG2, 以抗生素BSD最佳筛选剂量筛选出稳定整合1.3-fold HBV基因的HepG2细胞系, 1.3-fold HBV基因组扩增产物使用0.8%琼脂糖凝胶电泳检测, Sanger测序鉴定各候选阳性单克隆细胞的侧翼序列对应的基因组插入位置, ELISA法检测HBsAg、HBeAg含量, PCR法检测HBVDNA、cccDNA、HBx表达水平.
本研究成功筛选出1株能稳定、高效地表达HBsAg、HBeAg、HBVDNA、HBx、cccDNA标志物的优势单克隆株, 命名为HepGA14, 经鉴定其入HepG2基因组位置为1:166461695-166461715.
本研究构建并筛选出了1株能稳定、高效表达HBV生物标志物的优势单克隆株, 将在乙肝"治愈"药物的研制及肝硬化、肝癌、肝衰竭发病机制的研究上有较广阔的应用价值.
本研究只观察了连续培养20代范围内HepGA14的表达规律, 对其长期传代(>100代)后的表达稳定性及活性仍有待持续观察. 本项目组下一步, 将对于1.3倍HBV基因组插入HepG2后对宿主本身的细胞形态、功能及活性的影响; 对HepGA14转录出的产物是否具有与体内感染相一致的再感染性及致病功能; 对干扰素、核苷(酸)类似物及中药等抗病毒药物的应答敏感性等进行深入研究, 从而为实现攻克HBV做出积极贡献.
学科分类: 胃肠病学和肝病学
手稿来源地: 广西壮族自治区
同行评议报告学术质量分类
A级 (优秀): 0
B级 (非常好): B, B, B
C级 (良好): 0
D级 (一般): 0
E级 (差): 0
科学编辑: 张砚梁 制作编辑:张砚梁
| 1. | Hu J, Lin YY, Chen PJ, Watashi K, Wakita T. Cell and Animal Models for Studying Hepatitis B Virus Infection and Drug Development. Gastroenterology. 2019;156:338-354. [PubMed] [DOI] |
| 3. | Dandri M, Lütgehetmann M, Petersen J. Experimental models and therapeutic approaches for HBV. Semin Immunopathol. 2013;35:7-21. [PubMed] [DOI] |
| 4. | Allweiss L, Dandri M. Experimental in vitro and in vivo models for the study of human hepatitis B virus infection. J Hepatol. 2016;64:S17-S31. [PubMed] [DOI] |
| 5. | Ramboer E, De Craene B, De Kock J, Vanhaecke T, Berx G, Rogiers V, Vinken M. Strategies for immortalization of primary hepatocytes. J Hepatol. 2014;61:925-943. [PubMed] [DOI] |
| 6. | Wilkening S, Stahl F, Bader A. Comparison of primary human hepatocytes and hepatoma cell line Hepg2 with regard to their biotransformation properties. Drug Metab Dispos. 2003;31:1035-1042. [PubMed] [DOI] |
| 7. | Andersson TB, Kanebratt KP, Kenna JG. The HepaRG cell line: a unique in vitro tool for understanding drug metabolism and toxicology in human. Expert Opin Drug Metab Toxicol. 2012;8:909-920. [PubMed] [DOI] |
| 8. | Yan H, Liu Y, Sui J, Li W. NTCP opens the door for hepatitis B virus infection. Antiviral Res. 2015;121:24-30. [PubMed] [DOI] |
| 9. | Sun Y, Qi Y, Peng B, Li W. NTCP-Reconstituted In Vitro HBV Infection System. Methods Mol Biol. 2017;1540:1-14. [PubMed] [DOI] |
| 10. | Sells MA, Chen ML, Acs G. Production of hepatitis B virus particles in Hep G2 cells transfected with cloned hepatitis B virus DNA. Proc Natl Acad Sci USA. 1987;84:1005-1009. [PubMed] [DOI] |
| 11. | Acs G, Sells MA, Purcell RH, Price P, Engle R, Shapiro M, Popper H. Hepatitis B virus produced by transfected Hep G2 cells causes hepatitis in chimpanzees. Proc Natl Acad Sci USA. 1987;84:4641-4644. [PubMed] [DOI] |
| 12. | Priyambada SA, Misaki R, Okamoto T, Okamoto Y, Ohashi T, Ueda K, Matsuura Y, Fujiyama K. Cell surface N-glycan alteration in HepAD38 cell lines expressing Hepatitis B virus. Virus Res. 2017;238:101-109. [PubMed] [DOI] |
| 13. | Ladner SK, Otto MJ, Barker CS, Zaifert K, Wang GH, Guo JT, Seeger C, King RW. Inducible expression of human hepatitis B virus (HBV) in stably transfected hepatoblastoma cells: a novel system for screening potential inhibitors of HBV replication. Antimicrob Agents Chemother. 1997;41:1715-1720. [PubMed] [DOI] |
| 14. | Ying C, De Clercq E, Neyts J. Selective inhibition of hepatitis B virus replication by RNA interference. Biochem Biophys Res Commun. 2003;309:482-484. [PubMed] [DOI] |