修回日期: 2024-07-27
接受日期: 2024-08-14
在线出版日期: 2024-08-28
先前的研究已经表明长链非编码RNAs(long non-coding RNAs, lncRNAs)在包括胃癌在内的多种癌症进展中的重要调节因子的作用. 然而, LINC00662的生物学功能及其调控结胃癌进展的潜在机制尚不清楚.
探讨LINC00662能否靶向miR-615-5p调控胃癌细胞增殖、迁移和侵袭.
体外培养人胃上皮细胞GES1和胃癌细胞系(SNU-1、AGS和HS-746T), RT-qPCR法检测LINC00662和miR-615-5p表达. SNU-1细胞转染LINC00662小干扰RNA、miR-615-5p模拟物或抑制剂; CCK-8、克隆形成、Transwell分析细胞增殖、迁移和侵袭, 蛋白质印迹法检测细胞中基质金属蛋白酶(matrix metalloproteinase, MMP)-2、MMP-9和β-连环蛋白(β-catenin)蛋白表达. 双荧光素酶报告实验验证互作关系.
与GES1细胞比较, 胃癌细胞系中LINC00662表达上调而miR-615-5p表达下调(P<0.05). 下调LINC00662或上调miR-615-5p后, SNU-1细胞A值、克隆数、迁移数、侵袭数、MMP2、MMP9蛋白表达下调(P<0.05). 同时, 下调LINC00662降低β-catenin蛋白表达(P<0.05). LINC00662靶向结合miR-615-5p, 且下调LINC00662的SNU-1细胞中miR-615-5p表达升高(P<0.05). miR-615-5p抑制剂逆转下调LINC00662对SNU-1细胞增殖、迁移和侵袭的影响.
LINC00662通过靶向miR-615-5p和激活wnt/β-catenin通路来加速胃癌细胞增殖、迁移和侵袭.
核心提要: 我们的研究展示了LINC00662在胃癌组织和细胞SNU-1、AGS和HS-746T中显著高表达. 功能分析表明了LINC00662下调可以抑制胃癌细胞SNU-1的增殖, 迁移, 和侵袭通过靶向miR-615-5p.
引文著录: 孙兵, 王超雄, 黄坚, 方军. LINC00662通过miR-615-5p/wnt/β-catenin通路调控胃癌细胞增殖、迁移和侵袭. 世界华人消化杂志 2024; 32(8): 600-607
Revised: July 27, 2024
Accepted: August 14, 2024
Published online: August 28, 2024
Previous studies have shown the role of long non-coding RNAs (lncRNAs) as important regulators in the progression of various cancers, including gastric cancer. However, the biological function of LINC00662 and the mechanism underlying its regulation of gastric cancer progression are still unclear.
To investigate whether LINC00662 can target miR-615-5p to regulate gastric cancer cell proliferation, migration, and invasion.
Human gastric epithelial cells (GES1) and gastric cancer cell lines (SNU-1, AGS, and HS-746T) were cultured in vitro, and the expression levels of LINC00662 and miR-615-5p in these cell types were detected by RT-qPCR. SNU-1 cells were transfected with LINC00662 small interfering RNA, or miR-615-5p mimic or inhibitor, and cell proliferation, migration, and invasion were evaluated by CCK-8, clone formation, and Transwell assays. Western blot analysis was performed to examine the protein expression of matrix metalloproteinase (MMP)-2, MMP-9, and β-catenin. Dual-luciferase reporter experiment was used to verify the regulatory relationship between LINC00662 and miR-615-5p.
Compared with GES1 cells, LINC00662 was up-regulated and miR-615-5p was down-regulated in gastric cancer cell lines (P < 0.05). After down-regulating LINC00662 or up-regulating miR-615-5p, cell proliferation, migration, and invasion, and MMP2 and MMP9 protein levels in SNU-1 cells were decreased (P < 0.05). β-catenin protein level in SNU-1 cells with down-regulated LINC00662 expression was decreased (P < 0.05). LINC00662 targets miR-615-5p, and miR-615-5p expression was increased by LINC00662 down-regulation (P < 0.05). miR-615-5p inhibitor reversed the effect of down-regulating LINC00662 on SNU-1 cell proliferation, migration, and invasion.
LINC00662 promotes gastric cancer cell proliferation, migration, and invasion by targeting miR-615-5p and activating the Wnt/β-catenin pathway.
- Citation: Sun B, Wang CX, Huang J, Fang J. LINC00662 regulates gastric cancer cell proliferation, migration, and invasion via the miR-615-5p/Wnt/β-catenin pathway. Shijie Huaren Xiaohua Zazhi 2024; 32(8): 600-607
- URL: https://www.wjgnet.com/1009-3079/full/v32/i8/600.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v32.i8.600
胃癌是常见的消化系统恶性肿瘤[1], 探究影响胃癌细胞恶性表型的分子机制可为治疗靶点的选择提供新途径. LINC00662属于长链非编码RNA(long non-coding RNA,lncRNA), 其在多种癌组织中表达上调, 且发挥促癌基因作用[2-5]. 既往研究显示, LINC00662在胃癌组织中高表达, 且其过表达可促进胃癌细胞增殖和转移[6]. lncRNA具有微小RNA(microRNA, miRNA)的反应元件并可竞争结合miRNA从而影响蛋白表达. 但目前, LINC00662影响胃癌发展的分子机制尚未明确. LncBase Predicted v.2在线软件预测显示, LINC00662靶向结合miR-615-5p. 据报道, miR-615-5p在多种癌组织中显著低表达, 且发挥抑癌基因作用[7-9]. 然而, LINC00662能否靶向miR-615-5p影响胃癌进展尚不清楚. 本研究在于揭示LINC00662/miR-615-5p轴对胃癌细胞增殖、迁移和侵袭的影响.
人胃上皮细胞GES1和胃癌细胞系(SNU-1、AGS和HS-746T), 中国科学院上海细胞库; TRIzol、逆转录试剂盒和PCR试剂盒, 大连宝生物; RPMI-1640培养液、FBS, 美国Gibco; Lipofectamine 2000试剂盒, 美国Invitrogen; 双荧光素酶活性检测试剂盒, 北京索莱宝; PCR引物、LINC00662小干扰RNA(si-LINC00662)、miR-615-5p模拟物(mimics)、miR-615-5p抑制剂(anti-miR-615-5p)及对照, 上海生工; 兔抗人基质金属蛋白酶(matrix metalloproteinase, MMP)-2、MMP9、β-连环蛋白(β-catenin)、甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase, GAPDH)、山羊抗兔二抗, 美国Abcam.
1.2.1 细胞培养: GES1、SNU-1、AGS和HS-746T细胞在含10% FBS的RPMI-1640培养液中培养.
1.2.2 RT-qPCR法: 收集细胞, 用TRIzol提取总RNA, 逆转录为cDNA后, 行PCR扩增. 2-△△Ct法计算LINC00662和miR-615-5p的相对表达量.
1.2.3 细胞转染: SNU-1细胞接种至6孔板中, 用LipofectamineTM 2000脂质体法, 分别转染si-NC、si-LINC00662、miR-NC、miR-615-5p mimics、si-LINC00662与anti-miR-NC、si-LINC00662与anti-miR-615-5p, 记为si-NC组、si-LINC00662组、miR-NC组、miR-615-5p组、si-LINC00662+anti-miR-NC组、si-LINC00662+anti-miR-615-5p组.
1.2.4 CCK-8: 细胞接种至96孔板中, 培养24 h后, 与CCK-8孵育2 h, 用酶标仪测吸光度(A)值.
1.2.5 克隆形成实验: 细胞接种至6孔板中, 培养14 d后, 用多聚甲醛固定和结晶紫染色, 显微镜下计数.
1.2.6 Transwell: Transwell小室的上室预包被或不包被Matrigel基质胶, 分别用于侵袭和迁移检测. 上室接种用无血清培养基重悬的SNU-1细胞, 下室充满完全培养基, 24 h后, 下室表面细胞用多聚甲醛固定和结晶紫染色, 显微镜观察, 记数.
1.2.7 蛋白质印迹法: RIPA提取总蛋白, 经SDS-PAGE电泳后, 转至PVDF膜, 膜与anti-MMP2(1:500)、anti-MMP9(1:500)、anti-β-catenin(1:1000)和anti-GAPDH(1:1000)孵育过夜, 再与二抗(1:1000)孵育2 h. 避光显影, 分析蛋白表达.
1.2.8 双荧光素酶报告实验: 含miR-615-5p结合位点或突变位点的LINC00662序列插入pGL3载体, 构建LINC00662野生型或突变型荧光素酶载体(WT/MUT-LINC00662). SNU-1细胞共转染miR-615-5p mimics/miR-NC与WT/MUT-LINC00662. 48 h后收集细胞, 检测荧光素酶活性.
统计学处理 SPSS 22.0软件进行统计学分析. 计量资料以均数±标准差(mean±SD)表示. 组间比较用独立样本t检验或单因素方差分析. 以P<0.05表示差异有统计学意义.
与GES1细胞比较, SNU-1、AGS和HS-746T细胞中LINC00662表达上调且miR-615-5p表达下调(P<0.05), 见表1.
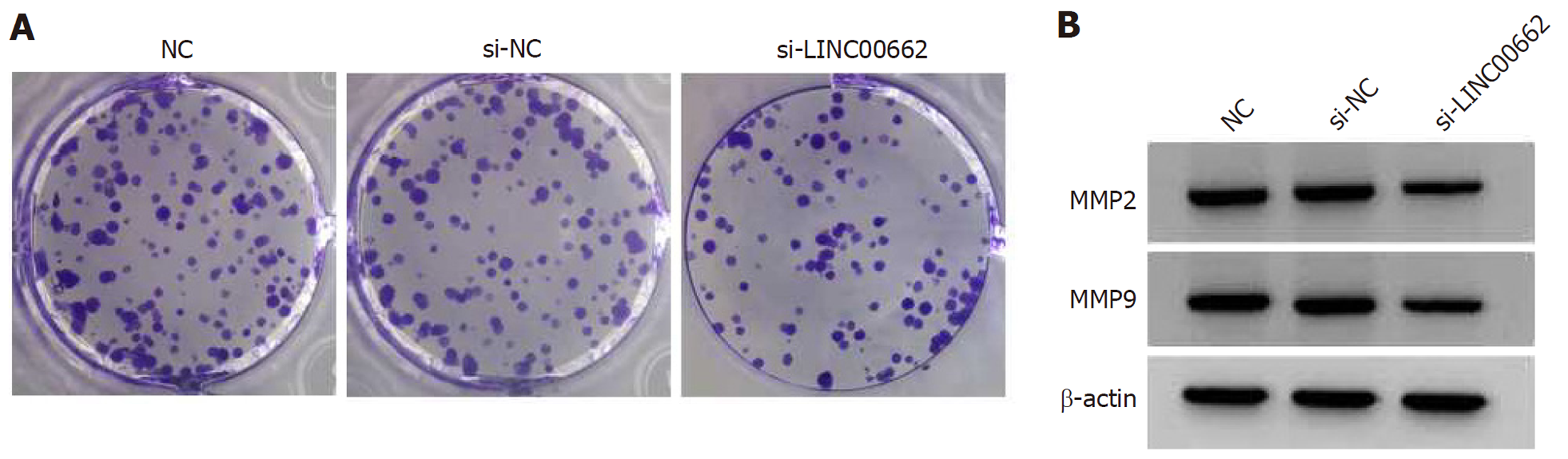
si-LINC00662组SNU-1细胞中LINC00662表达量明显低于NC组和si-NC组(P<0.05). 与NC组或si-NC组比较, si-LINC00662组SNU-1细胞A值、克隆数、迁移数、侵袭数、MMP2、MMP9蛋白表达降低(P<0.05), 见图1和表2.
| 组别 | LINC00662 | MMP2 | MMP9 | A值 | 细胞迁移数量(个) | 细胞侵袭数量(个) | 细胞克隆数量(个) |
| NC | 1.00±0.11 | 0.87±0.07 | 0.79±0.06 | 1.124±0.10 | 245±18.37 | 173±12.03 | 142±10.34 |
| si-NC | 1.01±0.08 | 0.85±0.08 | 0.77±0.05 | 1.118±0.10 | 241±19.73 | 169±14.51 | 138±9.38 |
| si-LINC00662 | 0.47±0.03a | 0.42±0.03a | 0.35±0.03a | 0.573±0.05a | 117±9.12a | 75±6.73a | 71±6.10a |
| F | 132.820 | 143.041 | 238.114 | 120.132 | 176.554 | 207.343 | 185.072 |
| P | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 |
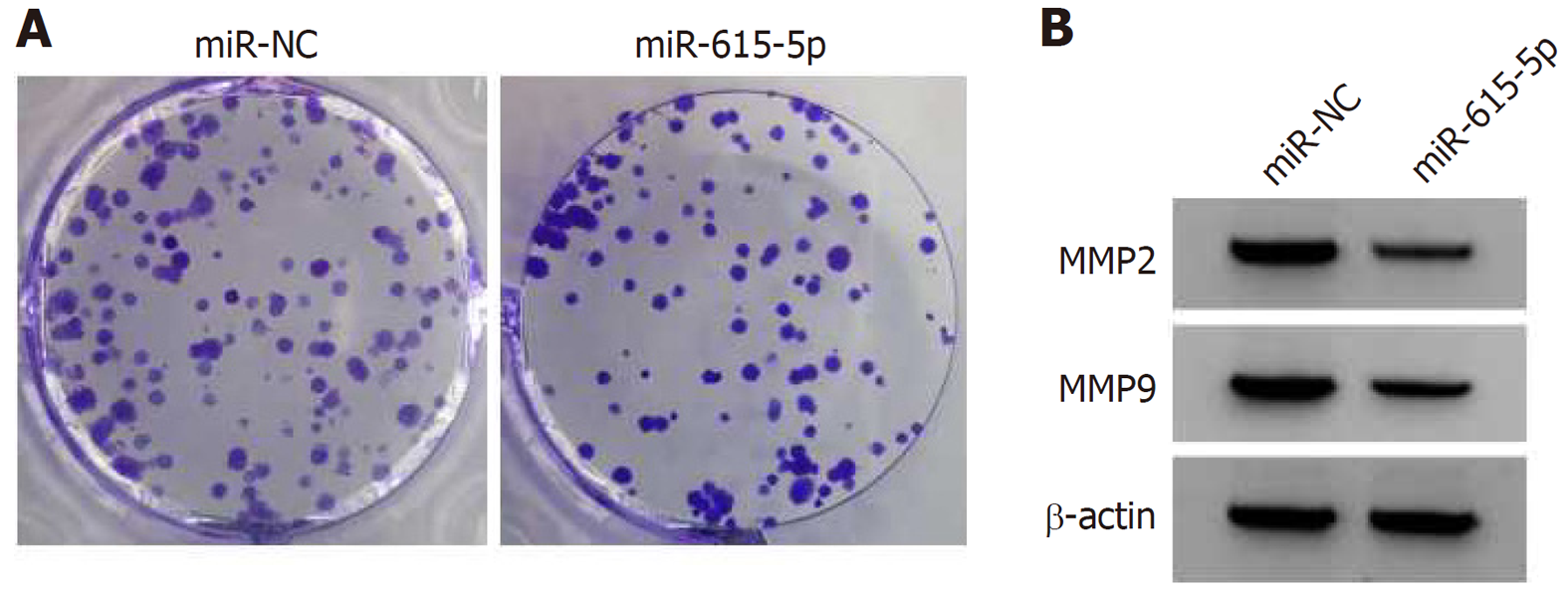
miR-615-5p组SNU-1细胞中miR-615-5p表达量明显高于miR-NC组(P<0.05). 与miR-NC组比较, miR-615-5p组SNU-1细胞A值、克隆数、迁移数、侵袭数、MMP2、MMP9蛋白表达降低(P<0.05), 见图2和表3.
LncBase Predicted v.2软件预测LINC00662和miR-615-5p存在结合位点, 见图3. miR-615-5p mimics降低WT-LINC00662组的荧光素酶活性(P<0.05), 见表4. 与si-NC组比较, si-LINC00662组LINC00662表达降低(0.42±0.04比1.00±0.11, t = 14.866, P<0.05), miR-615-5p表达量升高(2.01±0.18比1.00±0.08, t = 15.382, P<0.05).
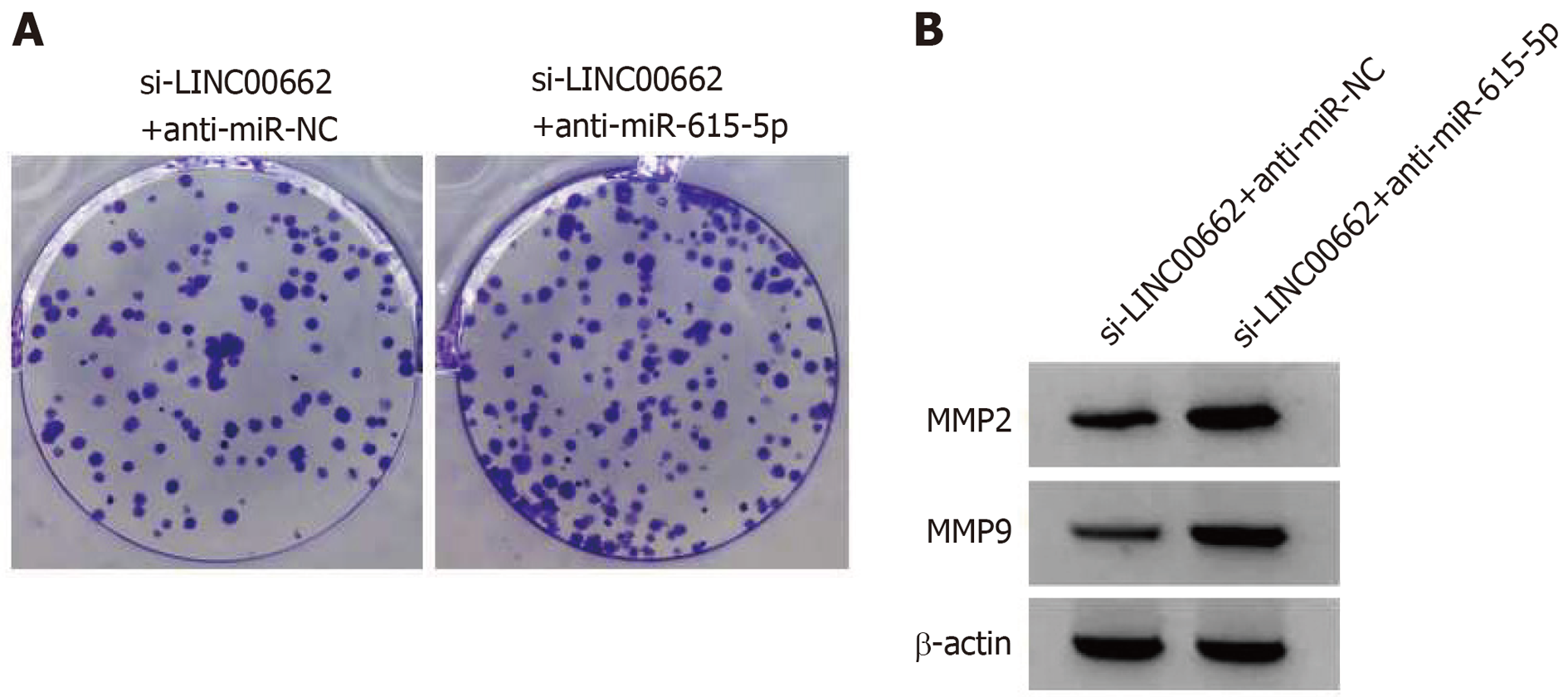
si-LINC00662+anti-miR-615-5p组SNU-1细胞中miR-615-5p表达量明显降低(P<0.05). 与si-LINC00662+anti-miR-NC组比较, si-LINC00662+anti-miR-615-5p组SNU-1细胞A值、克隆数、迁移数、侵袭数、MMP2、MMP9蛋白表达升高(P<0.05), 见图4和表5.
| 组别 | miR-615-5p | MMP2 | MMP9 | A值 | 细胞迁移数量(个) | 细胞侵袭数量(个) | 细胞克隆数量(个) |
| si-LINC00662+anti-miR-NC | 1.00±0.10 | 0.40±0.03 | 0.34±0.03 | 0.581±0.05 | 115±10.02 | 79±6.81 | 74±6.34 |
| si-LINC00662+anti-miR-615-5p | 0.43±0.04a | 0.92±0.08a | 0.81±0.07a | 1.053±0.10a | 227±18.23a | 142±12.17a | 137±10.37a |
| t | 15.877 | 18.258 | 18.514 | 12.665 | 16.152 | 13.552 | 15.550 |
| P | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 |
胃癌的发展被认为与基因的异常表达有关. LncRNA是可通过竞争性吸附miRNA调控miRNA靶基因的表达和下游信号通路来影响肿瘤细胞恶性表型. 研究已表明, TP53TG1[10]、LINC00942[11]和CRYM-AS1[12]等多种lncRNA在胃癌组织中异常表达, 为胃癌的治疗提供了潜在分子靶点.
作为一种lncRNA, LINC00662参与调控多种肿瘤细胞的恶性表型. LINC00662在宫颈癌组织和细胞系中表达升高, LINC00662过表达促进了宫颈癌细胞增殖、迁移、侵袭和放射抗性, 为宫颈癌的治疗提供了潜在分子靶点[13]. 食管鳞癌中LINC00662表达上调, 其通过竞争性结合miR-340-5p进而正向调控HOXB2来影响食管鳞癌的发展进程[14]. 沉默LINC00662可通过靶向miR-497-5p进而抑制EglN2的表达来降低乳腺癌细胞活力, 并阻碍乳腺癌细胞迁移和侵袭[15]. 与既往报道一致[6], 本研究显示, 下调LINC00662可阻碍胃癌细胞增殖、迁移和侵袭, 提示LINC00662可能成为胃癌治疗的分子靶点. 细胞迁移和侵袭受多种基因分子的调控, 其中MMP2和MMP9扮演重要作用[16]. 本研究结果显示, 下调LINC00662可降低MMP2和MMP9蛋白表达, 提示LINC00662调控MMP2和MMP9表达影响胃癌细胞迁移和侵袭.
进一步的分析显示, LINC00662靶向miR-615-5p. miR-615-5p在多种肿瘤的发展中起调控作用. 研究显示[17], miR-615-5p下调可以促进肺腺癌细胞增殖并阻碍其凋亡; 上调miR-615-5p可阻碍食管鳞癌细胞侵袭和迁移[18]. 本研究结果表明, miR-615-5p在胃癌细胞系中低表达, 上调miR-615-5p抑制胃癌细胞的增殖、迁移和侵袭能力, 这说明miR-615-5p在胃癌中也发挥抑癌基因作用; 同时, 下调miR-615-5p逆转了LINC00662敲低对胃癌细胞进展的抑制作用, 提示LINC00662靶向miR-615-5p来影响胃癌细胞增殖、迁移和侵袭.
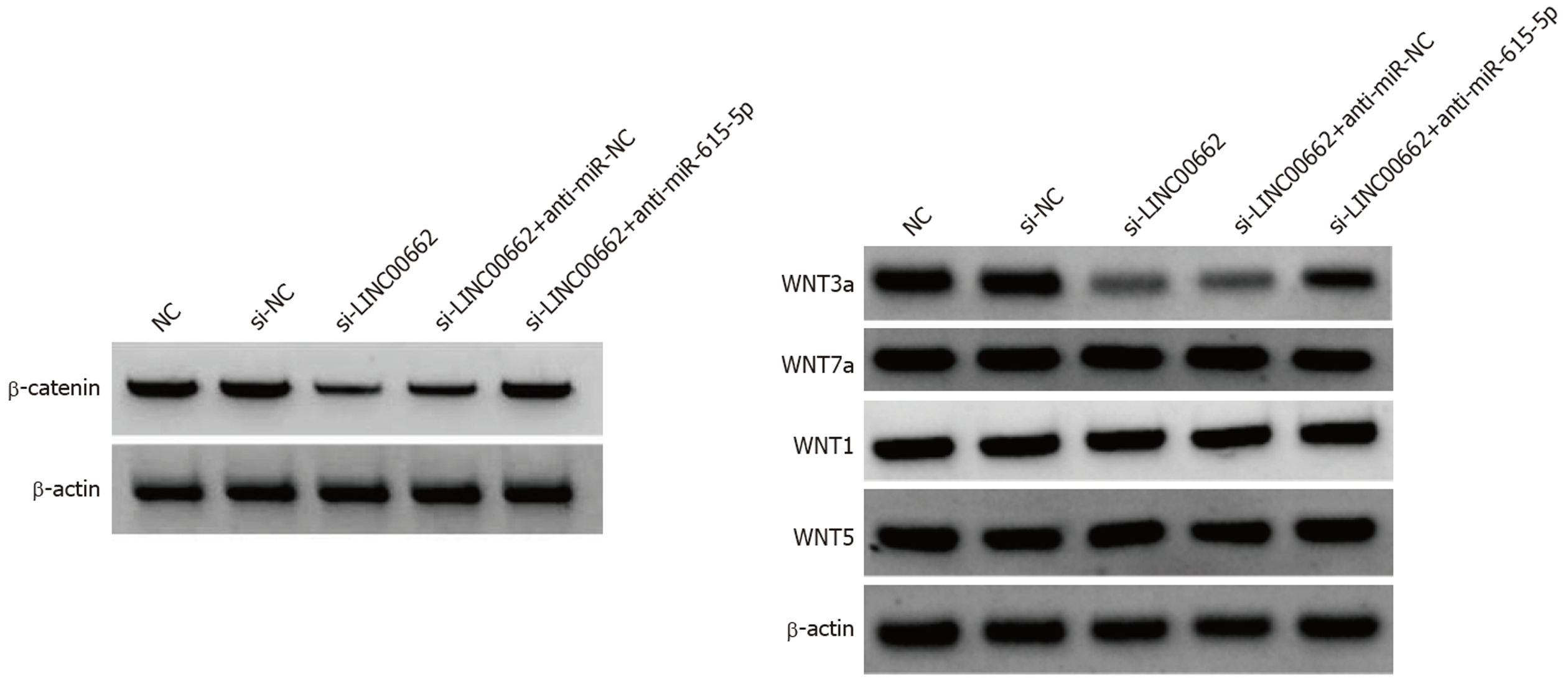
wnt/β-catenin通路是传递生长发育信息的重要通路, 其异常活化与肿瘤的发生有关[19]. Wang等[20]研究显示, LNCRNA ZEB2-AS1通过激活wnt/β-catenin通路促进胃癌细胞增殖和转移, 这证实了wnt/β-catenin通路的活性与胃癌进展有关. 本研究显示, 下调LINC00662可抑制胃癌细胞中wnt/β-catenin通路活性, 这种作用可以被miR-615-5p抑制剂所逆转, 进一步提示LINC00662靶向miR-615-5p来激活wnt/β-catenin信号通路.
综上, 胃癌细胞系中LINC00662表达上调, 而miR-615-5p表达下调. LINC00662靶向miR-615-5p促进wnt/β-catenin通路活性, 进而加速胃癌细胞增殖、迁移和侵袭. 这些发现为胃癌潜在分子靶点的开发提供了新思路.
大量的文献已经证实了长链非编码RNAs(long non-coding RNAs, lncRNAs)在包括胃癌在内的许多恶性肿瘤进展中发挥了必要调节因子作用. 然而, LINC00662的功能和潜在的调控机制在胃癌进展尚不清楚.
探索LINC00662是否可以调控胃癌的进展, 为临床治疗胃癌提供有效靶点.
以miR-615-5p为切入点探索LINC00662在胃癌中的作用和机制, 这可能是胃癌的治疗靶点.
RT-qPCR法分析了LINC00662、miR-615-5p在胃癌组织及癌旁组织的表达; 细胞增殖、迁移及侵袭经CCK8、克隆形成实验、与Transwell实验分析; 金属蛋白酶(matrix metalloproteinase, MMP)-2、MMP-9、β-连环蛋白(β-catenin)蛋白表达经Western blot检测; 采用双荧光素酶报告分析LINC00662与miR-615-5p的靶向关系.
我们实验数据证实了LINC00662在胃癌中的作用和潜在的机制. 结果展示了LINC00662在胃癌组织中高表达, 而miR-615-5p低表达;功能实验验证si- LINC00662可以降低SNU-1细胞A值、克隆数、迁移数、侵袭数、MMP-2和MMP-9蛋白表达以及β-catenin蛋白表达; LINC00662可靶向结合miR-615-5p并负向调控它的表达; miR-615-5p下调可逆转干扰LINC00662对SNU-1细胞增殖、迁移和侵袭的的抑制作用.
这项研究揭示了LINC00662在胃癌组织和细胞中上调, 沉默LINC00662可通过促进miR-615-5p表达来抑制胃癌细胞增殖、迁移及侵袭. 因此, 我们认为LINC00662/miR-615-5p轴可对调节胃癌进展至关重要, 并且可能是胃癌的治疗靶点.
本研究了LINC00662/miR-615-5p轴可调控胃癌生物恶性行为, 为临床治疗胃癌提供有效靶点.
学科分类: 胃肠病学和肝病学
手稿来源地: 浙江省
同行评议报告学术质量分类
A级 (优秀): A
B级 (非常好): 0
C级 (良好): C
D级 (一般): D
E级 (差): 0
科学编辑: 张砚梁 制作编辑:张砚梁
| 1. | Tan MC, Morgan DR. Gastric Cancer Trends in the United States: In Context and Possible Explanations. Clin Gastroenterol Hepatol. 2023;21:3234-3235. [PubMed] [DOI] |
| 2. | Wang S, Qiao C, Fang R, Yang S, Zhao G, Liu S, Li P. LncRNA CASC19: a novel oncogene involved in human cancer. Clin Transl Oncol. 2023;25:2841-2851. [PubMed] [DOI] |
| 3. | Baljon KJ, Ramaiah P, Saleh EAM, Al-Dolaimy F, Al-Dami FH, Gandla K, Alkhafaji AT, Abbas AHR, Alsaalamy AH, Bisht YS. LncRNA PVT1: as a therapeutic target for breast cancer. Pathol Res Pract. 2023;248:154675. [PubMed] [DOI] |
| 4. | Fang L, Huang H, Lv J, Chen Z, Lu C, Jiang T, Xu P, Li Y, Wang S, Li B, Li Z, Wang W, Xu Z. m5C-methylated lncRNA NR_033928 promotes gastric cancer proliferation by stabilizing GLS mRNA to promote glutamine metabolism reprogramming. Cell Death Dis. 2023;14:520. [PubMed] [DOI] |
| 5. | Zhang Y, Chen H, Yuan R, Wang Y, Zhang D, Zeng Q, Jiang W, Zhang R, Chen T, Chai C, Guo B. PDK1-stabilized LncRNA SPRY4-IT1 promotes breast cancer progression via activating NF-κB signaling pathway. Mol Carcinog. 2023;62:1009-1024. [PubMed] [DOI] |
| 6. | Tao F, Qi L, Liu G. Long intergenic non-protein coding RNA 662 accelerates the progression of gastric cancer through up-regulating centrosomal protein 55 by sponging microRNA-195-5p. Bioengineered. 2022;13:3007-3018. [PubMed] [DOI] |
| 7. | Jiao Y, Fu Y, Gong Y, Wang G, Chen S, Cai G, Wu S, Tang L. Circ_0067997 boosted the growth while repressed the apoptosis of SGC-7901/DDP cells via repressing miR-615-5p/AKT1 pathway. Cancer Biomark. 2023;37:27-38. [PubMed] [DOI] |
| 8. | Zhou W, Song W, Lu M. circ_0006789 promotes cervical cancer development via the miR-615-5p/HSF1 axis. Discov Oncol. 2024;15:165. [PubMed] [DOI] |
| 9. | Sun D, Gong L, Wang X, Chen S, Yi J, Liu X. Pro-inflammatory Cytokines Promote the Occurrence and Development of Colitis-associated Colorectal Cancer by Inhibiting miR-615-5p. Inflamm Bowel Dis. 2023;29:1854-1864. [PubMed] [DOI] |
| 10. | Fang D, Ou X, Sun K, Zhou X, Li Y, Shi P, Zhao Z, He Y, Peng J, Xu J. m6A modification-mediated lncRNA TP53TG1 inhibits gastric cancer progression by regulating CIP2A stability. Cancer Sci. 2022;113:4135-4150. [PubMed] [DOI] |
| 11. | Zhu Y, Zhou B, Hu X, Ying S, Zhou Q, Xu W, Feng L, Hou T, Wang X, Zhu L, Jin H. LncRNA LINC00942 promotes chemoresistance in gastric cancer by suppressing MSI2 degradation to enhance c-Myc mRNA stability. Clin Transl Med. 2022;12:e703. [PubMed] [DOI] |
| 12. | Zhang P, Chen C, Zhang J, Yu X. LncRNA CRYM-AS1 Inhibits Gastric Cancer Progression via Epigenetically Regulating CRYM. Ann Clin Lab Sci. 2022;52:249-259. [PubMed] |
| 13. | Wei J, Wang L, Sun Y, Bao Y. LINC00662 contributes to the progression and the radioresistance of cervical cancer by regulating miR-497-5p and CDC25A. Cell Biochem Funct. 2020;38:1139-1151. [PubMed] [DOI] |
| 14. | Zhang Z, Liang X, Ren L, Zhang S, Li S, Wan T, Xu D, Lv S. LINC00662 promotes cell viability and metastasis in esophageal squamous cell carcinoma by sponging miR-340-5p and upregulating HOXB2. Thorac Cancer. 2020;11:2306-2315. [PubMed] [DOI] |
| 15. | Cheng L, Xing Z, Zhang P, Xu W. Long non-coding RNA LINC00662 promotes proliferation and migration of breast cancer cells via regulating the miR-497-5p/EglN2 axis. Acta Biochim Pol. 2020;67:229-237. [PubMed] [DOI] |
| 16. | Liu Y, Sun M, Zhang B, Zhao W. KIF18A improves migration and invasion of colorectal cancer (CRC) cells through inhibiting PTEN signaling. Aging (Albany NY). 2023;15:9182-9192. [PubMed] [DOI] |
| 17. | Pan ZH, Guo XQ, Shan J, Luo SX. LINC00324 exerts tumor-promoting functions in lung adenocarcinoma via targeting miR-615-5p/AKT1 axis. Eur Rev Med Pharmacol Sci. 2018;22:8333-8342. [PubMed] [DOI] |
| 18. | Yang B, Xie R, Wu SN, Gao CC, Yang XZ, Zhou JF. MicroRNA-615-5p targets insulin-like growth factor 2 and exerts tumor-suppressing functions in human esophageal squamous cell carcinoma. Oncol Rep. 2018;39:255-263. [PubMed] [DOI] |
| 19. | Du Q, Zhang M, Gao A, He T, Guo M. Epigenetic silencing ZSCAN23 promotes pancreatic cancer growth by activating Wnt signaling. Cancer Biol Ther. 2024;25:2302924. [PubMed] [DOI] |
| 20. | Wang F, Zhu W, Yang R, Xie W, Wang D. LncRNA ZEB2-AS1 contributes to the tumorigenesis of gastric cancer via activating the Wnt/β-catenin pathway. Mol Cell Biochem. 2019;456:73-83. [PubMed] [DOI] |