修回日期: 2023-11-21
接受日期: 2023-12-20
在线出版日期: 2023-12-28
肝癌在全球癌症致死率中位列第三, 但其治疗效果仍未达到理想水平. 肝癌研究中的挑战之一是肝癌高度时空异质性、侵袭转移能力和抗药性. 因此, 深刻理解和模拟肝癌的免疫微环境对于研究至关重要. 本文探讨了肝癌研究中的临床前模型, 包括细胞系模型、患者来源性肿瘤移植小鼠模型、基因编辑小鼠模型、化学诱导模型、人源化小鼠模型、类器官模型和基于患者源性器官样肿瘤球模型. 各模型在模拟肝癌生物学行为和免疫微环境方面有各自的优势和不足. 通过深入了解现有模型的局限性, 我们有望推动下一代癌症模型的发展, 使其更精确地反映肝癌的特征, 为优化治疗策略、药物疗效预测和安全性评估提供支持, 促进个性化和精准化治疗. 此外, 文章还展望了未来的趋势和挑战, 以促进肝癌生物学和临床前研究的进展.
核心提要: 本文综合评估了当前各类常用的肝癌临床前模型, 强调了在高度异质和复杂的肝癌免疫微环境中, 各类模型的优势和局限性, 并展望未来临床治疗和基础研究的趋势,为肝癌的免疫精准化治疗提供重要视角和科学依据.
引文著录: 宋飞, 陈钟. 免疫精准化治疗背景下肝癌临床前模型的应用与展望. 世界华人消化杂志 2023; 31(24): 989-1000
Revised: November 21, 2023
Accepted: December 20, 2023
Published online: December 28, 2023
Hepatocellular carcinoma (HCC), ranking as the third leading cause of cancer-related mortality globally, continues to pose challenges in achieving optimal treatment outcomes. The complex nature of HCC, characterized by high spatiotemporal heterogeneity, invasive potential, and drug resistance, presents difficulties in its research. Consequently, an in-depth understanding and accurate simulation of the immune microenvironment of HCC are of paramount importance. This article comprehensively explores the application of preclinical models in HCC research, encompassing cell line models, patient-derived xenograft mouse models, genetically engineered mouse models, chemically induced models, humanized mouse models, organoid models, and microfluidic chip-based patient derived organotypic spheroids models. Each model possesses its distinct advantages and limitations in replicating the biological behavior and immune microenvironment of HCC. By scrutinizing the limitations of existing models, this paper aims to propel the development of next-generation cancer models, enabling more precise emulation of HCC characteristics. This will, in turn, facilitate the optimization of treatment strategies, drug efficacy prediction, and safety assessments, ultimately contributing to the realization of personalized and precision therapies. Additionally, this article also provides insights into future trends and challenges in the fields of tumor biology and preclinical research.
- Citation: Song F, Chen Z. Preclinical liver cancer models in the context of immunoprecision therapy: Application and perspectives. Shijie Huaren Xiaohua Zazhi 2023; 31(24): 989-1000
- URL: https://www.wjgnet.com/1009-3079/full/v31/i24/989.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v31.i24.989
肝癌是全球范围内第三大癌症类型, 每年新发病例超过840000例, 年死亡病例超过780000例, 占全球所有癌症死亡数的近9%[1,2]. 尽管手术、介入、放射、化疗、靶向和免疫治疗等多种手段的开发, 如索拉非尼、乐伐替尼的上市以及免疫治疗的进步[3,4], 使得肝癌的治疗有了新的突破. 然而, 肝癌的整体治疗效果仍不理想, 五年生存率仅有18%, 复发率高达70%[5,6].
免疫和靶向治疗的局限性部分归因于临床前模型模拟肝癌特性时所遇到的挑战. 这些挑战包括肝癌的复杂异质性、侵袭转移能力和抗药性[7]. 当前的临床前模型如患者源性类器官(patient-derived organoids, PDO)和患者源性异种移植瘤模型(patient-derived xenografts, PDX), 虽然在评估新药疗效上取得了一定的成效, 但它们在模拟肿瘤免疫微环境方面却显得力不从心[8,9]. 由于肿瘤免疫微环境在肝癌发展和疗效中的重要作用, 对于这一领域的深入理解和模拟显得至关重要. 近期研究显示, 结合免疫疗法和抗血管生成药物可显著提升晚期肝癌患者的生存率, 这一发现突显了发展能够精确模拟肝癌免疫微环境和生物学特性的临床前模型的重要性, 为评估和优化治疗策略提供了必要的基础[10,11].
这篇专家评述的目的是在当前肝癌治疗的研究背景下, 系统性地梳理和讨论现有广泛使用的临床前模型的优缺点, 以及它们在肝癌研究中的应用. 我们期望, 通过了解现有模型的局限性, 能推动下一代癌症模型的发展, 以更好地模拟肝癌的生物学行为和免疫微环境, 从而为优化治疗策略、预测药物的疗效和安全性, 以及实现患者的个性化和精准化治疗提供帮助. 此外, 我们还将前瞻性地讨论未来的发展趋势和挑战, 为肿瘤生物学及临床前癌症研究的未来发展方向提供参考.
在现代肿瘤研究中, 临床前模型起到了至关重要的作用. 不同的模型在模拟肿瘤生物学行为、评估新型治疗策略及预测药物疗效方面具有独特的优势, 而各种模型的综合运用则可为我们提供更为全面的研究视角. 为了满足不同的临床应用场景和科研目的, 各类不断被开发出来[12,13], 如图1所示. 现有临床前模型大概分为以下几类: (1)肝癌体外细胞培养模型: 这是最早使用的临床前模型, 其中最常见的就是二维细胞培养模型. 近年来, 也出现了三维细胞培养模型, 如类器官(organoids), 它们可以更好地模拟体内的肿瘤微环境[12,14]; (2)体内动物模型: 动物模型是一种重要的临床前模型, 包括自发肿瘤模型、化学诱导肿瘤模型、移植肿瘤模型(包括同种异位移植模型和异种移植模型)以及基因工程肿瘤模型等、人源化小鼠模型[15-17]; (3)新型肿瘤模型: 这是一种新型的临床前模型, 如微流控芯片(Microfluidic Chip)或生物打印技术构建的体外肿瘤模型[18-20], 可以在体外模拟肿瘤微环境和药物治疗过程.
2.1.1 肝癌细胞系模型: 肝癌细胞系作为最早使用的传统临床前模型, 因其基因型和表型的一致性、较低的维护费用、易于基因编辑性等优点, 在科学研究中扮演着重要的角色. 目前, 有大约40种不同的肝癌细胞系, 但最常用的是HepaRG、Huh 7、SK-Hep-1、Hep 3B和HepG2, 它们是从各种肝肿瘤中获得的[21]. 对这些文章的荟萃分析表明, 每种细胞类型都来自一种特定类型的癌症. 例如, Huh 7细胞系具有肝细胞癌的特征[22], MT-CHC01来源于肝内胆管癌细胞[23]和HepG2来源于肝母细胞癌[24]. 长期培养的肝癌细胞系在癌症和肝炎病毒感染的研究中具有广泛应用, 如HepG2和Huh7细胞系用于研究HDV的复制以及HBV的表达/复制[25], 而HepaRG细胞系则用于探讨HBV病毒进入宿主细胞的机制[26,27].
2.1.2 肝癌类器官模型: 基于肝衍生的类器官培养系统被应用于多种肝脏疾病模型中, 包括单基因疾病、酒精性肝炎、非酒精性脂肪性肝病、原发性硬化性胆管炎、病毒性肝炎和原发性肝癌等. 与传统细胞系和PDX模型相比, 类器官技术很好结合了这两种系统的优势[28,29]. 2017年《Nature Medicine》报道了一项研究[30], 其中构建了三种常见的原发性肝癌类器官(肝细胞癌、胆管癌和混合样癌). 通过长期培养扩增, 原发性肝癌衍生的类器官保留了原始肿瘤的组织学结构、基因表达和基因组景观, 并且适合于生物标志物鉴定和药物筛选测试. 作为体外模型, 肝癌类器官很好的保留了肿瘤内和患者间的遗传和表型异质性, 并且适合高通量药物的筛选. 研究表明[9], 使用3D类器官模型进行分子靶向药物敏感性测试得到的药物半数抑制浓度高于2D模型, 更接近人体真实有效血药浓度[9]. 这一有趣的发现从侧面证实了类器官药物反应的真实性. 此外, 类器官在测试多种药物组合方面也展现出优势[31]. 通过将现有的治疗方案与其他药物结合, 可以筛选出可能增强对某些靶向药物敏感性并降低耐药性问题的候选药物. 利用肝细胞癌患者源性类器官证实, 索拉非尼对CD44+PDO的疗效显著不足, 而Hedgehog信号通路抑制剂GANT61能够扭转对索拉非尼产生的耐药[32]. 总的来说, 对于评估靶向药物的敏感性方面, 类器官展示出了巨大的潜力和广阔的应用前景.
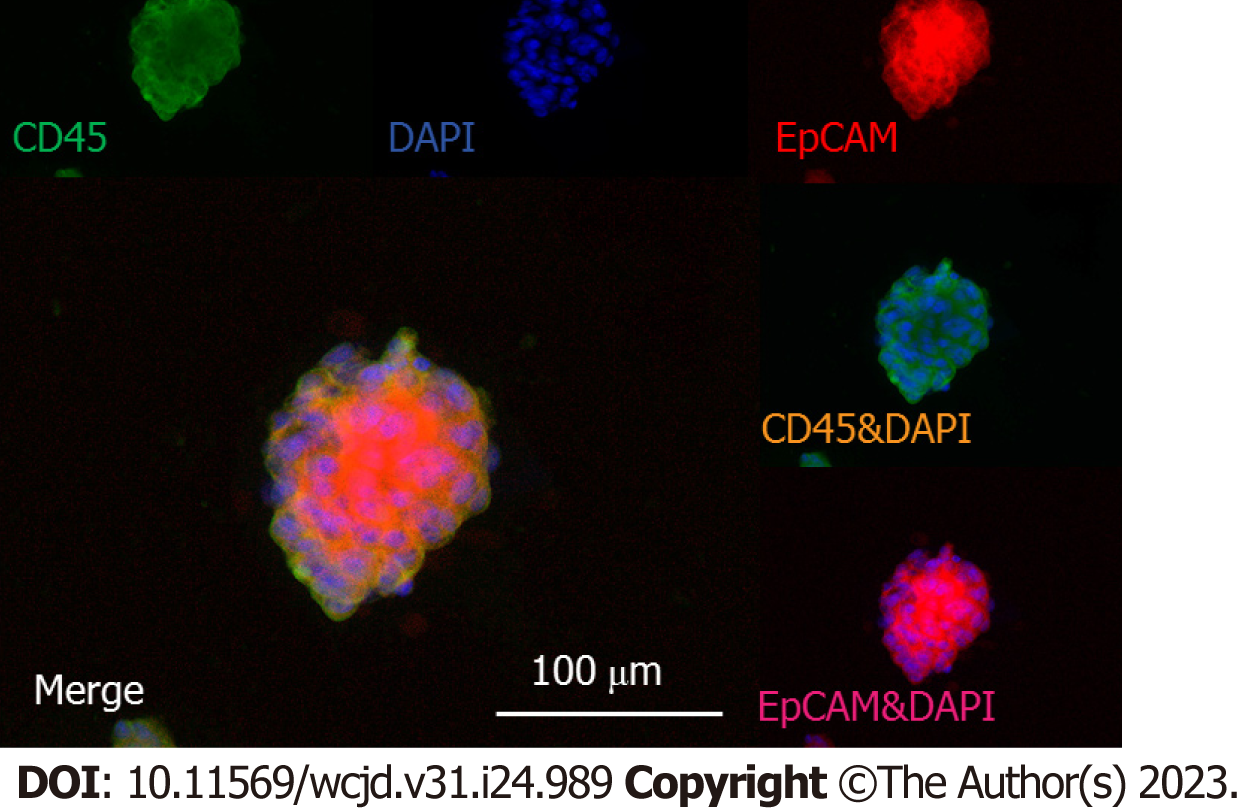
2.1.3 肝癌患者来源器官肿瘤球模型: 为了增强类器官的功能性, 研究者们提出了一种新颖的体外微流体培养系统, 该系统由胶原水凝胶中的上皮细胞和基质细胞组成[19]. 他们最近的一项研究验证了, 肿瘤产生的球状体(40-100 μm)含有相对类似比例的T淋巴细胞和髓样细胞群. 他们利用患者源性器官样肿瘤球(PDOTs)和鼠源性器官样肿瘤球状体(MDOTs)来评估对程序性死亡受体1(programmed death receptor1, PD-1)/细胞程序性死亡-配体1(programmed cell death 1 ligand 1, PD-L1)靶向疗法的短期反应, 而通过对培养上清中的细胞因子检测也可以作为评估免疫疗效的手段. PDOTs模型能够很好地再现体内免疫微环境(如图2). 此外, 这个模型以相对相似的比例反映了免疫微环境中的T淋巴细胞(CD3+、CD4+和CD8+)和髓样细胞(CD11b+和/或CD11c+)群体[33]. 最近的研究表明, CDK4/6抑制剂与抗PD-1抗体联合使用可以增加肿瘤细胞的死亡并减少趋化因子[34], 并且借助PDOTs模型证实了CDK4/6抑制剂增强抗PD-1抗体的免疫疗效.
在以往的研究中, Yong Beom Cho教授团队构建了来自肿瘤患者的器官样肿瘤球模型, 利用EpCAM和CD3抗体分别对肿瘤细胞和免疫细胞进行标识, 从而鉴定出PDOTs中存在的效应CD3+T细胞, 这类细胞在免疫疗法后的反应过程中具有必要的作用[18]. 因此, 我们能够清晰地确认在这个模型中, 肝肿瘤细胞和免疫细胞得到了成功的共培养, 并且他们的空间排列模式有效地模拟了肿瘤微环境. 这个模型对于深入理解肿瘤微环境并筛选免疫治疗药物具有巨大的研究潜力[35,36]. 此外, 这种微流控系统通过精细的隔离和设计良好的溶质扩散控制, 有效的模拟了体内肿瘤与血管、肿瘤与基质以及肿瘤与免疫微环境之间的相互作用. 此外, 这些培养系统可以用作药物的筛选工具. 因此, 它们有着实现高通量药物筛选的巨大潜力.
2.2.1 基因工程小鼠模型: 癌症基因组测序和突变分析为我们提供了丰富的信息, 揭示了许多高频突变的癌症基因, 例如MET, MYC, PTEN, RB1, TP53和CTNNB1, 以及涉及的信号通路, 如p53, WNT, NF-κB和TGF-β等[37-39]. 基因工程小鼠模型能进一步帮助我们理解这些癌症驱动基因在癌症发展中的作用. 利用Cre-LoxP重组, 我们可以在小鼠中诱导肝肿瘤, 如在AlfpCre+Trp53Δ2-10/Δ2-10小鼠中[40]. 同时, 利用流体动力学转染的Crispr-Cas9系统, 我们可以在几个月内在小鼠肝脏中诱导肿瘤 [41]. 有研究证明[42], 具有c-MYC和E2F1双转基因的小鼠在诱发肝癌的过程中, 发病率比只有一个基因转基因的小鼠要高.在MET过表达且CTNNB1缺失的小鼠中, 大多数在一个月内就出现了肝肿瘤[43]. 此外, 通过流体动力学注射将CRISPR-Cas9和睡美人系统构建的突变体输送到小鼠肝脏中, PTEN和p53被敲除, MET和CTNNB1被过表达, 这样的多基因组合突变的小鼠在六周内就出现了癌症[44]. 这些结果表明, 多基因突变能更快地、更高效地诱导肿瘤的形成.
尽管对PD-1通路的抑制作为一种治疗方法效果有限, 然而, 借助新型HCC基因工程小鼠模型揭示了CD8+T细胞在抑制外源抗原中的扮演的关键作用[45].研究进一步揭示了β-catenin在免疫逃逸过程中的激活作用, 并说明了β-catenin能通过削弱树突细胞的募集来促进免疫逃逸, 且CCL5能恢复免疫监视[46]. 总的来说, 基因工程小鼠模型为我们提供了一个研究平台, 使我们能够理解特定基因突变对肿瘤发生的影响, 并寻找可能的基因治疗的新靶点.
2.2.2 化学诱导的小鼠模型: 毒性化合物, 特别是肝癌诱导剂, 如二乙基硝胺(DEN)和黄曲霉毒素B1(AFB1), 会对肝脏产生直接或间接的影响, 破坏其稳态并引发肝细胞癌[47]. 这些化合物被广泛应用于构建小鼠肝癌模型, 帮助研究人员模拟人类疾病, 探索病因并评估潜在的治疗方案[48]. 首先, 基于DEN的化学诱导模型在小鼠肝癌研究中占有一席之地. 在一项研究中, 当小鼠在幼年期接触DEN, 然后在生长过程中反复给予四氯化碳(CCL4), 5个月后, 所有小鼠都能成功诱发肝癌[49]. 然而, 不同的年龄、性别和遗传背景会影响肿瘤发展的速度和方式. 例如, 一项实验研究发现, 幼年小鼠比成年小鼠更容易产生肝癌, 因为其肝细胞复制更快[50]. 其次, AFB1也是HCC的主要风险因素[51]. 举例来说, 研究发现, 7日龄的C57BL/6J和DBA/2J小鼠在接受相同剂量的AFB1注射后, DBA/2J小鼠的HCC发生率高达90%, 而C57BL/6J小鼠的发病率则只有27%[52]. 这种差异可能与这两种小鼠品系的基因特性有关, 这为我们找到AFB1诱导的肝细胞癌的潜在靶点提供了可能性.
然而, 这些化学诱导模型的有效性并非毫无问题. 一方面, 化学诱导的模型在一定程度上可以模拟肝细胞癌的发展过程, 包括肝损伤、肝硬化和肿瘤形成[48]. 但由于这种方式可能过于人为, 无法真实反映疾病发展的实际情况. 另一方面, 这些模型的制备时间长, 死亡率高, 并且癌症的发生时间、部位和数量存在个体差异. 据一项研究, 使用DEN和CCL4的传统化学诱导方法, 小鼠死亡率较高, 且癌症发生的时间和位置存在明显的个体差异[53]. 总之, 尽管化学诱导模型在肝癌研究中发挥了重要作用, 但我们仍需谨慎对待其局限性, 并积极寻找其他更有效的模型.
2.2.3 小鼠植入模型: 小鼠植入模型是研究肝细胞癌最常见的方法之一, 通过将肝癌细胞系或肿瘤组织片段注射到小鼠体内, 产生可靠、可重复的数据, 尤其在新抗癌药物和治疗策略的测试中广泛应用[54,55]. 按照肿瘤是否植入原始组织进行分类, 植入模型可分为原位模型和异位模型[56]. 异位模型通过皮下注射细胞或肿瘤组织, 便于直接观察肿瘤的生长和对抗癌药物的反应[57]. 然而, 原位模型在肝癌研究中更为普遍, 由于肝脏的复杂肿瘤微环境, 原位模型能更真实地模拟肝细胞癌的生理环境[58]. 不过, 原位植入需要更高技术挑战, 且需要注意小鼠肝脏微环境与人类的差异.
使用患者肝癌细胞系或肿瘤组织的异种移植模型, 必须在免疫缺陷小鼠中进行, 以避免产生排斥反应. 而无胸腺裸鼠和严重联合免疫缺陷小鼠则被广泛用于这种方法[59]. 在医学研究中, 非肥胖糖尿病严重联合免疫缺陷小鼠(non-obese diabetic severe combined immunodeficiency, NOD-SCID mice)以及基于NOD-SCID的NOD/LtSz SCID白细胞介素2受体共同γ链缺陷型(interleukin-2 receptor gamma chain null, IL2rγnull), 也称为NSG小鼠, 和NOD/Shi SCID IL2rβnull型(interleukin-2 receptor beta chain null, IL2rβnull), 也称为NOG小鼠, 在各类生物医学研究中得到了广泛的应用[60-62]. BALB/c小鼠中, 带有重组激活基因2(RAG2)基因失活(Rag2-null)和白细胞介素2受体γ链无效(IL2rγ-null)的品系, 以及新近开发的经过γ射线辐射免疫抑制(gamma irradiation immunosuppressed, GIS)的肿瘤异种移植小鼠模型, 均为新兴的免疫缺陷小鼠模型[63,64].
但这些模型均有其局限性, 同种异体移植小鼠虽然具有完整的免疫系统, 但其植入的组织或细胞来源于小鼠, 无法全面反映人类HCC的特性. 即使小鼠与人类有相似的基因, 人类和小鼠的免疫系统之间也存在显著的表型和功能差异. 因此, 我们需要在使用这些模型时充分认识到它们的局限性, 并寻求改进和更为有效的研究方法.
2.2.4 人源化小鼠模型: 人源化小鼠模型, 如NOG-TK-Tg小鼠[65]被设计来模拟人类的生理环境, 这是通过将人类的细胞、肿瘤、基因或功能性免疫系统植入到小鼠体内实现的[66]. 这些模型提供了一个高度复制人类肝脏功能的环境, 如表达肝脏特异性酶和成熟的人类肝脏基因表达谱, 以及呈现出人类特异性药物代谢模式[67-69].
对于免疫治疗研究, HIS小鼠模型的出现提供了重现人类免疫反应的可能性[70-72]. 例如, Hu PBL小鼠通过将外周血单核细胞移植到免疫缺陷小鼠中而产生, 用于研究病毒感染(如HIV或EB病毒)或移植物抗宿主(GVH)反应[60,70]. Hu HSC小鼠是通过注射人CD34+HSC并进行亚致死γ辐射, 以消除小鼠的HSC, 从而促进人HSC的移植, 该模型已成功应用于异种移植模型的开发[73,74]. 同时, BLT模型是通过将人胎儿胸腺/肝组织移植到小鼠肾包膜, 然后注射CD34+造血/祖细胞, 进行全身照射产生的[75]. 使用抗人分化簇45(抗hCD45)抗体处理的NSG小鼠和hCD45耗竭的人源化小鼠已用于移植HCC患者来源的异种移植物肿瘤组织[76]. 研究发现, 肿瘤微环境与肝癌之间存在复杂的相互作用, 肝癌细胞可能会改变肿瘤微环境以促进肿瘤生长[67,77-79].
然而, 尽管HIS人源化小鼠模型已在肿瘤生长、潜在靶点识别以及预测免疫治疗药物的效果和副作用方面取得了突出成绩, 但这些模型仍无法完全模拟肝细胞癌在人体肝脏环境中的生长[80,81]. 目前的研究正在探索新的人源化小鼠模型, 例如结合人肝嵌合小鼠与HIS人源化小鼠的模型, 以期能更好地模拟肿瘤的生长和进展[82].
临床前模型是生物医学研究的基础, 它们的使用有助于解析疾病的基础机制, 药物的作用方式, 以及进行新疗法的预筛选和优化. 然而, 这些模型都有其特定的优势和局限性, 如表1所示.
| 模型类型 | 优点 | 局限性 |
| 细胞系模型 | 易于培养和扩展-成本低-提供大量细胞供研究使用-适合高通量筛选 | 缺乏体内复杂的微环境-无法完全模拟肿瘤异质性和肿瘤-免疫细胞相互作用 |
| 类器官模型 | 保留了原始肿瘤组织的一些特征和结构 | 培养难度高-缺乏血管和免疫细胞的存在 |
| 小鼠异种移植模型 | 能够在活体动物中研究肿瘤的生长、治疗响应和转移能力 | 成本高, 周期长-小鼠免疫系统的差异限制了免疫治疗的研究 |
| 基因编辑小鼠模型 | 能够模拟人类肿瘤中的特定基因突变-研究基因在肿瘤发展中作用 | 成本高, 周期长-需要较长的时间和复杂的基因编辑技术 |
| 人源化小鼠模型 | 通过肿瘤和人类免疫系统的相互作用-更接近人体免疫环境的模型 | 成本高, 周期长, 成功率低-模型的复杂性和制备过程较为复杂 |
| 化学诱导肝癌小鼠模型 | 可以模拟肝癌发生的化学致癌过程-具有可控的诱导肝癌的能力 | 成本高, 周期长-无法考虑遗传因素对肝癌发展的影响-诱导过程可能与人类肝癌的发展过程有所差异 |
| PDOTS模型 | 成本低、周期短-能够保留肿瘤组织异质性和免疫微环境 | 对大规模研究的可行性有限-器官样本的获取和处理存在困难 |
总的来说, 细胞系模型是最常用的模型之一, 它们的优势在于可以在控制的实验环境下进行定量的生物化学和分子生物学实验[83,84]. 但是, 细胞系模型无法准确地反映复杂的生理环境和肿瘤的微环境, 包括免疫反应和细胞间相互作用. 小鼠异种移植模型(Xenograft Models)通过使用人类肿瘤细胞或组织, 更好地模拟了人类疾病的生物学特性[85,86]. 然而, 由于宿主小鼠的免疫系统被抑制, 这些模型不能准确地模拟人体的肿瘤免疫微环境[87]. 患者源性肿瘤异种移植模型能较好地保留原患者肿瘤的组织结构和基因表达谱, 提供了一个优秀的疗效预测平台[88,89]. 然而, 建立PDX模型需要较长时间, 成本较高, 仍然存在免疫环境模拟不足的问题[55,90]. 基因编辑小鼠模型在生理条件下研究特定基因在疾病发生和发展中的作用, 提供了一个重要的工具[91,92]. 然而, 基因编辑小鼠模型的制备时间长, 成本高, 并且不能完全反映疾病的遗传异质性[93,94]. 人源化小鼠模型模拟了人体肿瘤的免疫微环境, 为免疫治疗策略的评估提供了一个更为逼真的平台[95,96]. 但是, 人源化模型的制备复杂, 且可能存在人小鼠种间差异带来的影响[97,98]. 类器官模型克服了单细胞系统的缺陷, 可以在体外模拟生理环境, 包括细胞-细胞相互作用和细胞-基质相互作用[99,100]. 然而, 这些模型还无法完全模拟体内环境, 例如血流和免疫系统的影响[101,102]. PDOTs直接利用患者的肿瘤切片, 在体外环境中模拟肿瘤微环境[19,103]. PDOTs模型保留了原肿瘤的细胞异质性和结构, 可以更准确地评估药物的疗效. 甚至可以借助3D打印技术或者微流控芯片技术[104], 进行药物的高通量筛选. 尽管各种临床前模型都存在一定的局限性, 但他们的结合使用可以为我们提供了一个全方位的工具来理解疾病的复杂性, 以及为未来的临床研究打下基础.
在肝癌基础研究中, 整合多模态数据是未来提高临床前模型预测准确性和可靠性的关键因素. 随着越来越多高通量测序技术的应用, 基于转录组学、蛋白质组学、代谢组学、影像组学以及这些年逐渐兴起的单细胞组学、空间组学往往产生了大量的高维度数据. 利用机器学习以及生物信息学技术筛选出与临床表型密切相关的分子靶点, 并构建临床预测模型或者具有某些特定预测功能的"Panels". 近年来, 对于肝癌的分类和预测模型研究取得了一系列的进展. 复旦大学附属中山医院肝外科团队通过整合多组学的研究, 识别了肝癌的三种亚型: 微环境失调型、肿瘤增殖型和肿瘤代谢型, 并据此提出了一个创新的肝癌"肿瘤-微环境"预测模型[105,106]. 南通大学附属医院陈钟团队, 基于以往的研究, 构建了与泛凋亡相关的预测模型[107]. 根据此模型, 肝癌被进一步分类为三个亚型: SFN+PDK4-型、SFN-PDK4+型和SFN/PDK4 intermediate expression型. 值得注意的是, 这三个亚型在免疫治疗和靶向治疗的敏感性上表现出显著的差异性. 相比以往对单一靶点、单一组学的筛选, 随着单细胞转录组测序、空间转录组、单细胞ATAC测序、空间代谢组学等先进测序技术的普及, 通过对这些高维数据的解读结合必要的临床前模型的验证, 更能有望克服肝癌的时空异质性带来的干扰, 从而实现患者的个体化、精准化的免疫治疗.
在不断深化的多模态数据分析和精准医学的驱动下, 未来的临床前模型发展将不仅仅局限于肿瘤的静态特征描述, 而是趋向于动态、多维的治疗响应预测. 随着个性化医疗的需求日益增长, 结合实时监测和实验模型的可持续性将成为研究重点. 例如, 动态模拟技术的融入, 可以实时跟踪药物在肿瘤微环境中的分布和效应, 捕捉疗效的即时变化, 为临床决策提供更加精准的数据支持. 此外, 随着仿生技术和三维打印技术的进步, 未来的临床前模型将更加生物仿真, 能够更真实地复制肿瘤微环境和器官间的相互作用, 提供更为全面的治疗效果评估. 这些技术进步预示着一个新时代的到来, 临床前模型不再是简单的药物筛选工具, 而是成为了评估治疗策略、指导个性化治疗方案、并预测治疗结果的复合系统.
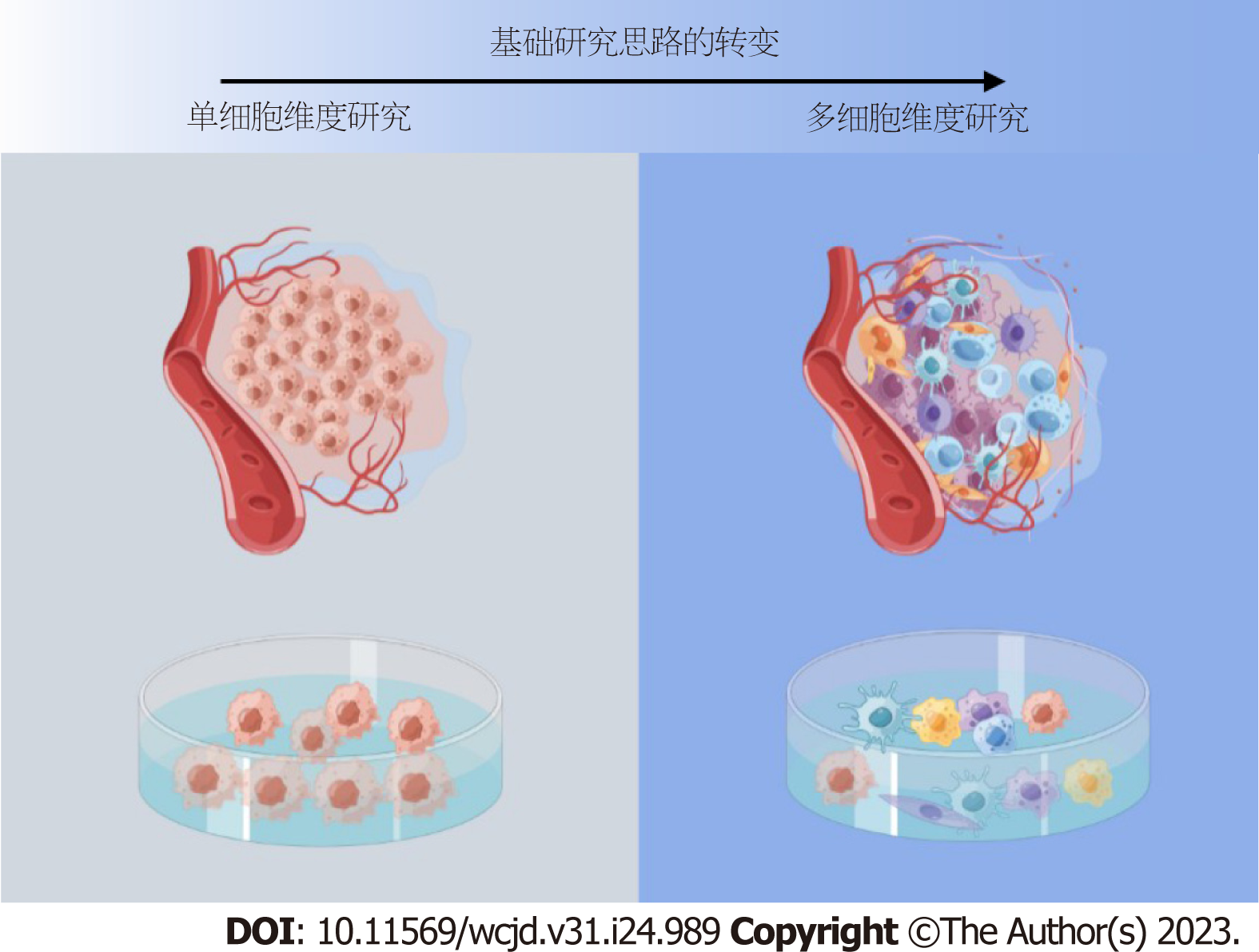
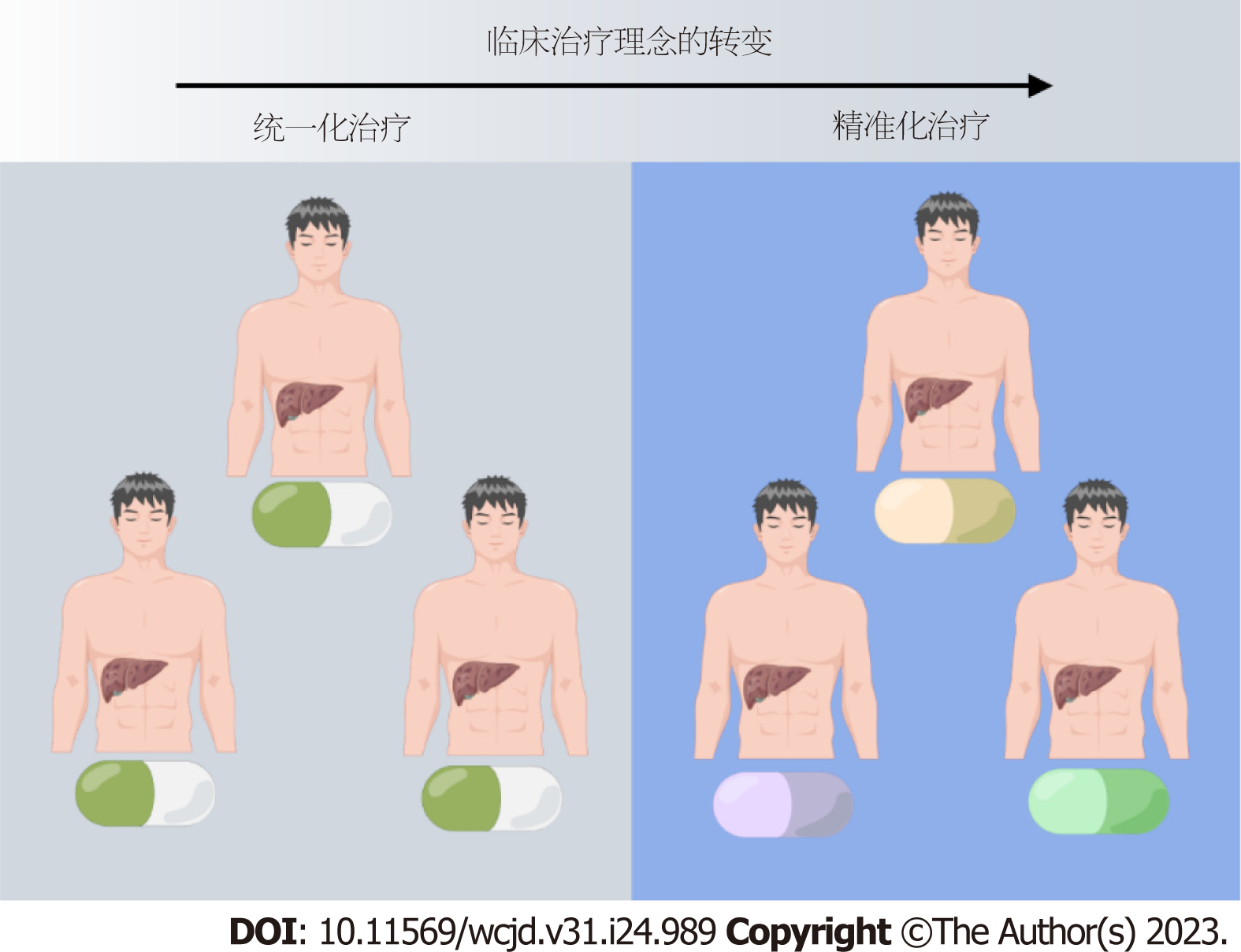
展望未来, 除了基础研究思路由单个细胞维度的研究向多细胞维度的改变, 如图3, 我们所抱持的临床治疗理念也正在发生根本性的转变, 从以往的一体化模式, 逐渐转向以个性化和精准化为特色的医疗模式, 如图4. 这一转变的推动力在于肝癌的显著异质性, 这一特性使得肝癌区别于其他类型的肿瘤. 首先, 从基因层面上看, 肝癌患者本身的个体异质性极强, 各个患者的基因特征各不相同. 其次, 肿瘤内部也存在显著的空间异质性, 这主要体现在不同部位的肿瘤微环境差异. 最后, 对于同一患者而言, 其肿瘤在不同的治疗阶段也会由于肿瘤亚克隆等因素产生干性和耐药性等表型的变化. 这些因素共同导致同一种治疗方案在不同的患者以及不同的治疗阶段产生显著的效果差异. 因此, 如何运用临床前模型的特性, 创新性地构建能与个性化和精准化治疗相匹配的临床前模型, 是我们需要深入思考和探讨的问题.
综上而言, 本文全面回顾了肝癌临床前模型的各项优势和局限, 其中包括经典的细胞系模型、患者来源性肿瘤移植小鼠模型、基因编辑小鼠模型、化学诱导肝癌小鼠模型、人源化小鼠模型、类器官模型以及PDOTs模型等. 我们充分认识到, 各种模型均在疾病机理的探索、新药筛选和精准化治疗策略的研究中起到了不可或缺的作用, 但同样面临着在肿瘤异质性、免疫微环境模拟及药物筛选高通量化等方面的挑战. 因此, 如何在新的研究中更好地优化和应用这些模型, 将最终实现肝癌患者的个性化精准化治疗, 是我们当前及未来需要深入思考和探索的问题.
学科分类: 胃肠病学和肝病学
手稿来源地: 江苏省
同行评议报告学术质量分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): C, C, C
D级 (一般): D
E级 (差): 0
科学编辑: 张砚梁 制作编辑:张砚梁
| 1. | Siegel RL, Miller KD, Jemal A. Cancer Statistics, 2017. CA Cancer J Clin. 2017;67:7-30. [PubMed] [DOI] |
| 2. | Candia J, Bayarsaikhan E, Tandon M, Budhu A, Forgues M, Tovuu LO, Tudev U, Lack J, Chao A, Chinburen J, Wang XW. The genomic landscape of Mongolian hepatocellular carcinoma. Nat Commun. 2020;11:4383. [PubMed] [DOI] |
| 3. | Paik DT, Chandy M, Wu JC. Patient and Disease-Specific Induced Pluripotent Stem Cells for Discovery of Personalized Cardiovascular Drugs and Therapeutics. Pharmacol Rev. 2020;72:320-342. [PubMed] [DOI] |
| 4. | Jilaveanu LB, Puligandla M, Weiss SA, Wang XV, Zito C, Flaherty KT, Boeke M, Neumeister V, Camp RL, Adeniran A, Pins M, Manola J, DiPaola RS, Haas NB, Kluger HM. Tumor Microvessel Density as a Prognostic Marker in High-Risk Renal Cell Carcinoma Patients Treated on ECOG-ACRIN E2805. Clin Cancer Res. 2018;24:217-223. [PubMed] [DOI] |
| 5. | Toh TB, Lim JJ, Hooi L, Rashid MBMA, Chow EK. Targeting Jak/Stat pathway as a therapeutic strategy against SP/CD44+ tumorigenic cells in Akt/β-catenin-driven hepatocellular carcinoma. J Hepatol. 2020;72:104-118. [PubMed] [DOI] |
| 6. | Hayes CN, Zhang P, Chayama K. The Role of Lipids in Hepatocellular Carcinoma. Brisbane (AU): Codon Publications. 2019-Oct-24. [PubMed] |
| 7. | Losic B, Craig AJ, Villacorta-Martin C, Martins-Filho SN, Akers N, Chen X, Ahsen ME, von Felden J, Labgaa I, DʹAvola D, Allette K, Lira SA, Furtado GC, Garcia-Lezana T, Restrepo P, Stueck A, Ward SC, Fiel MI, Hiotis SP, Gunasekaran G, Sia D, Schadt EE, Sebra R, Schwartz M, Llovet JM, Thung S, Stolovitzky G, Villanueva A. Intratumoral heterogeneity and clonal evolution in liver cancer. Nat Commun. 2020;11:291. [PubMed] [DOI] |
| 8. | Pamarthy S, Sabaawy HE. Patient derived organoids in prostate cancer: improving therapeutic efficacy in precision medicine. Mol Cancer. 2021;20:125. [PubMed] [DOI] |
| 9. | Xie C, Gu A, Khan M, Yao X, Chen L, He J, Yuan F, Wang P, Yang Y, Wei Y, Tang F, Su H, Chen J, Li J, Cen B, Xu Z. Opportunities and challenges of hepatocellular carcinoma organoids for targeted drugs sensitivity screening. Front Oncol. 2022;12:1105454. [PubMed] [DOI] |
| 10. | Meng X, Wu T, Hong Y, Fan Q, Ren Z, Guo Y, Yang X, Shi P, Yang J, Yin X, Luo Z, Xia J, Zhou Y, Xu M, Liu E, Jiang G, Li S, Zhao F, Ma C, Ma C, Hou Z, Li J, Wang J, Wang F. Camrelizumab plus apatinib as second-line treatment for advanced oesophageal squamous cell carcinoma (CAP 02): a single-arm, open-label, phase 2 trial. Lancet Gastroenterol Hepatol. 2022;7:245-253. [PubMed] [DOI] |
| 11. | Finn RS, Ikeda M, Zhu AX, Sung MW, Baron AD, Kudo M, Okusaka T, Kobayashi M, Kumada H, Kaneko S, Pracht M, Mamontov K, Meyer T, Kubota T, Dutcus CE, Saito K, Siegel AB, Dubrovsky L, Mody K, Llovet JM. Phase Ib Study of Lenvatinib Plus Pembrolizumab in Patients With Unresectable Hepatocellular Carcinoma. J Clin Oncol. 2020;38:2960-2970. [PubMed] [DOI] |
| 12. | Bleijs M, van de Wetering M, Clevers H, Drost J. Xenograft and organoid model systems in cancer research. EMBO J. 2019;38:e101654. [PubMed] [DOI] |
| 13. | Honkala A, Malhotra SV, Kummar S, Junttila MR. Harnessing the predictive power of preclinical models for oncology drug development. Nat Rev Drug Discov. 2022;21:99-114. [PubMed] [DOI] |
| 14. | Prior N, Inacio P, Huch M. Liver organoids: from basic research to therapeutic applications. Gut. 2019;68:2228-2237. [PubMed] [DOI] |
| 15. | Pyo DH, Hong HK, Lee WY, Cho YB. Patient-derived cancer modeling for precision medicine in colorectal cancer: beyond the cancer cell line. Cancer Biol Ther. 2020;21:495-502. [PubMed] [DOI] |
| 16. | Lin YL, Li Y. Study on the hepatocellular carcinoma model with metastasis. Genes Dis. 2020;7:336-350. [PubMed] [DOI] |
| 17. | Koga Y, Ochiai A. Systematic Review of Patient-Derived Xenograft Models for Preclinical Studies of Anti-Cancer Drugs in Solid Tumors. Cells. 2019;8. [PubMed] [DOI] |
| 18. | Hong HK, Yun NH, Jeong YL, Park J, Doh J, Lee WY, Cho YB. Establishment of patient-derived organotypic tumor spheroid models for tumor microenvironment modeling. Cancer Med. 2021;10:5589-5598. [PubMed] [DOI] |
| 19. | Jenkins RW, Aref AR, Lizotte PH, Ivanova E, Stinson S, Zhou CW, Bowden M, Deng J, Liu H, Miao D, He MX, Walker W, Zhang G, Tian T, Cheng C, Wei Z, Palakurthi S, Bittinger M, Vitzthum H, Kim JW, Merlino A, Quinn M, Venkataramani C, Kaplan JA, Portell A, Gokhale PC, Phillips B, Smart A, Rotem A, Jones RE, Keogh L, Anguiano M, Stapleton L, Jia Z, Barzily-Rokni M, Cañadas I, Thai TC, Hammond MR, Vlahos R, Wang ES, Zhang H, Li S, Hanna GJ, Huang W, Hoang MP, Piris A, Eliane JP, Stemmer-Rachamimov AO, Cameron L, Su MJ, Shah P, Izar B, Thakuria M, LeBoeuf NR, Rabinowits G, Gunda V, Parangi S, Cleary JM, Miller BC, Kitajima S, Thummalapalli R, Miao B, Barbie TU, Sivathanu V, Wong J, Richards WG, Bueno R, Yoon CH, Miret J, Herlyn M, Garraway LA, Van Allen EM, Freeman GJ, Kirschmeier PT, Lorch JH, Ott PA, Hodi FS, Flaherty KT, Kamm RD, Boland GM, Wong KK, Dornan D, Paweletz CP, Barbie DA. Ex Vivo Profiling of PD-1 Blockade Using Organotypic Tumor Spheroids. Cancer Discov. 2018;8:196-215. [PubMed] [DOI] |
| 20. | Meng F, Meyer CM, Joung D, Vallera DA, McAlpine MC, Panoskaltsis-Mortari A. 3D Bioprinted In Vitro Metastatic Models via Reconstruction of Tumor Microenvironments. Adv Mater. 2019;31:e1806899. [PubMed] [DOI] |
| 21. | Nikolic M, Sustersic T, Filipovic N. In vitro Models and On-Chip Systems: Biomaterial Interaction Studies With Tissues Generated Using Lung Epithelial and Liver Metabolic Cell Lines. Front Bioeng Biotechnol. 2018;6:120. [PubMed] [DOI] |
| 22. | Monteil M, Migianu-Griffoni E, Sainte-Catherine O, Di Benedetto M, Lecouvey M. Bisphosphonate prodrugs: synthesis and biological evaluation in HuH7 hepatocarcinoma cells. Eur J Med Chem. 2014;77:56-64. [PubMed] [DOI] |
| 23. | Cavalloni G, Peraldo-Neia C, Varamo C, Casorzo L, Dell'Aglio C, Bernabei P, Chiorino G, Aglietta M, Leone F. Establishment and characterization of a human intrahepatic cholangiocarcinoma cell line derived from an Italian patient. Tumour Biol. 2016;37:4041-4052. [PubMed] [DOI] |
| 24. | López-Terrada D, Cheung SW, Finegold MJ, Knowles BB. Hep G2 is a hepatoblastoma-derived cell line. Hum Pathol. 2009;40:1512-1515. [PubMed] [DOI] |
| 25. | Ni Y, Lempp FA, Mehrle S, Nkongolo S, Kaufman C, Fälth M, Stindt J, Königer C, Nassal M, Kubitz R, Sültmann H, Urban S. Hepatitis B and D viruses exploit sodium taurocholate co-transporting polypeptide for species-specific entry into hepatocytes. Gastroenterology. 2014;146:1070-1083. [PubMed] [DOI] |
| 26. | Mutz P, Metz P, Lempp FA, Bender S, Qu B, Schöneweis K, Seitz S, Tu T, Restuccia A, Frankish J, Dächert C, Schusser B, Koschny R, Polychronidis G, Schemmer P, Hoffmann K, Baumert TF, Binder M, Urban S, Bartenschlager R. HBV Bypasses the Innate Immune Response and Does Not Protect HCV From Antiviral Activity of Interferon. Gastroenterology. 2018;154:1791-1804.e22. [PubMed] [DOI] |
| 27. | Watashi K, Liang G, Iwamoto M, Marusawa H, Uchida N, Daito T, Kitamura K, Muramatsu M, Ohashi H, Kiyohara T, Suzuki R, Li J, Tong S, Tanaka Y, Murata K, Aizaki H, Wakita T. Interleukin-1 and tumor necrosis factor-α trigger restriction of hepatitis B virus infection via a cytidine deaminase activation-induced cytidine deaminase (AID). J Biol Chem. 2013;288:31715-31727. [PubMed] [DOI] |
| 28. | Drost J, Clevers H. Organoids in cancer research. Nat Rev Cancer. 2018;18:407-418. [PubMed] [DOI] |
| 29. | Kim J, Koo BK, Knoblich JA. Human organoids: model systems for human biology and medicine. Nat Rev Mol Cell Biol. 2020;21:571-584. [PubMed] [DOI] |
| 30. | Broutier L, Mastrogiovanni G, Verstegen MM, Francies HE, Gavarró LM, Bradshaw CR, Allen GE, Arnes-Benito R, Sidorova O, Gaspersz MP, Georgakopoulos N, Koo BK, Dietmann S, Davies SE, Praseedom RK, Lieshout R, IJzermans JNM, Wigmore SJ, Saeb-Parsy K, Garnett MJ, van der Laan LJ, Huch M. Human primary liver cancer-derived organoid cultures for disease modeling and drug screening. Nat Med. 2017;23:1424-1435. [PubMed] [DOI] |
| 31. | Kang Y, Deng J, Ling J, Li X, Chiang YJ, Koay EJ, Wang H, Burks JK, Chiao PJ, Hurd MW, Bhutani MS, Lee JH, Weston BR, Maitra A, Ikoma N, Tzeng CD, Lee JE, DePinho RA, Wolff RA, Pant S, McAllister F, Katz MH, Fleming JB, Kim MP. 3D imaging analysis on an organoid-based platform guides personalized treatment in pancreatic ductal adenocarcinoma. J Clin Invest. 2022;132. [PubMed] [DOI] |
| 32. | Wang S, Wang Y, Xun X, Zhang C, Xiang X, Cheng Q, Hu S, Li Z, Zhu J. Hedgehog signaling promotes sorafenib resistance in hepatocellular carcinoma patient-derived organoids. J Exp Clin Cancer Res. 2020;39:22. [PubMed] [DOI] |
| 33. | Balko JM, Sosman JA. A Critical Need for Better Cancer Immunotherapy Models: Are Organotypic Tumor Spheroid Cultures the Answer? Cancer Discov. 2018;8:143-145. [PubMed] [DOI] |
| 34. | Deng J, Wang ES, Jenkins RW, Li S, Dries R, Yates K, Chhabra S, Huang W, Liu H, Aref AR, Ivanova E, Paweletz CP, Bowden M, Zhou CW, Herter-Sprie GS, Sorrentino JA, Bisi JE, Lizotte PH, Merlino AA, Quinn MM, Bufe LE, Yang A, Zhang Y, Zhang H, Gao P, Chen T, Cavanaugh ME, Rode AJ, Haines E, Roberts PJ, Strum JC, Richards WG, Lorch JH, Parangi S, Gunda V, Boland GM, Bueno R, Palakurthi S, Freeman GJ, Ritz J, Haining WN, Sharpless NE, Arthanari H, Shapiro GI, Barbie DA, Gray NS, Wong KK. CDK4/6 Inhibition Augments Antitumor Immunity by Enhancing T-cell Activation. Cancer Discov. 2018;8:216-233. [PubMed] [DOI] |
| 35. | Mao C, Yeh S, Fu J, Porosnicu M, Thomas A, Kucera GL, Votanopoulos KI, Tian S, Ming X. Delivery of an ectonucleotidase inhibitor with ROS-responsive nanoparticles overcomes adenosine-mediated cancer immunosuppression. Sci Transl Med. 2022;14:eabh1261. [PubMed] [DOI] |
| 36. | Cañadas I, Thummalapalli R, Kim JW, Kitajima S, Jenkins RW, Christensen CL, Campisi M, Kuang Y, Zhang Y, Gjini E, Zhang G, Tian T, Sen DR, Miao D, Imamura Y, Thai T, Piel B, Terai H, Aref AR, Hagan T, Koyama S, Watanabe M, Baba H, Adeni AE, Lydon CA, Tamayo P, Wei Z, Herlyn M, Barbie TU, Uppaluri R, Sholl LM, Sicinska E, Sands J, Rodig S, Wong KK, Paweletz CP, Watanabe H, Barbie DA. Tumor innate immunity primed by specific interferon-stimulated endogenous retroviruses. Nat Med. 2018;24:1143-1150. [PubMed] [DOI] |
| 37. | Schulze K, Imbeaud S, Letouzé E, Alexandrov LB, Calderaro J, Rebouissou S, Couchy G, Meiller C, Shinde J, Soysouvanh F, Calatayud AL, Pinyol R, Pelletier L, Balabaud C, Laurent A, Blanc JF, Mazzaferro V, Calvo F, Villanueva A, Nault JC, Bioulac-Sage P, Stratton MR, Llovet JM, Zucman-Rossi J. Exome sequencing of hepatocellular carcinomas identifies new mutational signatures and potential therapeutic targets. Nat Genet. 2015;47:505-511. [PubMed] [DOI] |
| 38. | Xu LX, He MH, Dai ZH, Yu J, Wang JG, Li XC, Jiang BB, Ke ZF, Su TH, Peng ZW, Guo Y, Chen ZB, Chen SL, Peng S, Kuang M. Genomic and transcriptional heterogeneity of multifocal hepatocellular carcinoma. Ann Oncol. 2019;30:990-997. [PubMed] [DOI] |
| 39. | Shibata T, Aburatani H. Exploration of liver cancer genomes. Nat Rev Gastroenterol Hepatol. 2014;11:340-349. [PubMed] [DOI] |
| 40. | Katz SF, Lechel A, Obenauf AC, Begus-Nahrmann Y, Kraus JM, Hoffmann EM, Duda J, Eshraghi P, Hartmann D, Liss B, Schirmacher P, Kestler HA, Speicher MR, Rudolph KL. Disruption of Trp53 in livers of mice induces formation of carcinomas with bilineal differentiation. Gastroenterology. 2012;142:1229-1239.e3. [PubMed] [DOI] |
| 41. | Liu F, Song Y, Liu D. Hydrodynamics-based transfection in animals by systemic administration of plasmid DNA. Gene Ther. 1999;6:1258-1266. [PubMed] [DOI] |
| 42. | Calvisi DF, Conner EA, Ladu S, Lemmer ER, Factor VM, Thorgeirsson SS. Activation of the canonical Wnt/beta-catenin pathway confers growth advantages in c-Myc/E2F1 transgenic mouse model of liver cancer. J Hepatol. 2005;42:842-849. [PubMed] [DOI] |
| 43. | Tward AD, Jones KD, Yant S, Cheung ST, Fan ST, Chen X, Kay MA, Wang R, Bishop JM. Distinct pathways of genomic progression to benign and malignant tumors of the liver. Proc Natl Acad Sci USA. 2007;104:14771-14776. [PubMed] [DOI] |
| 44. | Cao X, Zhang Y, Zhou Q, Sun S, He M, Wang X, Ma P, Yang X, Lv L, Zhan L. Establishment of a Novel Mouse Hepatocellular Carcinoma Model for Dynamic Monitoring of Tumor Development by Bioluminescence Imaging. Front Oncol. 2022;12:794101. [PubMed] [DOI] |
| 45. | Ott PA, Hodi FS, Kaufman HL, Wigginton JM, Wolchok JD. Combination immunotherapy: a road map. J Immunother Cancer. 2017;5:16. [PubMed] [DOI] |
| 46. | Ruiz de Galarreta M, Bresnahan E, Molina-Sánchez P, Lindblad KE, Maier B, Sia D, Puigvehi M, Miguela V, Casanova-Acebes M, Dhainaut M, Villacorta-Martin C, Singhi AD, Moghe A, von Felden J, Tal Grinspan L, Wang S, Kamphorst AO, Monga SP, Brown BD, Villanueva A, Llovet JM, Merad M, Lujambio A. β-Catenin Activation Promotes Immune Escape and Resistance to Anti-PD-1 Therapy in Hepatocellular Carcinoma. Cancer Discov. 2019;9:1124-1141. [PubMed] [DOI] |
| 47. | Verna L, Whysner J, Williams GM. N-nitrosodiethylamine mechanistic data and risk assessment: bioactivation, DNA-adduct formation, mutagenicity, and tumor initiation. Pharmacol Ther. 1996;71:57-81. [PubMed] [DOI] |
| 48. | Connor F, Rayner TF, Aitken SJ, Feig C, Lukk M, Santoyo-Lopez J, Odom DT. Mutational landscape of a chemically-induced mouse model of liver cancer. J Hepatol. 2018;69:840-850. [PubMed] [DOI] |
| 49. | Berger ML, Bhatt H, Combes B, Estabrook RW. CCl4-induced toxicity in isolated hepatocytes: the importance of direct solvent injury. Hepatology. 1986;6:36-45. [PubMed] [DOI] |
| 50. | Vesselinovitch SD, Mihailovich N. Kinetics of diethylnitrosamine hepatocarcinogenesis in the infant mouse. Cancer Res. 1983;43:4253-4259. [PubMed] |
| 51. | Fox JG, Feng Y, Theve EJ, Raczynski AR, Fiala JL, Doernte AL, Williams M, McFaline JL, Essigmann JM, Schauer DB, Tannenbaum SR, Dedon PC, Weinman SA, Lemon SM, Fry RC, Rogers AB. Gut microbes define liver cancer risk in mice exposed to chemical and viral transgenic hepatocarcinogens. Gut. 2010;59:88-97. [PubMed] [DOI] |
| 52. | Leenders MW, Nijkamp MW, Borel Rinkes IH. Mouse models in liver cancer research: a review of current literature. World J Gastroenterol. 2008;14:6915-6923. [PubMed] [DOI] |
| 53. | McGlynn KA, Hunter K, LeVoyer T, Roush J, Wise P, Michielli RA, Shen FM, Evans AA, London WT, Buetow KH. Susceptibility to aflatoxin B1-related primary hepatocellular carcinoma in mice and humans. Cancer Res. 2003;63:4594-4601. [PubMed] |
| 54. | Hernandez-Gea V, Toffanin S, Friedman SL, Llovet JM. Role of the microenvironment in the pathogenesis and treatment of hepatocellular carcinoma. Gastroenterology. 2013;144:512-527. [PubMed] [DOI] |
| 55. | Hidalgo M, Amant F, Biankin AV, Budinská E, Byrne AT, Caldas C, Clarke RB, de Jong S, Jonkers J, Mælandsmo GM, Roman-Roman S, Seoane J, Trusolino L, Villanueva A. Patient-derived xenograft models: an emerging platform for translational cancer research. Cancer Discov. 2014;4:998-1013. [PubMed] [DOI] |
| 56. | Boetto J, Peyre M, Kalamarides M. Mouse Models in Meningioma Research: A Systematic Review. Cancers (Basel). 2021;13. [PubMed] [DOI] |
| 57. | Hu B, Li H, Guo W, Sun YF, Zhang X, Tang WG, Yang LX, Xu Y, Tang XY, Ding GH, Qiu SJ, Zhou J, Li YX, Fan J, Yang XR. Establishment of a hepatocellular carcinoma patient-derived xenograft platform and its application in biomarker identification. Int J Cancer. 2020;146:1606-1617. [PubMed] [DOI] |
| 58. | Nadarevic T, Giljaca V, Colli A, Fraquelli M, Casazza G, Miletic D, Štimac D. Computed tomography for the diagnosis of hepatocellular carcinoma in adults with chronic liver disease. Cochrane Database Syst Rev. 2021;10:CD013362. [PubMed] [DOI] |
| 59. | Duchosal MA, Eming SA, McConahey PJ, Dixon FJ. The hu-PBL-SCID mouse model. Long-term human serologic evolution associated with the xenogeneic transfer of human peripheral blood leukocytes into SCID mice. Cell Immunol. 1992;139:468-477. [PubMed] [DOI] |
| 60. | Ito M, Hiramatsu H, Kobayashi K, Suzue K, Kawahata M, Hioki K, Ueyama Y, Koyanagi Y, Sugamura K, Tsuji K, Heike T, Nakahata T. NOD/SCID/gamma(c)(null) mouse: an excellent recipient mouse model for engraftment of human cells. Blood. 2002;100:3175-3182. [PubMed] [DOI] |
| 61. | Shultz LD, Lyons BL, Burzenski LM, Gott B, Chen X, Chaleff S, Kotb M, Gillies SD, King M, Mangada J, Greiner DL, Handgretinger R. Human lymphoid and myeloid cell development in NOD/LtSz-scid IL2R gamma null mice engrafted with mobilized human hemopoietic stem cells. J Immunol. 2005;174:6477-6489. [PubMed] [DOI] |
| 62. | Ito M, Kobayashi K, Nakahata T. NOD/Shi-scid IL2rgamma(null) (NOG) mice more appropriate for humanized mouse models. Curr Top Microbiol Immunol. 2008;324:53-76. [PubMed] [DOI] |
| 63. | Traggiai E, Chicha L, Mazzucchelli L, Bronz L, Piffaretti JC, Lanzavecchia A, Manz MG. Development of a human adaptive immune system in cord blood cell-transplanted mice. Science. 2004;304:104-107. [PubMed] [DOI] |
| 64. | Dhanasekaran R, Bandoh S, Roberts LR. Molecular pathogenesis of hepatocellular carcinoma and impact of therapeutic advances. F1000Res. 2016;5. [PubMed] [DOI] |
| 65. | Hasegawa M, Kawai K, Mitsui T, Taniguchi K, Monnai M, Wakui M, Ito M, Suematsu M, Peltz G, Nakamura M, Suemizu H. The reconstituted 'humanized liver' in TK-NOG mice is mature and functional. Biochem Biophys Res Commun. 2011;405:405-410. [PubMed] [DOI] |
| 66. | Wang M, Yao LC, Cheng M, Cai D, Martinek J, Pan CX, Shi W, Ma AH, De Vere White RW, Airhart S, Liu ET, Banchereau J, Brehm MA, Greiner DL, Shultz LD, Palucka K, Keck JG. Humanized mice in studying efficacy and mechanisms of PD-1-targeted cancer immunotherapy. FASEB J. 2018;32:1537-1549. [PubMed] [DOI] |
| 67. | Zhao Y, Shuen TWH, Toh TB, Chan XY, Liu M, Tan SY, Fan Y, Yang H, Lyer SG, Bonney GK, Loh E, Chang KTE, Tan TC, Zhai W, Chan JKY, Chow EK, Chee CE, Lee GH, Dan YY, Chow PK, Toh HC, Lim SG, Chen Q. Development of a new patient-derived xenograft humanised mouse model to study human-specific tumour microenvironment and immunotherapy. Gut. 2018;67:1845-1854. [PubMed] [DOI] |
| 68. | Azuma H, Paulk N, Ranade A, Dorrell C, Al-Dhalimy M, Ellis E, Strom S, Kay MA, Finegold M, Grompe M. Robust expansion of human hepatocytes in Fah-/-/Rag2-/-/Il2rg-/- mice. Nat Biotechnol. 2007;25:903-910. [PubMed] [DOI] |
| 69. | Zhou ZF, Peng F, Li JY, Ye YB. Intratumoral IL-12 Gene Therapy Inhibits Tumor Growth In A HCC-Hu-PBL-NOD/SCID Murine Model. Onco Targets Ther. 2019;12:7773-7784. [PubMed] [DOI] |
| 70. | Bhargavan B, Woollard SM, McMillan JE, Kanmogne GD. CCR5 antagonist reduces HIV-induced amyloidogenesis, tau pathology, neurodegeneration, and blood-brain barrier alterations in HIV-infected hu-PBL-NSG mice. Mol Neurodegener. 2021;16:78. [PubMed] [DOI] |
| 71. | Dierksheide JE, Baiocchi RA, Ferketich AK, Roychowdhury S, Pelletier RP, Eisenbeis CF, Caligiuri MA, VanBuskirk AM. IFN-gamma gene polymorphisms associate with development of EBV+ lymphoproliferative disease in hu PBL-SCID mice. Blood. 2005;105:1558-1565. [PubMed] [DOI] |
| 72. | Washburn ML, Bility MT, Zhang L, Kovalev GI, Buntzman A, Frelinger JA, Barry W, Ploss A, Rice CM, Su L. A humanized mouse model to study hepatitis C virus infection, immune response, and liver disease. Gastroenterology. 2011;140:1334-1344. [PubMed] [DOI] |
| 73. | Czechowicz A, Kraft D, Weissman IL, Bhattacharya D. Efficient transplantation via antibody-based clearance of hematopoietic stem cell niches. Science. 2007;318:1296-1299. [PubMed] [DOI] |
| 74. | Lan P, Tonomura N, Shimizu A, Wang S, Yang YG. Reconstitution of a functional human immune system in immunodeficient mice through combined human fetal thymus/liver and CD34+ cell transplantation. Blood. 2006;108:487-492. [PubMed] [DOI] |
| 75. | Melkus MW, Estes JD, Padgett-Thomas A, Gatlin J, Denton PW, Othieno FA, Wege AK, Haase AT, Garcia JV. Humanized mice mount specific adaptive and innate immune responses to EBV and TSST-1. Nat Med. 2006;12:1316-1322. [PubMed] [DOI] |
| 76. | Zhou SL, Yin D, Hu ZQ, Luo CB, Zhou ZJ, Xin HY, Yang XR, Shi YH, Wang Z, Huang XW, Cao Y, Fan J, Zhou J. A Positive Feedback Loop Between Cancer Stem-Like Cells and Tumor-Associated Neutrophils Controls Hepatocellular Carcinoma Progression. Hepatology. 2019;70:1214-1230. [PubMed] [DOI] |
| 77. | Song M, He J, Pan QZ, Yang J, Zhao J, Zhang YJ, Huang Y, Tang Y, Wang Q, He J, Gu J, Li Y, Chen S, Zeng J, Zhou ZQ, Yang C, Han Y, Chen H, Xiang T, Weng DS, Xia JC. Cancer-Associated Fibroblast-Mediated Cellular Crosstalk Supports Hepatocellular Carcinoma Progression. Hepatology. 2021;73:1717-1735. [PubMed] [DOI] |
| 78. | Craig AJ, von Felden J, Garcia-Lezana T, Sarcognato S, Villanueva A. Tumour evolution in hepatocellular carcinoma. Nat Rev Gastroenterol Hepatol. 2020;17:139-152. [PubMed] [DOI] |
| 79. | Asai A, Yasuoka H, Matsui M, Tsuchimoto Y, Fukunishi S, Higuchi K. Programmed Death 1 Ligand Expression in the Monocytes of Patients with Hepatocellular Carcinoma Depends on Tumor Progression. Cancers (Basel). 2020;12. [PubMed] [DOI] |
| 80. | Tsuchida T, Lee YA, Fujiwara N, Ybanez M, Allen B, Martins S, Fiel MI, Goossens N, Chou HI, Hoshida Y, Friedman SL. A simple diet- and chemical-induced murine NASH model with rapid progression of steatohepatitis, fibrosis and liver cancer. J Hepatol. 2018;69:385-395. [PubMed] [DOI] |
| 81. | Kametani Y, Ohno Y, Ohshima S, Tsuda B, Yasuda A, Seki T, Ito R, Tokuda Y. Humanized Mice as an Effective Evaluation System for Peptide Vaccines and Immune Checkpoint Inhibitors. Int J Mol Sci. 2019;20. [PubMed] [DOI] |
| 82. | Domenicali M, Caraceni P, Giannone F, Baldassarre M, Lucchetti G, Quarta C, Patti C, Catani L, Nanni C, Lemoli RM, Bernardi M. A novel model of CCl4-induced cirrhosis with ascites in the mouse. J Hepatol. 2009;51:991-999. [PubMed] [DOI] |
| 83. | Li J, Zhao W, Akbani R, Liu W, Ju Z, Ling S, Vellano CP, Roebuck P, Yu Q, Eterovic AK, Byers LA, Davies MA, Deng W, Gopal YN, Chen G, von Euw EM, Slamon D, Conklin D, Heymach JV, Gazdar AF, Minna JD, Myers JN, Lu Y, Mills GB, Liang H. Characterization of Human Cancer Cell Lines by Reverse-phase Protein Arrays. Cancer Cell. 2017;31:225-239. [PubMed] [DOI] |
| 84. | Zhao W, Li J, Chen MM, Luo Y, Ju Z, Nesser NK, Johnson-Camacho K, Boniface CT, Lawrence Y, Pande NT, Davies MA, Herlyn M, Muranen T, Zervantonakis IK, von Euw E, Schultz A, Kumar SV, Korkut A, Spellman PT, Akbani R, Slamon DJ, Gray JW, Brugge JS, Lu Y, Mills GB, Liang H. Large-Scale Characterization of Drug Responses of Clinically Relevant Proteins in Cancer Cell Lines. Cancer Cell. 2020;38:829-843.e4. [PubMed] [DOI] |
| 85. | Goyama S, Wunderlich M, Mulloy JC. Xenograft models for normal and malignant stem cells. Blood. 2015;125:2630-2640. [PubMed] [DOI] |
| 86. | Nardone RM, MacLeod RA, Capes-Davis A. Cancer: Authenticate new xenograft models. Nature. 2016;532:313. [PubMed] [DOI] |
| 87. | Lai Y, Wei X, Lin S, Qin L, Cheng L, Li P. Current status and perspectives of patient-derived xenograft models in cancer research. J Hematol Oncol. 2017;10:106. [PubMed] [DOI] |
| 88. | Byrne AT, Alférez DG, Amant F, Annibali D, Arribas J, Biankin AV, Bruna A, Budinská E, Caldas C, Chang DK, Clarke RB, Clevers H, Coukos G, Dangles-Marie V, Eckhardt SG, Gonzalez-Suarez E, Hermans E, Hidalgo M, Jarzabek MA, de Jong S, Jonkers J, Kemper K, Lanfrancone L, Mælandsmo GM, Marangoni E, Marine JC, Medico E, Norum JH, Palmer HG, Peeper DS, Pelicci PG, Piris-Gimenez A, Roman-Roman S, Rueda OM, Seoane J, Serra V, Soucek L, Vanhecke D, Villanueva A, Vinolo E, Bertotti A, Trusolino L. Interrogating open issues in cancer precision medicine with patient-derived xenografts. Nat Rev Cancer. 2017;17:254-268. [PubMed] [DOI] |
| 89. | Aparicio S, Hidalgo M, Kung AL. Examining the utility of patient-derived xenograft mouse models. Nat Rev Cancer. 2015;15:311-316. [PubMed] [DOI] |
| 90. | Zanella ER, Grassi E, Trusolino L. Towards precision oncology with patient-derived xenografts. Nat Rev Clin Oncol. 2022;19:719-732. [PubMed] [DOI] |
| 91. | Brown ZJ, Heinrich B, Greten TF. Mouse models of hepatocellular carcinoma: an overview and highlights for immunotherapy research. Nat Rev Gastroenterol Hepatol. 2018;15:536-554. [PubMed] [DOI] |
| 92. | Rudin CM, Poirier JT, Byers LA, Dive C, Dowlati A, George J, Heymach JV, Johnson JE, Lehman JM, MacPherson D, Massion PP, Minna JD, Oliver TG, Quaranta V, Sage J, Thomas RK, Vakoc CR, Gazdar AF. Molecular subtypes of small cell lung cancer: a synthesis of human and mouse model data. Nat Rev Cancer. 2019;19:289-297. [PubMed] [DOI] |
| 93. | Hill W, Caswell DR, Swanton C. Capturing cancer evolution using genetically engineered mouse models (GEMMs). Trends Cell Biol. 2021;31:1007-1018. [PubMed] [DOI] |
| 94. | Kersten K, de Visser KE, van Miltenburg MH, Jonkers J. Genetically engineered mouse models in oncology research and cancer medicine. EMBO Mol Med. 2017;9:137-153. [PubMed] [DOI] |
| 95. | Chuprin J, Buettner H, Seedhom MO, Greiner DL, Keck JG, Ishikawa F, Shultz LD, Brehm MA. Humanized mouse models for immuno-oncology research. Nat Rev Clin Oncol. 2023;20:192-206. [PubMed] [DOI] |
| 96. | De La Rochere P, Guil-Luna S, Decaudin D, Azar G, Sidhu SS, Piaggio E. Humanized Mice for the Study of Immuno-Oncology. Trends Immunol. 2018;39:748-763. [PubMed] [DOI] |
| 97. | Choi Y, Lee S, Kim K, Kim SH, Chung YJ, Lee C. Studying cancer immunotherapy using patient-derived xenografts (PDXs) in humanized mice. Exp Mol Med. 2018;50:1-9. [PubMed] [DOI] |
| 98. | Horowitz NB, Mohammad I, Moreno-Nieves UY, Koliesnik I, Tran Q, Sunwoo JB. Humanized Mouse Models for the Advancement of Innate Lymphoid Cell-Based Cancer Immunotherapies. Front Immunol. 2021;12:648580. [PubMed] [DOI] |
| 99. | Lau HCH, Kranenburg O, Xiao H, Yu J. Organoid models of gastrointestinal cancers in basic and translational research. Nat Rev Gastroenterol Hepatol. 2020;17:203-222. [PubMed] [DOI] |
| 100. | LeSavage BL, Suhar RA, Broguiere N, Lutolf MP, Heilshorn SC. Next-generation cancer organoids. Nat Mater. 2022;21:143-159. [PubMed] [DOI] |
| 101. | Dedhia PH, Bertaux-Skeirik N, Zavros Y, Spence JR. Organoid Models of Human Gastrointestinal Development and Disease. Gastroenterology. 2016;150:1098-1112. [PubMed] [DOI] |
| 102. | Aboulkheyr Es H, Montazeri L, Aref AR, Vosough M, Baharvand H. Personalized Cancer Medicine: An Organoid Approach. Trends Biotechnol. 2018;36:358-371. [PubMed] [DOI] |
| 103. | Sun Y, Revach OY, Anderson S, Kessler EA, Wolfe CH, Jenney A, Mills CE, Robitschek EJ, Davis TGR, Kim S, Fu A, Ma X, Gwee J, Tiwari P, Du PP, Sindurakar P, Tian J, Mehta A, Schneider AM, Yizhak K, Sade-Feldman M, LaSalle T, Sharova T, Xie H, Liu S, Michaud WA, Saad-Beretta R, Yates KB, Iracheta-Vellve A, Spetz JKE, Qin X, Sarosiek KA, Zhang G, Kim JW, Su MY, Cicerchia AM, Rasmussen MQ, Klempner SJ, Juric D, Pai SI, Miller DM, Giobbie-Hurder A, Chen JH, Pelka K, Frederick DT, Stinson S, Ivanova E, Aref AR, Paweletz CP, Barbie DA, Sen DR, Fisher DE, Corcoran RB, Hacohen N, Sorger PK, Flaherty KT, Boland GM, Manguso RT, Jenkins RW. Targeting TBK1 to overcome resistance to cancer immunotherapy. Nature. 2023;615:158-167. [PubMed] [DOI] |
| 104. | Aref AR, Campisi M, Ivanova E, Portell A, Larios D, Piel BP, Mathur N, Zhou C, Coakley RV, Bartels A, Bowden M, Herbert Z, Hill S, Gilhooley S, Carter J, Cañadas I, Thai TC, Kitajima S, Chiono V, Paweletz CP, Barbie DA, Kamm RD, Jenkins RW. 3D microfluidic ex vivo culture of organotypic tumor spheroids to model immune checkpoint blockade. Lab Chip. 2018;18:3129-3143. [PubMed] [DOI] |
| 105. | Gao Q, Zhu H, Dong L, Shi W, Chen R, Song Z, Huang C, Li J, Dong X, Zhou Y, Liu Q, Ma L, Wang X, Zhou J, Liu Y, Boja E, Robles AI, Ma W, Wang P, Li Y, Ding L, Wen B, Zhang B, Rodriguez H, Gao D, Zhou H, Fan J. Integrated Proteogenomic Characterization of HBV-Related Hepatocellular Carcinoma. Cell. 2019;179:561-577.e22. [PubMed] [DOI] |
| 106. | Dong LQ, Peng LH, Ma LJ, Liu DB, Zhang S, Luo SZ, Rao JH, Zhu HW, Yang SX, Xi SJ, Chen M, Xie FF, Li FQ, Li WH, Ye C, Lin LY, Wang YJ, Wang XY, Gao DM, Zhou H, Yang HM, Wang J, Zhu SD, Wang XD, Cao Y, Zhou J, Fan J, Wu K, Gao Q. Heterogeneous immunogenomic features and distinct escape mechanisms in multifocal hepatocellular carcinoma. J Hepatol. 2020;72:896-908. [PubMed] [DOI] |
| 107. | Song F, Wang CG, Mao JZ, Wang TL, Liang XL, Hu CW, Zhang Y, Han L, Chen Z. PANoptosis-based molecular subtyping and HPAN-index predicts therapeutic response and survival in hepatocellular carcinoma. Front Immunol. 2023;14:1197152. [PubMed] [DOI] |