修回日期: 2021-12-22
接受日期: 2022-01-20
在线出版日期: 2022-02-08
胆管癌恶性程度高, 预后差. 靶向治疗是胆管癌的重要研究方向, 探索新的分子靶点对于胆管癌靶向治疗至关重要.
用生物信息学分析方法挖掘胆管癌的枢纽基因, 为胆管癌的靶向治疗提供潜在分子靶点.
从GEO数据库中下载2组胆管癌表达谱芯片数据, 采用GEO2R在线分析工具筛选胆管癌肿瘤组织与正常组织差异表达基因, 对差异表达基因作GO富集分析、KEGG通路分析、蛋白质相互作用网络分析, 利用Cytoscape软件筛选枢纽基因.使用GEPIA数据库对枢纽基因在胆管癌组织中的表达量进行验证.
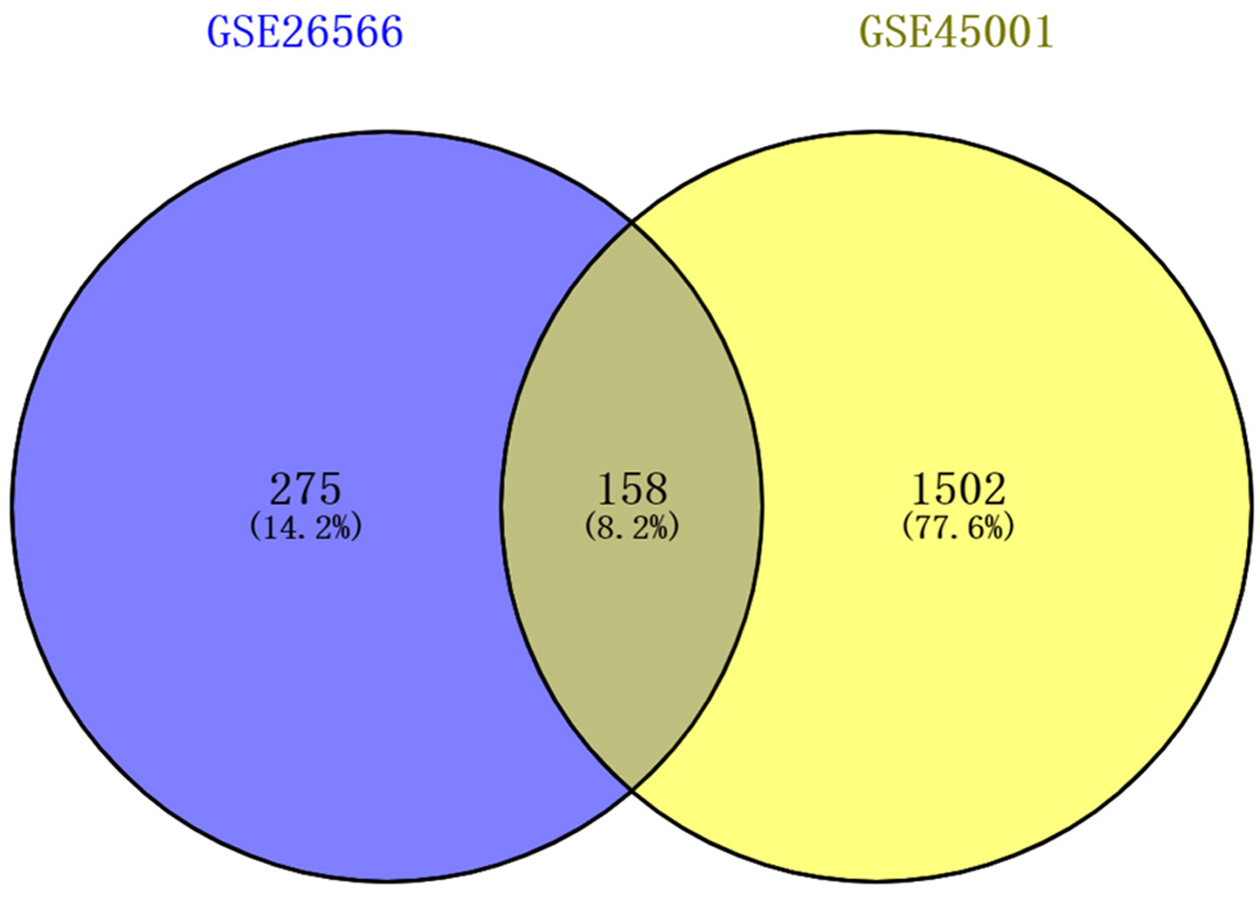
共得到共同差异表达基因158个. GO富集分析结果显示, 差异基因主要参与细胞对锌离子反应、细胞增殖与粘附、代谢以及蛋白质聚合等生物学过程, 主要存在外泌体、胞外区、弹性纤维等区域, 主要分子功能与结合肝素、半胱氨酸型内肽酶抑制剂活性、蛋白质同源二聚化、受体结合及磷酸吡哆醛结合等相关. KEGG通路分析结果显示, 差异基因主要参与矿物质吸收、代谢、PPAR信号通路及脂肪酸降解等过程. 基于String数据库构建蛋白质相互作用网络图, Cytoscape软件CytoHubba插件筛选枢纽基因, 皆为上调基因. GEPIA数据库验证枢纽基因在胆管癌组织中表达量显著高于正常组织.
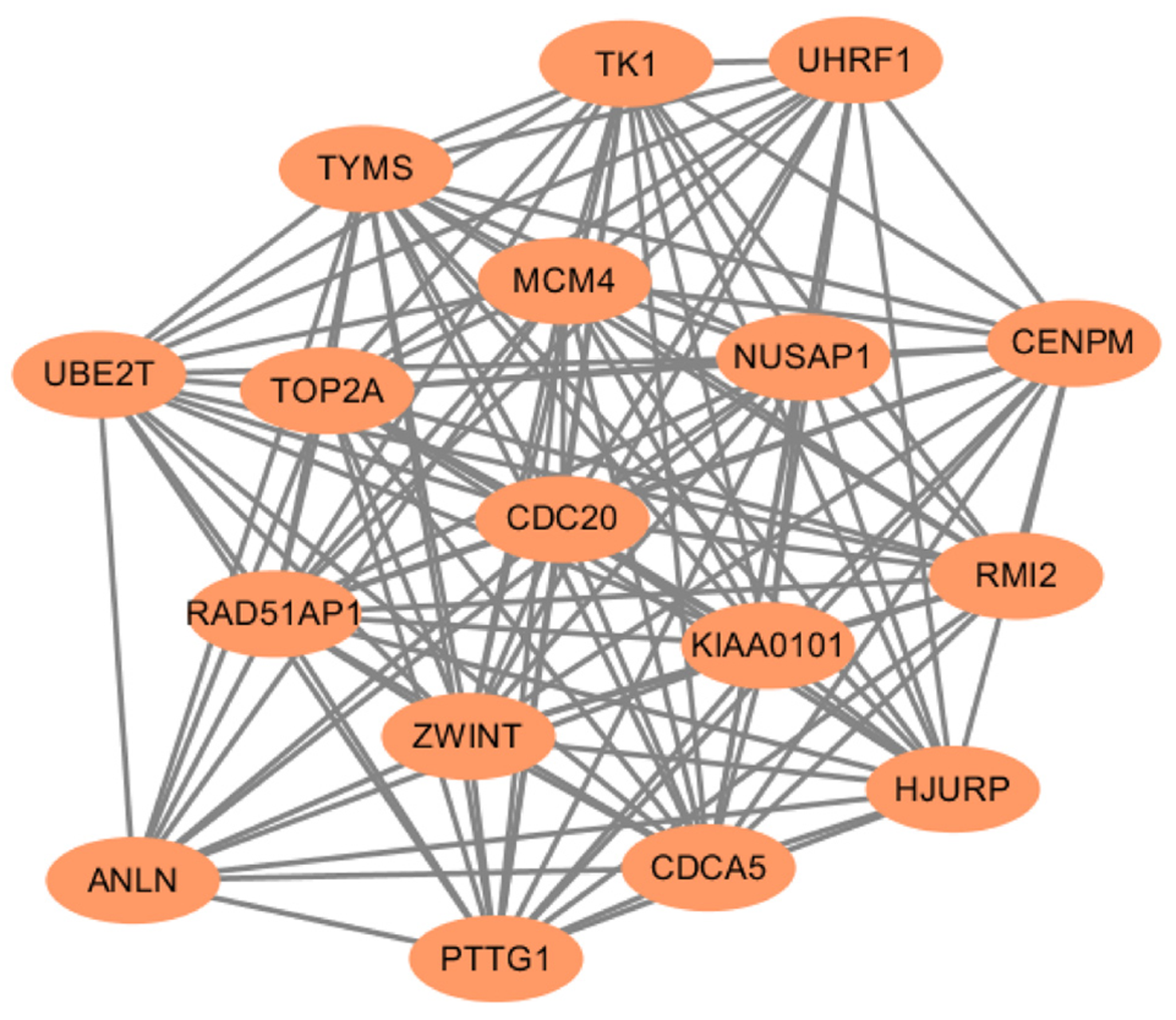
本研究获取了8个与胆管癌相关的枢纽基因, 分别是NUSAP1, TOP2A, RAD51AP1, MCM4, KIAA0101, CDCA5, TYMS, ZWINT. 这些基因为深入研究胆管癌的靶向治疗提供了新思路, 有望成为新的分子治疗靶点.
核心提要: 本研究利用生物信息学方法, 探索胆管癌与正常组织的差异表达基因, 分析其参与的主要生物学过程与信号通路, 通过筛选与验证最终得到8个枢纽基因, 为深入研究胆管癌靶向治疗分子靶点提供一定理论基础.
引文著录: 于珍, 吴静, 张磊, 刘树业. 基于GEO数据库对胆管癌潜在分子靶点的筛选及生物信息学分析. 世界华人消化杂志 2022; 30(3): 128-135
Revised: December 22, 2021
Accepted: January 20, 2022
Published online: February 8, 2022
Cholangiocarcinoma is a highly malignant tumor with a poor prognosis. Targeted therapy is important for the treatment of cholangiocarcinoma, and it is therefore of great clinical importance to identify novel molecular targets for targeted therapy of this malignancy.
To identify potential molecular targets for the treatment of cholangiocarcinoma and identify the key genes involved in cholangiocarcinoma by bioinformatics analysis.
We downloaded two sets of cholangiocarcinoma expression profile data from GEO database. GEO2R online analysis tool was used to screen differentially expressed genes in cholangiocarcinoma tumor tissues and normal tissues, and we performed GO enrichment analysis, KEGG pathway analysis, and protein interaction network for differentially expressed genes. We used Cytoscape software to calculate key genes. The GEPIA database was used to verify the expression of hub genes in cholangiocarcinoma tissues.
A total of 158 differentially expressed genes were identified. GO enrichment analysis showed that these differential genes were mainly involved in the cellular response to zinc ion, negative regulation of growth, cell adhesion, metabolic process, and protein homotetramerization. They were enriched in exosomes, extracellular spaces, elastic fibers, and organelle membranes. The main molecular functions are related to heparin binding, cysteine-type endopeptidase inhibitor activity, protein homodimerization activity, receptor binding, and pyridoxal phosphate binding. KEGG pathway analysis showed that differential genes are mainly involved in processes such as mineral absorption, carbon and propanoate metabolism, PPAR signaling pathway, and fatty acid degradation. A protein interaction network diagram was constructed based on the String database, and the CytoHubba plug-in of the Cytoscape software was used to calculate the key genes. The key genes were all up-regulated ones. GEPIA analysis verified that the expression of key genes in cholangiocarcinoma tissues was significantly higher than that in normal tissues.
In this study, eight key genes related to cholangiocarcinoma were identified, including NUSAP1, TOP2A, RAD51AP1, MCM4, KIAA0101, CDCA5, TYMS, and ZWINT. These genes provide new ideas for in-depth study of the targeted therapy of cholangiocarcinoma, and are expected to become new molecular therapeutic targets.
- Citation: Yu Z, Wu J, Zhang L, Liu SY. Potential molecular target screening and bioinformatics analysis of cholangiocarcinoma based on GEO database. Shijie Huaren Xiaohua Zazhi 2022; 30(3): 128-135
- URL: https://www.wjgnet.com/1009-3079/full/v30/i3/128.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v30.i3.128
胆管癌是一种异质程度极高的恶性肿瘤, 多原发于胆管上皮细胞[1]. 其发病率逐年增加, 患者5年生存率较低, 小于10%[2]. 胆管癌侵袭性强, 其发生可能与胆汁淤积、感染及炎症相关, 但具体的分子发病机制尚不完全清楚[3]. 胆管癌的早期症状隐匿, 不易发现, 多数患者确诊时已是晚期[4]. 对于晚期胆管癌患者而言, 治疗方法有限, 预后较差. 使用标准化疗方案(吉西他滨和顺铂)治疗的患者中位总生存期仍不到1年[5]. 靶向治疗可有助于改善晚期肿瘤患者的治疗效果, 提高患者生活质量, 因而获得广泛关注. 而目前, 用于胆管癌的靶向分子疗法尚未见批准[6]. 开发有前景的分子靶点, 对于提高胆管癌靶向治疗的应用前景至关重要.随着高通量测序技术的不断完善和发展, 利用生物信息学技术挖掘胆管癌基因组潜在分子靶点成为可能. 本研究中, 我们利用基因表达综合(gene expression omnibus, GEO)数据库中的2组胆管癌表达谱芯片数据进行一系列生物信息学分析, 获得胆管癌与正常组织的差异表达基因, 探讨枢纽基因在胆管癌发生发展中的作用, 为胆管癌的靶向治疗提供潜在分子靶点.
胆管癌数据集来源于GEO数据库, 编号分别为GSE26566和GSE45001. GSE26566包含104例胆管癌组织样本和6例正常胆管组织样本. GSE45001包括10个胆管癌组织样本和10例非肿瘤组织样本.
1.2.1 差异表达基因筛选: 2组原始数据集采用GEO2R (http://www.ncbi.nlm.nih.gov/geo/geo2r/)在线分析工具进行数据处理, 处理后数据进行差异基因筛选. 调整后的P值(adj.P)<0.05和|logFC|>1.5作为纳入标准. 运用SangerBox在线软件作火山图. 用Venny 2.1.0对上述2个数据集的差异基因绘制Venn图取交集.
1.2.2 差异基因功能分析: 使用DAVID(http://david.ncifcrf.gov)在线分析对筛选出来的158个差异基因进行GO富集分析, 包括生物学过程(biological process, BP)、细胞组分和定位(cellular component, CC)以及分子功能(molecular function, MF), 同时进行KEGG信号通路分析.设置P<0.05.
1.2.3 蛋白质互作网络与枢纽基因分析: 使用STRING 11.5数据库(https://sring-db.org/)预测胆管癌差异基因编码蛋白间的蛋白质互作网络(protein protein interaction network, PPI). 通过Cytoscape 3.7.0软件MCODE插件获得PPI中最显著的核心模块, 设置参数degree cutoff = 2、node score cutoff = 0.2、k-score = 2及max.depth = 100[7].利用CytoHubba插件Degree算法筛选出核心模块中前10位有较高连接度的枢纽基因[8].
1.2.4 枢纽基因表达验证: 使用GEPIA数据库(https://gepia.cancer-pku.cn/)对枢纽基因的mRNA在胆管癌组织及正常组织中实际表达水平进行验证.
统计学处理 采用Wilcoxon检验分析胆管癌组织和正常组表达数据. P<0.05差异有统计学意义.
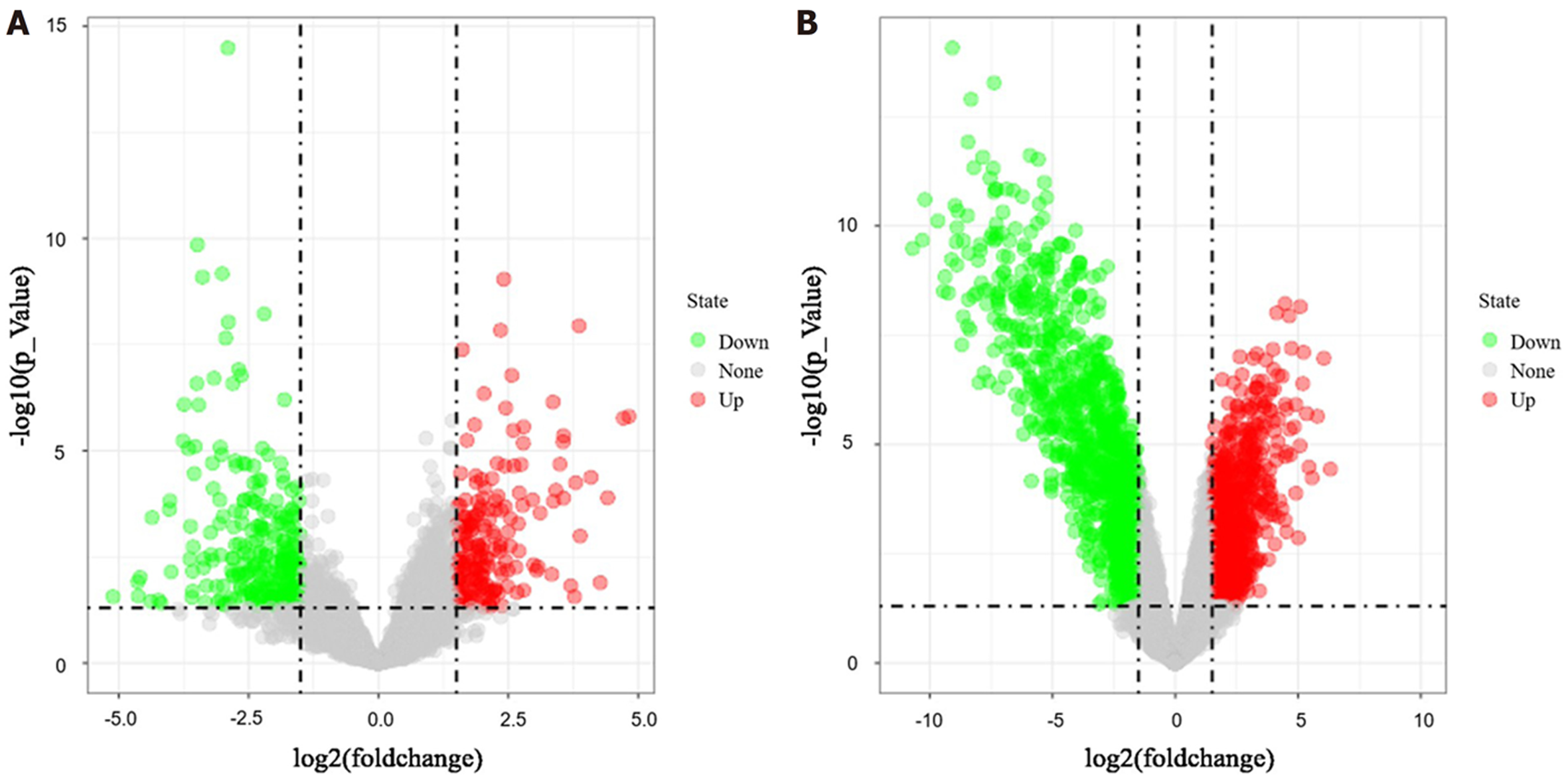
经GEO2R分析, 分别从数据集GSE26566和GSE45001中提取到差异基因433个和1660个, 绘制火山图(图1), 其中绿色为下调基因、红色为上调基因. 取2个数据集差异表达基因的交集, 制作Venn图(图2), 得到相同差异表达基因158个, 其中上调基因53个, 下调基因105个.
GO富集分析结果显示这些差异基因主要参与细胞对锌离子反应、增长的负调控, 并且参与细胞粘附、代谢以及蛋白质聚合等生物过程; 主要细胞成分位于外泌体、胞外区、弹性纤维、细胞器膜基质及细胞外基质; 主要分子功能与结合肝素、半胱氨酸型内肽酶抑制剂活性、蛋白质同源二聚化、受体结合及磷酸吡哆醛结合等相关(表1). KEGG信号通路分析显示, 这些差异基因主要参与矿物质吸收、碳代谢、丙酸代谢、代谢途径、PPAR信号通路、脂肪酸降解、细胞色素P450对异生物质的代谢、化学致癌、ECM-受体相互作用和糖酵解/糖异生等通路(表2).
| 类型 | GO号 | 注释 | 基因数 | P值 |
| 生物学过程 | GO: 0071294 | 细胞对锌离子的反应 | 6 | 5.78E-07 |
| GO: 0045926 | 生长负调控 | 6 | 5.78E-07 | |
| GO: 0007155 | 细胞粘附 | 15 | 6.93E-05 | |
| GO: 0008152 | 代谢过程 | 9 | 1.39E-04 | |
| GO: 0051289 | 蛋白质同源四聚化 | 6 | 2.02E-04 | |
| 细胞组分 | GO: 0070062 | 胞外外泌体 | 49 | 8.12E-07 |
| GO: 0005615 | 胞外域 | 31 | 9.57E-07 | |
| GO: 0071953 | 弹性纤维 | 3 | 4.26E-04 | |
| GO: 0031090 | 细胞器膜 | 6 | 8.80E-04 | |
| GO: 0031012 | 细胞外基质 | 10 | 9.90E-04 | |
| 分子功能 | GO: 0008201 | 结合肝素 | 10 | 1.17E-05 |
| GO: 0004869 | 半胱氨酸型内肽酶抑制剂活性 | 4 | 0.003238 | |
| GO: 0042803 | 蛋白质同源二聚化活性 | 14 | 0.012004 | |
| GO: 0005102 | 结合受体 | 9 | 0.012698 | |
| GO: 0030170 | 结合磷酸吡哆醛 | 4 | 0.01368 |
| 条目 | 通路 | 基因数 | P值 |
| hsa04978 | 矿物质吸收 | 7 | 2.01E-05 |
| hsa01200 | 碳代谢 | 9 | 1.05E-04 |
| hsa00640 | 丙酸代谢 | 5 | 4.41E-04 |
| hsa01100 | 代谢途径 | 30 | 5.77E-04 |
| hsa03320 | PPAR信号通路 | 6 | 0.001749 |
| hsa00071 | 脂肪酸降解 | 5 | 0.002098 |
| hsa00980 | 细胞色素P450对异生素的代谢 | 6 | 0.002715 |
| hsa05204 | 化学致癌 | 6 | 0.003812 |
| hsa04512 | ECM-受体相互作用 | 6 | 0.005455 |
| hsa00010 | 糖酵解/糖异生 | 5 | 0.01122 |
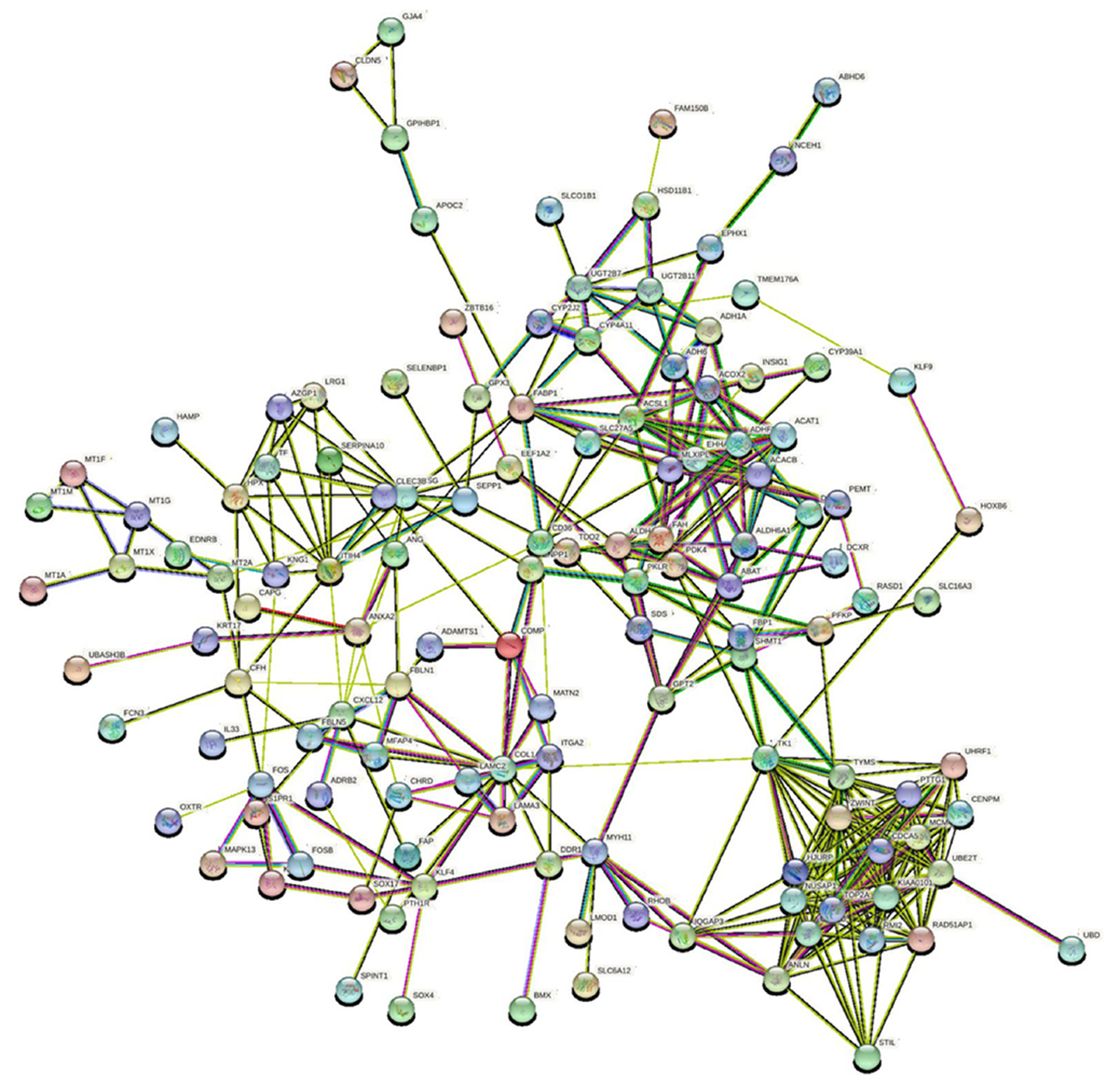
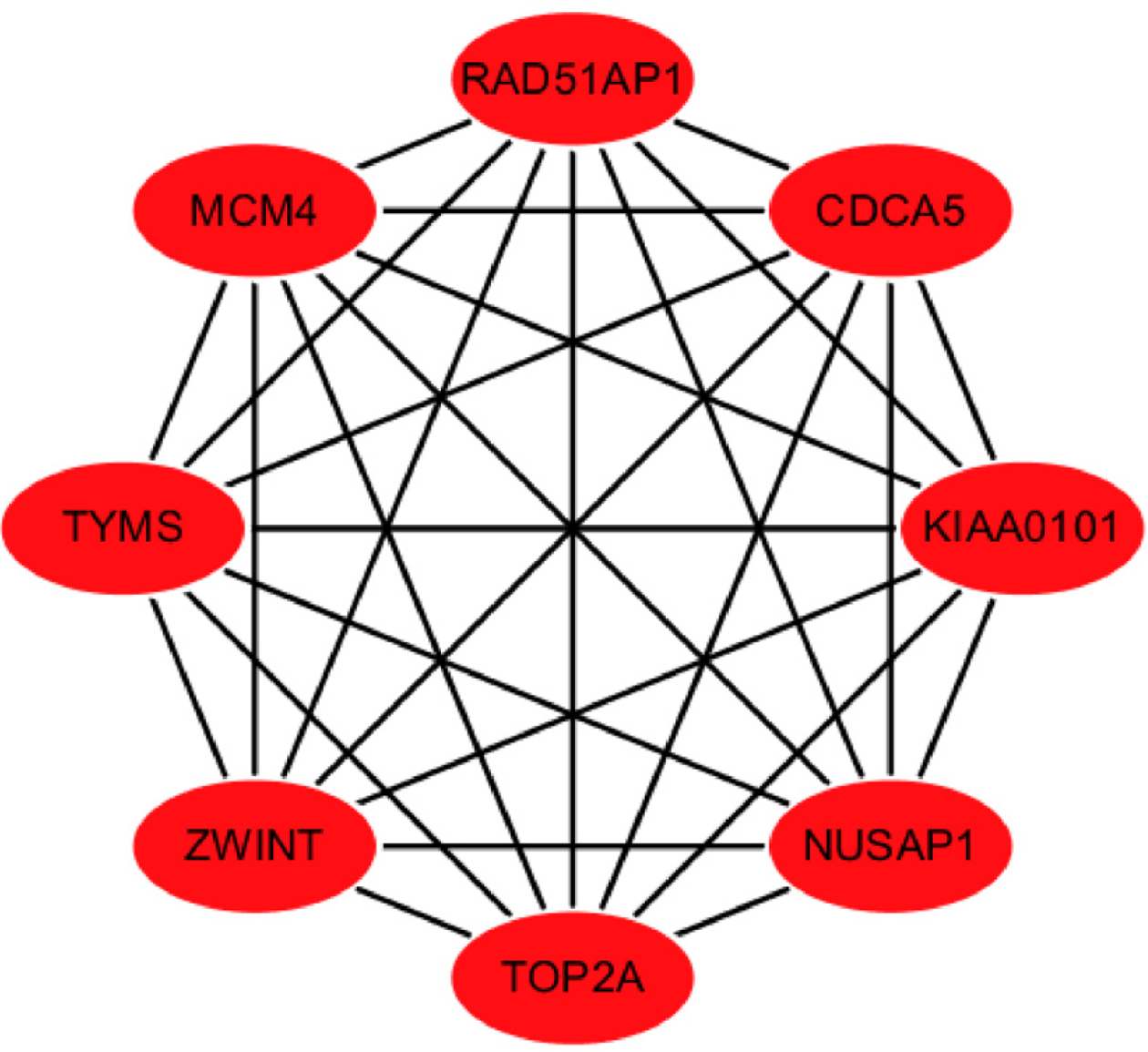
除去游离的蛋白节点, 共有130个蛋白质节点连接形成382条复杂的PPI网络(图3).最显著模块由17个重要节点及127条边的网络构成(图4). 通过Cytohubba插件Degree算法确认枢纽基因, 结果如图5, 显示枢纽基因分别为NUSAP1, TOP2A, RAD51AP1, MCM4, KIAA0101, CDCA5, TYMS, ZWINT, 皆为上调基因.
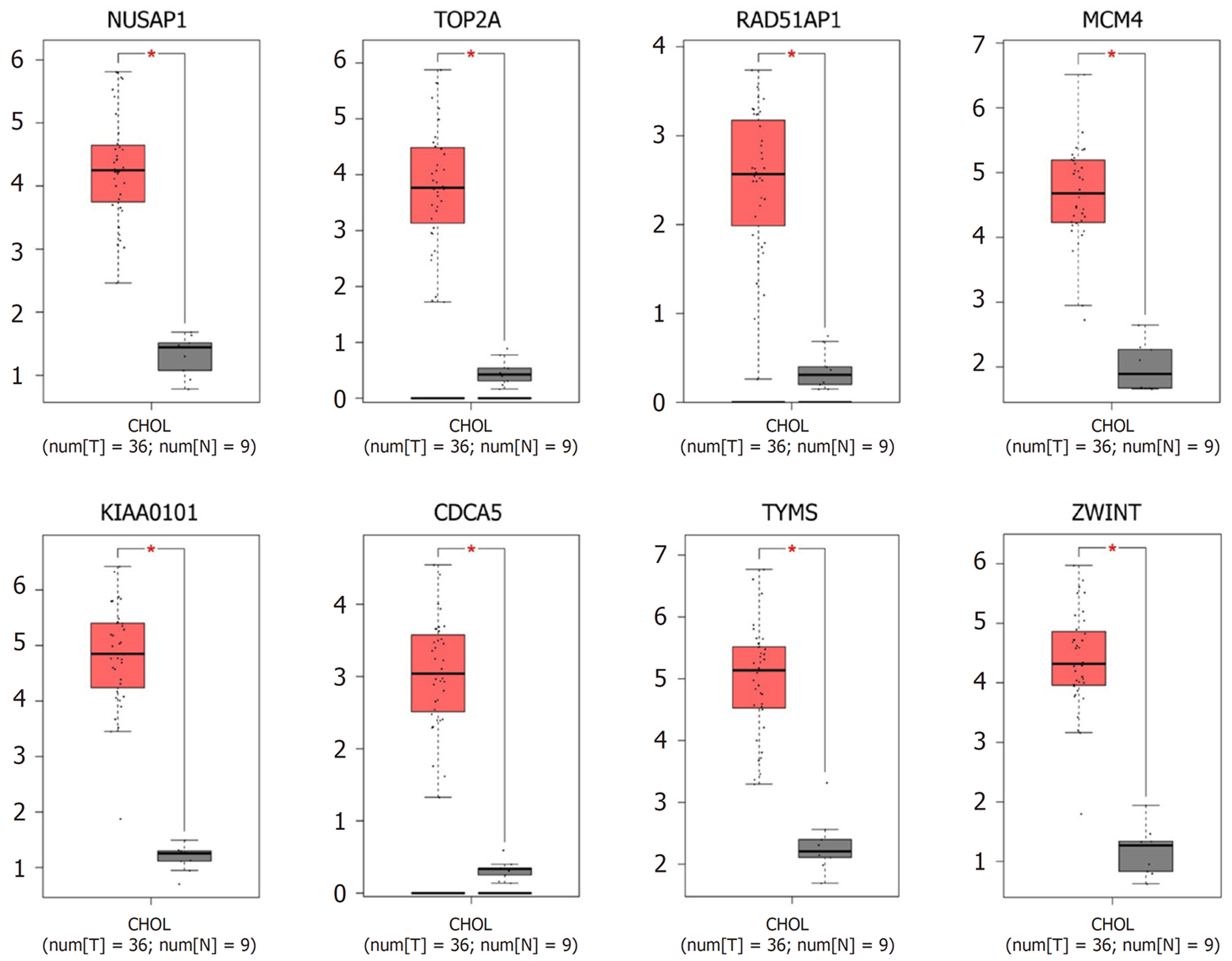
通过GEPIA数据库验证8个枢纽基因mRNA的表达水平. 如图6所示, 胆管癌组织中NUSAP1, TOP2A, RAD51AP1, MCM4, KIAA0101, CDCA5, TYMS和ZWINT的表达较正常组织均显著增加.
胆管癌是仅次于肝细胞癌的第二大常见肝脏原发性恶性肿瘤, 也是胆道最常见的恶性肿瘤, 根据解剖位置可分为肝内胆管癌、肝门周围胆管癌和远端胆管癌[9]. 胆管癌发病早期症状隐匿, 晚期恶性程度高, 预后差, 所以寻找有助于治疗胆管癌的分子靶点至关重要.
本研究中, 我们使用GEO2R分析了GEO数据库GSE26566和GSE45001两组数据集.获得差异基因158个, 其中上调基因53个, 下调基因105个. 通过DAVID在线工具对共同差异基因做GO富集分析, 结果显示差异基因主要参与细胞对锌离子反应、细胞增殖的调控及细胞粘附、代谢等生物过程, 富集于外泌体、胞外区、弹性纤维等区域, 主要分子功能与结合肝素、半胱氨酸型内肽酶抑制剂活性、蛋白质同源二聚化、受体结合及磷酸吡哆醛结合等相关. KEGG信号通路分析表明, 差异基因主要参与矿物质吸收、代谢、PPAR信号通路及脂肪酸降解等过程.
经Cytoscape软件筛选, 本研究共得到8个枢纽基因, 分别是NUSAP1, TOP2A, RAD51AP1, MCM4, KIAA0101, CDCA5, TYMS, ZWINT, 皆为上调基因. 经验证, 这8个枢纽基因在胆管癌组织中的表达较正常组织高. NUSAP1编码核仁和纺锤体相关蛋白, 参与调节纺锤体的组装和维持正常的胞质分裂, 因此是细胞有丝分裂和增殖的重要调节因子[10,11]. 研究发现[12-14], NUSAP1在多种肿瘤中存在过表达, 包括肝癌、肺腺癌和肾细胞癌. 另外, NUSAP1的上调与黑色素瘤和口腔鳞状细胞癌的恶性进展有关[15,16]. 因此, NUSAP1可能是一个潜在的治疗靶点.DNA拓扑异构酶Ⅱ-α(TOP2A)基因参与调节核酸空间结构的动态变化[17], 其表达与肿瘤侵袭、复发以及不同癌症的预后相关, 包括乳腺癌、睾丸畸胎瘤、膀胱移行细胞癌、脑膜瘤、神经胶质瘤、肝癌和子宫内膜癌[18-23]. RAD51AP1(RAD51-associated protein 1)基因编码一种DNA结合蛋白, 在RAD51介导的同源重组中至关重要[24]. Obama等[25]研究表明, RAD51AP1参与肝内胆管癌的细胞增殖和DNA修复, 其沉默有助于抑制胆管癌细胞的生长. MCM4是微染色体维持蛋白家族的成员之一, 具有复制解旋酶活性, 参与DNA复制和DNA解旋[26]. 有研究显示, MCM4的高表达会增加多种肿瘤发病风险, 如肝癌、食管癌、乳腺癌等[27-29]. KIAA0101编码一种保守的增殖细胞核抗原结合蛋白, 与DNA聚合酶竞争结合以调节DNA复制和损伤修复[30]. 据报道, KIAA0101基因在乳腺癌和非小细胞肺癌组织中存在表达上调, 敲低KIAA0101基因可抑制乳腺癌细胞的生长, 另外KIAA0101高表达也与非小细胞肺癌患者术后复发相关[31,32]. 细胞分裂周期相关蛋白5(CDCA5), 也称为Sororin, 在确保姐妹染色单体的准确分离方面发挥着关键作用[33]. 最近的研究表明[34-36], CDCA5的高表达与多种癌症的肿瘤发生和组织侵袭相关, 包括口腔鳞状细胞癌、非小细胞肺癌、尿路上皮细胞癌和胃癌. TYMS编码胸苷酸合酶, 通过催化dUMP的甲基化产生dTMP, 在DNA复制中发挥重要作用[37,38]. TYMS的高表达与许多实体瘤的发生存在关联, 包括肺癌、乳腺癌、胃癌、结肠直肠癌和肾细胞癌等[39-43]. ZWINT编码一种调控着丝点分裂的动粒蛋白, 在维持有丝分裂周期过程中起着至关重要的作用[44]. 越来越多的证据表明, ZWINT 在许多人类癌症中通常高度表达, 并且与预后不良和早期复发有关[45-47].
本研究通过生物信息学方法从胆管癌组织中筛选出8个枢纽基因, 与正常组织相比, NUSAP1, TOP2A, RAD51AP1, MCM4, KIAA0101, CDCA5, TYMS, ZWINT在胆管癌中的表达均上调. 结果提示, 这8个枢纽基因可能在胆管癌的发生发展中起到关键作用, 为胆管癌的靶向治疗提供了理论基础, 但这些基因在胆管癌中的具体机制和作用仍需要进一步实验验证.
胆管癌是胆道常见的恶性肿瘤, 预后不佳. 靶向治疗是改善胆管癌治疗效果的重要手段, 探索新的分子靶点对于胆管癌靶向治疗的研究十分必要.
寻找影响胆管癌发生和进展的枢纽基因对于开发胆管癌靶向治疗的靶点具有重要意义.
通过生物信息学方法探索胆管癌靶向治疗的潜在分子靶点.
利用GEO数据库分析2组胆管癌数据集, 得到共同差异表达基因. 通过GO富集分析与KEGG通路分析共同差异基因主要参与的生物学过程与信号通路. 基于String数据库构建蛋白质互作网络图, 经过Cytoscape软件筛选枢纽基因, 通过GEPIA数据库验证枢纽基因在胆管癌与正常组织中的表达量.
2组胆管癌数据集共包含158个共同差异表达基因. GO富集分析及KEGG通路分析结果显示, 差异基因主要参与细胞对锌离子反应, 细胞增殖与粘附等生物学过程, 涉及矿物质吸收、代谢、PPAR信号通路及脂肪酸降解等信号通路. 最终筛选得到了8个上调的枢纽基因, 在胆管癌组织中表达量显著高于正常组织.
本研究通过生物信息学方法获得了与胆管癌发生与进展息息相关的8个枢纽基因, 分别为NUSAP1, TOP2A, RAD51AP1, MCM4, KIAA0101, CDCA5, TYMS, ZWINT, 可能为胆管癌靶向治疗的潜在分子靶点.
这8个枢纽基因对胆管癌的影响和机制需进一步通过实验验证, 有望为胆管癌靶向治疗分子靶点的开发提供新思路.
学科分类: 胃肠病学和肝病学
手稿来源地: 天津市
同行评议报告学术质量分类
A级 (优秀): 0
B级 (非常好): B, B
C级 (良好): 0
D级 (一般): D
E级 (差): E
科学编辑: 张砚梁 制作编辑:张砚梁
| 1. | Khan AS, Dageforde LA. Cholangiocarcinoma. Surg Clin North Am. 2019;99:315-335. [PubMed] [DOI] |
| 2. | Primrose JN, Fox RP, Palmer DH, Malik HZ, Prasad R, Mirza D, Anthony A, Corrie P, Falk S, Finch-Jones M, Wasan H, Ross P, Wall L, Wadsley J, Evans JTR, Stocken D, Praseedom R, Ma YT, Davidson B, Neoptolemos JP, Iveson T, Raftery J, Zhu S, Cunningham D, Garden OJ, Stubbs C, Valle JW, Bridgewater J; BILCAP study group. Capecitabine compared with observation in resected biliary tract cancer (BILCAP): a randomised, controlled, multicentre, phase 3 study. Lancet Oncol. 2019;20:663-673. [PubMed] [DOI] |
| 3. | Labib PL, Goodchild G, Pereira SP. Molecular Pathogenesis of Cholangiocarcinoma. BMC Cancer. 2019;19:185. [PubMed] [DOI] |
| 4. | Rodrigues PM, Olaizola P, Paiva NA, Olaizola I, Agirre-Lizaso A, Landa A, Bujanda L, Perugorria MJ, Banales JM. Pathogenesis of Cholangiocarcinoma. Annu Rev Pathol. 2021;16:433-463. [PubMed] [DOI] |
| 5. | Tsimafeyeu I, Temper M. Cholangiocarcinoma: An Emerging Target for Molecular Therapy. Gastrointest Tumors. 2021;8:153-158. [PubMed] [DOI] |
| 6. | Sarkis Y, Al Soueidy A, Kourie HR. Will advanced cholangioca-rcinoma become a targetable malignancy? Crit Rev Oncol Hematol. 2021;159:103233. [PubMed] [DOI] |
| 7. | Wang J, Zhong J, Chen G, Li M, Wu FX, Pan Y. ClusterViz: A Cytoscape APP for Cluster Analysis of Biological Network. IEEE/ACM Trans Comput Biol Bioinform. 2015;12:815-822. [PubMed] [DOI] |
| 8. | Chin CH, Chen SH, Wu HH, Ho CW, Ko MT, Lin CY. cytoHubba: identifying hub objects and sub-networks from complex interactome. BMC Syst Biol. 2014;8 Suppl 4:S11. [PubMed] [DOI] |
| 9. | Rizvi S, Khan SA, Hallemeier CL, Kelley RK, Gores GJ. Cholangiocarcinoma - evolving concepts and therapeutic strategies. Nat Rev Clin Oncol. 2018;15:95-111. [PubMed] [DOI] |
| 10. | Zhao Y, He J, Li Y, Lv S, Cui H. NUSAP1 potentiates chemoresis-tance in glioblastoma through its SAP domain to stabilize ATR. Signal Transduct Target Ther. 2020;5:44. [PubMed] [DOI] |
| 11. | Wang H, Liu Z, Wu P, Wang H, Ren W. NUSAP1 Accelerates Osteosarcoma Cell Proliferation and Cell Cycle Progression via Upregulating CDC20 and Cyclin A2. Onco Targets Ther. 2021;14:3443-3454. [PubMed] [DOI] |
| 12. | Roy S, Hooiveld GJ, Seehawer M, Caruso S, Heinzmann F, Schneider AT, Frank AK, Cardenas DV, Sonntag R, Luedde M, Trautwein C, Stein I, Pikarsky E, Loosen S, Tacke F, Ringelhan M, Avsaroglu SK, Goga A, Buendia MA, Vucur M, Heikenwalder M, Zucman-Rossi J, Zender L, Roderburg C, Luedde T. microRNA 193a-5p Regulates Levels of Nucleolar- and Spindle-Associated Protein 1 to Suppress Hepatocarcinogenesis. Gastroenterology. 2018;155:1951-1966.e26. [PubMed] [DOI] |
| 13. | Zeng H, Ji J, Song X, Huang Y, Li H, Huang J, Ma X. Stemness Related Genes Revealed by Network Analysis Associated With Tumor Immune Microenvironment and the Clinical Outcome in Lung Adenocarcinoma. Front Genet. 2020;11:549213. [PubMed] [DOI] |
| 14. | Fang L, Zhang M, Chen L, Xiong H, Ge Y, Lu W, Wu X, Heng B, Yu D, Wu S. [Corrigendum] Downregulation of nucleolar and spindleassociated protein 1 expression suppresses cell migration, proliferation and invasion in renal cell carcinoma. Oncol Rep. 2021;45:793. [PubMed] [DOI] |
| 15. | Polini B, Carpi S, Doccini S, Citi V, Martelli A, Feola S, Santorelli FM, Cerullo V, Romanini A, Nieri P. Tumor Suppressor Role of hsa-miR-193a-3p and -5p in Cutaneous Melanoma. Int J Mol Sci. 2020;21. [PubMed] [DOI] |
| 16. | Okamoto A, Higo M, Shiiba M, Nakashima D, Koyama T, Miyamoto I, Kasama H, Kasamatsu A, Ogawara K, Yokoe H, Tanzawa H, Uzawa K. Down-Regulation of Nucleolar and Spindle-Associated Protein 1 (NUSAP1) Expression Suppresses Tumor and Cell Proliferation and Enhances Anti-Tumor Effect of Paclitaxel in Oral Squamous Cell Carcinoma. PLoS One. 2015;10:e0142252. [PubMed] [DOI] |
| 17. | Bossaert M, Pipier A, Riou JF, Noirot C, Nguyên LT, Serre RF, Bouchez O, Defrancq E, Calsou P, Britton S, Gomez D. Transcription-associated topoisomerase 2α (TOP2A) activity is a major effector of cytotoxicity induced by G-quadruplex ligands. Elife. 2021;10. [PubMed] [DOI] |
| 18. | Ogino M, Fujii T, Nakazawa Y, Higuchi T, Koibuchi Y, Oyama T, Horiguchi J, Shirabe K. Implications of Topoisomerase (TOP1 and TOP2α) Expression in Patients With Breast Cancer. In Vivo. 2020;34:3483-3487. [PubMed] [DOI] |
| 19. | Hendricks A, Gieseler F, Nazzal S, Bräsen JH, Lucius R, Sipos B, Claasen JH, Becker T, Hinz S, Burmeister G, Schafmayer C, Schrader C. Prognostic relevance of topoisomerase II α and minichromosome maintenance protein 6 expression in colorectal cancer. BMC Cancer. 2019;19:429. [PubMed] [DOI] |
| 20. | Bredel M, Slavc I, Birner P, Czech T, Haberler C, Ströbel T, Wolfsberger S, Budka H, Hainfellner JA. DNA topoisomerase IIalpha expression in optic pathway gliomas of childhood. Eur J Cancer. 2002;38:393-400. [PubMed] [DOI] |
| 21. | Hikita K, Yamakage Y, Okunaga H, Motoyama Y, Matsuyama H, Matsuoka K, Murata T, Nakayoshi T, Oda A, Kato K, Tanaka H, Asao N, Dan S, Kaneda N. (S)-Erypoegin K, an isoflavone isolated from Erythrina poeppigiana, is a novel inhibitor of topoisomerase IIα: Induction of G2 phase arrest in human gastric cancer cells. Bioorg Med Chem. 2021;30:115904. [PubMed] [DOI] |
| 22. | Zhang F, Wu H. MiR-599 targeting TOP2A inhibits the malignancy of bladder cancer cells. Biochem Biophys Res Commun. 2021;570:154-161. [PubMed] [DOI] |
| 23. | Coleman LW, Perkins SL, Bronstein IB, Holden JA. Expression of DNA toposiomerase I and DNA topoisomerase II-alpha in testicular seminomas. Hum Pathol. 2000;31:728-733. [PubMed] [DOI] |
| 24. | Pires E, Sharma N, Selemenakis P, Wu B, Huang Y, Alimbetov DS, Zhao W, Wiese C. RAD51AP1 mediates RAD51 activity through nucleosome interaction. J Biol Chem. 2021;297:100844. [PubMed] [DOI] |
| 25. | Obama K, Satoh S, Hamamoto R, Sakai Y, Nakamura Y, Furukawa Y. Enhanced expression of RAD51 associating protein-1 is involved in the growth of intrahepatic cholangiocarcinoma cells. Clin Cancer Res. 2008;14:1333-1339. [PubMed] [DOI] |
| 26. | Zheng R, Lai G, Li R, Hao Y, Cai L, Jia J. Increased expression of MCM4 is associated with poor prognosis in patients with hepatocellular carcinoma. J Gastrointest Oncol. 2021;12:153-173. [PubMed] [DOI] |
| 27. | Xu Y, Yang X, Si T, Yu H, Li Y, Xing W, Guo Z. MCM4 in human hepatocellular carcinoma: a potent prognostic factor associated with cell proliferation. Biosci Trends. 2021;15:100-106. [PubMed] [DOI] |
| 28. | Huang XP, Rong TH, Wu QL, Fu JH, Yang H, Zhao JM, Fang Y. MCM4 expression in esophageal cancer from southern China and its clinical significance. J Cancer Res Clin Oncol. 2005;131:677-682. [PubMed] [DOI] |
| 29. | Issac MSM, Yousef E, Tahir MR, Gaboury LA. MCM2, MCM4, and MCM6 in Breast Cancer: Clinical Utility in Diagnosis and Prognosis. Neoplasia. 2019;21:1015-1035. [PubMed] [DOI] |
| 30. | Tantiwetrueangdet A, Panvichian R, Sornmayura P, Leelaudomlipi S, Macoska JA. PCNA-associated factor (KIAA0101/PCLAF) overexpression and gene copy number alterations in hepatocellular carcinoma tissues. BMC Cancer. 2021;21:295. [PubMed] [DOI] |
| 31. | Lv W, Su B, Li Y, Geng C, Chen N. KIAA0101 inhibition suppresses cell proliferation and cell cycle progression by promoting the interaction between p53 and Sp1 in breast cancer. Biochem Biophys Res Commun. 2018;503:600-606. [PubMed] [DOI] |
| 32. | Cao H, Zheng J, Yao Y, Yang Q, Yan R, Sun W, Ruan K, Zhou J, Zhou J. Overexpression of KIAA0101 Promotes the Progression of Non-small Cell Lung Cancer. J Cancer. 2020;11:6663-6674. [PubMed] [DOI] |
| 33. | Shen A, Liu L, Chen H, Qi F, Huang Y, Lin J, Sferra TJ, Sankararaman S, Wei L, Chu J, Chen Y, Peng J. Cell division cycle associated 5 promotes colorectal cancer progression by activating the ERK signaling pathway. Oncogenesis. 2019;8:19. [PubMed] [DOI] |
| 34. | Nguyen MH, Koinuma J, Ueda K, Ito T, Tsuchiya E, Nakamura Y, Daigo Y. Phosphorylation and activation of cell division cycle associated 5 by mitogen-activated protein kinase play a crucial role in human lung carcinogenesis. Cancer Res. 2010;70:5337-5347. [PubMed] [DOI] |
| 35. | Tokuzen N, Nakashiro K, Tanaka H, Iwamoto K, Hamakawa H. Therapeutic potential of targeting cell division cycle associated 5 for oral squamous cell carcinoma. Oncotarget. 2016;7:2343-2353. [PubMed] [DOI] |
| 36. | Chang IW, Lin VC, He HL, Hsu CT, Li CC, Wu WJ, Huang CN, Wu TF, Li CF. CDCA5 overexpression is an indicator of poor prognosis in patients with urothelial carcinomas of the upper urinary tract and urinary bladder. Am J Transl Res. 2015;7:710-722. [PubMed] |
| 37. | Gallegos-Arreola MP, Zúñiga-González GM, Sánchez-López JY, Cruz AYN, Peralta-Leal V, Figuera LE, Puebla-Pérez AM, Ronquillo-Carreón CA, Puebla-Mora AG. TYMS 2R3R polymorphism and DPYD [IVS]14+1G>A gene mutation in Mexican colorectal cancer patients. Acta Biochim Pol. 2018;65:227-234. [PubMed] [DOI] |
| 38. | Xie D, Wang L, Xiao Q, Wu X, Zhang L, Yang Q, Wang L. New Insight into the Octamer of TYMS Stabilized by Intermolecular Cys43-Disulfide. Int J Mol Sci. 2018;19. [PubMed] [DOI] |
| 39. | Agulló-Ortuño MT, García-Ruiz I, Díaz-García CV, Enguita AB, Pardo-Marqués V, Prieto-García E, Ponce S, Iglesias L, Zugazagoitia J, López-Martín JA, Paz-Ares L, Nuñez JA. Blood mRNA expression of REV3L and TYMS as potential predictive biomarkers from platinum-based chemotherapy plus pemetrexed in non-small cell lung cancer patients. Cancer Chemother Pharmacol. 2020;85:525-535. [PubMed] [DOI] |
| 40. | Rahimi Z, Bozorgi Zarini M, Rahimi Z, Shakiba E, Vaisi-Raygani A, Moradi MT, Yari K. Variants of Genes Involved in Metabolism of Folate Among Patients with Breast Cancer: Association of TYMS 3R Allele with Susceptibility to Breast Cancer and Metastasis. Iran J Pathol. 2021;16:62-68. [PubMed] [DOI] |
| 41. | Ando H, Fukushima M, Eshima K, Hasui T, Shimizu T, Ishima Y, Huang CL, Wada H, Ishida T. A novel intraperitoneal therapy for gastric cancer with DFP-10825, a unique RNAi therapeutic targeting thymidylate synthase, in a peritoneally disseminated xenograft model. Cancer Med. 2019;8:7313-7321. [PubMed] [DOI] |
| 42. | Karimi L, Jaberi M, Asadi M, Zarredar H, Zafari V, Bornehdeli S, Niknam S, Kermani TA. Significance of microRNA-330-5p/TYMS Expression Axis in the Pathogenesis of Colorectal Tumorigenesis. J Gastrointest Cancer. 2021;. [PubMed] [DOI] |
| 43. | Mizutani Y, Wada H, Yoshida O, Fukushima M, Nonomura M, Nakao M, Miki T. Significance of thymidylate synthase activity in renal cell carcinoma. Clin Cancer Res. 2003;9:1453-1460. [PubMed] |
| 44. | Mou K, Zhang J, Mu X, Wang L, Liu W, Ge R. Zwint facilitates melanoma progression by promoting c-Myc expression. Exp Ther Med. 2021;22:818. [PubMed] [DOI] |
| 45. | Peng F, Li Q, Niu SQ, Shen GP, Luo Y, Chen M, Bao Y. ZWINT is the next potential target for lung cancer therapy. J Cancer Res Clin Oncol. 2019;145:661-673. [PubMed] [DOI] |
| 46. | Ying H, Xu Z, Chen M, Zhou S, Liang X, Cai X. Overexpression of Zwint predicts poor prognosis and promotes the proliferation of hepatocellular carcinoma by regulating cell-cycle-related proteins. Onco Targets Ther. 2018;11:689-702. [PubMed] [DOI] |
| 47. | Yang XY, Wu B, Ma SL, Yin L, Wu MC, Li AJ. Decreased Expression of ZWINT is Associated With Poor Prognosis in Patients With HCC After Surgery. Technol Cancer Res Treat. 2018;17:1533033818794190. [PubMed] [DOI] |