修回日期: 2021-06-07
接受日期: 2021-06-20
在线出版日期: 2021-09-28
消化系统肿瘤是最常见的恶性肿瘤之一, 然而其预后仍不好. 探索潜在生物标志物是肿瘤研究的重要方向. 我们拟利用生物信息学方法探索消化系统的潜在标志物.
探究在消化系统肿瘤中PDE2A表达与免疫浸润相关性, 探索PDE2A在消化系统肿瘤中的预后价值.
通过HPA、TIMER和UALCAN数据库分析PDE2A在不同消化系统肿瘤中的mRNA及蛋白表达, 利用GEPIA数据库探索PDE2A在不同消化系统肿瘤患者预后的影响. 通过TIMER数据库探索PDE2A表达与不同消化系统肿瘤免疫浸润相关性. 利用cBioPortal数据库分析PDE2A在不同消化系统肿瘤的遗传突变信息. 通过STRING数据库探索PDE2A的潜在蛋白相互作用网络.
PDE2A基因mRNA和蛋白在肝癌、胃癌、胰腺癌、结肠癌和食管癌组织中表达低于正常癌旁组织. PDE2A高表达的肝癌和胰腺癌患者较PDE2A低表达的患者预后良好. PDE2A表达与肝癌、胃癌、胰腺癌、结肠癌和食管癌不同免疫细胞如CD4+T淋巴细胞、CD8+T淋巴细胞、B细胞、巨噬细胞和中性粒细胞呈正相关或负相关. PDE2A基因在胃癌、胰腺癌、结肠癌及食管癌中存在错义突变等遗传改变.
PDE2A表达与消化系统肿瘤免疫浸润有关. PDE2A高表达的肝癌和胰腺癌患者预后良好. PDE2A基因可能为潜在的消化系统肿瘤免疫治疗靶点和预后标志物.
核心提要: PDE2A表达与消化系统肿瘤免疫浸润有关. PDE2A高表达的肝癌和胰腺癌患者预后良好. PDE2A基因可能为潜在的消化系统肿瘤免疫治疗靶点和预后标志物.
引文著录: 张钊熊, 韩强, 刘树业. 基于生物信息学分析PDE2A基因在消化系统肿瘤预后及免疫浸润的临床意义. 世界华人消化杂志 2021; 29(18): 1055-1063
Revised: June 7, 2021
Accepted: June 20, 2021
Published online: September 28, 2021
Gastrointestinal cancer is one of the most common malignant cancers, but its prognosis is still poor. Exploring potential biomarkers is an important aspect of cancer research. We intended to explore potential biomarkers for gastrointestinal cancer using bioinformatics methods.
To explore the correlation between the expression of PDE2A and immune infiltration in gastrointestinal cancer, and to investigate the prognostic value of PDE2A in gastrointestinal cancer.
The mRNA and protein expression of PDE2A in different gastrointestinal cancers was analyzed based on the HPA, TIMER, and UALCAN databases. GEPIA database was used to investigate the effect of PDE2A on prognosis of patients with different gastrointestinal cancers. The relationship between PDE2A expression and immune infiltration was analyzed using the TIMER database. Frequencies of PDE2A mutations were analyzed using cBioPortal database. Protein-protein interaction network analysis of PDE2A was based on the STRING database.
The mRNA and protein expression of PDE2A in liver hepatocellular carcinoma (LIHC), stomach adenocarcinoma (STAD), pancreatic adenocarcinoma (PAAD), colon adenocarcinoma (COAD), and esophageal carcinoma (ESCA) was lower than that in normal adjacent tissues. Patients with LIHC and PAAD with high PDE2A expression had a better prognosis. The expression of PDE2A was positively or negatively correlated with different immune cells such as CD4+ T cells, CD8+ T cells, B cells, macrophages, and neutrophils in LIHC, STAD, PAAD, COAD, and ESCA. Finally, there were missense mutations in PDE2A gene in STAD, PAAD, COAD, and ESCA.
The expression of PDE2A is related to immune infiltration in gastrointestinal cancer. Patients with LIHC and PAAD with high PDE2A expression have a better prognosis. PDE2A may be a potential immunotherapy target and prognostic marker for gastrointestinal cancer.
- Citation: Zhang ZX, Han Q, Liu SY. Clinical significance of PDE2A in prognosis and immune infiltration in gastrointestinal cancer based on bioinformatics analysis. Shijie Huaren Xiaohua Zazhi 2021; 29(18): 1055-1063
- URL: https://www.wjgnet.com/1009-3079/full/v29/i18/1055.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v29.i18.1055
癌症是全世界发病率和死亡率的主要原因, 给社会造成巨大的健康和经济负担[1]. 目前多种不同肿瘤的生存率仍不理想, 消化系统肿瘤是导致中国肿瘤患者死亡的主要原因之一[2]. 大样本、高通量和多肿瘤数据对我们找到致癌的关键基因十分必要. 一些医学公共数据库如TCGA (The Cancer Genome Atlas)数据库提供了丰富的组学数据, 使我们在不同组学层面进行探索成为可能.
越来越多的研究关注到肿瘤免疫和预后标志物的探索上. 例如, Ye等[3]发现CD96参与多种免疫应答, 调控免疫细胞浸润, 并影响各种癌症类型的恶性特性. 另有研究报道FOXD1可能为口腔鳞状细胞癌的潜在预后标志物和抗耐药治疗靶点[4]. 肿瘤免疫治疗和靶向药物治疗是肿瘤治疗的重要手段, 但目前对肿瘤免疫治疗和靶向药物治疗中出现的一些问题尚未完全阐明.
PDE2A基因(phosphodiesterase 2A), 中文名为磷酸二酯酶2A, 此基因位于染色体11q13.4上, 属于磷酸水解酶家族. PDE2A基因突变可引起不同的遗传病包括综合征性阵发性运动障碍和非典型Rett综合征[5,6]. 此外, 有研究提示PDE2A在恶性黑色素瘤细胞生长和侵袭中发挥重要作用[7]. 然而, PDE2A基因在消化系统肿瘤中尚未见报道. 本研究中, 我们探索了PDE2A在不同肿瘤类型中的表达情况. 此外, 我们探索了PDE2A表达与5种不同消化系统肿瘤(肝癌、胃癌、胰腺癌、结肠癌和食管癌)预后和免疫浸润的相关性, 以期为消化系统肿瘤的诊治提供理论依据.
(1)HPA数据库(https://http://www.proteinatlas.org/); (2)UALCAN数据库(http://ualcan.path.uab.edu/); (3)GEPIA数据库(http://gepia2.cancer-pku.cn/#index); (4)TIMER数据库(http://timer.cistrome.org/); (5)STRING数据库(https://string-db.org/).
1.2.1 基因表达分析: 通过HPA数据库(https://http://www.proteinatlas.org/)获取PDE2A基因在不同肿瘤中的相对表达水平. 此外, 采用TIMER数据库(http://timer.cistrome.org/)分析PDE2A在不同癌组织和正常癌旁组织的表达情况. 利用UALCAN数据库(http://ualcan.path.uab.edu/)探索PDE2A基因在肝癌、胃癌、胰腺癌、结肠癌和食管癌不同病理分期的表达情况.
1.2.2 蛋白表达分析: 采用HPA数据库探索PDE2A蛋白在不同消化系统肿瘤和正常癌旁组织中的表达水平.
1.2.3 生存预后分析: 利用GEPIA数据库(http://gepia2.cancer-pku.cn/#index)分析PDE2A mRNA表达对肝癌、胃癌、胰腺癌、结肠癌和食管癌患者预后的影响.
1.2.4 免疫浸润分析: 通过TIMER数据库中的"Immune association"模块探索PDE2A与5种不同消化系统肿瘤免疫浸润程度的相关性, 包括CD4+T淋巴细胞、CD8+T淋巴细胞、B细胞、巨噬细胞和中性粒细胞.
1.2.5 遗传突变分析: 通过cBioPortal数据库(https://www.cbioportal.org/)的"Cancer types summary"模块分析PDE2A基因在所有TCGA肿瘤类型的突变类型和拷贝数改变(Copy number alteration, CNV)信息. 此外, 利用"Mutations"模块分析PDE2A基因在肝癌、胃癌、胰腺癌、结肠癌和食管癌中的遗传突变信息.
1.2.6 蛋白相互作用分析: 利用STRING数据库(https://string-db.org/)探索PDE2A的潜在蛋白相互作用网络.
统计学处理 肿瘤组和正常组表达数据两组比较采用Wilcoxon检验. 肿瘤患者的生存分析采用COX回归分析. 免疫分析采用Spearman相关分析. P<0.05为差异有统计学意义.
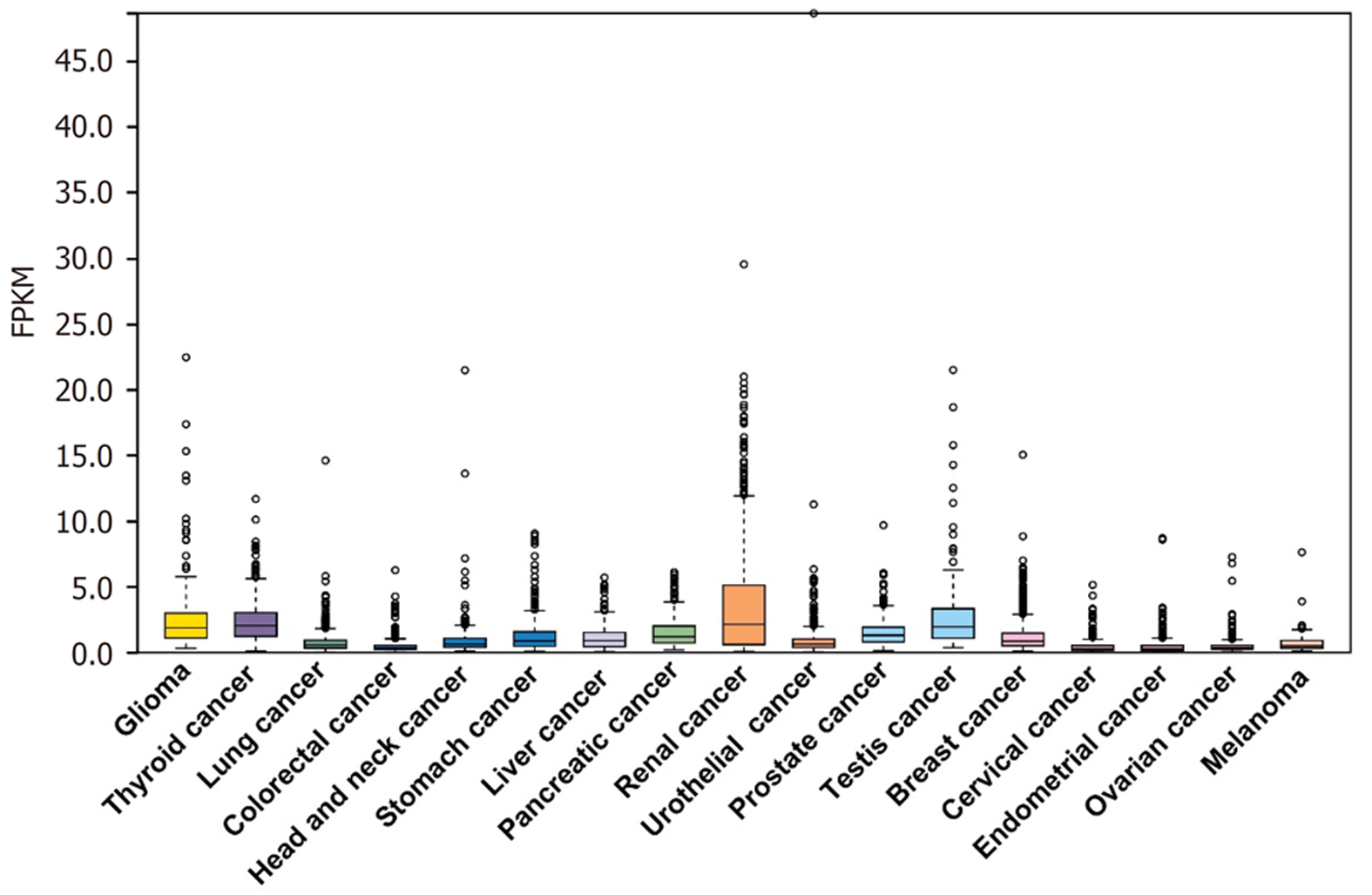
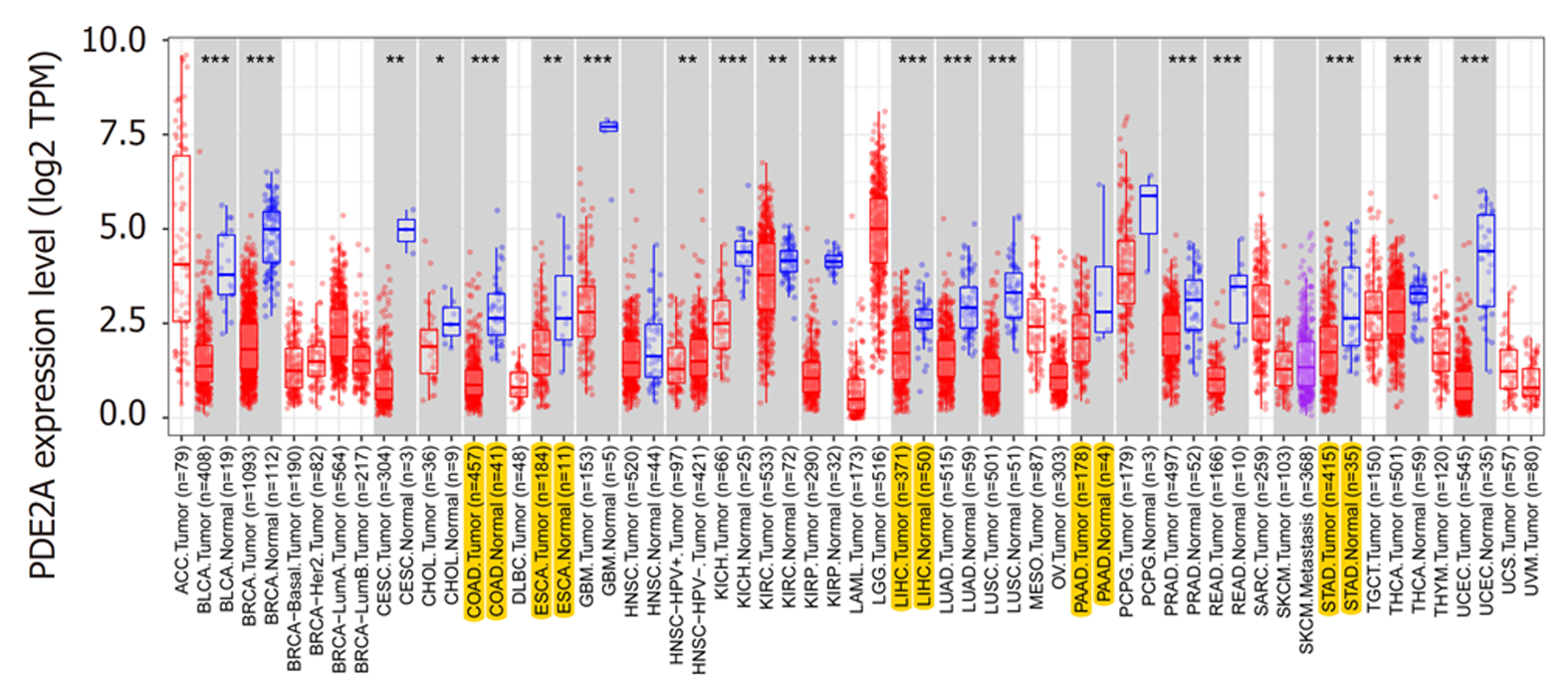
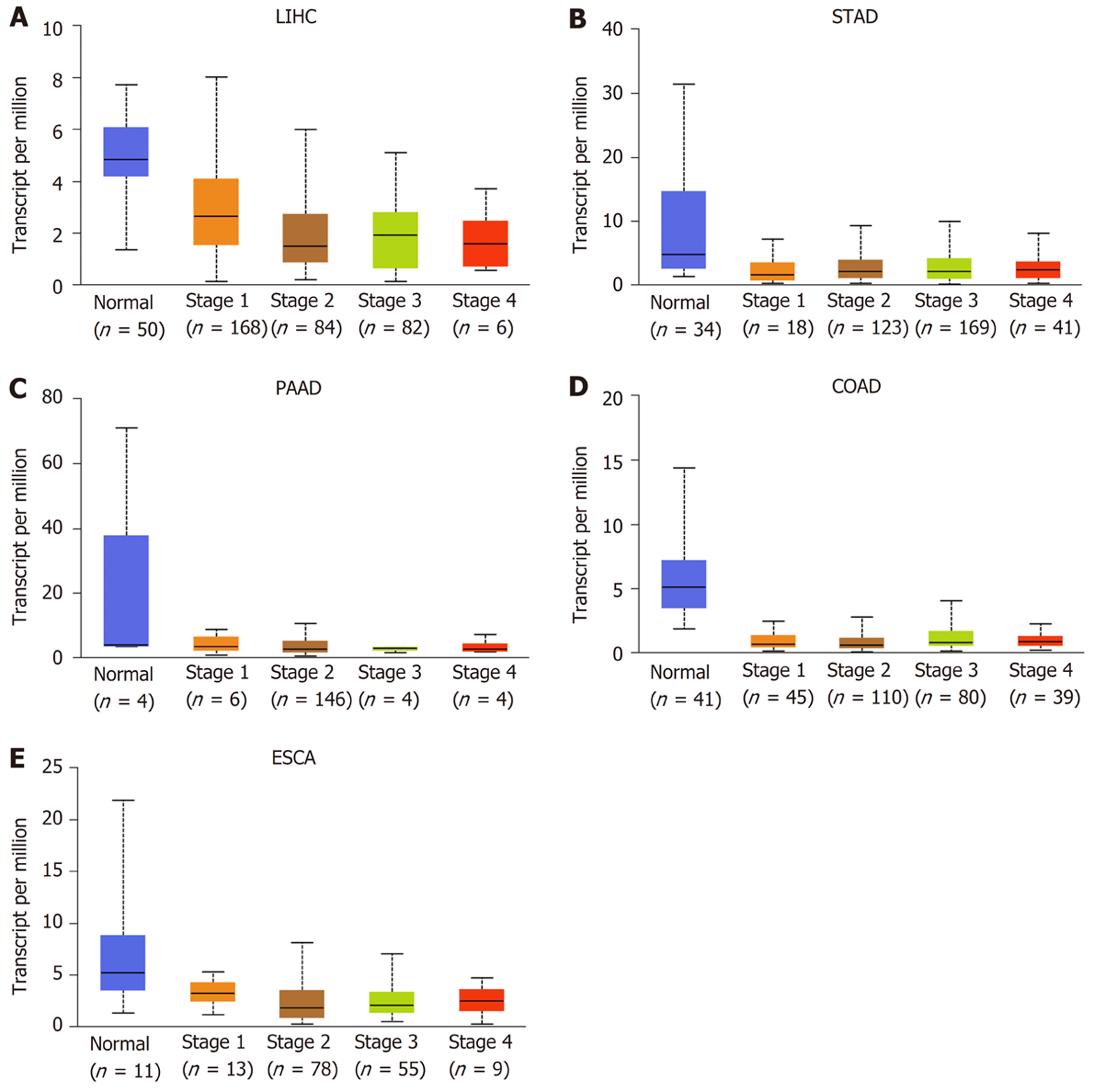
我们通过HPA数据集分析了PDE2A基因在不同肿瘤组织中的表达(图1). 此外, 我们进一步分析了PDE2A在不同肿瘤组织和正常癌旁组织中的表达, TIMER数据库结果提示PDE2A基因在肝癌、胃癌、胰腺癌、结肠癌和食管癌组织中表达显著低于正常癌旁组织(图2). 最后, 我们通过UALCAN数据库分析了不同消化系统肿瘤不同病理分期中PDE2A基因的表达(图3A-E).
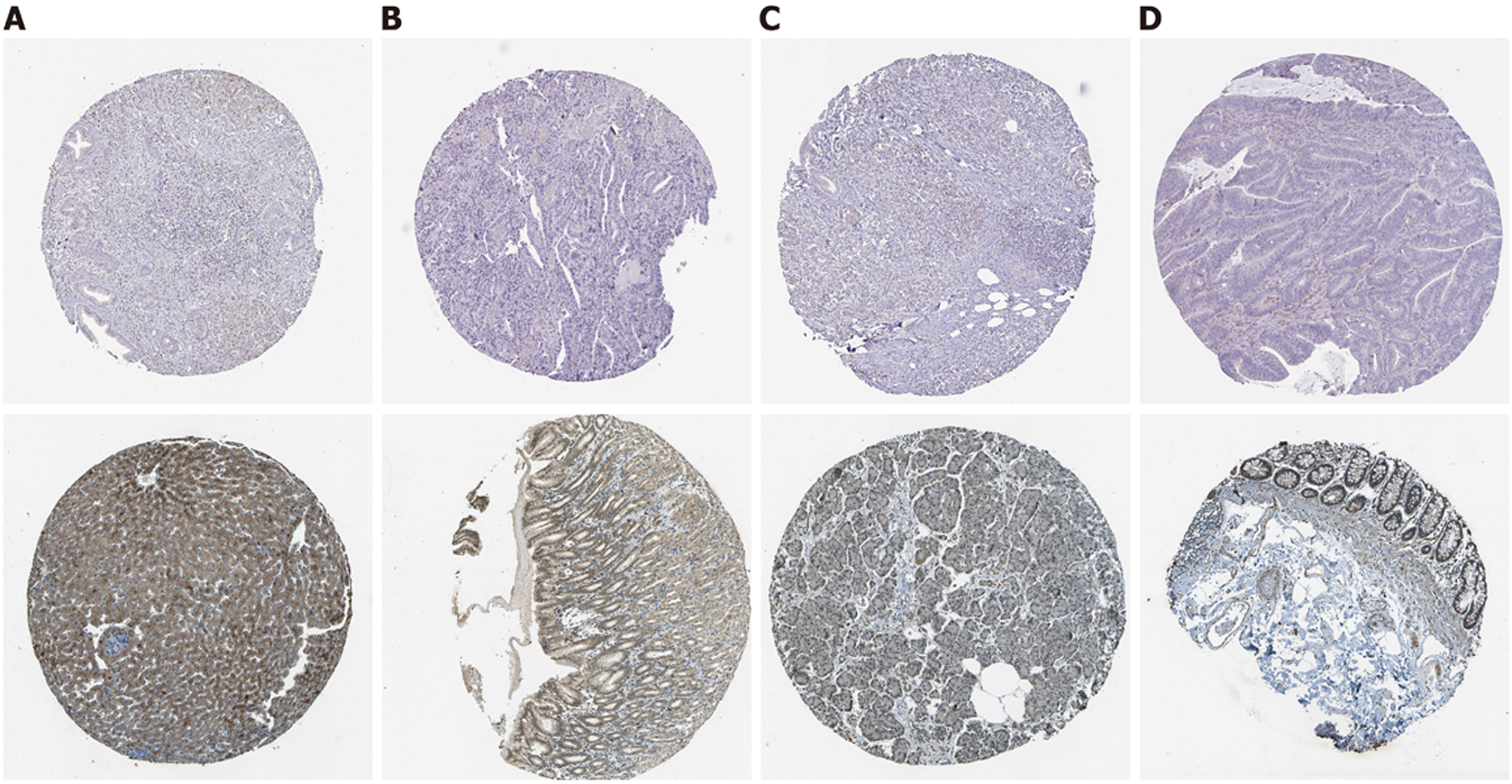
我们通过分析HPA数据库探索了PDE2A蛋白在不同消化系统中的表达情况. HPA数据库结果提示PDE2A蛋白在肝癌、胃癌、胰腺癌和结肠癌组织中呈阴性表达或低表达, 在正常肝组织、胃组织、胰腺组织和结直肠组织中呈中等表达或高表达(图4A-D).
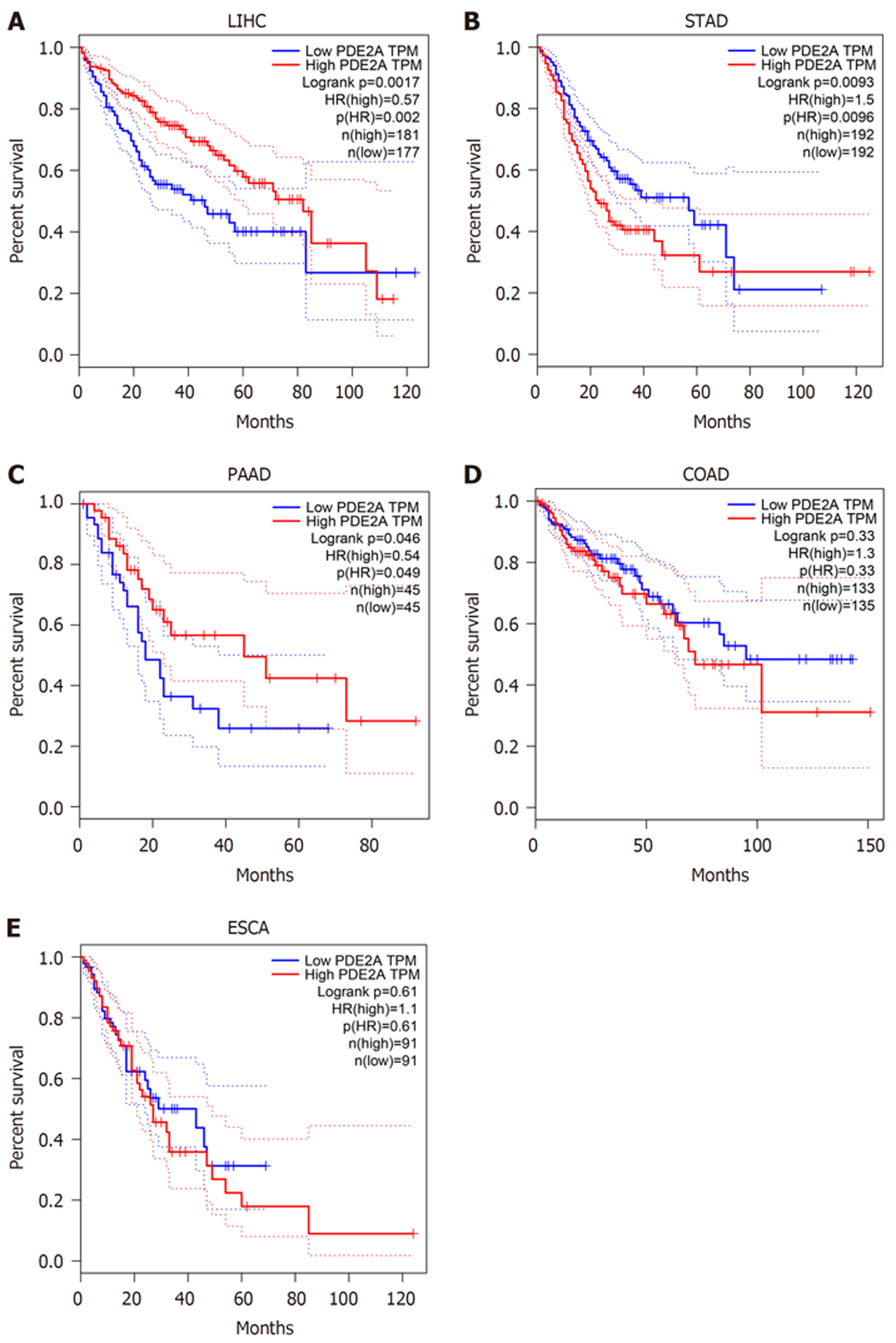
为了进一步研究PDE2A在不同消化系统肿瘤中的预后价值, 我们分析了GEPIA数据库. 结果提示PDE2A高表达的肝癌和胰腺癌患者较PDE2A低表达的患者预后良好(图5A、图5C). 然而PDE2A高表达的胃癌患者较PDE2A低表达的患者预后差(图5B). PDE2A表达对结肠癌和食管癌患者预后无影响(图5D、图5E).
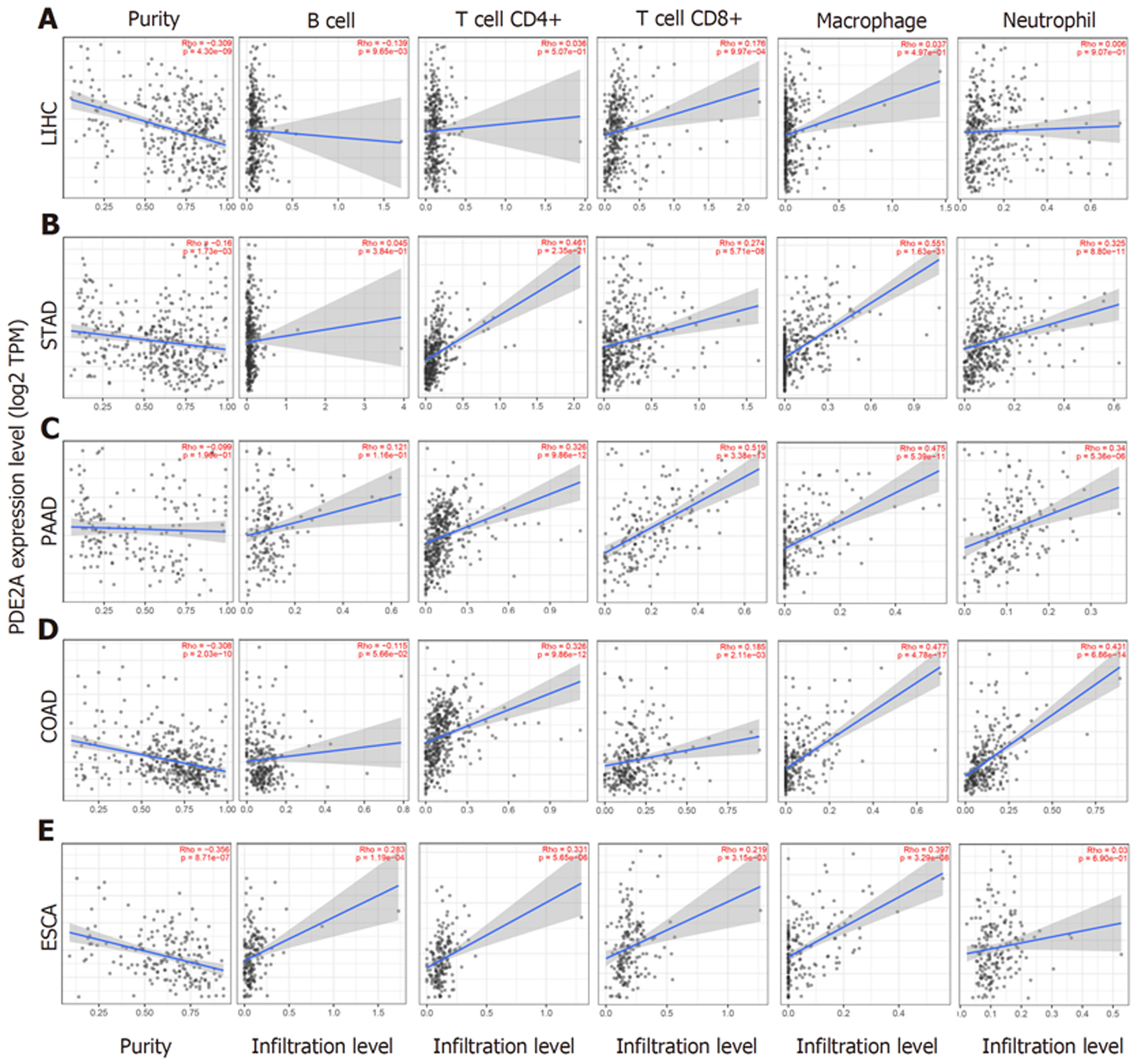
为了探索PDE2A表达与肝癌、胃癌、胰腺癌、结肠癌和食管癌不同免疫细胞的相关性, 我们分析了TIMER数据库. 结果提示PDE2A表达与肝癌、胃癌、胰腺癌、结肠癌和食管癌不同免疫细胞如CD4+T淋巴细胞、CD8+T淋巴细胞、B细胞、巨噬细胞和中性粒细胞呈正相关或负相关(图6A-E).
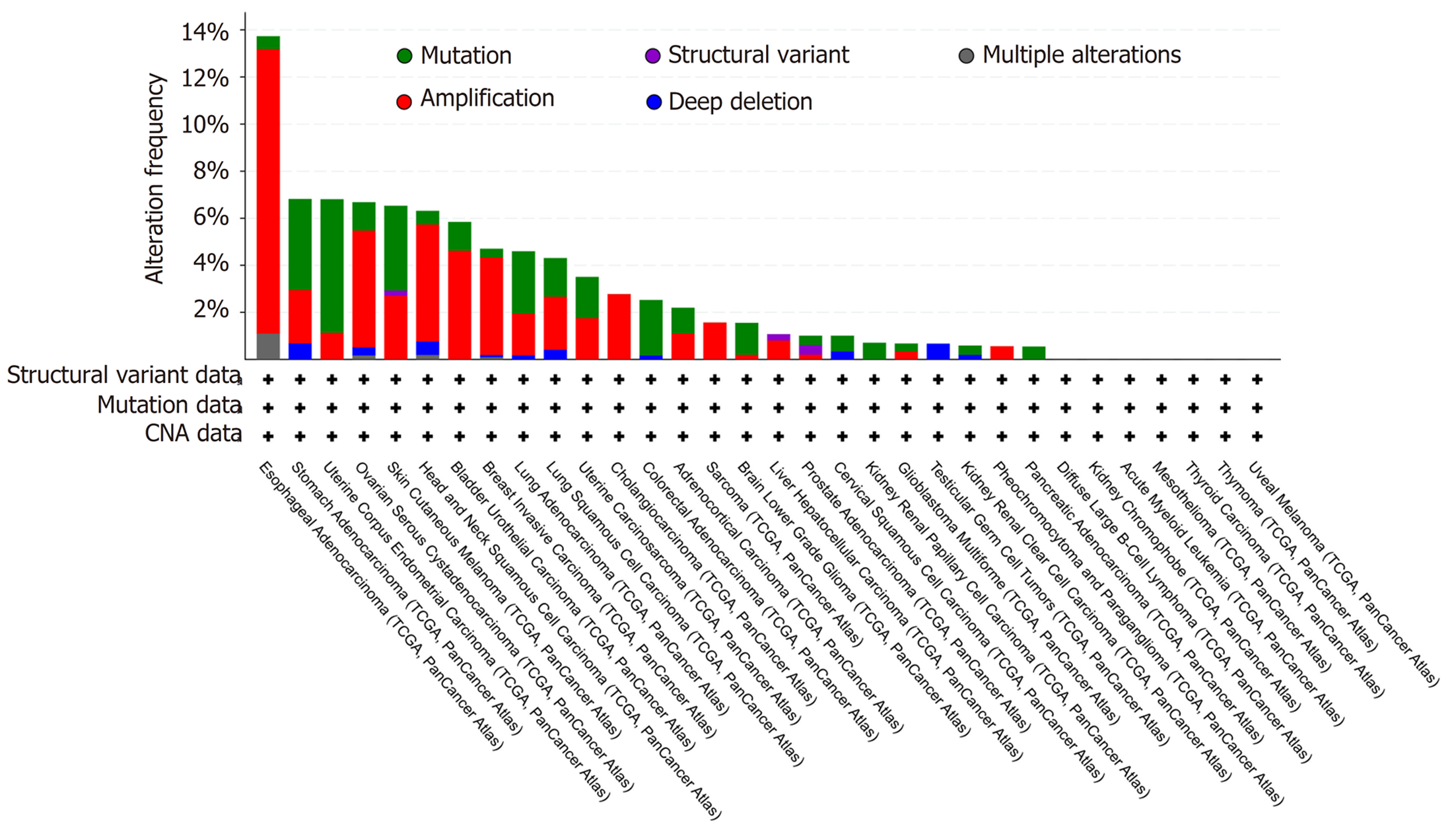
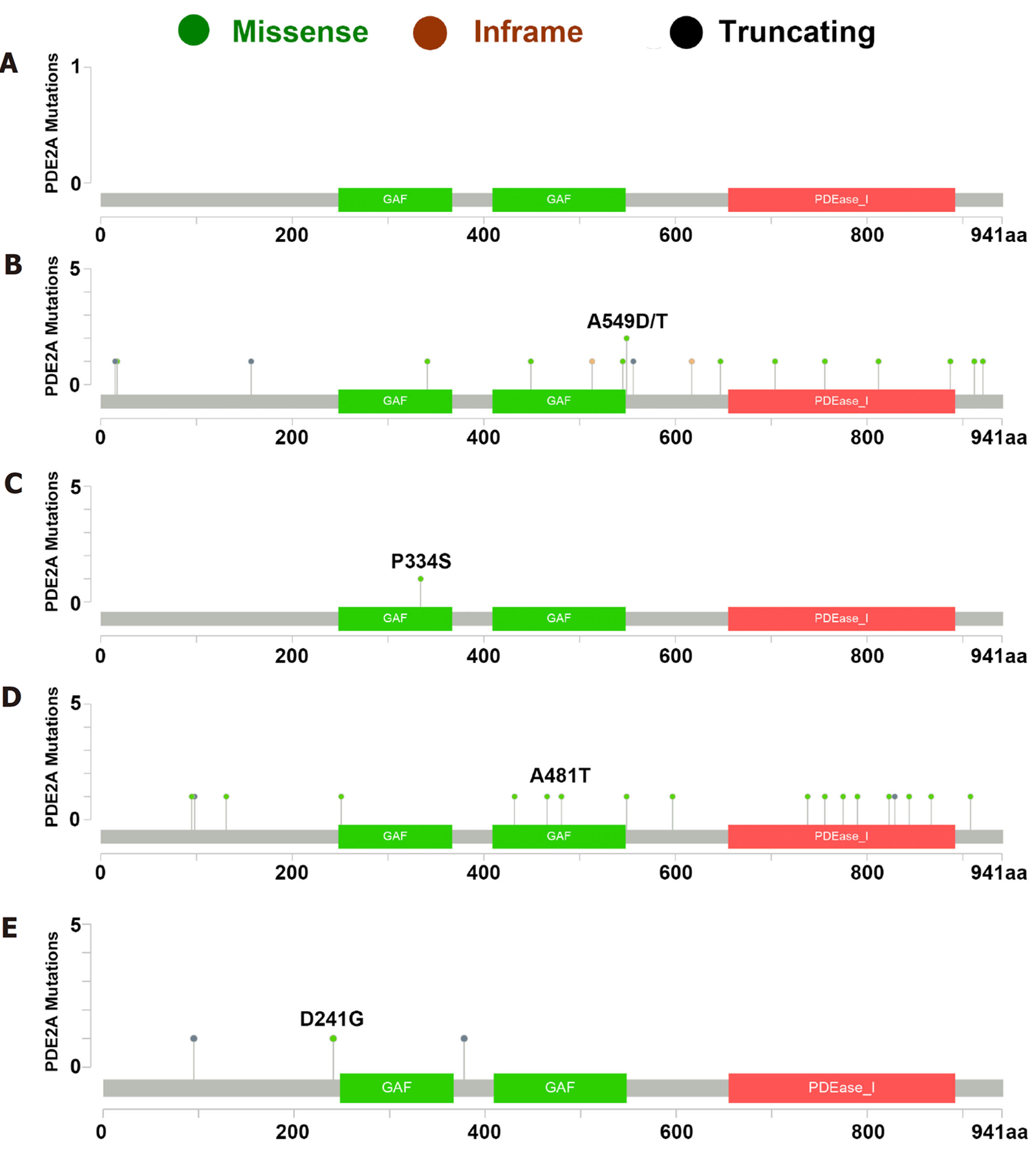
我们通过cBioPortal数据库分析了PDE2A基因在不同肿瘤中的遗传突变信息, 结果提示PDE2A基因在不同肿瘤中存在结构变异、突变及染色体缺失等遗传改变(图7). 我们进一步通过"Mutations"模块分析了PDE2A基因在肝癌、胃癌、胰腺癌、结肠癌和食管癌中的突变情况, 结果提示PDE2A基因胃癌、胰腺癌、结肠癌及食管癌中存在错义突变等遗传改变(图8B-E), 然而在肝癌中无基因突变发生(图8A).
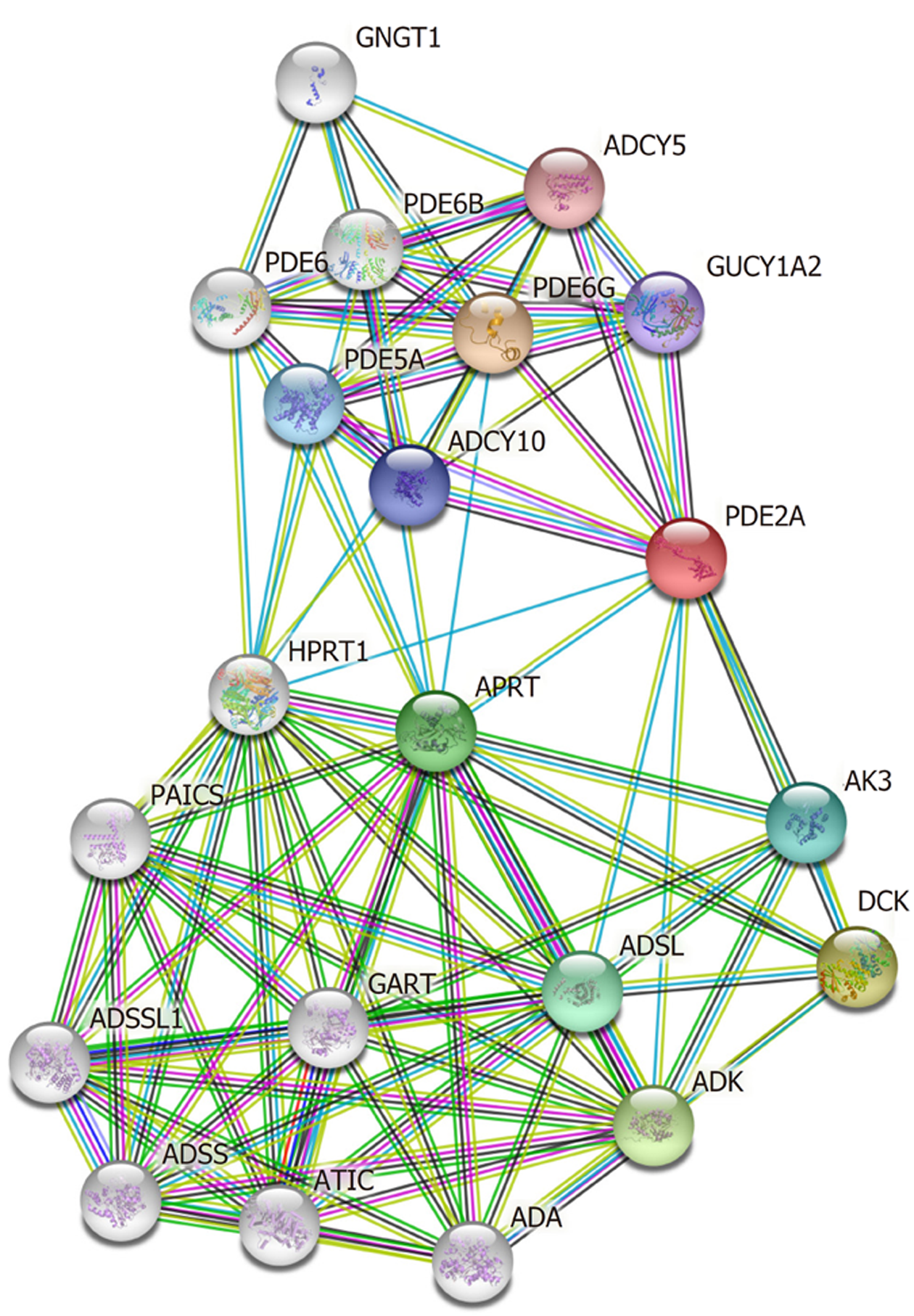
为了探索PDE2A的潜在蛋白相互作用网络, 我们分析了STRING数据库. 结果提示PDE2A的潜在相互作用蛋白包括ADCY10、GUCY1A2、APRT和HPRT1等(图9).
目前针对消化系统肿瘤的治疗方法主要包括外科手术、化疗和免疫治疗[8]. 晚期消化系统肿瘤患者的生存率仍较低, 从分子角度寻找更好的消化系统肿瘤免疫治疗标志物和诊治标志物十分必要. Kang等[9]发现LIMK1可促进胃癌腹膜转移且可能是胃癌治疗的靶点. 另一研究提示血清外泌体中的miR-638可能为肝癌重要的独立预后标志物[10]. Peng等[11]发现CMTM6和PD-L1共表达与结直肠癌免疫微环境激活和预后良好相关. 上述研究对我们明确消化系统肿瘤的免疫机制和预后判断提供了有力证据, 但我们对消化系统肿瘤免疫和预后标志物的研究仍不足.
目前已有多篇研究发现PDE2A参与不同肿瘤的病理进展. 不同研究发现PDE2A可作为宫颈腺癌早期诊断和预后评估的生物标志物[12,13]. 另一学者的结果提示长链非编码RNA LINC01537通过促进PDE2A表达影响温伯格效应, 进而影响肺癌的能量代谢[14]. 以上研究均提示PDE2A在肿瘤中发挥重要作用, 然而PDE2A在消化系统肿瘤如肝癌等尚未见报道.
我们的研究首次探索了PDE2A在消化系统肿瘤中的表达, 基因分析结果提示PDE2A基因和蛋白在肝癌、胃癌、胰腺癌、结肠癌和食管癌患者中表达低于正常癌旁组织. 然而, Ding等的研究与我们的发现相反. 他们的结果提示PDE2A在宫颈癌组织和HeLa细胞中呈高表达[12]. 我们认为导致PDE2A在不同肿瘤中表达差异的原因可能为不同肿瘤间存在异质性. 此外, 我们还发现PDE2A高表达的肝癌和胰腺癌患者预后良好, 然而PDE2A高表达的胃癌患者预后较差. 我们认为有如下原因: 首先, 纳入研究患者的数量不够大, 其次, 其他因素可能影响了肿瘤患者的预后.
免疫治疗在消化系统肿瘤如肝癌和肠癌中取得了显著效果[15,16]. 大量的证据表明, 免疫细胞浸润可影响肿瘤的发生和复发, 是决定免疫治疗反应和临床疗效的重要因素[17]. 因此, 越来越多的学者将研究重点关注到肿瘤的免疫方向. 我们的结果发现PDE2A表达与肝癌、胃癌、胰腺癌、结肠癌和食管癌不同免疫细胞如CD4+T淋巴细胞、CD8+T淋巴细胞等呈正相关或负相关, 提示PDE2A表达水平可反应消化道肿瘤的免疫状态. 肿瘤浸润性淋巴细胞包括T淋巴细胞和B淋巴细胞, 具有抗肿瘤功能. 其中CD8 + T淋巴细胞能特异性识别主要组织相容性复合体抗原, 广泛应用于肿瘤靶向治疗[18]. 我们的结果可能为消化系统肿瘤免疫治疗的研究提供潜在依据. 我们同样分析了PDE2A基因在消化系统肿瘤中的遗传突变, 结果提示PDE2A基因胃癌、胰腺癌、结肠癌及食管癌中存在错义突变等遗传改变. 上述结果可能为消化系统疾病靶向药物的开发提供潜在线索.
然而, 我们的研究仍有一些不足. 首先, 我们的研究主要是基于数据库挖掘, 我们拟开展更多的体内外实验验证实验. 其次, PDE2A在不同消化系统肿瘤如肝癌、胃癌、胰腺癌、结肠癌和食管癌中呈低表达, 但其低表达的分子机制尚不清楚. 我们拟在后续研究探索PDE2A在消化系统肿瘤中的具体分子机制.
综上所述, 本研究利用生物信息学方法分析了PDE2A基因在消化系统肿瘤预后及免疫浸润的临床意义. 我们发现PDE2A mRNA和蛋白在肝癌、胃癌、胰腺癌、结肠癌和食管癌患者呈低表达. 此外, PDE2A表达与不同消化系统肿瘤的免疫浸润有关. PDE2A高表达的肝癌和胰腺癌患者预后良好. PDE2A可能为潜在的消化系统肿瘤免疫治疗和预后标志物.
消化系统肿瘤的预后尚不理想. 生物标志物是肿瘤研究的重要方向之一, 探索新的消化系统肿瘤相关标志物十分必要.
通过生物信息学探索PDE2A在消化系统肿瘤的潜在临床意义.
探究PDE2A是否与消化系统肿瘤免疫浸润有关, 探索PDE2A是否影响消化系统肿瘤患者的预后.
通过生物信息学分析PDE2A在不同消化系统肿瘤中的mRNA及蛋白表达. 探索PDE2A对消化系统肿瘤患者预后的影响. 分析PDE2A表达与消化系统肿瘤免疫浸润的相关性. 探索PDE2A在不同消化系统肿瘤的遗传突变情况.
PDE2A基因的mRNA及蛋白在肝癌、胃癌和胰腺癌等消化系统肿瘤中呈低表达. PDE2A高表达的肝癌和胰腺癌患者预后良好. PDE2A表达与不同消化系统肿瘤的免疫浸润有关. PDE2A基因胃癌、胰腺癌、结肠癌及食管癌中存在错义突变等遗传改变.
PDE2A基因在肝癌、胃癌、胰腺癌、结肠癌和食管癌患者呈低表达且与免疫浸润有关.
PDE2A基因可能为潜在的消化系统肿瘤免疫治疗和预后标志物.
学科分类: 胃肠病学和肝病学
手稿来源地: 天津市
同行评议报告学术质量分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): C
D级 (一般): D, D
E级 (差): 0
科学编辑: 刘继红 制作编辑:张砚梁
| 1. | Siegel RL, Miller KD, Fuchs HE, Jemal A. Cancer Statistics, 2021. CA Cancer J Clin. 2021;71:7-33. [PubMed] [DOI] |
| 2. | 郑 荣寿, 孙 可欣, 张 思维, 曾 红梅, 邹 小农, 陈 茹, 顾 秀瑛, 魏 文强, 赫 捷. 2015年中国恶性肿瘤流行情况分析. 中华肿瘤杂志. 2019;41:19-28. [DOI] |
| 3. | Ye W, Luo C, Liu F, Liu Z, Chen F. CD96 Correlates With Immune Infiltration and Impacts Patient Prognosis: A Pan-Cancer Analysis. Front Oncol. 2021;11:634617. [PubMed] [DOI] |
| 4. | Chen S, Yang M, Wang C, Ouyang Y, Chen X, Bai J, Hu Y, Song M, Zhang S, Zhang Q. Forkhead box D1 promotes EMT and chemoresistance by upregulating lncRNA CYTOR in oral squamous cell carcinoma. Cancer Lett. 2021;503:43-53. [PubMed] [DOI] |
| 5. | Doummar D, Dentel C, Lyautey R, Metreau J, Keren B, Drouot N, Malherbe L, Bouilleret V, Courraud J, Valenti-Hirsch MP, Minotti L, Dozieres-Puyravel B, Bär S, Scholly J, Schaefer E, Nava C, Wirth T, Nasser H, de Salins M, de Saint Martin A, Warde MTA, Kahane P, Hirsch E, Anheim M, Friant S, Chelly J, Mignot C, Rudolf G. Biallelic PDE2A variants: a new cause of syndromic paroxysmal dyskinesia. Eur J Hum Genet. 2020;28:1403-1413. [PubMed] [DOI] |
| 6. | Haidar Z, Jalkh N, Corbani S, Abou-Ghoch J, Fawaz A, Mehawej C, Chouery E. A Homozygous Splicing Mutation in PDE2A in a Family With Atypical Rett Syndrome. Mov Disord. 2020;35:896-899. [PubMed] [DOI] |
| 7. | Hiramoto K, Murata T, Shimizu K, Morita H, Inui M, Manganiello VC, Tagawa T, Arai N. Role of phosphodiesterase 2 in growth and invasion of human malignant melanoma cells. Cell Signal. 2014;26:1807-1817. [PubMed] [DOI] |
| 8. | Pan R, Zhu M, Yu C, Lv J, Guo Y, Bian Z, Yang L, Chen Y, Hu Z, Chen Z, Li L, Shen H; China Kadoorie Biobank Collaborative Group. Cancer incidence and mortality: A cohort study in China, 2008-2013. Int J Cancer. 2017;141:1315-1323. [PubMed] [DOI] |
| 9. | Kang X, Li W, Liu W, Liang H, Deng J, Wong CC, Zhao S, Kang W, To KF, Chiu PWY, Wang G, Yu J, Ng EKW. LIMK1 promotes peritoneal metastasis of gastric cancer and is a therapeutic target. Oncogene. 2021;40:3422-3433. [PubMed] [DOI] |
| 10. | Yokota Y, Noda T, Okumura Y, Kobayashi S, Iwagami Y, Yamada D, Tomimaru Y, Akita H, Gotoh K, Takeda Y, Tanemura M, Murakami T, Umeshita K, Doki Y, Eguchi H. Serum exosomal miR-638 is a prognostic marker of HCC via downregulation of VE-cadherin and ZO-1 of endothelial cells. Cancer Sci. 2021;112:1275-1288. [PubMed] [DOI] |
| 11. | Peng QH, Wang CH, Chen HM, Zhang RX, Pan ZZ, Lu ZH, Wang GY, Yue X, Huang W, Liu RY. CMTM6 and PD-L1 coexpression is associated with an active immune microenvironment and a favorable prognosis in colorectal cancer. J Immunother Cancer. 2021;9. [PubMed] [DOI] |
| 12. | Ding H, Xiong XX, Fan GL, Yi YX, Chen YR, Wang JT, Zhang W. The New Biomarker for Cervical Squamous Cell Carcinoma and Endocervical Adenocarcinoma (CESC) Based on Public Database Mining. Biomed Res Int. 2020;2020:5478574. [PubMed] [DOI] |
| 13. | Feng G, Beilei Z, Caizhi C, Wen Z. Analysis of CASP12 diagnostic and prognostic values in cervical cancer based on TCGA database. Biosci Rep. 2019;39. [PubMed] [DOI] |
| 14. | Gong W, Yang L, Wang Y, Xian J, Qiu F, Liu L, Lin M, Feng Y, Zhou Y, Lu J. Analysis of Survival-Related lncRNA Landscape Identifies A Role for LINC01537 in Energy Metabolism and Lung Cancer Progression. Int J Mol Sci. 2019;20. [PubMed] [DOI] |
| 17. | Liu X, Wu S, Yang Y, Zhao M, Zhu G, Hou Z. The prognostic landscape of tumor-infiltrating immune cell and immunomodulators in lung cancer. Biomed Pharmacother. 2017;95:55-61. [PubMed] [DOI] |
| 18. | Duewell P, Steger A, Lohr H, Bourhis H, Hoelz H, Kirchleitner SV, Stieg MR, Grassmann S, Kobold S, Siveke JT, Endres S, Schnurr M. RIG-I-like helicases induce immunogenic cell death of pancreatic cancer cells and sensitize tumors toward killing by CD8(+) T cells. Cell Death Differ. 2014;21:1825-1837. [PubMed] [DOI] |