修回日期: 2020-06-27
接受日期: 2020-07-15
在线出版日期: 2020-08-28
肝细胞癌(hepatocellular carcinoma, HCC)是临床上常见的实体恶性肿瘤, 预后较差. 本文拟采用生物信息学技术分析PPP1R105基因在肝细胞癌中的表达相关信号通路及其与患者预后关系. 同时, 采用免疫组织化学法对PPP1R105生物信息数据进行验证.
探讨PPP1R10基因(Ki-67或MKI67)在HCC中的表达及其临床意义.
在TCGA数据中, 检索PPP1R105基因在各个肿瘤组织中的相对表达情况; 分析PPP1R105基因在肝细胞癌组织和癌旁组织中的相对表达水平, 并比较是否存在差异. 应用蛋白相互作用数据库STRING构建PPP1R105基因编码蛋白相互作用网络. 比较PPP1R105高低表达组患者无疾病进展生存(disease free survival, DFS)和总生存(overall survival, OS)是否存在差异. 选取我院手术治疗的原发性肝细胞癌患者81例进行回顾性分析, 采用免疫组织化学法检测肝细胞癌组织中PPP1R105蛋白表达情况, 并与患者的临床特征进行相关性分析.
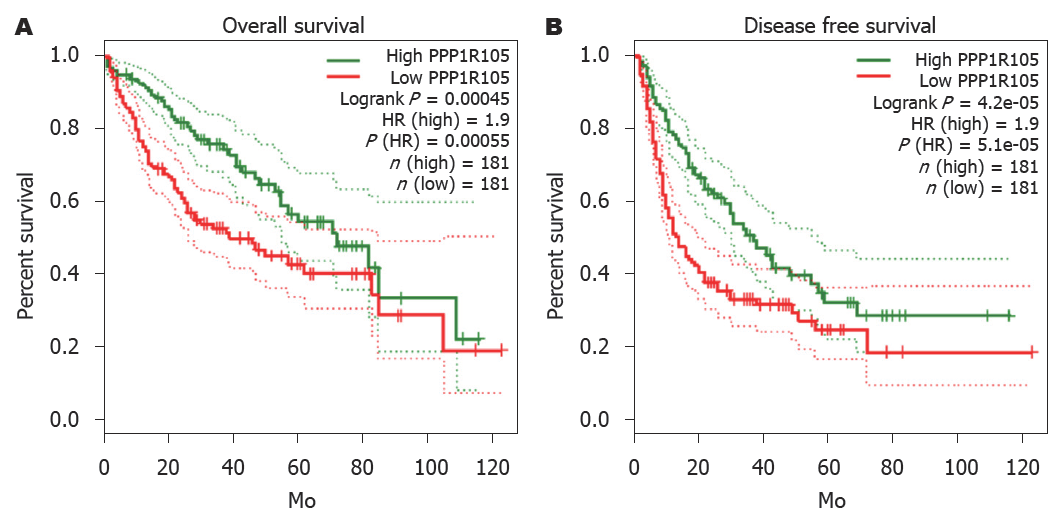
PPP1R105基因mRNA在不同肿瘤中表达水平差异并不显著, 其表达不具备肿瘤间特异性. 在肝细胞癌中, 癌组织PPP1R105mRNA表达水平显著高于癌旁正常肝组织(P<0.05), 且表达水平与患者临床分期有关(P<0.05). 蛋白相互作用网络区域聚集指数为0.99, 蛋白相互作用网络富集明显(P<0.01). PPP1R105及其相关基因信号通路主要富集于细胞周期、细胞衰老、病毒致癌及p53信号通路. KIF18B与PPP1R105正相关表达最为显著(rpearson = 0.91, P<0.05); 而MCELL基因与PPP1R105负相关表达最为显著(rpearson = -0.59, P<0.05); PPP1R105高表达组OS (HR = 1.90, P<0.01)和DFS(HR = 1.90, P<0.01)显著低于低表达组. PPP1R105蛋白主要表达于细胞核质及核仁, 81例患者中高表达者30例(37.0%). PPP1R105蛋白高表达患者肿瘤大于5 cm及TNM分期Ⅲa期患者比例显著高于MIK67蛋白低表达者(P<0.05).
肝细胞癌患者PPP1R105高表达, 其与患者总生存和无疾病进展生存较短有关, 可作为肝细胞癌患者预后不良分子标志物.
核心提要: PPP1R105基因mRNA在肝细胞癌(hepatocellular carcinoma, HCC)组织中表达水明显上调, 其信号通路主要富集于细胞周期、细胞衰老、病毒致癌及p53. PPP1R105高表达组无疾病进展生存和总生存显著低于低表达组. PPP1R105高表达可能是HCC患者预后不良的危险因素.
引文著录: 孙建贺, 侯计平, 康永振. PPP1R105基因在肝细胞癌中的表达相关信号通路及其与患者预后关系. 世界华人消化杂志 2020; 28(16): 765-776
Revised: June 27, 2020
Accepted: July 15, 2020
Published online: August 28, 2020
Hepatocellular carcinoma (HCC) is a common malignant tumor with a poor prognosis. In this study, we evaluated the expression of the PPP1R105 gene in HCC and its relationship with prognosis through bioinformatics analysis. Then, we used immunohistochemistry to verify the expression of PPP1R105 protein.
To investigate the expression of PPP1R105 (Ki-67 or MKI67) in HCC and its clinical significance.
The relative expression data of the PPP1R105 gene in various tumor tissues were retrieved from the tumor protein database and TCGA database and compared between cancer and paired normal tissues. The protein-protein interaction network (PPI) of PPP1R105 was constructed by using the STRING database. The progression free survival (DFS) and overall survival (OS) between the high and low PPP1R105 expression groups were compared. Eighty-one HCC patients who underwent surgery were retrospectively included in the study, and PPP1R105 protein expression was detected by immunohistochemistry. The relationship between PPP1R105 protein expression and patients' features was evaluated.
The expression levels of PPP1R105 mRNA in different tumors were not significantly different. In HCC, the expression level of PPP1R105 mRNA was significantly higher than that of normal liver tissue, and the expression level was related with clinical stage. The aggregation index of PPI was 0.99, and the enrichment of protein interaction network was significant (P < 0.01). PPP1R105 was mainly enriched in cell cycle, cell aging, virus carcinogenesis, and p53 signaling pathway. The positive correlation between KIF18B and PPP1R105 was the most significant (rperson = 0.91, P < 0.05), while the negative correlation between MCELL and PPP1R105 was the most significant (rperson = -0.59, P < 0.05). DFS (HR = 1.90, P < 0.01) and OS (HR = 1.90, P < 0.01) in the high PPP1R105 expression group were significantly shorter than those in the low expression group. PPP1R105 protein was mainly expressed in the cytoplasm and nucleolus of the cells. Of the included 81 patients, 30 (37.0%) showed high expression of PPP1R105 protein. The proportion of patients with high expression of PPP1R105 protein was higher than that of patients with low expression of MIK67 protein (P < 0.05).
PPP1R105 is highly expressed in patients with HCC, which is related to a poor DFS and OS. PPP1R105 can be used as a poor prognostic molecular marker in HCC.
- Citation: Sun JH, Hou JP, Kang YZ. Clinical significance of expression of PPP1R105 in hepatocellular carcinoma. Shijie Huaren Xiaohua Zazhi 2020; 28(16): 765-776
- URL: https://www.wjgnet.com/1009-3079/full/v28/i16/765.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v28.i16.765
在全球范围内, 肝癌是消化系统常见的恶性肿瘤, 尤其是东亚人群, 由于乙肝慢性肝炎患者数量巨大, 乙肝-肝硬化-肝癌的发生率显著高于欧美国家[1,2]. 目前, 肝细胞癌(hepatocellular carcinoma, HCC)已经成为一个重大的全球健康问题. HCC在亚洲和非洲等地区发病率较高, 主要归因于黄曲霉毒素的膳食暴露及慢性乙型肝炎病毒感染[3,4], 而西方欧美国家发病率相对较低[5].
众所周知, 转移是大多数癌症患者的最终死因, 转移和复发也是HCC患者死亡和治疗失败的主要原因. 近年来研究显示, PPP1R105 (Ki-67或MKI67)编码种与细胞增殖有关的核蛋白. PPP1R105编码蛋白(也称为MKI67)是细胞增殖的标志物, 在细胞静休期, PPP1R1057抗原只能在细胞核内检测到, 而在有丝分裂过程中, 大部分蛋白质被重新定位到染色体表面PPP1R105蛋白存在于细胞周期的所有活跃阶段(G1、S、G2和有丝分裂), 但静息(静止)细胞(G0)中不存在PPP1R105蛋白. 已有研究显示, 在多种肿瘤中高表达并与患者的预后不良有关[6,7]. PPP1R105编码蛋白是一种核蛋白, 并作为肿瘤细胞增殖相关标记物. 已有研究也发现PPP1R105与肿瘤转移密切相关[8]. 但PPP1R105作为肝细胞癌患者预后分子标记物及其作为肝细胞癌治疗靶点的研究报道较少.
1.1.1 PPP1R105表达水平分析: 在肿瘤蛋白数据库[9]和TCGA数据中(https://portal.gdc.cancer.gov/), 检索PPP1R105基因在各个肿瘤组织中的相对表达情况; 在TCGA数据库在线分析软件GEPIA[10](http://gepia.cancer-pku.cn/detail.php)中分析PPP1R105基因在肝细胞癌组织和癌旁组织中的相对表达情况, 并比较是否存在差异. 同时分析其PPP1R105 mRNA相对表达水平与患者临床分期的关系.
1.1.2 PPP1R105蛋白-蛋白相互作用网络: 应用蛋白相互作用数据库STRING[11](http://string-db.org/cgi/input.pl)构建PPP1R105基因编码蛋白相互作用网络, 网络构建条件为, 蛋白间相互作用信度0.7, 相互作用蛋白不高于20个(前20), 相互作用来源为共表达、基因功能和比邻关系.
1.1.3 共表达分析: 采用Pearson相关检验对PPP1R105共表达基因进行了聚类分析, 同时对PPP1R105正相关和负相关共表达的top2基因进行了相关性检验.
1.1.4 生存分析: TCGA数据库中, 根据PPP1R105 mRNA在肝细胞癌组织中表达的中位数将患者分为PPP1R105高表达(>PPP1R105 mRNA中位数)和低表达(≤PPP1R105 mRNA中位数)组. 依据风险比例模型绘制高低表达组患者的生存曲线, 对曲线进行log-rank检验, 计算风险比.
1.2.1 IHC检测PPP1R105表达: 选取我院手术治疗的原发性肝细胞癌患者81例进行回顾性分析, 同时于病理科选取对应患者的组织蜡块标本, 采用免疫组织化学法检测肝细胞癌组织中PPP1R105蛋白表达情况, 并与患者的临床特征进行相关性分析. PPP1R105蛋白表达按试剂盒说明进行(PPP1R105兔多克隆抗体, 购自Abcam中国公司, 上海市浦东新区伽利略路338号).
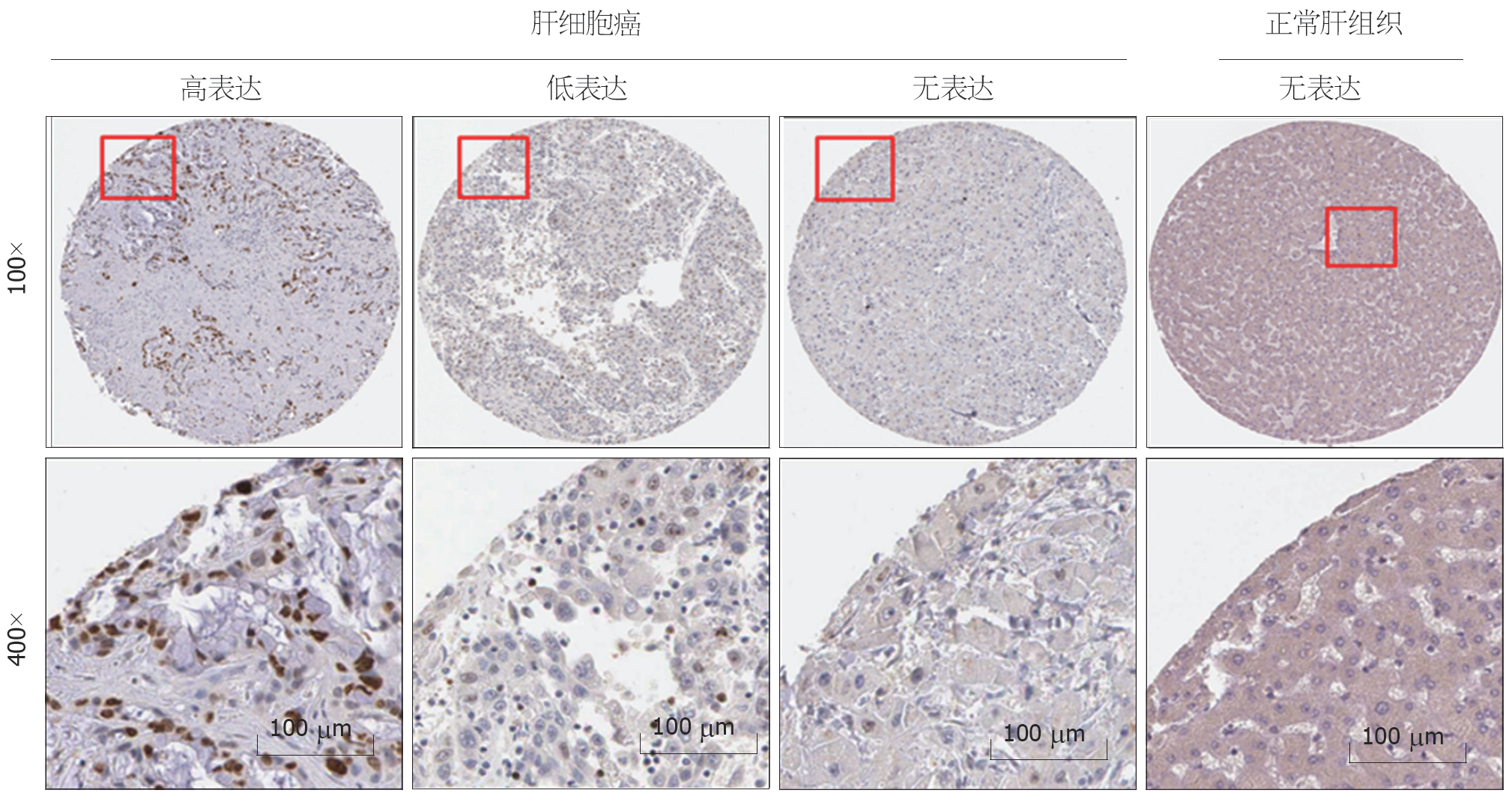
1.2.2 PPP1R105高低表达标准: 选取2个高倍视野, 通过基于强度和异质性的快速评分法(Q-score) 进行半定量评分, 具体评分标准及分值见表1. 每份样本的Q-score得分是染色强度和阳性染色细胞百分比评分的总和, 总评分范围为0-7分, Q-score≥2分为高表达, Q-score<2分为低表达[12].
| 标准(染色强度) | 分值 |
| 不着色 | 0 |
| 浅黄 | 1 |
| 棕黄 | 2 |
| 棕褐色 | 3 |
| 阳性染色肿瘤细胞百分比 | |
| 0% | 0 |
| 1%-25% | 1 |
| 26%-50% | 2 |
| 51%-75% | 3 |
| 76%-100% | 4 |
统计学处理 采用SPSS 22.0软件进行数据分析, 计量资料应用mean±SD表示, ANVOA检验; 计数资料采用n表示, χ2检验; 生存分析采用K-M法, P<0.05为存在统计学差异.
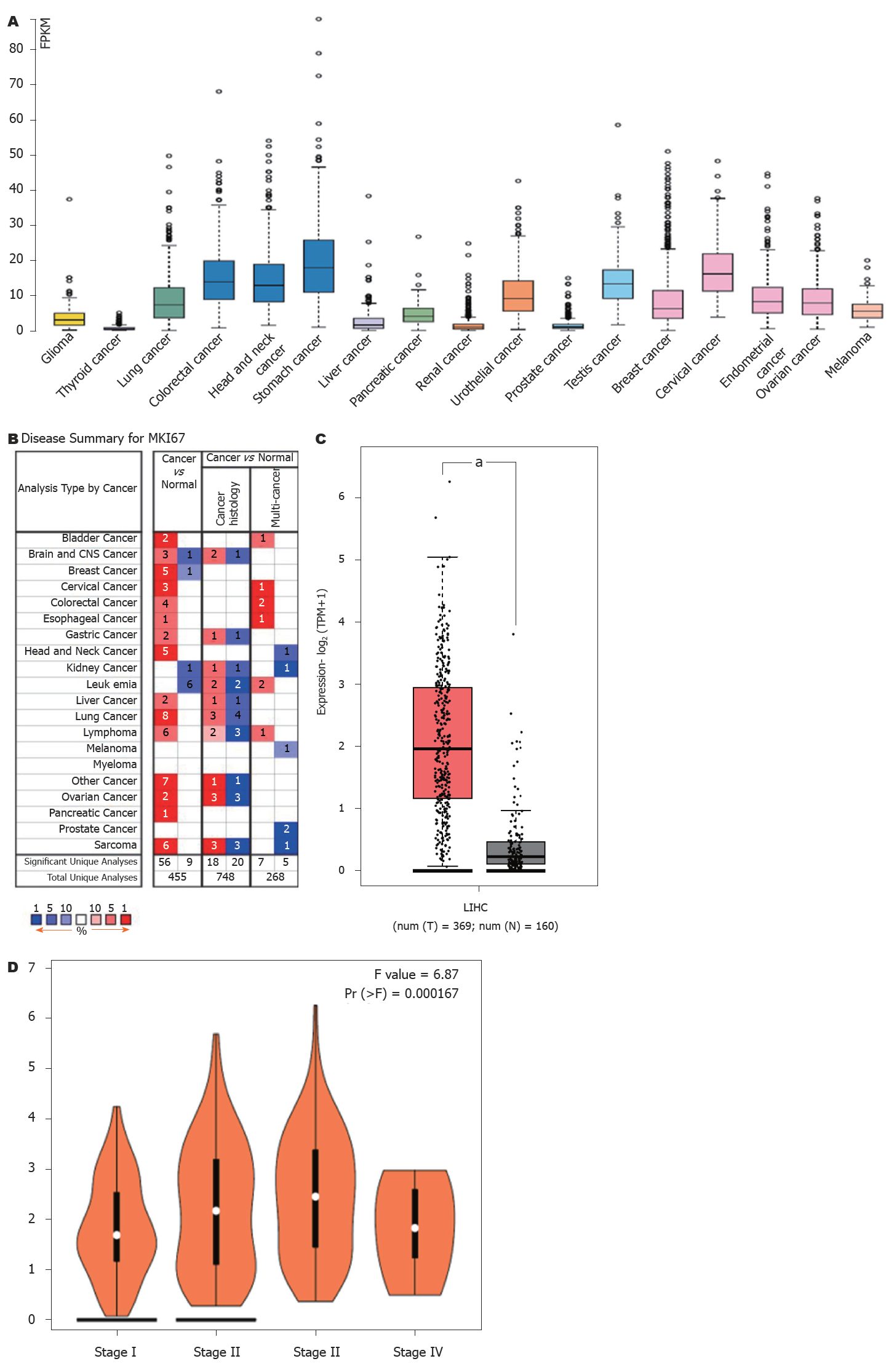
PPP1R105基因mRNA在不同肿瘤中表达水平差异并不显著, 其表达特不具备肿瘤间特异性(图1A). 但在大多数肿瘤组织中, 其表达水平均高于对应的正常组织(图1B). 在肝细胞癌中, 癌组织中PPP1R105 mRNA表达水平显著高于癌旁正常肝组织(图1C)且表达水平与患者临床分期有关(图1D).
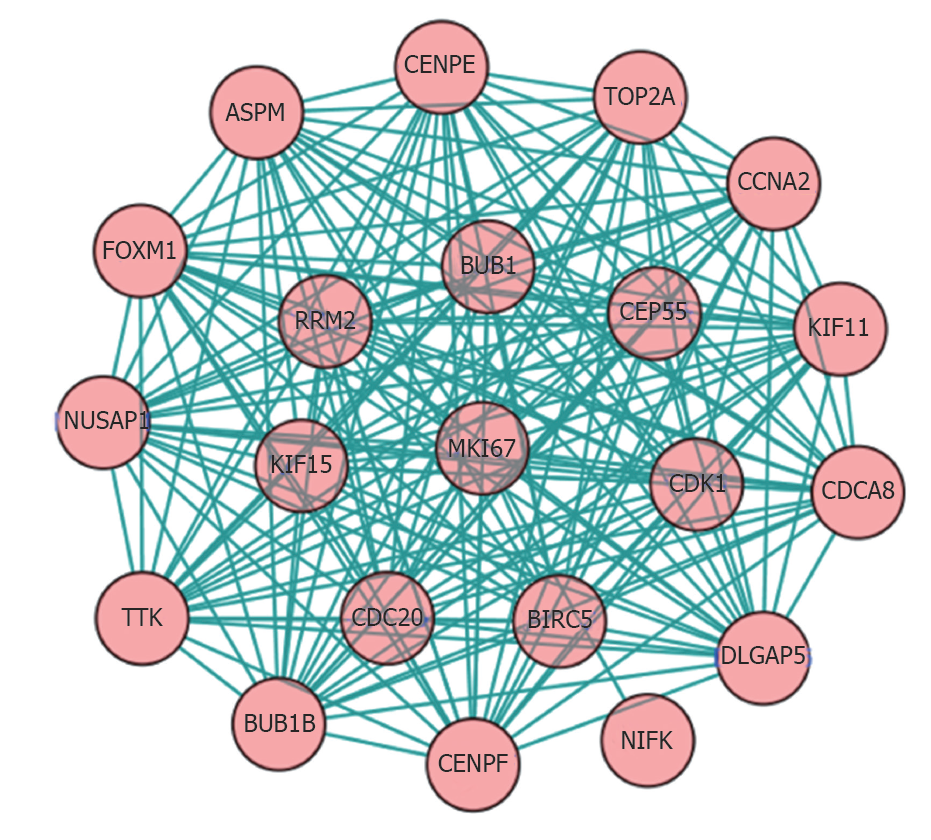
PPP1R105基因编码蛋白相互作用网络中筛选出20个相关基因, 相互作用关系为191, 平均相互作用指数为18.2, 区域聚集指数为0.99, 蛋白相互作用网络富集明显(P<0.01), 图2.
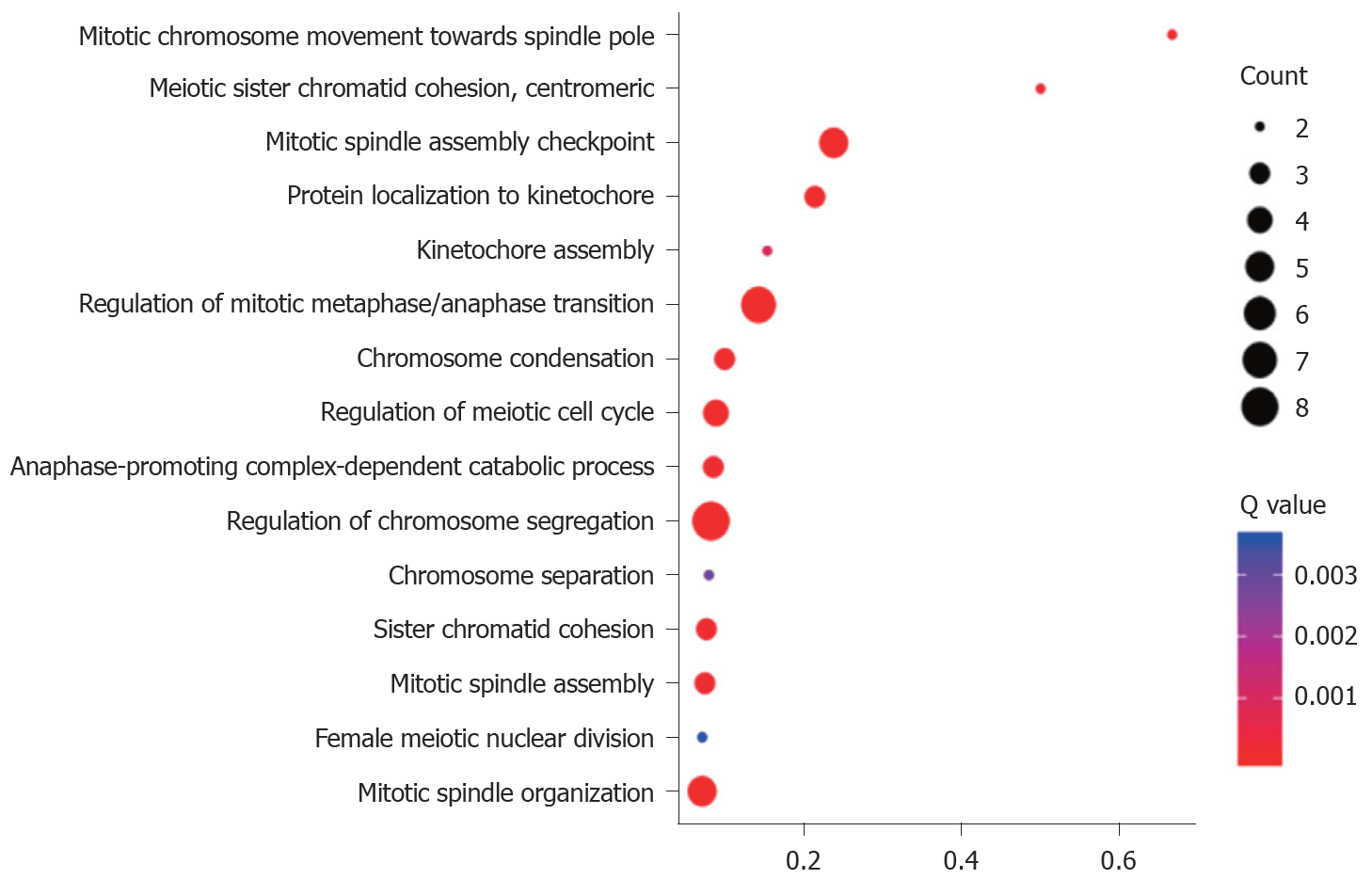
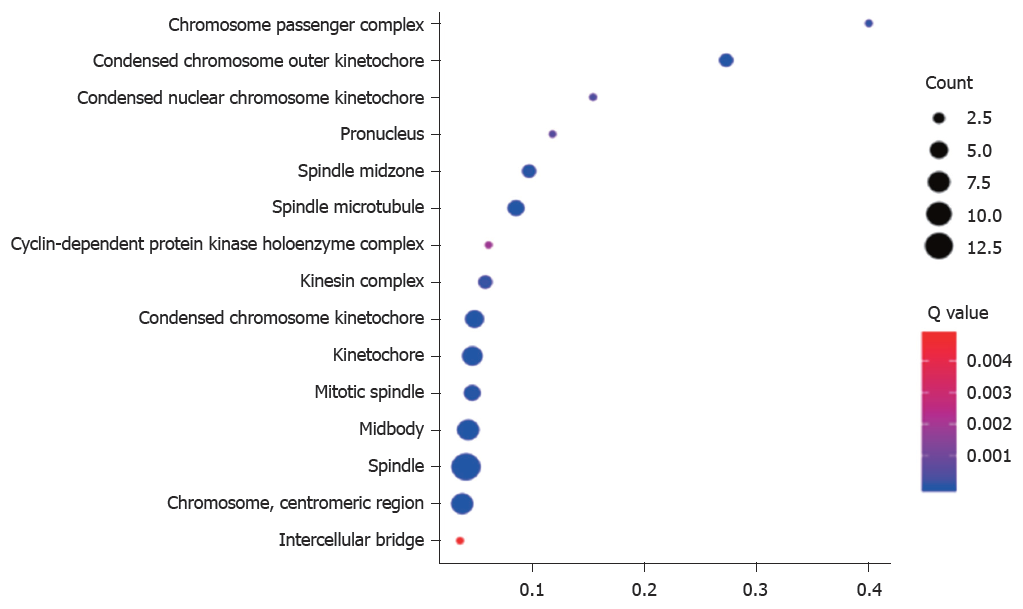
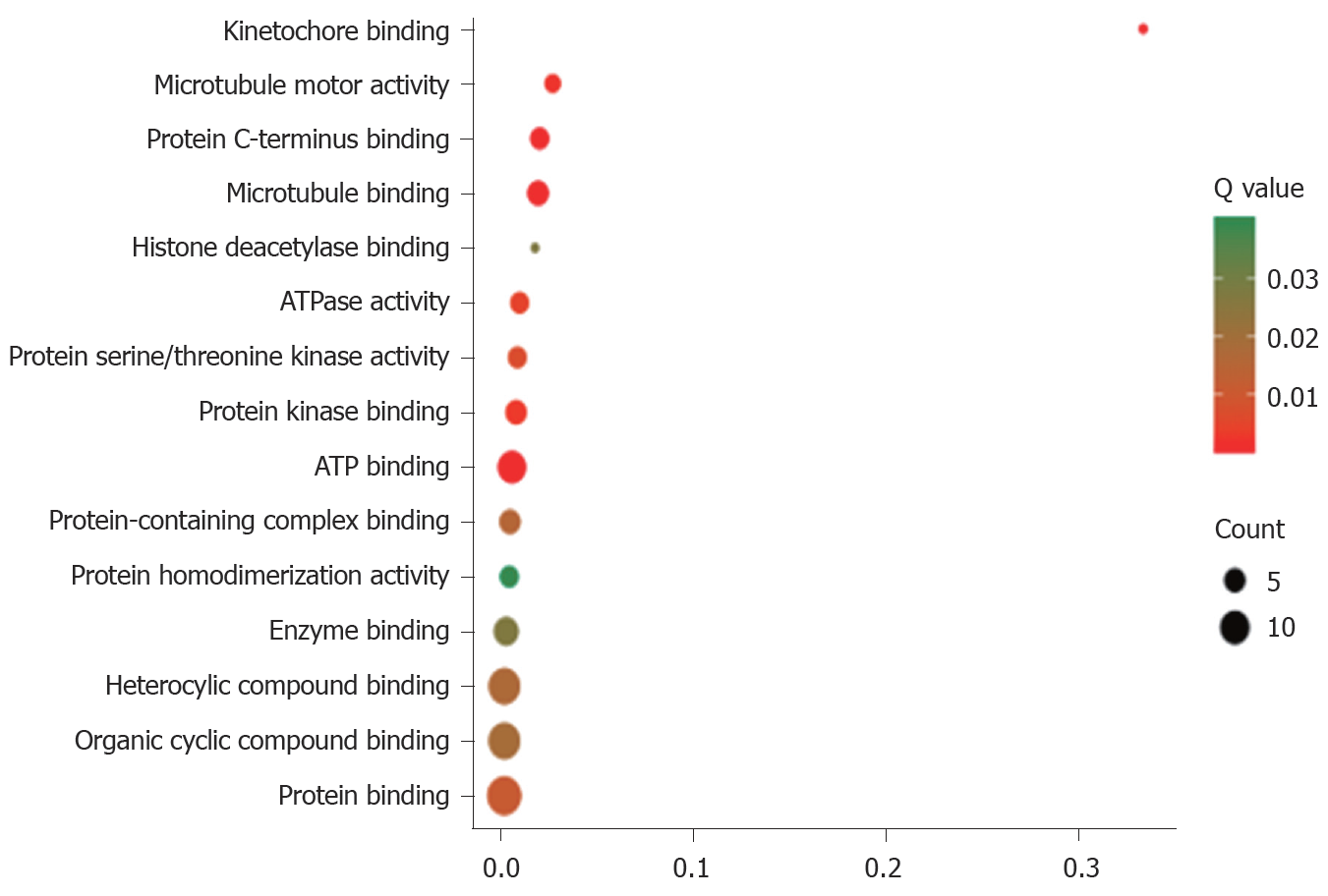
PPP1R105及其相关基生物学功能、细胞成分和分子功能富集见表2. 因生物学过程主要富集于有丝分裂染色体向纺锤体极运动、减数分裂姐妹染色单体结合, 着丝粒和有丝分裂纺锤体装配检查点, 图3. 细胞成分主要富集于染色体复合体、纺锤中间区和纺锤体微管等, 图4. 分子功能主要富集于微管运动活性、蛋白C末端结合和组蛋白脱乙酰酶结合等, 图5.
| GO功能富集 | 基因数 | P值 | 基因比例 |
| 生物学过程 | |||
| 有丝分裂染色体向纺锤体极运动 | 2 | 0.00012 | 0.666667 |
| 减数分裂姐妹染色单体结合, 着丝粒 | 2 | 0.00018 | 0.5 |
| 有丝分裂纺锤体装配检查点 | 5 | 2.09E-09 | 0.238095 |
| 蛋白质定位 | 3 | 9.88E-06 | 0.214286 |
| 有丝分裂中后期转换的调控 | 7 | 7.25E-12 | 0.142857 |
| 减数分裂细胞周期的调控 | 4 | 3.00E-06 | 0.088889 |
| 后期促进复杂依赖性分解代谢过程 | 3 | 0.0001 | 0.085714 |
| 染色体分离的调控 | 8 | 6.50E-12 | 0.082474 |
| 染色体分离 | 2 | 0.0029 | 0.08 |
| 姐妹染色单体粘连 | 3 | 0.00013 | 0.076923 |
| 有丝分裂纺锤体组装 | 3 | 0.00014 | 0.075 |
| 有丝分裂纺锤体组织 | 5 | 2.49E-07 | 0.071429 |
| 雌核减数分裂 | 2 | 0.0036 | 0.071429 |
| 细胞成分 | |||
| 染色体复合体 | 2 | 0.0001 | 0.4 |
| 纺锤中间区 | 3 | 3.28E-05 | 0.096774 |
| 纺锤体微管 | 4 | 1.90E-06 | 0.085106 |
| 细胞周期蛋白依赖性蛋白激酶全酶复合物 | 2 | 0.0019 | 0.060606 |
| 运动蛋白复合物 | 3 | 0.00011 | 0.057692 |
| 凝聚染色体动粒 | 5 | 9.02E-07 | 0.048077 |
| 动粒 | 6 | 6.22E-08 | 0.046154 |
| 有丝分裂纺锤体 | 4 | 1.46E-05 | 0.045977 |
| 中身 | 7 | 9.80E-09 | 0.042424 |
| 主轴 | 13 | 2.09E-16 | 0.040373 |
| 染色体, 着丝粒区 | 7 | 1.65E-08 | 0.037037 |
| 细胞间桥 | 2 | 0.0048 | 0.035088 |
| 分子功能 | |||
| 微管运动活性 | 3 | 0.0019 | 0.027273 |
| 蛋白C末端结合 | 4 | 0.00084 | 0.020619 |
| 微管结合 | 5 | 0.00084 | 0.019763 |
| 组蛋白脱乙酰酶结合 | 2 | 0.0271 | 0.018182 |
| atp酶活性 | 4 | 0.0046 | 0.010204 |
| 蛋白丝氨酸/苏氨酸激酶活性 | 4 | 0.0067 | 0.009009 |
| 蛋白激酶结合 | 5 | 0.0028 | 0.008347 |
| ATP结合 | 9 | 0.00084 | 0.006156 |
| 含蛋白质复合物结合 | 5 | 0.0155 | 0.005165 |
| 蛋白质均聚活性 | 4 | 0.0397 | 0.004819 |
| 酶结合 | 7 | 0.0271 | 0.003186 |
| 杂环化合物结合 | 12 | 0.0171 | 0.002262 |
| 有机环状化合物结合 | 12 | 0.0188 | 0.00223 |
| 蛋白质结合 | 14 | 0.0112 | 0.00212 |
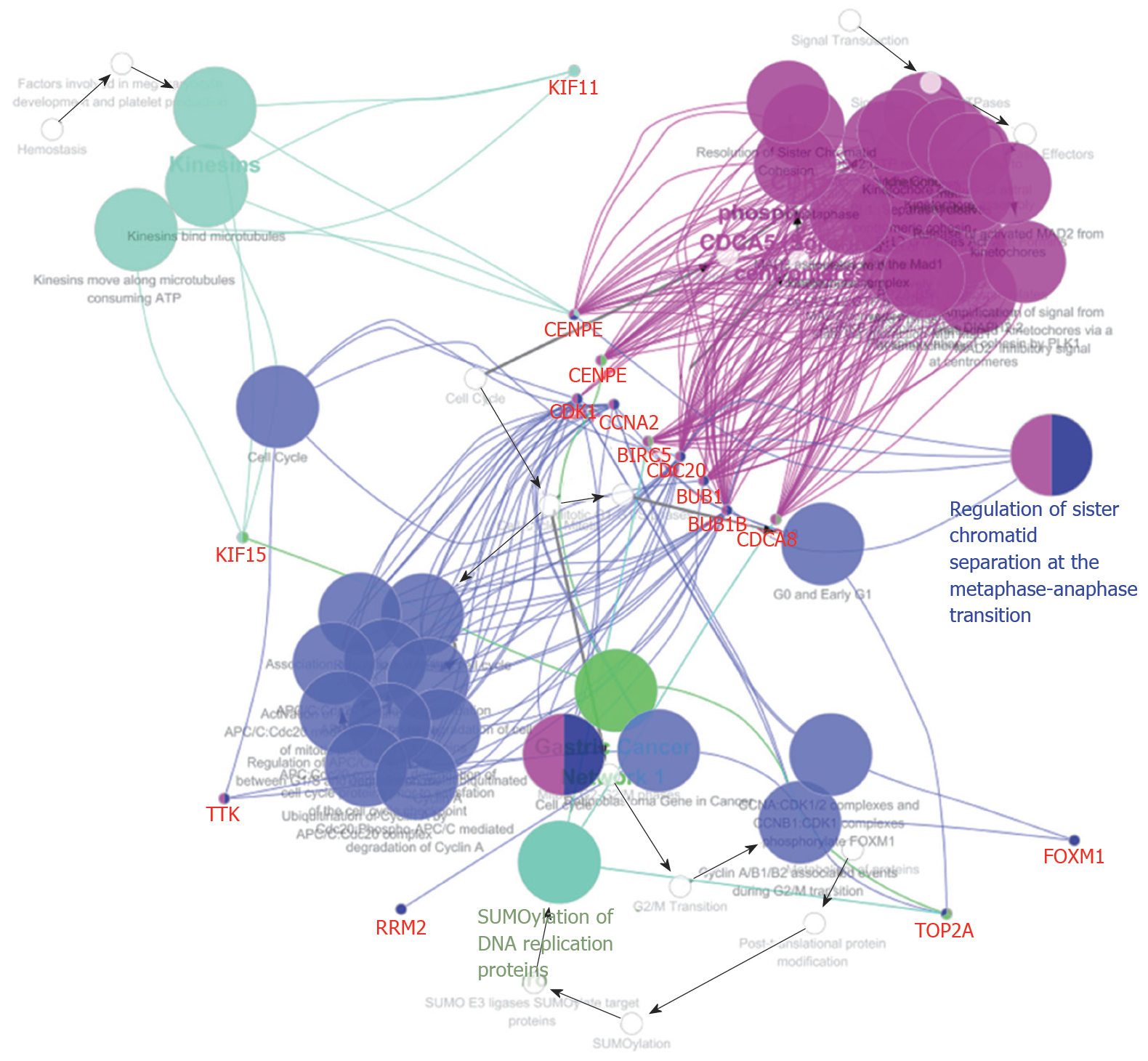
PPP1R105及其相关基因KEGG信号通路富集见表3. PPP1R105及其相关基因信号通路主要富集于细胞周期、细胞衰老、病毒致癌及p53信号通路, 图6.
| 信号通路 | 基因数 | 背景基因 | P值 | 基因比例 |
| 细胞周期 | 6 | 123 | 7.26E-08 | 0.04878 |
| 孕酮介导的卵母细胞成熟 | 3 | 94 | 0.0015 | 0.031915 |
| 卵母细胞减数分裂 | 3 | 116 | 0.0018 | 0.025862 |
| 细胞衰老 | 3 | 156 | 0.0031 | 0.019231 |
| 病毒致癌 | 3 | 183 | 0.004 | 0.016393 |
| p53信号通路 | 2 | 68 | 0.0084 | 0.029412 |
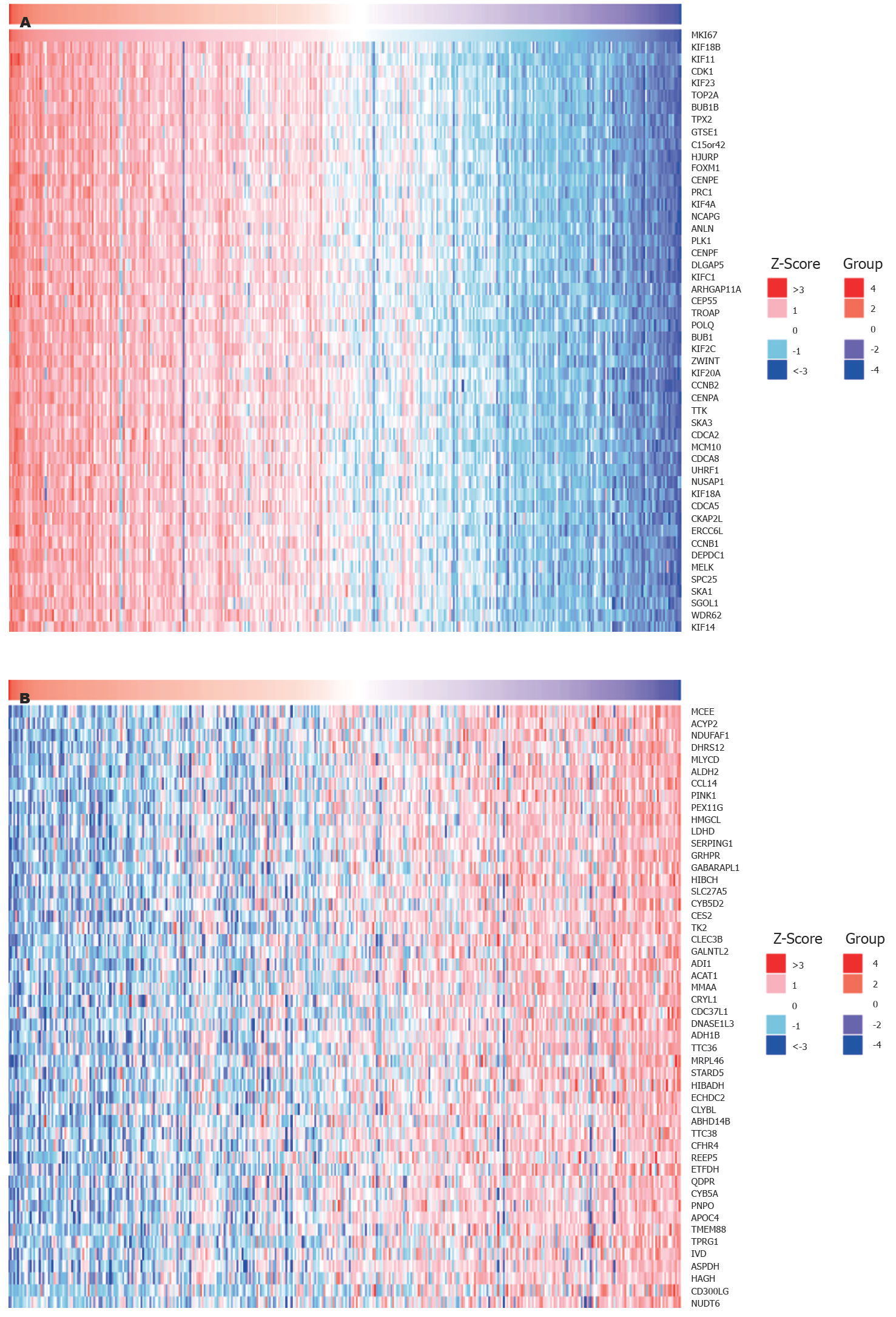
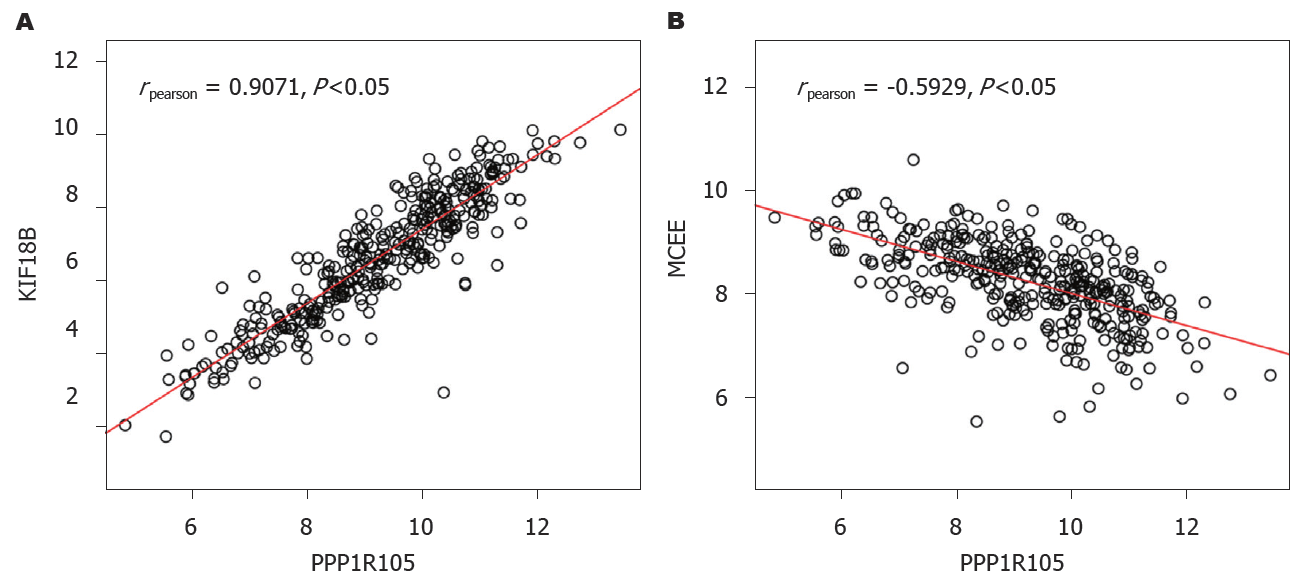
对与PPP1R105共表达基进行分析, 并采用聚类热图(图7)进行表示. 同时发现KIF18B与PPP1R105正相关表达最为显著(rpearson = 0.91, P<0.05); 而MCELL基因与PPP1R105负相关表达最为显著(rpearson = -0.59, P<0.05), 图8.
TCGA数据库中, 根据PPP1R105 mRNA 在肝细胞癌组织中表达的中位数将患者分为PPP1R105高表达(>PPP1R105 mRNA中位数)和低表达(≤PPP1R105 mRNA中位数)组. PPP1R105高表达组DFS (HR = 1.90, P<0.01)和OS (HR = 1.90, P<0.01)显著低于低表达组, 图9.
免疫组织化学染色显示, PPP1R105蛋白主要表达于细胞质及细胞核, 阳性表达呈现黄棕色颗粒状, 均匀分布于细胞核内. 81例患者中高表达者30例(37.0%)低表达者51 (63.0%)例, 图10.
PPP1R105蛋白高表达患者肿瘤大于5 cm及TNM分期Ⅲa期患者比例显著高于MIK67蛋白低表达者, 且差异有统计学意义(P<0.05), 表4.
| 分组特征 | n = 81 | PPP1R105蛋白 | χ2 | P值 | |
| 高表达(n = 30) | 低表达(n = 51) | ||||
| 性别[n, (%)] | 0.24 | 0.63 | |||
| 男性 | 72 | 26 | 46 | ||
| 女性 | 9 | 4 | 5 | ||
| 年龄(yr) | 0.81 | 0.37 | |||
| ≤60 | 53 | 20 | 23 | ||
| >60 | 28 | 10 | 18 | ||
| 肿瘤大小(cm) | 5.18 | 0.02 | |||
| ≤5 | 29 | 6 | 23 | ||
| >5 | 52 | 24 | 28 | ||
| 组织分化程度 | 2.86 | 0.09 | |||
| 高中分化 | 60 | 19 | 41 | ||
| 低分化 | 21 | 11 | 10 | ||
| AFP(μg/L) | 1.89 | 0.17 | |||
| ≤400 | 46 | 20 | 26 | ||
| >400 | 35 | 10 | 25 | ||
| TNM分期 | |||||
| Ⅰ/Ⅱ | 56 | 16 | 40 | 5.58 | 0.02 |
| Ⅲa | 25 | 14 | 11 | ||
| 位置 | 2.72 | 0.10 | |||
| 左侧 | 19 | 4 | 15 | ||
| 右侧 | 62 | 26 | 36 | ||
PPP1R105基因定位于人10号染色体, 编码一种核蛋白, 该蛋白与细胞增殖相关, 可能是细胞增殖所必需的蛋白因子. 在细胞分裂增殖的过程中, 除G0期外PPP1R105蛋白在其他细胞周期各个阶段均有不同程度的表达, 因此PPP1R105蛋白被认为是反映细胞增殖活性的标志物.
在本研究中, 我们发现PPP1R105基因mRNA在不同肿瘤中表达水平差异并不显著, 但在大多数肿瘤组织中均呈现较高水平的表达, 这与肿瘤细胞增殖活跃有关. 在肝细胞癌中, 癌组织中PPP1R105 mRNA表达水平显著高于癌旁正常肝组织, 且表达水平与患者临床分期有关. 提示肝癌中, PPP1R105 mRNA呈现上调, 且分期越晚, 表达水平越高. 同时信号通路富集也证实PPP1R105信号通路富集于细胞周期. 从上述多个方面证实PPP1R105基因编码蛋白主要生物学功能与细胞分裂增殖有关. 进一步免疫组化显示, PPP1R105高表达患者肿瘤较大、TNM分期较晚. 这些特征均为肝细胞癌预后不良的危险因素. 生存分析也同时证实, PPP1R105高表达患者总生存期和无疾病进展生存期均低于低表达者, 提示PPP1R105是肝细胞癌预后不良的独立危险因素.
尽管PPP1R105的表达与细胞增殖密切相关, 但在分子功能研究中, PPP1R105在细胞周期不同阶段的生物学功能也不尽相同. 在早期研究中, 注射抗PPP1R105抗体可抑制小鼠3T3细胞的增殖[13]. 同时, 小干扰RNA下调IM-9多发性髓鞘瘤和RT-4膀胱癌细胞系中PPP1R105基因表达后, 细胞的增殖能力明显降低[14,15]. 然而, 新近的研究表明, PPP1R105缺失对人类细胞周期进程不同阶段影响不同, 其功能与G1/S检查点的状态有关. 人成纤维细胞(WI-38、IMR90和HFF)以及非肿瘤来源的二倍体细胞(hTERT-RPE1和hTERT-BJ)在PPP1R105缺失时能够诱导细胞产生周期蛋白依赖性激酶抑制剂检查点蛋白p21[16]. 因此, 这些细胞被称为"PPP1R105敏感细胞"与"PPP1R105敏感"细胞相比, 肿瘤细胞系(HeLa、U2OS和293T细胞)在PPP1R105缺失时不诱导p21或显示S期改变[17]. 因此认为PPP1R105蛋白的生物学功能是细胞周期依赖的.
本研究对PPP1R105在肿瘤尤其是肝细胞癌中的表达情况及生物学功能进行生物信息学分析, 证实PPP1R105主要与细胞周期有关. 同时证实, PPP1R105是肝细胞癌预后不良的危险因素. 同时, 随着大数据和人工智能在医学尤其是肿瘤领域的应用, 大数据深入挖掘有望为恶性肿瘤的诊断和治疗提供新的思路和方法.
PPP1R105基因mRNA在多种恶性肿瘤中表达水平上调, PPP1R105基因mRNA在肝细胞癌(hepatocellular carcinoma, HCC)中表达、生物学功能及其表达水平与患者预后关系研究鲜有报道.
通过本研究进一步明确PPP1R10基因mRNA在HCC中的表达水平、相关生物学功能、信号通路及其作为HCC预后分子标志物的可行性.
评价PPP1R10基因mRNA在HCC中的表达及其临床意义.
对比PPP1R105基因在HCC及其他各个肿瘤组织中的相对表达情况. 构建PPP1R105基因编码蛋白相互作用网络. 比较PPP1R105高低表达组患者无疾病进展生存(disease free survival, DFS)和总生存(overall survival, OS)是否存在差异. 采用免疫组织化学法检测肝细胞癌组织中PPP1R105蛋白表达情况, 并与患者的临床特征进行相关性分析.
PPP1R105基因mRNA在HCC癌组织达水平显著上调, 并与患者的DFS和OS降低有关. PPP1R105及其相关基因信号通路主要富集于细胞周期、细胞衰老、病毒致癌及p53信号通路. PPP1R105蛋白高表达患者肿瘤大于5 cm及TNM分期Ⅲa期患者比例显著高于MIK67蛋白低表达者.
肝细胞癌患者PPP1R105高表达, 其与患者总生存和无疾病进展生存较短有关, 可作为肝细胞癌患者预后不良分子标志物.
PPP1R105可能是HCC预后不良发独立危险因素, 进一步对PPP1R105相关信号通路进行研究, 为开发抑制PPP1R105表达的HCC靶向治疗药物提供了新的靶点.
学科分类: 胃肠病学和肝病学
手稿来源地: 天津市
同行评议报告学术质量分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): C, C
D级 (一般): D, D
E级 (差): 0
科学编辑: 张晗 制作编辑:刘继红
| 2. | Siegel RL, Miller KD, Jemal A. Cancer statistics, 2019. CA Cancer J Clin. 2019;69:7-34. [PubMed] [DOI] |
| 3. | Mouchli MA, Singh S, Loftus EV, Boardman L, Talwalkar J, Rosen CB, Heimbach JK, Wiesner RH, Hasan B, Poterucha JJ, Kymberly WD. Risk Factors and Outcomes of De Novo Cancers (Excluding Nonmelanoma Skin Cancer) After Liver Transplantation for Primary Sclerosing Cholangitis. Transplantation. 2017;101:1859-1866. [PubMed] [DOI] |
| 4. | Lee S, Jung Y, Bae Y, Yun SP, Kim S, Jo H, Seo HI. Prevalence and risk factors of nonalcoholic fatty liver disease in breast cancer patients. Tumori. 2017;103:187-192. [PubMed] [DOI] |
| 5. | Kamath GR, Taioli E, N Egorova N, Llovet JM, Perumalswami PV, Weiss JJ, Schwartz M, Ewala S, Bickell NA. Liver Cancer Disparities in New York City: A Neighborhood View of Risk and Harm Reduction Factors. Front Oncol. 2018;8:220. [PubMed] [DOI] |
| 8. | Mohamed WS, Omar MM, Khayri TM, Fakhr IM. Assessment of the Proliferative Marker Ki-67 and p53 Protein Expression in HBV- and HCV-related Hepatocellular Carcinoma Cases in Egypt. Int J Health Sci (Qassim). 2008;2:27-34. [PubMed] [DOI] |
| 9. | Su AI, Wiltshire T, Batalov S, Lapp H, Ching KA, Block D, Zhang J, Soden R, Hayakawa M, Kreiman G, Cooke MP, Walker JR, Hogenesch JB. A gene atlas of the mouse and human protein-encoding transcriptomes. Proc Natl Acad Sci USA. 2004;101:6062-6067. [PubMed] [DOI] |
| 10. | Tang Z, Li C, Kang B, Gao G, Li C, Zhang Z. GEPIA: a web server for cancer and normal gene expression profiling and interactive analyses. Nucleic Acids Res. 2017;45:W98-W102. [PubMed] [DOI] |
| 11. | Szklarczyk D, Gable AL, Lyon D, Junge A, Wyder S, Huerta-Cepas J, Simonovic M, Doncheva NT, Morris JH, Bork P, Jensen LJ, Mering CV. STRING v11: protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets. Nucleic Acids Res. 2019;47:D607-D613. [PubMed] [DOI] |
| 12. | 彭 绍华, 杨 剑锋, 谢 平平, 邓 虹, 厉 浩, 冯 德云. 肝细胞肝癌组织中survivin和caspase 3、Ki67的表达及意义. 肿瘤. 2005;25:243-245, 253. [DOI] |
| 13. | Starborg M, Gell K, Brundell E, Höög C. The murine Ki-67 cell proliferation antigen accumulates in the nucleolar and heterochromatic regions of interphase cells and at the periphery of the mitotic chromosomes in a process essential for cell cycle progression. J Cell Sci. 1996;109:143-153. [PubMed] [DOI] |
| 14. | Schlüter C, Duchrow M, Wohlenberg C, Becker MH, Key G, Flad HD, Gerdes J. The cell proliferation-associated antigen of antibody Ki-67: a very large, ubiquitous nuclear protein with numerous repeated elements, representing a new kind of cell cycle-maintaining proteins. J Cell Biol. 1993;123:513-522. [PubMed] [DOI] |
| 15. | Kausch I, Lingnau A, Endl E, Sellmann K, Deinert I, Ratliff TL, Jocham D, Sczakiel G, Gerdes J, Böhle A. Antisense treatment against Ki-67 mRNA inhibits proliferation and tumor growth in vitro and in vivo. Int J Cancer. 2003;105:710-716. [PubMed] [DOI] |
| 16. | Fischer M, Grossmann P, Padi M, DeCaprio JA. Integration of TP53, DREAM, MMB-FOXM1 and RB-E2F target gene analyses identifies cell cycle gene regulatory networks. Nucleic Acids Res. 2016;44:6070-6086. [PubMed] [DOI] |
| 17. | Sun X, Bizhanova A, Matheson TD, Yu J, Zhu LJ, Kaufman PD. Ki-67 Contributes to Normal Cell Cycle Progression and Inactive X Heterochromatin in p21 Checkpoint-Proficient Human Cells. Mol Cell Biol. 2017;37:e00569-16. [PubMed] [DOI] |