修回日期: 2016-07-15
接受日期: 2016-07-31
在线出版日期: 2016-09-18
区分D基因型重组类型, 了解青海地区乙型肝炎病毒(hepatitis B virus, HBV)感染者C/D基因型重组情况.
收集217份青海地区慢性乙型肝炎感染者样品, PCR扩增535-1460 nt和1779-2400 nt 2个片段的乙型肝炎病毒基因序列, 分2区(593-799 nt、799-1450 nt和1799-2400 nt)构建进化树, 确定青海地区HBV基因重组类型. 利用 INNO-LiPA HBV分型试剂检测青海地区的C/D基因型重组样品, 计算符合率.
青海地区利用HBV不同区域进化树分型, 结果显示各基因型比例为CD1(61.9%)、CD2(8.4%)、C(27.0%)和B(2.8%). 基因型分布在藏族和汉族乙型肝炎感染者中的差异具有显著的统计学意义(χ2 = 17.9, P<0.01), 藏族乙型肝炎感染者中C/D基因型重组和C基因型比例为91.5.0%和8.5%; 在汉族中的比例为68.5%和32.3%. C/D基因型重组和C基因型HBV e抗原阴阳性的差异(χ2 = 0.28, P>0.05)以及病毒载量的差异(t = 1.125, P>0.05)均没有统计学意义. C/D重组样品INNO-LiPA分型试剂检测结果为D基因型的理论符合率为87.8%.
青海地区乙型肝炎感染者以CD1(10-799 nt)基因型重组以主, 对其临床意义应进一步的研究分析. 该地区的HBV分型检测应注意基因重组对分型试剂的影响.
核心提要: 青海地区乙型肝炎病毒(hepatitis B virus, HBV)基因型以CD基因型重组为主, 且存在两种重组类型, 在该地区藏族和汉族HBV感染者中的分布也具有显著的差异.
引文著录: 黄维金, 辜文洁, 王佑春. 青海地区乙型肝炎感染者C/D基因型重组分布. 世界华人消化杂志 2016; 24(26): 3779-3789
Revised: July 15, 2016
Accepted: July 31, 2016
Published online: September 18, 2016
To distinguish two forms of hepatitis B virus (HBV) C/D recombinant and investigate the distribution of HBV C/D recombinant genotypes in Qinghai Province.
A total of 217 chronic HBV infected serum samples were collected from Qinghai Province. Two fragments of HBV, 535-1460 nt and 1779-2400 nt, were amplified and sequenced. The two forms of C/D recombinant were confirmed by the phylogenetic tree constructed based on 593-799 nt, 799-1450 nt and 1799-2400 nt. The samples of C/D recombinant were determined by INNO-LiPA HBV genotyping assay.
The distribution of HBV genotypes in Qinghai Province detected by the phylogenetic tree based on different fragments was as follows: CD1 (61.9%); CD2 (8.4%); C (27.0%) and B (2.8%). Ethnically, the C/D recombinant had a higher prevalence in Tibetan patients (91.5%) than in Han populations (62.6%). The distribution of HBV genotypes in Tibetan patients was significantly different from that in Han populations (χ2 = 17.9, P < 0.01). Clinically, there was no significant difference in viral load between Tibetan and Han populations (F = 0.68, P > 0.05), and between males and females. There was no significant difference in HBeAg positive/negative ratios or HBV viral load between the C/D recombinant genotype and genotype C (χ2 = 0.28, P > 0.05; t = 1.125, P > 0.05). The majority (87.8%) of samples of C/D recombinant were reported to be genotype D by INNO-LiPA HBV genotyping assay.
The CD1 (10-799 nt) recombinant of HBV is the dominant genotype in Qinghai Province. The clinical outcomes of different C/D recombinant genotypes should be further studied, and the results of INNO-LiPA HBV genotyping assay should be reinterpreted in Qinghai Province.
- Citation: Huang WJ, Gu WJ, Wang YC. Distribution of hepatitis B virus C/D recombinant genotypes in Qinghai Province. Shijie Huaren Xiaohua Zazhi 2016; 24(26): 3779-3789
- URL: https://www.wjgnet.com/1009-3079/full/v24/i26/3779.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v24.i26.3779
我国是乙型肝炎病毒(hepatitis B virus, HBV)感染的高流行区, 1992年的调查显示我国1-59岁人群的乙型肝炎表面抗原(hepatitis B surface antigen, HbsAg)携带率为9.8%, 据此推算中国约有1.2亿人携带HBsAg. 随着乙型肝炎疫苗纳入儿童计划免疫管理, 2006年的研究显示我国1-4岁儿童的HBsAg流行率已降到1.0%[1,2], 已感染人群中至少15%-25%最终死于HBV 相关的肝病, 大部分为肝细胞癌[3]. 乙型肝炎的预后和抗病毒治疗效果与基因型有关[4-6]. HBV根据核苷酸的同源性划分为A-I基因型[7,8], 我国已发现的基因型有A、B、C、D和I. 我国北部(长江以北)以C基因型为主, 南部以B基因型为主, D基因型主要分布在新疆、西藏, I基因型分布在我国广西[7], 基因型分布具有地区和民族特点[9,10]. 青海地区的 D 基因型为C/D基因型重组[9,11], 被区分为CD1和CD2两种类型[9], CD1的10-799 nt为D基因型, 其他部分基因序列为C基因型; CD2的10-1499为D基因型, 其他部分基因序列为C基因型. HBV基因型检测试剂不能区分重组情况[12], 本研究跨C/D基因型的重组位点扩增HBV基因序列, 分区构建进化树鉴别C/D基因重组类型, 对青海地区的HBV基因型进行了分析.
2008-03采集自青海省传染病医院的乙型肝炎感染者血清217份, 样品长期保存在零下20 ℃, 其血清背景资料如表1.
| 民族 | n | 男 | 女 | 年龄 |
| 藏族 | 59 | 39 | 19 | 34(6-68) |
| 汉族 | 149 | 101 | 47 | 30(2-80) |
| 回族 | 8 | 2 | 6 | 32(9-44) |
| 蒙族 | 1 | 0 | 1 | 23 |
| 合计 | 217 | 142 | 73 | 31(2-80) |
1.2.1 乙型肝炎标志物检测: 乙型肝炎的血清学标志物利用北京万泰生物药业股份有限公司生产的乙型肝炎五项ELISA诊断试剂, 按照各试剂的说明书操作. HBV核酸定量检测试剂采用深中山大学达安基因股份有限公司(达安基因)生产的乙型肝炎荧光定量PCR检测试剂盒检测, 操作均按照说明书进行.
1.2.2 HBV C/D基因型重组鉴别: 采用全血DNA试剂盒提取病毒DNA, 按照试剂说明书操作. C/D基因型重组鉴别的PCR片段扩增: 采用HU(5'TGCTCAAGGAACCTCTATG3')和HL(5'GGGTCGTCCGVGGGATTCA3')引物, 扩增包括重组位点两侧的序列(535-1460 nt), 重组位点两侧构建进化树分型, 鉴别两种重组类型CD1(10-799 nt)或 CD2(10-1499 nt); 扩增1779-2400 nt的前C区, 扩增引物为PC1(5'GGCTGTAGGCATAAATTGGT3')和PC2(5'CTGCGAGGCGAGGGAGTTCTTCTTC3'), 鉴别C/D基因型重组和纯D基因型[13]. PCR采用的Taq酶为HotStar Taq DNA Polymerase.
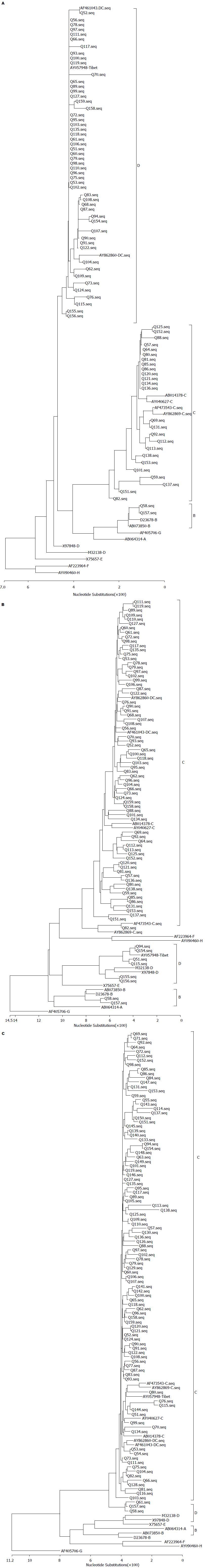
1.2.3 序列分析划分基因型: 测序结果, 利用DNAStar软件包的MegAlign进行分析, 构建进化树划分基因型. 根据青海地区C/D基因型重组存在CD1(10-799 nt)和CD2(10-1499 nt)两种类型, 如593-799 nt、799-1450 nt和1799-2400 nt均为C基因型, 则该序列为C基因型; 如果593-799 nt为D基因型, 799-1450 nt和1799-2400 nt为C基因型则为CD1; 如果593-799 nt和799-1450 nt均为D基因型, 1799-2400 nt为C基因型,则为CD2; 如果593-799 nt, 799-1450 nt和1799-2400 nt均为D基因型, 则为未重组D基因型.
1.2.4 INNO-LiPA HBV Genotyping试剂基因分型: 对检测为C/D重组的样品, 利用INNO-LiPA HBV Genotyping作为对比试剂进行检测. INNO-LiPA HBV Genotyping试剂由比利时INNOGENETICS公司生产, 按试剂盒说明书进行操作, 扩增P基因B和C区的巢式PCR产物, 经反相膜杂交, 检测其基因型.
统计学处理 t检验、方差分析采用Statistic软件计算, 其他描述性统计数据利用Microsoft Office Excel或Graphpad prism5.0软件计算.
由于部分血清量不足, 完成了125份样品的5项乙型肝炎血清学指标的检测. 将血清学指标分为Ⅰ-Ⅳ4种类型:Ⅰ(HBsAg+、HBeAg-、HBeAb-、HBcAb+)、Ⅱ(HBsAg+、HBeAg-、HBeAb+、HBcAb+)、Ⅲ(HBsAg+、HBeAg+、HBeAb-、HBcAb+)和Ⅳ(HBsAg+、HBeAg+、HBeAb-、HBcAb-). 采用达安基因的HBV核酸定量试剂检测 HBV DNA(表2).
| 血清学模式 | n | 男-女 | 年龄 | 病毒载量 |
| Ⅰ | 11 | 9-2 | 35 | 1.57×104 |
| Ⅱ | 22 | 14-8 | 30 | 2.21×104 |
| Ⅲ | 88 | 70-17 | 31 | 5.29×106 |
| Ⅳ | 4 | 2-2 | 38 | 3.79×108 |
2.2.1 分区构建进化树划分基因型的结果: 采用跨过重组区域的引物扩增HBV基因, 扩增PCR产物直接测序, 序列测定后, 分别构建593-799 nt、799-1450 nt和1799-2400 nt不同的区域的进化树, 划分基因型. 基因进化树分型结果如图1. 青海地区的基因分布为CD1(61.9%), CD2(8.4%), C(27.0%)和B(2.8%). 2份样品PCR扩增阴性, 未能分型. 青海地区的HBV基因型主要以C/D重组基因型(70.2%)和C基因型为主, 两者合计约占97.2%. CD1和CD2占C/D重组株的比例分别为 88%和12%. 青海地区的B基因型均为Ba亚型. 如图1所示Q58和Q157在前C区(1799-2400 nt)基因进化树分型为C基因型, 在其他区域为B基因型, 归为Ba亚型.
2.2.2 INNO-LiPA基因型检测C/D重组株的结果: C/D重组株采用 INNO-LiPA基因型试剂检测, 按照INNO-LiPA试剂的检测区域, 理论上应为D基因型. 实际检测结果87.8%为D基因型, 12.2%为CD基因型, 2.8%为其他基因型.
基因型分布结果表现出了民族间的显著差异, 藏族中的C/D重组基因型和C基因型比例为91.5%和8.5%; 而汉族中的C/D重组基因型和C基因型比例分别为62.6%和32.3%, B基因型占4.1%; 回族中的C/D重组基因型和C基因型的比例分别为62.5%和37.5%. 不同民族之间基因型分布差异具有显著的统计学意义(χ2 = 17.9, P<0.01).
在藏族和汉族之间病毒载量的几何均数差异无显著的统计学意义(F = 0.68, P>0.05), 藏族男女之间和汉族男女的病毒载载量差异均无显著的统计学意义(t = 3.55, P>0.05; t = 0.549, P>0.05). C/D重组基因型感染者病毒载量低于C基因型感染者, 但差异无显著统计意义(t = 1.125, P = 0.25). Ⅰ和Ⅱ血清学模式为e抗原阴性, Ⅲ和Ⅳ血清学模式为e抗原阳性, 按照e抗原阴阳性分组, C基因型和C/D重组基因型之间的e抗原差异没有显著的统计学意义(χ2 = 0.28, P>0.05)(表3).
| 基因型 | 血清学模式 | 病毒载量(IU/mL) | |
| Ⅰ+Ⅱ | Ⅲ+Ⅳ | ||
| C | 5 | 18 | 1.85×106 |
| C/D重组 | 26 | 70 | 1.13×106 |
关于青藏高原地区HBV基因型分布和C/D重组亚型的分布有不同的报告数据. 如果不区分C/D重组类型, 该地区的C/D重组株可被报告为D基因型. Zhou等[9]研究发现青藏高原地区的乙型肝炎流行株为C/D基因型重组株, 占该地区乙型肝炎感染者的51.9%, 结果和曹占良等[14]研究报告拉萨地区乙型肝炎感染者中D基因型占53.4%的比例类似, 后者未区分重组类型; 另外Zhou等[9]报告青海地区C/D重组基因型比例为68.5%, 与本研究发现青海地区C/D重组基因型比例为70.2%的结果类似. Chen等[15]研究结果显示西宁地区的D基因型比例为34/56(60.7%), Chen等[15]的研究也未区分重组类型. 以上研究均表明青藏高原地区以D基因型或C/D重组基因型为主, 但是这一结果与Zhang等[16]的研究数据存在较大的差距, 后者报告该地区以C基因型为主, 占53.0%.
另外, 在C/D重组亚型分布方面也存在结果差异, Cui等[17]首先在2002年报告了西藏地区的D基因型主要为一种新的C/D基因型重组类型, 即CD2重组株. 我们于2003年在青海藏民中分离出另外一种C/D基因型重组类型[11], 即CD1重组株. 后来Zhou等[9]研究发现CD1和CD2两种重组类型是青藏高原地区乙型肝炎感染者的主要基因型. 本研究结果也表明我国青海地区HBV感染者以CD1和CD2两种重组基因型为主, 占70.2%, 重组类型中又以CD1为主, 占C/D重组基因型的88%, 略低于Zhou等[9]报告的92%, 也略低于我国其他研究者[18]报告的青海同德地区C/D重组株比例为96.3%, 后者未区分重组类型. 以上结果均与Zhang等[16]的研究结果存在较大差异, Zhang等[16]研究发现C/D重组情况是该地区的少数情况, 占比为6.2%, 且为CD2重组类型. 分析不同研究分型结果差异的原因, 可能和样品选择及分型方法的差异有关. 本研究选择来自青海省传染病医院的样品, 具有青海地区的代表性, 对CD1和CD2重组类型采用HBV分区基因序列分析法进行鉴别, 考虑了HBV重组区域附近的整体基因序列情况, Zhou等[9]采用的方法为PCR-RFLP法, 具有简单快速的特点, 由于考虑了重组基因型之间的个别保守位点, 对亚型检测有较好的区分能力, 但是不能准确区分特殊基因型[19]. 相比之下, Zhang[16]的结果和大多研究结果不太一致, 很可能是由于其扩增HBV的RT部分序列, 不能区分C/D基因型重组情况. 本研究的方法还可鉴别检测我国的B/C基因型重组情况, 结果显示青海地区B基因型比例为2.4%, 符合我国主要流行株特点, 在前核心区发生了和C基因型的重组, 为B基因型的Ba重组株[20]. 对本研究检测C/D重组类型的样品, 采用了INNO-LiPA HBV分型试剂进行对比验证, INNO-LiPA HBV分型试剂是一种准确快速的分型试剂[12,21], 已在我国获批上市, 用于临床乙型肝炎基因型检测. 但INNO-LiPA分型试剂不能区分C/D基因型重组的情况, C/D基因型重组的样品理论上检测均应为D基因型. 有报告INNO-LiPA HBV分型试剂结果和测序结果的符合率为81%[12]和82%[21], 本研究对C/D重组株的检测结果与理论基因型D的检出符合率为87.8%, 表明该研究采用的分区构建进化树的方法准确性较好, 而且可准确区分CD重组情况和具体的重组类型. 同样, 传统采用S基因划分基因型的方法也不能区别CD1和CD2两种重组类型, 对于青藏高原地区HBV基因型研究可以采用本研究方法或采用全因序列分析的方法更为准确[12]. 采用全基因序列分析的方法还可全面了解HBV基因序列特点[22], 发现新的基因型重组情况[2,11,23,24], 了解HBV的基因进化趋势[25].
很多研究[4-6,26-29]表明, 不同HBV基因型感染后的预后和抗病毒治疗效果不同: 在急性感染慢性化的比例中, A、D基因型高于B、C基因型; HBeAg自发阴转在C、D基因型感染者中的比例低于A、B基因型; 基本核心区(basic core promoter, BCP)A1762T/G1764A突变联合前S基因缺失和高病毒滴度的情况在C基因型感染者中的比例高于B基因型HBV感染者; 单纯BCPA1762T/G1764A突变率在D基因型比A基因型高; 在严重的终末期肝脏疾病, 如HBV相关的原发性肝细胞癌中C和D基因型比例高. HBV基因型与治疗相关性研究中也发现, 对干扰素治疗反应性有基因型之间的差异: A、B基因型好于C、D基因型; 核苷类似物抗病毒药物使用后, 发生聚合酶区变异的比例在B基因型亚组之间有不同, Ba组比Bj组出现的频率要高. 所以检测HBV基因型对乙型肝炎感染者的预后估计和用药指导具有一定的临床意义[4]. 本研究对青海地区的C/D重组病毒的临床特点初步分析发现, 乙型肝炎感染者中C/D基因重组与C基因型之间e抗原阴/阳性和HBV病毒载量差异没有显著的统计学意义, 这与西藏拉萨的研究结果一致[26], 可能与选择的样品为慢性感染者样品有关. 为做好该地区的HBV防治工作, 获得更多与C/D基因重组相关的临床进展和预后信息, 值得对该地区HBV引起的肝癌、肝硬化病例进行深入分析, 比较C/D基因型重组分布, 以及对干扰素等治疗的反应情况. 有报告C和D基因型的临床表现不同[5], 且C和D基因型的感染者较B基因型易发生肝病进展, D基因型临床肝功能酶指标高于B和C基因型[26], D基因型干扰素治疗效果差[30]. 由于本研究的样品为保存血样, 未进行肝功能检测和临床HBV感染分期, 也有报告重组株的病毒滴度低于非重组株[24], C/D基因型重组与C基因型HBV感染是否预后不同, 应进一步的积累数据, 深入分析.
青海省传染病医院(青海省第四人民医院)检验科王芝主任等, 负责收集样品和整理样品信息.
乙型肝炎病毒(hepatitis B virus, HBV)的基因型与乙型肝炎预后和治疗效果相关, HBV的基因型分布具有地区差异, 而HBV存在不同的重组株, 对HBV基因型分布和重组情况分析, 可以为该地区的HBV防控提供重要的数据.
龚作炯, 教授,武汉大学人民医院感染科; 王友亮, 副研究员, 中国人民解放军军事医学科学院生物工程研究所
常规的HBV基因型分型方法不能鉴别不同的HBV基因重组株, 在青海、西藏等重组株流行较高的地区, 更应该注意分型方法或试剂的研究与选择, 以准确分析该地区的基因型特征.
已有多个报道青藏高原地区存在HBV重组株, 但是由于检测方法的差异或者未区分重组类型, 导致对于重组株的比例分布等数据差异较大, 本文区分了不同的重组类型, 结果更具有代表性.
本文重点分析了青海的基因型重组情况, 所用方法可区分不同的重组类型, 为其他HBV 基因型重组研究提供了借鉴.
研究发现HBV基因重组会影响基因型检测结果的准确性, 在HBV分型方法的研究和分型试剂的评价中引起重视.
本文对HBV分子流行病学调查有重要的意义, 也为青海地区诊治和预防乙型肝炎提供理论依据.
手稿来源: 自由投稿
学科分类: 胃肠病学和肝病学
手稿来源地: 北京市
同行评议报告分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): C, C
D级 (一般): 0
E级 (差): 0
编辑: 于明茜 电编:李瑞芳
| 1. | Liang X, Bi S, Yang W, Wang L, Cui G, Cui F, Zhang Y, Liu J, Gong X, Chen Y. Reprint of: Epidemiological serosurvey of Hepatitis B in China--declining HBV prevalence due to Hepatitis B vaccination. Vaccine. 2013;31 Suppl 9:J21-J28. [PubMed] [DOI] |
| 2. | Cui Y, Jia J. Update on epidemiology of hepatitis B and C in China. J Gastroenterol Hepatol. 2013;28 Suppl 1:7-10. [PubMed] [DOI] |
| 4. | Lin CL, Kao JH. The clinical implications of hepatitis B virus genotype: Recent advances. J Gastroenterol Hepatol. 2011;26 Suppl 1:123-130. [PubMed] [DOI] |
| 7. | Li GJ, Hue S, Harrison TJ, Yang JY, Chen QY, Wang XY, Fang ZL. Hepatitis B virus candidate subgenotype I1 varies in distribution throughout Guangxi, China and may have originated in Long An county, Guangxi. J Med Virol. 2013;85:799-807. [PubMed] [DOI] |
| 8. | Okamoto H, Tsuda F, Sakugawa H, Sastrosoewignjo RI, Imai M, Miyakawa Y, Mayumi M. Typing hepatitis B virus by homology in nucleotide sequence: comparison of surface antigen subtypes. J Gen Virol. 1988;69:2575-2583. [PubMed] [DOI] |
| 9. | Zhou B, Xiao L, Wang Z, Chang ET, Chen J, Hou J. Geographical and ethnic distribution of the HBV C/D recombinant on the Qinghai-Tibet Plateau. PLoS One. 2011;6:e18708. [PubMed] [DOI] |
| 10. | Pourkarim MR, Amini-Bavil-Olyaee S, Kurbanov F, Van Ranst M, Tacke F. Molecular identification of hepatitis B virus genotypes/subgenotypes: revised classification hurdles and updated resolutions. World J Gastroenterol. 2014;20:7152-7168. [PubMed] [DOI] |
| 11. | 黄 维金, 张 华远, 王 佑春, 林 京香, 吴 星, 周 诚, 李 河民. 乙型肝炎病毒全基因克隆及序列分析发现-D/C基因型重组株. 中华微生物学和免疫学杂志. 2003;23:418-422. |
| 12. | Osiowy C, Giles E. Evaluation of the INNO-LiPA HBV genotyping assay for determination of hepatitis B virus genotype. J Clin Microbiol. 2003;41:5473-5477. [PubMed] [DOI] |
| 13. | Thakur V, Kazim SN, Guptan RC, Malhotra V, Sarin SK. Molecular epidemiology and transmission of hepatitis B virus in close family contacts of HBV-related chronic liver disease patients. J Med Virol. 2003;70:520-528. [PubMed] [DOI] |
| 14. | 曹 占良, 高 英堂, 刘 糟, 景 丽, 吉 宗, 刘 彤, 刘 持佳, 杜 智. 基因芯片技术检测西藏粉萨地区的乙型肝炎病毒基因型. 世界华人消化杂志. 2007;15:3500-3506. [DOI] |
| 15. | Chen X, Gao J, Ji Z, Zhang W, Zhang L, Xu R, Zhang J, Li F, Li S, Hu S. A description of the hepatitis B virus genomic background in a high-prevalence area in China. Virol J. 2014;11:101. [PubMed] [DOI] |
| 16. | Zhang Q, Liao Y, Chen J, Cai B, Su Z, Ying B, Lu X, Tao C, Wang L. Epidemiology study of HBV genotypes and antiviral drug resistance in multi-ethnic regions from Western China. Sci Rep. 2015;5:17413. [PubMed] [DOI] |
| 17. | Cui C, Shi J, Hui L, Xi H, Zhuoma G. The dominant hepatitis B virus genotype identified in Tibet is a C/D hybrid. J Gen Virol. 2002;83:2773-2777. [PubMed] [DOI] |
| 18. | 张 杰, 姜 双应, 王 学文, 王 锋, 边 涛, 牟 振云, 张 勇. 青海同德藏族人群感染HBV基因型、血清型调查. 中华实验和临床病毒学杂志. 2009;23:2-4. |
| 20. | Sugauchi F, Orito E, Ichida T, Kato H, Sakugawa H, Kakumu S, Ishida T, Chutaputti A, Lai CL, Ueda R. Hepatitis B virus of genotype B with or without recombination with genotype Cover the precore region plus the core gene. J Virol. 2002;76:5985-5992. [PubMed] [DOI] |
| 22. | 黄 维金, 周 诚, 王 佑春, 张 华远, 吴 星, 梁 争论, 李 河民. 乙型肝炎病毒中国流行株B、C、D/C及A基因型的全基因克隆与序列分析. 世界华人消化杂志. 2009;17:2978-2983. [DOI] |
| 23. | Wang Z, Liu Z, Zeng G, Wen S, Qi Y, Ma S, Naoumov NV, Hou J. A new intertype recombinant between genotypes C and D of hepatitis B virus identified in China. J Gen Virol. 2005;86:985-990. [PubMed] [DOI] |
| 24. | Liao H, Li X, Liu Y, Xu Z, Huang P, Nian X, Liu X, Xu D. Intergenotype recombinant analysis of full-length hepatitis B virus genomes from 516 Chinese patients with different illness categories. J Med Virol. 2016; Jun 22. [Epub ahead of print]. [PubMed] [DOI] |
| 25. | Araujo NM. Hepatitis B virus intergenotypic recombinants worldwide: An overview. Infect Genet Evol. 2015;36:500-510. [PubMed] [DOI] |
| 29. | Tong S, Revill P. Overview of hepatitis B viral replication and genetic variability. J Hepatol. 2016;64:S4-S16. [PubMed] [DOI] |
| 30. | Tian Q, Jia J. Hepatitis B virus genotypes: epidemiological and clinical relevance in Asia. Hepatol Int. 2016; Jun 14. [Epub ahead of print]. [PubMed] [DOI] |