修回日期: 2016-03-16
接受日期: 2016-03-29
在线出版日期: 2016-05-08
目的: 筛选胃癌患者与慢性胃炎患者及健康志愿者的唾液蛋白质组差异表达谱, 并建立胃癌患者与慢性胃炎唾液无创伤分子诊断(判别)模型.
方法: 采集57例胃癌患者(术前且无放化疗)和28例慢性胃炎患者的唾液标本, 用WCX(弱阳离子交换型)纳米磁珠联合基质辅助激光解析离子化飞行时间质谱(matrix assisted laser desorption ionization time-of-flight mass spectrometry, MALDI-TOF-MS)技术进行检测, 获得各标本的蛋白指纹图谱. 用Biomarker Wizard软件分析所获得的蛋白指纹图谱找出差异蛋白, 再用Biomarker Patterns 5.0.2建立鉴别诊断模型.
结果: 优化筛选建立了由m/z为4267.09、6564.85、2138.14等3个差异蛋白峰组成的胃癌与慢性胃炎的鉴别诊断模型, 经临床回代检验该分子诊断模型对鉴别胃癌与慢性胃炎的灵敏度和特异度分别为96%(55/57)和86%(24/28), 通过交叉验证法进一步验证诊断模型的可靠性, 结果该模型的灵敏度和特异度分别为89%(51/57)和75%(21/28).
结论: 基于WCX结合MALDI-TOF-MS技术建立的唾液蛋白组分子诊断模型为胃癌与慢性胃炎的鉴别诊断提供了一种敏感无创的新方法.
核心提示: 唾液进行疾病诊断具有无创、微量、易于收集和储存、操作简便快捷、易于被患者接受等明显优势. 本文研究结果显示唾液蛋白指纹图谱的检测对于鉴别胃癌和慢性胃炎具有较好的敏感性、准确性和特异性, 选用唾液标本研究胃癌与胃炎的鉴别诊断, 是探索胃癌无创伤分子诊断新方法.
引文著录: 谢梦洲, 贺佐梅, 黄飞娟, 周小青, 吴正治. 胃癌与慢性胃炎的唾液蛋白指纹图谱鉴别诊断模型. 世界华人消化杂志 2016; 24(13): 1976-1983
Revised: March 16, 2016
Accepted: March 29, 2016
Published online: May 8, 2016
AIM: To screen differential protein expression profiles in saliva between gastric carcinoma and chronic gastritis, and to establish a model for differentiating these groups.
METHODS: Saliva samples from 57 patients with gastric carcinoma and 28 patients with gastritis were purified with weak cationic-exchange magnetic beads (MB-WCX) and then analyzed by matrix-assisted laser desorption ionization time-of-flight mass spectrometry (MALDI-TOF-MS). Subsequently, we compared the saliva peptide signatures of the two groups and obtained differently expressed peptides by using Biomarker Wizard. Then, we established two diagnostic models to distinguish the gastric carcinoma group from the gastritis group and normal control group by using Biomarker Patterns 5.0.2.
RESULTS: A diagnostic pattern for discriminating between the gastric carcinoma group and gastritis group using three peptides (m/z 4267.09, 6564.85, 2138.14) was established with a 96% (55/57) sensitivity and an 86% (24/28) specificity. A sensitivity of 89%(51/57) and a specificity of 75% (21/28) were obtained in cross validation.
CONCLUSION: The novel molecular diagnostic model obtained by saliva protein fingerprinting for distinguishing gastric carcinoma from chronic gastritis is sensitive and noninvasive.
- Citation: Xie MZ, He ZM, Huang FJ, Zhou XQ, Wu ZZ. Comparative study of saliva peptide signatures between gastric carcinoma and chronic gastritis. Shijie Huaren Xiaohua Zazhi 2016; 24(13): 1976-1983
- URL: https://www.wjgnet.com/1009-3079/full/v24/i13/1976.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v24.i13.1976
早期胃癌患者大多无症状, 或者仅有一些非特异性消化道症状, 临床上与慢性胃炎常难以鉴别, 但预后却截然不同. 目前, 胃癌的确诊主要依赖于胃镜及病理活检, 虽然特异性高, 但患者耐受性低, 很难作为早期广泛筛查胃癌的手段[1]. 因此, 针对普通人群, 研究无创、便捷、准确的胃癌快速筛查技术对胃癌防治意义重大. 唾液中含有2300多种蛋白质和多肽[2], 且采集简便、无创, 无疑是无创分子诊断引人注目的领域. 本研究旨在基于唾液蛋白质组学技术探索建立鉴别胃癌(gastric carcinoma)与慢性胃炎(chronic gastritis)的无创性分子诊断模型.
本研究自2014-12/2015-04共收集符合诊断和纳入标准的胃癌患者57例, 全部病例来源于湖南省肿瘤医院胃十二指肠胰腺外科, 均经病理检查确诊. 患者年龄24-73岁, 中位年龄56岁, 男39例, 女18例, 肿瘤类型(低分化腺癌35例, 中-低分化腺癌5例, 高分化腺癌1例, 中分化腺癌2例, 其他类型14例); 慢性胃炎28例, 为湖南省中医附一院脾胃病科2014-12/2015-02住院患者, 所有病例均经胃镜检查确诊, 患者年龄27-68岁, 中位年龄55岁, 男19例, 女9例, 慢性浅表性胃炎26例, 慢性萎缩性胃炎2例. 该研究方案符合人体试验伦理学标准, 并得到伦理委员会的批准, 受试者在受试前已知情, 且获得书面同意. 诊断标准: 胃癌与慢性胃炎(包括慢性浅表性胃炎和慢性萎缩性胃炎)的诊断均根据《内科学》的诊断标准[3]; 纳入标准: (1)符合诊断标准, 年龄在20-75岁之间; (2)未经手术和放化疗; (3)自愿且能够配合参加的受试对象. 以上3项必须全部符合才能纳入. 排除标准: (1)不符合上述诊断标准者; (2)年龄<20或>75岁者; (3)患有口腔局部及唾液腺的炎症、消化道溃疡、肿瘤及先天性疾病; 患有舍格伦综合征、囊性纤维病; 患有其他系统严重并发疾病. 质量控制: (1)釆用统一诊断标准、统一调查表格、统一调查方法; (2)调査者在调査时严格按照"标准化"执行, 减少调查者偏倚, 确保资料的一致性和真实性; (3)电子胃镜及病理结果均由1 mo内三级甲等医院诊断. 蛋白指纹图谱(液体芯片)试剂盒(WCX磁珠、Wash Buffer、Elution Buffer、U9裂解液)及MALDI-TIF-MS(蛋白指纹图谱仪Ⅰ型), 均为湖州赛尔迪生物医药科技有限公司产品, 试剂盒批号为K20150501; dH2O(HPLC级)、CHCA为Sigma公司产品.
1.2.1 样品收集与预处理: 按照本课题专用临床观察表进行临床观察记录. 取材前1 d晚上临睡前清水漱口3次(不再进任何食物和药物), 采用非刺激性唾液采集方式, 第2天晨起后漱口前空腹取材. 由经过培训的课题组专人取材, 前5 min内的唾液自然吞下后开始收集, 患者将已经过消毒的无菌小圆柱形棉花含入口中, 口腔唾液积聚至一定量后, 将该棉花吐入置于冰浴中的15 mL唾液离心管内, 4 ℃下静置1 h后, 3000 r/min、4 ℃下离心10 min, 冰浴上分装在1 mL EP管中, 每管200 μL, 于-80 ℃冰箱冷冻保存. 实验时取出样本, 冰上解冻, 所有检测唾液样本均1次冻融. 取5 μL唾液加入10 μL U9裂解液, 混合孵育30 min后, 加入185 μL Wash Buffer稀释(唾液最终上样量为2.5 μL).
1.2.2 纳米磁珠预处理、上样和洗脱: (1)取WCX纳米磁珠50 μL加入到200 μLPCR管, 置于磁铁上孵育1 min(注意避免由于孵育时间过长导致磁珠结块), 去除上清液; (2)再依次加入100 μL Wash Buffer洗脱5 min, 在磁铁上孵育1 min, 去除上清液; 再重复步骤(2)操作1次; (3)向每个装有纳米磁珠的PCR管中加入100 μL处理好的唾液样品, 混匀后, 置于室温孵育30 min, 将PCR管置于磁铁上孵育1 min, 去除上清液; (4)每管加入100 μL Wash Buffer洗脱5 min, 然后将PCR管置于磁铁上孵育1 min, 去除上清液; 再重复步骤(4)操作一次; (5)在PCR管中加入10 μL Elution Buffer洗脱5 min(不能少于5 min), 将PCR管置于磁铁上孵育1 min, 取5 μL上清液移至另一个PCR管中; (6)装有5 μL上清液的PCR管中加入5 μL CHCA(基质)饱和溶液, 充分混匀(混合到样品颜色发灰, 而没有明显的沉淀并及时上样), 取2 μL混合溶液(1 μL唾液样品+1 μL基质)加样到Au/Steel芯片上, 待干后放入仪器读取.
1.2.3 芯片检测、数据采集和参数设置: 采用蛋白指纹图谱仪Ⅰ型(湖州赛尔迪生物医药科技有限公司)质谱仪读取芯片信息. 设置激光强度为190, 灵敏度为5, 收集数据的质荷比(m/z)范围为2000-25000 m/z, 优化范围为2000-15000 m/z, 信号收集位置40-60, 平均每点收集20次, 收集总点为100次. 已知多肽标准芯片标准(all-in-one), 激光离子流0.5. 用Ciphergen Proteinchip Software 3.2.1软件自动采集数据, 纵坐标为峰强度(蛋白相对含量), 横坐标为蛋白质质荷比(m/z).
统计学处理 对位于2000-25000 m/z峰值, 用Biomarker Wizard软件过滤噪音. 设置初始的噪音过滤值为5, 二次信噪比为2, 以10%为最小阈值进行聚类, 经上述数据预处理后, 采用t检验比较2组唾液蛋白质质谱数据(由Biomarker Wizard软件完成), 找出2组之间具有统计学意义的差异表达蛋白质峰. 用Biomarker Pattern Software 5.0.2采用决策树算法计算出多个变量(m/z为蛋白质质谱峰)变化对两样本的判别价值, 确定最佳的筛选模型(即诊断模型).
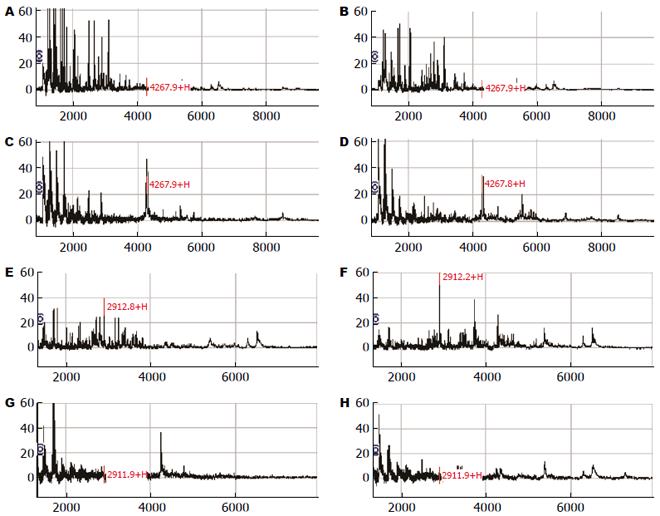
2.1.1 筛选差异蛋白质峰: 胃癌组与胃炎组两组共85份唾液标本, 经过标准化后, 在相对分子质量为2000-25000 m/z范围内共检测到371个蛋白质峰, 两组比值>2(胃癌组/慢性胃炎组>2, 或者慢性胃炎组/胃癌组>2)的共有23个差异蛋白质峰有统计学意义(P<0.05)(表1), 其中有3个蛋白峰胃癌组表达下调(典型下调图, 图1A-D, m/z = 4267), 14个蛋白峰胃癌组表达上调(典型上调图, 图1E-H, m/z = 2912), 共有17个差异蛋白质峰有显著差异(P<0.01).
| 序号 | m/z | 胃癌组(mean±SD) | 胃炎组(mean±SD) | 胃癌/胃炎 | 胃炎/胃癌 | P值 | 胃癌组表达上调/下调 |
| 1 | 4267.098 | 3.582±4.283 | 15.647±11.537 | 0.229 | 4.368 | 0.000 | Down |
| 2 | 2135.405 | 2.830±2.720 | 6.398±3.923 | 0.442 | 2.261 | 0.000 | Down |
| 3 | 2606.828 | 1.364±1.538 | 4.078±3.763 | 0.334 | 2.990 | 0.000 | Down |
| 4 | 6564.857 | 3.180±3.334 | 0.950±1.695 | 3.345 | 0.299 | 0.000 | Up |
| 5 | 13053.090 | 0.892±1.096 | 0.245±0.504 | 3.645 | 0.274 | 0.000 | Up |
| 6 | 5438.674 | 3.591±3.827 | 1.736±2.425 | 2.069 | 0.483 | 0.000 | Up |
| 7 | 2912.397 | 10.019±12.771 | 2.980±2.681 | 3.362 | 0.297 | 0.001 | Up |
| 8 | 12566.820 | 0.722±0.810 | 0.193±0.301 | 3.737 | 0.268 | 0.001 | Up |
| 9 | 2957.555 | 5.646±5.465 | 2.217±2.940 | 2.546 | 0.393 | 0.001 | Up |
| 10 | 6294.777 | 3.305±3.464 | 1.016±1.694 | 3.255 | 0.307 | 0.002 | Up |
| 11 | 3619.969 | 4.391±4.535 | 1.737±1.316 | 2.528 | 0.396 | 0.002 | Up |
| 12 | 6523.113 | 5.030±5.651 | 1.520±2.762 | 3.309 | 0.302 | 0.004 | Up |
| 13 | 5369.372 | 6.567±7.468 | 3.186±6.010 | 2.061 | 0.485 | 0.004 | Up |
| 14 | 3872.659 | 1.770±1.557 | 0.733±1.128 | 2.416 | 0.414 | 0.005 | Up |
| 15 | 10733.280 | 2.017±2.493 | 0.879±2.306 | 2.295 | 0.436 | 0.006 | Up |
| 16 | 2217.586 | 5.424±11.102 | 11.909±20.074 | 0.455 | 2.195 | 0.007 | Down |
| 17 | 2979.671 | 2.543±2.346 | 1.111±1.625 | 2.289 | 0.437 | 0.007 | Up |
| 18 | 2127.584 | 0.486±1.818 | 1.553±2.107 | 0.313 | 3.194 | 0.013 | Down |
| 19 | 2084.888 | 1.519±2.707 | 0.179±1.326 | 8.470 | 0.118 | 0.022 | Up |
| 20 | 2945.206 | 3.143±4.087 | 1.324±1.526 | 2.374 | 0.421 | 0.026 | Up |
| 21 | 2330.624 | 1.417±1.823 | 0.391±1.274 | 3.621 | 0.276 | 0.027 | Up |
| 22 | 3677.940 | 2.576±3.387 | 0.980±1.090 | 2.628 | 0.381 | 0.030 | Up |
| 23 | 2265.565 | 1.682±2.344 | 0.475±1.161 | 3.539 | 0.283 | 0.040 | Up |
2.1.2 建立诊断模型: 用Biomarker Pattern Software 5.0.2采用决策树算法计算出多个变量(m/z蛋白质质谱峰)变化对两样本的判别价值, 确定最佳的筛选模型(图2), 最终选定m/z为4267.09、6564.85、2138.14建立胃癌组与胃炎组的判别模型, 由4267.09、6564.85、2138.14 m/z 3个差异峰组成的诊断模型树状图. 当满足条件: (1)4268.09 m/z≤5.08; (2)5438.67 m/z>5.08、6564.85 m/z>0.88且2138.14 m/z≤7.10则提示为胃癌患者; 当满足条件: (1)4268.08 m/z>5.08且6564.85≤0.88; (2)4267.09m/z>5.08、6564.85>0.88且2138.14 m/z>7.10则提示为慢性胃炎患者. 该模型的灵敏度和特异度分别为96%(55/57)和86%(24/28)(表2).
| 分组 | n | 正确诊断 | 错误诊断 | 正确率(%) |
| 胃癌组 | 57 | 55 | 2 | 96 |
| 慢性胃炎组 | 28 | 24 | 4 | 86 |
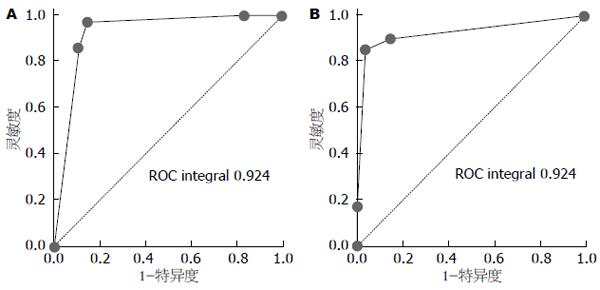
对所建立的胃癌与胃炎的鉴别诊断模型采用十字交叉法进行验证, 结果该模型的灵敏度和特异度分别89%(51/57)和75%(21/28)(表3), 十字交叉验证的ROC曲线值为0.924(图3), 进一步的分析验证了所建立模型的准确性.
| 分组 | n | 正确诊断 | 错误诊断 | 正确率(%) |
| 胃癌组 | 57 | 51 | 6 | 89 |
| 慢性胃炎组 | 28 | 21 | 7 | 75 |
众所周知, 胃癌的发病模式多为慢性胃炎-萎缩性胃炎-非完全性肠化生(小肠)-完全性肠化生(大肠)-中度异型增生-重度异型增生-胃癌, 慢性胃炎和慢性萎缩性胃炎是胃癌发生发展的一个重要阶段[4-6], 胃炎和胃癌的初期临床表现难以鉴别, 因此胃癌早期与慢性胃炎的鉴别诊断尤为重要. 胃癌诊断的金标准是内镜加活检[7], 但该检查属于侵入性检查, 检查时患者普遍痛苦体验强烈, 况且大量胃癌患者早期无明显症状, 主动就医性差, 难以作为胃癌普遍筛查手段; 血清胃泌素(serum gastrin, SG)水平的检查在胃癌诊断中具有重要作用, 但其在人体内的水平受种族和地域的影响较大[8]; B超多用于观察胃的邻近脏器, CT在组织特异性方面存在不足, 对胃黏膜的充血、水肿、浅表隆起或凹陷性病变不敏感[9]. 因此, 目前仍缺乏能够用于大众人群的胃癌早期筛查方法.
胃癌早期诊断是提高胃癌患者5年生存率的关键. 胃癌的发生发展常通过原癌通路, 包括幽门螺杆菌(Helicobacter pylori, H. pylori)感染型胃炎、慢性萎缩性胃炎、肠化生、原位癌等[10]. 慢性萎缩性胃炎和胃癌组织基因的表达既有相似性, 又有异质性, 肿瘤基因表达与癌前状态和癌前病变相关, 基因表达的相似性提示慢性萎缩性胃炎与胃癌这两者病理发展的相关性和延续性[11]. H. pylori感染是慢性胃炎和胃癌的共同致病因素[12-14], 而EBV病毒与H. pylori联合感染, 是肠型胃癌的重要危险因素[15]. 在慢性萎缩性胃炎患者体内检测到jhp0945, 是发展为胃癌的重要分子标志物[16]. 胃癌PGⅠ和PGⅠ/Ⅱ水平明显低于萎缩性胃炎组以及正常对照组[17]. RNA家族的分子有很多成员参与了胃癌发生发展的信息传导、调控等多条途径[18,19]. Ji等[20]通过检测p53β和△133p53两个亚型mRNA分别在慢性浅表性胃炎组织细胞、慢性萎缩性胃炎组织细胞、胃癌组织细胞的表达水平, 结果发现慢性浅表性胃炎组织细胞中仅有p53β mRNA表达, 慢性萎缩性胃炎组织细胞中同时有p53β和△133p53 mRNA的表达, 胃癌细胞系中没有p53β和△133p53 mRNA的表达. 分别采集25名慢性萎缩性胃炎患者、胃癌患者, 以及正常健康人的血清标本, 进行蛋白质组学分析, 构建并验证胃癌前病变及胃癌的诊断模型, 结果其敏感性和特异性均>65%, 高于现有的肿瘤标志物筛查水平[21].
寻找胃癌的生物标志物是目前研究胃癌早期诊断、分期、治疗靶点以及预后评估的热点. 蛋白质组学技术的发展为唾液生物标志物的发现提供了强大武器. 也已发现唾液中含有2300多种蛋白质[2], 利用唾液进行疾病诊断具有无创、微量、易于收集和储存、操作简便快捷、易于被患者接受等明显优势[22]. 已有研究证明, 唾液中含有H. pylori及H. pylori抗体IgG[23]、KYNA(具有抗结肠癌作用)[24]等180余种生物标志物[25]. 唾液蛋白的研究目前多采用的方法, 质谱技术是蛋白质组学研究中的前沿技术.
本项研究采用的WCX结合MALDI-TOF-MS蛋白质学技术在方法学上具有高通量、高敏感性、高特异性优势, 其检测的标本可以是血液[26]、尿液[27]、唾液等人体各种体液. 检测的疾病涵盖了糖尿病[28]、子宫内膜异位[29]、肺结核[30]等各系统疾病. 本研究应用WCX结合MALDI-TOF-MS技术分析胃癌与慢性胃炎患者唾液在蛋白质组水平上的差异, 发现了23个具有显著性意义的差异蛋白质峰, 经Biomarker Pattern Software 5.0.2系统软件筛选、采用决策树算法建立了由4267.09、6564.85、2138.14 m/z 3个显著差异蛋白峰组成的鉴别诊断模型, 经临床回代检验该模型的灵敏度和特异度分别达96%和86%; 对所建立的胃癌与胃炎的鉴别诊断模型采用十字交叉法进行验证, 结果该模型的灵敏度和特异度分别为89%和75%, 显示该模型对胃癌和慢性胃炎的鉴别具有较高的诊断效率. 本研究结果提示, 唾液蛋白质组学无创伤分子诊断方法对于鉴别诊断胃癌和慢性胃炎具有重要临床价值, 值得进一步深入研究并在临床推广应用.
慢性胃炎和胃癌的初期临床表现难以鉴别, 但预后却截然不同, 故胃癌与慢性胃炎的鉴别诊断尤为重要. 现有的胃癌诊断多为有创、侵入性检查, 患者主动就医性差, 因而寻找一种无创、简便、快速的胃癌诊断新方法势在必行.
姜春萌, 教授, 主任, 大连医科大学附属第二医院消化科
寻找胃癌的生物标志物是目前研究胃癌早期诊断、分期、治疗靶点以及预后评估的热点, 而蛋白质组学技术的发展为唾液生物标志物的发现提供了强大武器.
唾液是与血清成分相似的人体组织液, 在消化系统疾病诊断方面, 显示着与血液诊断相似的特异性及敏感性, 甚至在某些方面优于血清学的检查. 已发现唾液中含有2300多种蛋白质, 其中有180余种生物标志物可用于相关疾病的诊断.
唾液最主要和最重要的成分是蛋白质, 而蛋白质是生命活动的执行者和体现者, 是生物体最重要的结构部分, 是生命本质的物质基础. 选用唾液标本研究胃癌与胃炎的鉴别诊断, 探索胃癌无创伤分子诊断新方法.
胃癌与胃炎的唾液蛋白指纹图谱鉴别诊断模型具有较高的诊断效率, 对于鉴别诊断胃癌和慢性胃炎具有重要临床价值, 值得进一步深入研究并在临床推广应用.
蛋白指纹图谱技术: 是由蛋白质芯片及分析仪器-表面加强激光解析电离飞行时间质谱两部分组成, 可以将样品中的蛋白质成分的变化记录下来, 绘制成蛋白指纹质谱图, 并显示样品中各种蛋白的分子量、含量等信息. 通过这项技术, 将来人们有可能拥有自己的生命光盘, 记录下个人有生以来不同阶段的蛋白指纹图谱, 从分子水平了解并观察自己的生理变化, 或作为治疗前后的比对参照.
本文选用胃癌、慢性胃炎患者的唾液标本作为研究对象, 具有一定的创新性, 使用前沿的蛋白质组学技术, 获得了较好的研究结果, 是胃癌普查的有希望的新方法.
编辑: 郭鹏 电编:都珍珍
| 1. | 华 子辰, 朱 正伦, 朱 正纲. 血清肿瘤标志物在胃癌诊疗中的应用. 上海交通大学学报(医学版). 2014;34:1411-1416. |
| 2. | Bălănescu P, Lădaru A, Bălănescu E, Băicuş C, Dan GA. Systemic sclerosis biomarkers discovered using mass-spectrometry-based proteomics: a systematic review. Biomarkers. 2014;19:345-355. [PubMed] [DOI] |
| 3. | 陆 再英, 钟 南山. 内科学(第7版). 北京: 人民卫生出版社 2008; 382-385, 396-400. |
| 4. | Nishizawa T, Suzuki H, Akimoto T, Maehata T, Morizane T, Kanai T, Yahagi N. Effects of preoperative proton pump inhibitor administration on bleeding after gastric endoscopic submucosal dissection: A systematic review and meta-analysis. United European Gastroenterol J. 2016;4:5-10. [PubMed] [DOI] |
| 5. | Kim GH, Liang PS, Bang SJ, Hwang JH. Screening and Surveillance for Gastric Cancer in the United States: Is it needed? Gastrointest Endosc. 2016; Feb 29. [Epub ahead of print]. [PubMed] [DOI] |
| 6. | Ohira M, Toyokawa T, Sakurai K, Kubo N, Tanaka H, Muguruma K, Yashiro M, Onoda N, Hirakawa K. Current status in remnant gastric cancer after distal gastrectomy. World J Gastroenterol. 2016;22:2424-2433. [PubMed] [DOI] |
| 7. | Mahadevan D, Sudirman A, Kandasami P, Ramesh G. Laparoscopic staging in gastric cancer: An essential step in its management. J Minim Access Surg. 2010;6:111-113. [PubMed] [DOI] |
| 8. | Zhang XM, Li JX, Zhang GY, Li XH, Gu H. The value of serum pepsinogen levels for the diagnosis of gastric diseases in Chinese Han people in midsouth China. BMC Gastroenterol. 2014;14:3. [PubMed] [DOI] |
| 9. | Kim JH, Eun HW, Hong SS, Kim YJ, Han JK, Choi BI. Gastric cancer detection using MDCT compared with 2D axial CT: diagnostic accuracy of three different reconstruction techniques. Abdom Imaging. 2012;37:541-548. [PubMed] [DOI] |
| 10. | Gomes C, Almeida A, Ferreira JA, Silva L, Santos-Sousa H, Pinto-de-Sousa J, Santos LL, Amado F, Schwientek T, Levery SB. Glycoproteomic analysis of serum from patients with gastric precancerous lesions. J Proteome Res. 2013;12:1454-1466. [PubMed] [DOI] |
| 12. | Yakoob J, Abbas Z, Khan R, Salim SA, Abrar A, Awan S, Ahmad Z. Helicobacter pylori: correlation of the virulence marker iceA allele with clinical outcome in a high prevalence area. Br J Biomed Sci. 2015;72:67-73. [PubMed] |
| 13. | Ferreira Júnior M, Batista SA, Vidigal PV, Cordeiro AA, Oliveira FM, Prata LO, Diniz AE, Barral CM, Barbuto RC, Gomes AD. Infection with CagA-positive Helicobacter pylori strain containing three EPIYA C phosphorylation sites is associated with more severe gastric lesions in experimentally infected Mongolian gerbils (Meriones unguiculatus). Eur J Histochem. 2015;59:2489. [PubMed] [DOI] |
| 14. | Yazbek PB, Trindade AB, Chin CM, Dos Santos JL. Challenges to the treatment and new perspectives for the eradication of Helicobacter pylori. Dig Dis Sci. 2015;60:2901-2912. [PubMed] [DOI] |
| 15. | Cárdenas-Mondragón MG, Torres J, Flores-Luna L, Camorlinga-Ponce M, Carreón-Talavera R, Gomez-Delgado A, Kasamatsu E, Fuentes-Pananá EM. Case-control study of Epstein-Barr virus and Helicobacter pylori serology in Latin American patients with gastric disease. Br J Cancer. 2015;112:1866-1873. [PubMed] [DOI] |
| 16. | Gong Y, Peng X, He L, Liang H, You Y, Zhang J. The distribution of jhp0940, jhp0945, jhp0947, jhp0949 and jhp0951 genes of Helicobacter pylori in China. BMC Gastroenterol. 2015;15:115. [PubMed] [DOI] |
| 17. | 汪 畅. 血清胃蛋白酶原、胃泌素-17和幽门螺杆菌IgG抗体在萎缩性胃炎和早期胃癌诊断中的价值. 蚌埠: 蚌埠医学院 2013; . |
| 18. | Yang Q, Zhang RW, Sui PC, He HT, Ding L. Dysregulation of non-coding RNAs in gastric cancer. World J Gastroenterol. 2015;21:10956-10981. [PubMed] [DOI] |
| 19. | Huang YK, Yu JC. Circulating microRNAs and long non-coding RNAs in gastric cancer diagnosis: An update and review. World J Gastroenterol. 2015;21:9863-9886. [PubMed] [DOI] |
| 20. | Ji W, Zhang N, Zhang H, Ma J, Zhong H, Jiao J, Gao Z. Expression of p53β and Δ133p53 isoforms in different gastric tissues. Int J Clin Exp Pathol. 2015;8:10468-10474. [PubMed] |
| 21. | Li P, Ma D, Zhu ST, Tang XD, Zhang ST. Serum peptide mapping in gastric precancerous lesion and cancer. J Dig Dis. 2014;15:239-245. [PubMed] [DOI] |
| 22. | Martí-Álamo S, Mancheño-Franch A, Marzal-Gamarra C, Carlos-Fabuel L. Saliva as a diagnostic fluid. Literature review. J Clin Exp Dent. 2012;4:e237-e243. [PubMed] [DOI] |
| 23. | Krishnaswamy RT, David CM, Govindaiah S, Krishnaprasad RB, Jogigowda SC. Salivary IgG assay to detect Helicobacter pylori infection in an Indian adult population. Indian J Dent Res. 2012;23:694-695. [PubMed] [DOI] |
| 24. | Walczak K, Dąbrowski W, Langner E, Zgrajka W, Piłat J, Kocki T, Rzeski W, Turski WA. Kynurenic acid synthesis and kynurenine aminotransferases expression in colon derived normal and cancer cells. Scand J Gastroenterol. 2011;46:903-912. [PubMed] [DOI] |
| 25. | Al-Tarawneh SK, Border MB, Dibble CF, Bencharit S. Defining salivary biomarkers using mass spectrometry-based proteomics: a systematic review. OMICS. 2011;15:353-361. [PubMed] [DOI] |
| 26. | Matysiak J, Światły A, Hajduk J, Matysiak J, Kokot ZJ. Influence of honeybee sting on peptidome profile in human serum. Toxins (Basel). 2015;7:1808-1820. [PubMed] [DOI] |
| 27. | Fu G, Liu N, Chu L, Zhang M. Screening high abundance of peptide for making examination possible in human urine. Ann Clin Lab Sci. 2015;45:264-269. [PubMed] |
| 28. | Ai T, Chen F, Zhou S, Zhang J, Zheng H, Zhou Y, Hu W, Liu X, Li L, Lin J. Magnetic bead-based serum peptidome profiling in patients with gestational diabetes mellitus. Biomed Res Int. 2015;2015:586309. [PubMed] [DOI] |
| 29. | Zhao Y, Liu YN, Li Y, Tian L, Ye X, Cui H, Chang XH. Identification of biomarkers for endometriosis using clinical proteomics. Chin Med J (Engl). 2015;128:520-527. [PubMed] [DOI] |
| 30. | Liu J, Li Y, Wei L, Yang X, Xie Z, Jiang T, Wang C, Zhang X, Xu D, Chen Z. Screening and identification of potential biomarkers and establishment of the diagnostic serum proteomic model for the Traditional Chinese Medicine Syndromes of tuberculosis. J Ethnopharmacol. 2014;155:1322-1331. [PubMed] [DOI] |