修回日期: 2015-09-13
接受日期: 2015-09-21
在线出版日期: 2015-10-18
目的: 探讨联合单独或联合应用2对不同引物的16S rRNA序列分析法, 能否提高自发性细菌性腹膜炎(spontaneous bacterial peritonitis, SBP)腹水细菌检测的敏感性及准确性, 并且通过其检测细菌的结果来评价抗感染治疗效果.
方法: 收集77例肝硬化失代偿期合并腹水的标本, 其中SBP 61例, 非SBP 16例, 应用2对不同引物(MSQ-F/MSQ-R与27F/1492R)的组合, 对腹水细菌分别进行16S rRNA序列分析, 对腹水进行常规化验、细菌培养及生化方法鉴定细菌, 分析单独或联合应用2对不同引物的16S rRNA序列分析法在鉴定SBP腹水细菌中的应用价值, 并对腹水细菌状态进行动态评价.
结果: 与腹水细菌培养、生化鉴定的细菌结果相比, 16S rRNA序列分析法鉴定细菌的结果与之的符合率: 单用引物MSQ-F/MSQ-R: 属的符合率73.33%(11/15), 种的符合率66.67%(10/15); 单用27F/1492R引物: 属的符合率80.00%(12/15), 种的符合率73.3%(11/15); 引物MSQ-F/MSQ-R联合27F/1492R: 属的符合率86.67%(13/15), 种的鉴定的符合率80.00%(12/15), 应用2对引物的组合进行16S rRNA序列分析鉴定细菌, 可以得到更高的准确率. 5例SBP患者(腹水细菌培养阳性组2例, 临床感染组3例)经抗感染治疗后, 腹水细菌培养阴性, 但临床感染控制不理想的患者, 16S rRNA序列分析法检测腹水细菌仍为阳性.
结论: 联合应用2对不同引物的16S rRNA序列分析法, 可以提高SBP腹水细菌的鉴定能力, 并且可以动态应用于临床进行抗感染疗效评价.
核心提示: 本研究中应用2对不同的16S rRNA基因通用引物的组合, 对腹水细菌进行检测、鉴定, 结果发现该方法可提高自发性细菌性腹膜炎(spontaneous bacterial peritonitis)腹水细菌的鉴定能力; 可以检测抗生素治疗前后腹水的细菌, 能动态检测抗感染的效果, 评价抗感染治疗疗效.
引文著录: 赵莹莹, 邢卉春. 基于不同引物的16S rRNA序列分析法对SBP腹水细菌的鉴定及抗感染疗效评价的意义. 世界华人消化杂志 2015; 23(29): 4713-4719
Revised: September 13, 2015
Accepted: September 21, 2015
Published online: October 18, 2015
AIM: To explore a new method of polymerase chain reaction (PCR) based on 16S rRNA gene of bacteria with two different pairs of primers (used alone or in combination) and alignment sequencing, to improve the detection rate of bacteria in ascitic fluid and evaluate its clinical value in bacterial species identification and evaluation of antibiotic efficacy.
METHODS: Blood and ascitic fluid samples were collected from 77 spontaneous bacterial peritonitis (SBP) (n = 61) cirrhotic patients with ascites. Bacterial culture of ascitic fluid was conducted and the results were used as the gold standard in this study. Bacterial DNA in ascitic fluid was detected by PCR, based on the 16S rRNA gene, with two different pairs primers (MSQ-F/MSQ-R, 27F/1492R) and alignment sequencing assay. The results of bacterial species and positive rate between PCR method and bacterial culture of ascitic fluid were compared, and the results of bacterial species identified with different pair primers were also compared.
RESULTS: For products amplified with primers MSQ-F/MSQ-R, the coincidence rate of bacterial genus identification between PCR assay and ascitic fluid culture was 73.33% (11/15), and the coincidence rate of bacterial species identification was 66.67(10/15). For products amplified with primers27F/1492R, the coincidence rate of bacterial genus identification between PCR assay and ascitic fluid culture was 80.00% (12/15), and the coincidence rate of bacterial species identification was 73.33% (11/15). For products amplified with primers MSQ-F/MSQ-R combined with primers 27F/1492R, the coincidence rate of bacterial genus identification between PCR assay and ascitic fluid culture was 86.67% (13/15), and the coincidence rate of bacterial species identification was 80.00% (12/15). In five patients with SBP whose ascitic fluid culture was negative, PCR reaction was positive, which was consistent with clinical characteristics.
CONCLUSION: The method combining the two different pairs of primers for identifying bacteria based on the 16S rRNA gene and alignment sequencing has better identification ability and can evaluate antibiotic efficacy dynamically.
- Citation: Zhao YY, Xing HC. Clinical application of 16S rRNA sequencing analysis for diagnosis of spontaneous bacterial peritonitis and evaluation of antibiotic efficacy. Shijie Huaren Xiaohua Zazhi 2015; 23(29): 4713-4719
- URL: https://www.wjgnet.com/1009-3079/full/v23/i29/4713.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v23.i29.4713
失代偿期肝硬化合并腹水患者常见的并发症之一就是自发性细菌性腹膜炎(spontaneous bacterial peritonitis, SBP), 病死率高[1-5], 本实验室前期研究[6]采用16S rRNA基因序列分析方法, 探讨了其在快速诊断SBP中的应用价值, 结果显示该方法检测腹水细菌的灵敏度、特异度均为100%, 鉴定细菌的结果与腹水细菌培养、生化鉴定结果的属符合率为73.33%(11/15), 因此可以用于SBP的快速诊断, 结合序列测定可对检测阳性的细菌进行鉴定. 但单对引物对细菌鉴定的敏感性及准确性均有待进一步提高. 本研究结合前期工作, 选用2对不同的基于16S rRNA基因的引物形成组合后, 对腹水细菌进行PCR测序, 探讨单独或联合应用2对不同引物的16S rRNA序列分析法, 能否提高腹水细菌检测的敏感性及准确性, 并且观察该方法能否用于检测抗感染治疗过程中的细菌的动态变化, 从而进一步探讨其能否评价抗感染治疗效果.
选取均来自首都医科大学附属北京地坛医院住院患者, 为2012-03/2013-04的77例肝硬化失代偿期伴腹水的住院患者, 同时收集这些患者在抗感染治疗前、抗感染治疗1 wk后这两个时间点的所有临床资料. LH755全自动血液分析仪、全自动H7600生化分析仪(日立, 日本), 微生物鉴定分析仪、厌氧/需氧血培养瓶、Phoenix Spec Nephelometer比浊仪、BD-Phoenix 100微生物鉴定分析仪(BD, 美国), 精巧型恒温混匀仪(Eppendorf Thermomixer compact, 德国), PCR仪(PTC-240 MJ Rearch, 美国), 凝胶成像分析仪(WD-9413B, 六一牌, 北京六一仪器厂). Wizard® Genomic DNA Purification Kit(Promega, USA), PCR上下游引物、GoTaq酶(生工生物工程, 上海), Nuclerase Free Water、1000 bp、500 bp DNA Ladder Marker、10×Loading Buffer(宝生物工程, 大连), Gold View核酸染料(北京泛博生物化学有限公司), BD-Phoenix AST指示剂(BD, 美国).
1.2.1 SBP诊断标准: 排除腹腔脏器穿孔引起的继发性腹膜炎的情况下: (1)腹水中多形核白细胞计数(polymorphonuclear, PMN)≥0.25×109 cells/L的腹水[1]; (2)血性腹水(腹水中红细胞计数>10.00×103 cells/L)情况下, 腹水PMN数要根据腹水中红细胞数进行校正计算, 即每250个红细胞计数要减去1个PMN计数[2]; (3)腹水细菌培养阴性, 但是PMN>0.25×109 cells/L, 称为是变异性SBP[3]. 其中, 酒精性肝硬化腹水的患者, 出现发热、腹痛、腹水PMN升高就可以认为是SBP[1]. 有些腹水细菌培养结果为阴性, 但是血培养阳性, 且PMN升高, 也应可以诊断为SBP[2].
1.2.2 资料收集: 患者基本信息包括: 生命体征、腹腔感染的症状与体征, 是否合并肝性脑病、感染性休克等严重并发症; 除此之外, 还需收集患者的血常规、C-反应蛋白(C reactive protein, CRP)、降钙素原、血氨、肝肾功能等的变化值. 同时腹水床旁接种于血培养瓶及肝素锂抗凝管, 用于腹水细菌培养、腹水生化、鲎试验等检测、另外50 mL腹水标本用于提取细菌DNA并进行16S rRNA序列分析法; 将所有患者按照临床资料分为3组: (1)A组(腹水细菌培养阳性组, n = 15): 明确为腹腔感染[1]、腹水细菌培养阳性的SBP患者, 且排除继发性腹膜炎者; (2)B组(腹水细菌培养阴性且无临床感染组, n = 16): 腹水细菌培养阴性、临床诊疗过程证实无腹腔感染的患者; (3)C组(腹水细菌培养阴性组, n = 46): 腹水细菌培养阴性、有腹腔感染的局部或全身系统症状, 和/或腹部压痛、反跳痛等提示临床腹腔感染症状、体征, 部分患者细菌血培养结果为阳性, 经抗感染治疗后, 患者局部及全身体征消失, 腹水PMN及血中性粒细胞百分比降至正常. 本研究方案已通过伦理委员会的批准, 每位同意参加研究的患者都需要签署知情同意书.
1.2.3 腹水细菌培养, 生化鉴定方法进行细菌鉴定: 腹水20 mL, 床旁接种于血培养瓶, 然后置于BD-100微生物鉴定分析仪进行培养, 应用Phoenix Spec Nephelometer比浊仪及BD专利呈色反应(指示剂随细菌生长过程中的氧化还原反应而变色的反应)结合的双重检测, 进行细菌及药敏的鉴定. (指示剂为246003 AST Broth 4 0.00 DM、246009 AST-S Indicator 100.00 DM).
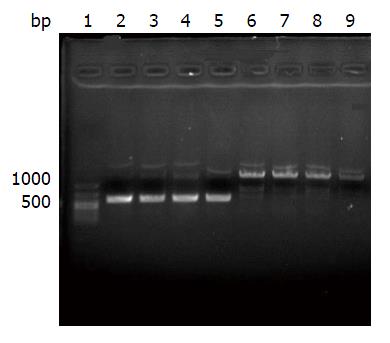
1.2.4 基于16S rRNA基因的PCR反应: (1)腹水细菌基因组DNA的提取: 按照试剂盒说明书提取; (2)PCR反应: 用于检测腹水的细菌基因组DNA. 参照相关文献[7-9], 以16S rRNA基因通用引物MSQ-F/MSQ-R、27F/1492R进行PCR反应, 前者的PCR产物大小约为500 bp, 后者的PCR产物长度约为1500 bp(表1); (3)PCR反应条件: MSQ-F/MSQ-R: 94 ℃预变性10 min、94 ℃变性45 s、56 ℃退火30 s、72 ℃延伸1 min、重复循环36次, 72 ℃ 10 min、4 ℃保存(抗生素治疗前后). 27F/1492R: A组于抗生素治疗前后进行; 94 ℃预变性10 min、94 ℃变性45 s、52 ℃退火30 s、72 ℃延伸1.5 min, 重复循环36次, 72 ℃ 10 min、4 ℃保存.
| 引物 | 序列(方向5'-3') | 位置(以E. coli)为准 | 备注 | 温度(℃) |
| MSQ-F | AGAGTTTGATCATGGCTCAG | 7-26 | 针对多数细菌 | 58 |
| MSQ-R | ACCGCGACTGCTGCTGGCAC | 531-512 | 针对肠道细菌 | 68 |
| 27F | AGAGTTTGATCCTGGCTCAG | 8-27 | 针对肠道相关细菌 | 57.8 |
| 1492R | GGYTACCTTGTTACGACTT-CTT | 1512-1292 | 针对肠道大部分细菌 | 54.3 |
1.2.5 PCR产物的进行序列测序及细菌鉴定: 将电泳验证结果为阳性的PCR产物的样本外送测序; 测序结果回报后均在网上用BLAST工具分析(http://blast.ncbi.nlm.nih.gov/Blast.cgi)与GenBank已知核酸序列(http://www.ncbi.nlm.nih.gov/Genbank/index.html)比对分析, 进行细菌种、属的鉴定. 并与腹水细菌培养的结果进行比较.
统计学处理 对计数资料进行分析, 计算16S rRNA序列分析法鉴定细菌的阳性率; 同时比较其与腹水细菌培养的生化方法鉴定的细菌种、属的符合率.
A组检测腹水细菌的阳性率为100%(15/15), C组检测腹水细菌的阳性率为76.09%(35/46). B组患者的PCR检测及腹水培养均为阴性; C组的腹水PMN≥0.25×109 cells/L的标本仅47.82%(22/46).
应用2对不同引物(16S rRNA与27F/1492R)进行基于16S rRNA基因的PCR反应后, 扩增产物进行序列测定, 经BLAST工具分析及GenBank已知核酸序比对后, 再将得到的细菌鉴定结果进行整理, 发现应用引物MSQ-F/MSQ-R进行16S rRNA序列分析与腹水细菌培养的生化鉴定方法相比, 二者菌属鉴定的符合率为73.33%(11/15), 菌种的符合率66.67%(10/15); 单用27F/1492R引物: 二者菌属鉴定符合率80.00%(12/15)菌种的符合率73.30%(11/15); 联合应用引物16S rRNA与27F/1492R组合: 二者菌属的符合率86.67%(13/15), 菌种的鉴定符合率80.00%(12/15)(图1).
通过比较腹水细菌培养的生化鉴定与16S rRNA序列分析检测方法, 结果发现与腹水培养的生化鉴定方法相比, 联合应用2对不同引物的组合进行16S rRNA序列分析法鉴定细菌(以MSQ-F/MSQ-R及27F/1492R为引物)的结果, 与腹水细菌培养、生化鉴定细菌的属的符合率更高, 可基本满足临床需要, 要高于单用一种引物对腹水细菌鉴定的方法(以MSQ-F/MSQ-R为引物的16S rRNA基因PCR扩增).
抗生素的使用过程中, 在使用的第3、5、7天时均要对抗生素疗效进行评价. 因此, 我们针对抗生素治疗前及治疗1 wk后的患者资料进行了分析.
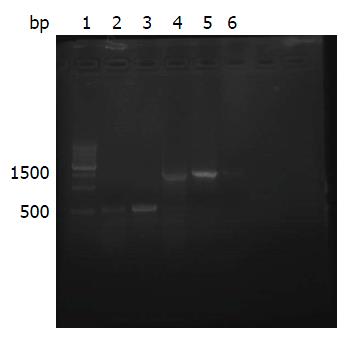
A组(腹水细菌培养阳性组)的患者经治疗后, 腹水细菌培养及再次进行基于16S rRNA基因的PCR反应均呈阴性的结果有13例; 仍呈阳性的有2例. 这2例在抗生素治疗前的腹水细菌培养结果分别为头状葡萄球菌、屎肠球菌; 应用抗生素后, 腹水细菌培养结果为阴性, 16S rRNA基因PCR的结果分别为克雷伯氏杆菌、大肠杆菌. C组(腹水细菌培养阴性组)的患者经治疗后, 用腹水再次进行基于16S rRNA基因的PCR反应, 有43例患者呈阴性结果; 而仍有3例患者呈阳性: 抗感染治疗前的细菌为克雷伯氏杆菌、大肠埃希氏菌、变形杆菌, 抗感染治疗后分别为克雷伯氏杆菌、大肠埃希氏菌、变形杆菌(图2).
这5例患者PCR反应仍阳性的患者, 经临床分析认为, 其仍存在感染, 调整抗感染治疗后, 发热、腹痛、腹膜刺激征症状明显缓解; 提示该5例患者确实仍然存在腹腔感染.
SBP患者早期大多数中毒症状不明显, 近半数患者无腹痛, 腹膜刺激征轻微甚至缺如[1,2]. 目前SBP的确诊主要依赖于腹水培养、腹水细菌学检查, 但腹水细菌培养阳性率很低, 即使腹水中PMN≥0.25×109 cells/L的腹水, 细菌培养阳性率也仅达65%[3-5], 临床对SBP的疗效评价主要依靠对患者生命体征状况、感染症状、体征及血常规、CRP、降钙素原、鲎试验、腹水细胞计数等综合评价[10-13]. 近年来, 随着分子生物学技术的发展, 许多研究者利用细菌16S rRNA基因恒定区设计引物, 可以对不同菌属、菌种的细菌进行分类鉴定[3,10,14,15]. Srinivasan等[14]利用16S rRNA基因测序法与传统的非16S rRNA基因相比, 鉴定细菌的属的符合率及种的符合率为均较高; 可鉴定临床的革兰氏阴性杆菌、革兰氏阳性球菌及革兰氏阴性球菌. Teng等[15]也证实应用16S rRNA基因测序法可以鉴定需氧革兰氏阴性杆菌. 潘红英等[16]应用16S rRNA基因PCR及基因芯片检测了腹水细菌基因组DNA. Hardick等[17]应用高分辨熔融分析技术诊断SBP. 全敏等[18]建立了SBP常见感染菌PCR-单链构象多态性(single-strand conformation polymorphism)图谱, 同时使用2对引物(ER10/ER11与P11P/P13P)同时扩增标准菌DNA. 本实验室前期研究, 进行了基于16S rRNA基础上的PCR方法在快速诊断自发性细菌性腹膜炎中应用价值的相关研究[6], 结果表明基于16S rRNA基因的方法检测腹水细菌感染的特异性、灵敏度、阳性预测值、阴性预测值均较高, 在快速诊断SBP中有一定的应用价值, 可以从属、种的水平鉴定细菌, 这些结论与既往的研究结论一致[14,15]; 但是测序结果与GenBank核酸数据库中的已知序列进行菌种鉴定时, 有4份标本的菌种与腹水细菌培养得到的生化鉴定菌种不符合, 为此我们设计了2对引物, 以期能提高检测效率及细菌鉴定能力.
本研究通过比较腹水细菌培养的生化鉴定与16S rRNA序列分析检测方法, 结果发现与腹水培养的生化鉴定方法相比, 联合应用2对不同引物的组合进行16S rRNA序列分析法鉴定细菌的结果, 与腹水细菌培养、生化鉴定细菌的属的符合率更高. 当然, 这也可能与2对引物自身的特点有关: 从MSQ-F/MSQ-R及27F/1492R的扩增产物的靶序列位置来看, 后者几乎覆盖16S rRNA细菌基因组DNA的全长, 联合应用2对引物组合进行反应时, 一旦其中1对引物基础上的16S rRNA序列分析法鉴定的结果与生化鉴定结果不一致时, 可以参考另外1对引物的测序结果, 有助于评定细菌的鉴定结果, 指导临床. 以细菌培养、生化鉴定结果为标准, 用2对不同引物组合的进行16S rRNA序列分析, 对细菌菌属鉴定的准确性较好. 5例自发性细菌性腹膜炎患者(A组2例, C组3例)经抗感染治疗后, 腹水细菌培养阴性, 但临床感染控制不理想的患者, 经PCR测序检测腹水细菌仍为阳性. 这5例患者虽然发热、腹痛、腹膜刺激征较用药前明显缓解, 腹水细菌培养阴性, 但血中性粒细胞百分比或降钙素原或CRP较前无明显变化(数值仍超出正常参考值), 用16S rRNA序列分析法检测获得了细菌阳性的结果, 提示基于16S rRNA基因的PCR测序可更灵敏地检测到腹水的细菌感染. 这可能因为16S rRNA基因序列分析法能检测的模板起始浓度低; 对标本的纯度要求不高, 可为未经任何处理的临床标本; 具有特异性强、灵敏度高等特点: 只需针对靶序列设计一段通用16S rRNA基因保守区引物即可进行反应. 但是由于不同的引物含有不同的GC含量、Tm值, 导致扩增的效率及产物会有所不同.
由此可见, 本研究与既往的应用16S rRNA基因恒定区设计引物, 来对不同菌属、菌种的细菌进行分类鉴定研究相比, 有以下几个优点: (1)简便的技术路线: 本研究只采用了普通实验室常用的PCR技术, 一般在2-4 h内完成扩增反应, 收集标本的当天便可以应用基于16S rRNA的PCR方法检测腹水是否有细菌感染, PCR方法阳性的再送去测序, 测序结果通常在次日即可回报; 操作更方便、简洁、省时、可重复性强; (2)费用低: 与高通量的深度测序或基因芯片相比, 本研究采用的方法在费用上更为大多数实验室能接受, 应用于临床则会使患者诊断费用大大降低; (3)安全性高: 整个实验过程中未应用明显放射性及毒性物质, 对环境及人体都不具有明显损害性.
本研究中应用的腹水细菌鉴定方法, 与其他方法相比, 有以下3个优点: (1)创新: 研究中应用2对不同的16S rRNA基因通用引物, 并将之进行组合应用于腹水细菌的检测、鉴定, 尤其是对抗感染治疗前后的腹水细菌也进行了检测及鉴定, 这是本研究不同于既往研究的创新之处; (2)准确率更高: 单独应用1对引物的研究方法, 检测腹水细菌的灵敏度、特异度均比较高, 鉴定细菌的结果与腹水细菌培养、生化鉴定的属符合率也可以满足临床的需要, 但是应用2对引物的组合进行16S rRNA基因序列分析鉴定细菌, 可以得到更高的准确率, 提高SBP腹水细菌的鉴定能力; (3)评价抗感染疗效: 通过检测抗生素治疗前后腹水的细菌, 能动态检测抗感染的效果, 评价抗感染治疗疗效.
16S rRNA基因序列分析法能用于鉴定所有病原细菌, 虽步骤相对多于传统的腹水细菌培养、生化鉴定方法, 但是其稳定性好、准确度高、适用面广、费用低、可重复性强等特点, 无论是在属还是种的层次上, 都有较高的准确率. 正是这些特点, 使本研究中应用的, 通过联合应用2对不同引物的16S rRNA基因序列分析法, 可以用于SBP的快速诊断及提高腹水细菌的鉴定能力, 同时可以通过其检测抗感染过程中的细菌, 来对SBP的抗感染治疗效果进行动态评价. 在SBP的诊断中, 该方法具有较高的细菌阳性率、及菌属、种鉴定的符合率, 而且费用低, 有希望改变目前SBP的腹水细菌培养阳性率不高、花费昂贵的现状.
自发性细菌性腹膜炎(spontaneous bacterial peritonitis, SBP)是失代偿期肝硬化合并腹水患者常见的并发症, 病死率高, 目前其确诊主要靠腹水细菌培养, 但阳性率很低. 近年许多研究利用细菌16S rRNA基因恒定区设计引物, 对细菌进行分类鉴定. 本实验室前期进行了16S rRNA基础上的PCR方法快速诊断SBP的相关研究, 表明该方法可以应用于SBP的早期快速诊断, 但检测效率及细菌鉴定能力还有待于提高. 本研究结合前期工作, 探讨联合应用2对不同引物的组合, 能否改进腹水细菌鉴定方法, 且观察其能否动态检测细菌.
张进祥, 副教授, 华中科技大学同济医学院附属协和医院
细菌16S rDNA通常有10个可变区, 其间以恒定区相间隔. 由于16S rDNA基因序列总长度适宜, 结构完整, 在细菌中含量多、有足够的信息量,故在细菌学研究中常被作为研究的靶分子. 通常设计保守区引物,即所有细菌的共同引物进行PCR,可判断细菌的存在与否; 通过对扩增产物序列的分析, 可以对细菌进行种属鉴定和分类.
Srinivasan等利用16S rRNA基因测序法鉴定细菌, 获得较高的属、种的符合率, 可鉴定革兰氏阴性杆菌、革兰氏阳性球菌及革兰氏阴性球菌. Teng等也证实应用16S rRNA基因测序法可以鉴定需氧革兰氏阴性杆菌. 潘红英等应用16S rRNA基因PCR及基因芯片检测了腹水细菌基因组DNA. Hardick等应用高分辨熔融分析技术联合16S PCR检测SBP的腹水细菌, 结果提示其有较高的敏感性、特异性及细菌种的鉴定符合率. 全敏等建立了SBP常见感染菌PCR-单链构象多态性图谱, 可以同时使用2对引物扩增标准菌DNA.
本研究应用基于不同引物的16S rRNA序列分析法对SBP腹水细菌鉴定, 技术路线简便, 省时、可重复性强、费用低、安全性高, 整个实验过程中未应用明显放射性及毒性物质. 应用2对不同的16S rRNA基因通用引物组合, 进行了腹水细菌的检测、鉴定, 尤其是对抗感染治疗前后的腹水细菌也进行了检测及鉴定, 这是不同于以往研究的创新之处; 除了得到更高的准确率、提高SBP腹水细菌的鉴定能力外, 还能动态检测抗感染的效果, 评价抗感染治疗疗效.
16S rRNA基因序列分析法能用于鉴定所有病原细菌, 其稳定性好、准确度高、适用面广、费用低、可重复性强等特点, 无论是在属还是种的层次上, 都有较高的准确率, 因此该方法可用于SBP的快速诊断及提高腹水细菌的鉴定能力, 同时还能检测抗感染过程中的细菌动态变化, 对抗感染治疗效果进行评价. 在SBP的诊断中, 该方法具有较高的细菌检出阳性率、及菌属、种鉴定的符合率、费用低,有希望改变目前SBP的腹水细菌培养阳性率不高、花费昂贵的现状.
自发性细菌性腹膜炎: 是失代偿期肝硬化合并腹水患者常见的并发症之一, 病死率高, 是在腹腔和邻近器官无直接细菌感染来源的情况下发生于腹腔的感染; 多形核白细胞计数: 多形核白细胞主要是中性粒细胞, 也包括包括少量嗜酸性粒细胞、嗜碱性粒细胞和肥大细胞; 鲎试验: 是检测内毒素血症的一种试验. 在内毒素性休克、急性化脓性胆管炎、重症肝炎、腹膜炎、肝硬化等临床病例中阳性率较高.
本文联合应用2对不同引物的16S rRNA序列分析法, 证明此方法可以提高SBP腹水细菌的鉴定能力, 并且可以动态应用于临床进行抗感染疗效评价. 研究内容具有一定的重要性, 为诊断SBP提供了依据. 本篇文章有足够的实用性. 研究结果较准确, 实验证据较充分. 讨论部分条理分明, 有系统的理论分析. 文章具有可读性.
编辑: 于明茜 电编:都珍珍
| 1. | Runyon BA. Management of adult patients with ascites due to cirrhosis: an update. Hepatology. 2009;49:2087-2107. [PubMed] [DOI] |
| 2. | Rimola A, García-Tsao G, Navasa M, Piddock LJ, Planas R, Bernard B, Inadomi JM. Diagnosis, treatment and prophylaxis of spontaneous bacterial peritonitis: a consensus document. International Ascites Club. J Hepatol. 2000;32:142-153. [PubMed] |
| 3. | Koulaouzidis A, Bhat S, Saeed AA. Spontaneous bacterial peritonitis. World J Gastroenterol. 2009;15:1042-1049. [PubMed] |
| 4. | Shizuma T, Fukuyama N. Investigation into bacteremia and spontaneous bacterial peritonitis in patients with liver cirrhosis in Japan. Turk J Gastroenterol. 2012;23:122-126. [PubMed] |
| 5. | Wiest R, Krag A, Gerbes A. Spontaneous bacterial peritonitis: recent guidelines and beyond. Gut. 2012;61:297-310. [PubMed] [DOI] |
| 6. | 赵 莹莹, 全 敏, 王 琦, 程 丹颖, 段 英, 李 贲, 杨 松, 吴 淑玲, 刘 顺爱, 邢 卉春. PCR方法在自发性腹膜炎快速诊断中的应用. 中华实验和临床感染病杂志(电子版). 2013;7:19-22. |
| 9. | Isoun TT. The histopathology of experimental disease produced in mice infected with Trypanosoma vivax. Acta Trop. 1975;32:267-272. [PubMed] |
| 10. | Hwang SM, Kim MS, Park KU, Song J, Kim EC. Tuf gene sequence analysis has greater discriminatory power than 16S rRNA sequence analysis in identification of clinical isolates of coagulase-negative staphylococci. J Clin Microbiol. 2011;49:4142-4149. [PubMed] [DOI] |
| 11. | 刘 志娟, 蔡 晧东, 张 艳华. 血清降钙素原在肝硬化并发自发性腹膜炎患者诊治中的变化. 中华实验和临床感染病杂志. 2012;6:238-241. |
| 12. | 全 敏, 邢 卉春. 肝硬化合并自发性细茵性腹膜炎诊断的研究进展. 中华实验和临床感染病杂志. 2012;6:156-158. |
| 13. | Koulaouzidis A. Diagnosis of spontaneous bacterial peritonitis: an update on leucocyte esterase reagent strips. World J Gastroenterol. 2011;17:1091-1094. [PubMed] [DOI] |
| 14. | Srinivasan R, Karaoz U, Volegova M, MacKichan J, Kato-Maeda M, Miller S, Nadarajan R, Brodie EL, Lynch SV. Use of 16S rRNA gene for identification of a broad range of clinically relevant bacterial pathogens. PLoS One. 2015;10:e0117617. [PubMed] [DOI] |
| 15. | Teng JL, Yeung MY, Yue G, Au-Yeung RK, Yeung EY, Fung AM, Tse H, Yuen KY, Lau SK, Woo PC. In silico analysis of 16S rRNA gene sequencing based methods for identification of medically important aerobic Gram-negative bacteria. J Med Microbiol. 2011;60:1281-1286. [PubMed] [DOI] |
| 16. | 潘 红英, 谌 翠容, 尚 志强, 孙 洪运, 陈 群伟, 徐 晶, 叶 荣夏, 娄 国强, 卢 德荣. 定量PCR联合基因芯片检测腹水细菌16S rRNA诊断自发性细菌性腹膜炎. 中华肝脏病杂志. 2011;19:297-300. |
| 17. | Hardick J, Won H, Jeng K, Hsieh YH, Gaydos CA, Rothman RE, Yang S. Identification of bacterial pathogens in ascitic fluids from patients with suspected spontaneous bacterial peritonitis by use of broad-range PCR (16S PCR) coupled with high-resolution melt analysis. J Clin Microbiol. 2012;50:2428-2432. [PubMed] [DOI] |
| 18. | 全 敏, 李 炜, 王 琦, 邢 卉春, 成 军. 自发性腹膜炎常见感染菌PCR-SSCP图谱的建立. 中华实验和临床感染病杂志. 2013;7:9-11. |