修回日期: 2015-02-11
接受日期: 2015-03-06
在线出版日期: 2015-04-18
目的: 研究小干扰RNA(small interfering RNA, siRNA)沉默人胰腺癌Capan-2、PANC-1细胞中叉头框蛋白C1(fork head box C1, FOXC1)基因对胰腺癌细胞增殖能力的影响及作用机制.
方法: 以胰腺癌细胞、原发胰腺癌组织为研究对象, 实时定量逆转录聚合酶链反应(quantitative real-time reverse transcription polymerase chain reaction, qRT-PCR)检测FOXC1 mRNA在胰腺癌细胞及组织中的表达情况; 将人胰腺癌细胞分为2组: FOXC1 siRNA组(实验组)、NC siRNA组(阴性对照组), 脂质体转染法将FOXC1 siRNA转染入胰腺癌细胞, qRT-PCR、Western blot技术检测胰腺癌细胞FOXC1 mRNA及蛋白表达变化, 5-乙炔基-2'-脱氧尿苷(5-ethynyl-2'-deoxyuridine, EdU)细胞增殖法检测各组胰腺癌细胞增殖情况; 流式细胞技术(flow cytometry, FCM)检测各组胰腺癌细胞的周期分布, Western blot技术分析周期相关蛋白P21、P53、Cyclin D1蛋白表达情况.
结果: qRT-PCR结果提示: 相比正常胰腺上皮细胞和癌旁胰腺组织, FOXC1 mRNA在胰腺癌细胞及胰腺癌组织中表达较高(P<0.05); qRT-PCR及Western blot结果显示FOXC1 siRNA有效沉默了胰腺癌细胞FOXC1基因的转录和表达, EdU细胞增殖实验提示: 沉默了胰腺癌细胞FOXC1基因表达后, 胰腺癌细胞胞的增殖能力明显下降, 较阴性对照组, 差异有统计学意义(P<0.05); FCM结果显示: 沉默了胰腺癌细胞FOXC1基因表达后胰腺癌细胞被阻滞在G0/G1期, 较阴性对照组, 差异有统计学意义(P<0.05); Western blot结果显示: 较阴性对照组, P21、P53表达水平无明显变化, Cyclin D1表达水平下降, 差异有统计学意义(P<0.05).
结论: FOXC1 siRNA能够有效沉默人胰腺癌Capan-2、PANC-1细胞FOXC1基因的表达, 抑制其增殖, 将细胞周期阻滞于G0/G1期, 提示FOXC1影响细胞增殖可能是通过调控细胞周期实现的, 其机制可能是部分通过调控细胞周期蛋白Cyclin D1的表达而实现.
核心提示: 本研究采用小干扰RNA技术, 有效沉默了胰腺癌细胞中转录因子叉头框蛋白C1(fork head box C1, FOXC1)基因表达, 阻滞细胞周期于G0/G1期, 抑制了胰腺癌细胞的增殖, 其机制可能部分通过调控细胞周期蛋白Cyclin D1的表达而实现, 可能为胰腺癌治疗提供参考靶点和理论依据.
引文著录: 喻超, 江建新, 孙诚谊. 沉默FOXC1基因对人胰腺癌细胞增殖的影响. 世界华人消化杂志 2015; 23(11): 1712-1720
Revised: February 11, 2015
Accepted: March 6, 2015
Published online: April 18, 2015
AIM: To investigate small interfering RNA (siRNA)-mediated silencing of fork head box protein C1 (FOXC1) gene on the proliferation human pancreatic cancer Capan-2 and PANC-1 cells and to explore the possible underlying mechanisms.
METHODS: Quantitative real-time reverse transcription-polymerase chain reaction (qRT-PCR) was used to examine the expression of FOXC1 mRNA in pancreatic cancer cell lines and primary carcinoma tissues from human patients. The human pancreatic cancer cells were divided into two groups: an FOXC1 siRNA group (experimental group) and an NC siRNA group (negative control group). qRT-PCR and Western blot were used to detect FOXC1 mRNA and protein expression in pancreatic cancer cells and to determine proliferation rate after transfection with 5-ethynyl-2'-deoxyuridine (EdU). Flow cytometry was used to examine cell cycle distribution. Western blot analysis was used to detect the expression of cell cycle related proteins P21, P53, and Cyclin D1.
RESULTS: FOXC1 mRNA expression in pancreatic cancer cells and pancreatic cancer tissues were significantly higher than in normal epithelial cells and matched tumor adjacent pancreatic tissue, respectively (P < 0.05). qRT-PCR and Western blot analysis showed that FOXC1 siRNA effectively silenced the transcription and expression of FOXC1 in pancreatic cancer cells. EdU cell proliferation experiments showed that compared with the control group, silencing FOXC1 gene expression in pancreatic cancer cells significantly decreased cell proliferation (P < 0.05). FCM results showed that compared with the control group, FOXC1 silencing arrested the tumor cells in G0/G1 phase (P < 0.05). Western blot analysis showed that compared with the control group, Cyclin D1 expression was significantly decreased (P < 0.05), while P21 and P53 expression was unchanged.
CONCLUSION: FOXC1 siRNA can effectively silence the FOXC1 gene expression in human pancreatic cancer Capan-2 PANC-1 cells. FOXC1 silencing inhibits pancreatic cancer cell proliferation and arrests cell cycle in G0/G1 phase, suggesting that FOXC1 alters cell proliferation possibly by regulation of the cell cycle through, in part, regulating the expression of Cyclin D1.
- Citation: Yu C, Jiang JX, Sun CY. Effect of siRNA-mediated FOXC1 gene silencing on proliferation of pancreatic cancer cells. Shijie Huaren Xiaohua Zazhi 2015; 23(11): 1712-1720
- URL: https://www.wjgnet.com/1009-3079/full/v23/i11/1712.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v23.i11.1712
胰腺癌恶性程度高, 预后差, 5年生存率不足5%, 其发生和发展与多种因素相关, 而多基因改变被认为是维持其恶性生物学行为的主要原因[1-7]. 肿瘤细胞的主要生物学特征之一是细胞的异常增殖, 这也是决定肿瘤恶性生物学行为的一个重要参数. 肿瘤细胞的异常增殖与肿瘤的浸润、转移以及肿瘤复发等行为密切相关. 因此, 抑制肿瘤增殖一直是肿瘤学研究的热点. 转录因子叉头框蛋白C1(fork head box C1, FOXC1)是叉头框转录因子基因家族(FOX家族)的一员, FOX家族蛋白参与许多细胞生物学进程, 在胚胎发育和肿瘤发生发展中发挥重要作用, 参与乳腺癌[8,9]、胃癌[10]等的发生与发展, 但在胰腺癌中的报告很少. 本研究以胰腺癌细胞和组织为研究对象, 通过实时定量逆转录聚合酶链反应(quantitative real-time reverse transcription polymerase chain reaction, qRT-PCR)检测FOXC1基因的表达情况, 并通过小干扰RNA(small interfering RNA, siRNA)沉默胰腺癌细胞中FOXC1基因表达, 观察其对胰腺癌细胞增殖的影响, 并探讨其可能的作用机制, 为胰腺癌的基因诊断与治疗提供实验依据.
人胰腺癌细胞系: Capan-2、PANC-1购自美国模式培养物集存库(American type culture collection, ATCC); 人胰腺导管上皮(HPDE)细胞(来源: Ontario Cancer Institute, Ontario, Canada), 原代人正常胰腺上皮细胞(Normal Epithelia, 来源: 上海睿星生物公司); 原发性胰腺癌组织: 住院并接受手术切除的胰腺癌组织和癌旁对应无瘤组织18例, 全部标本均经组织学确诊, 实验的设计与实施, 符合伦理学要求. 胎牛血清购自美国Gibco公司; 逆转录(RT)试剂盒、聚合酶链反应(PCR)相关SYBR Premix试剂、FOXC1上游、下游引物购自宝生物工程(大连)有限公司; Lipofectamine2000购自Invitrogen公司; 5-乙炔基-2'-脱氧尿苷(5-ethynyl-2'-deoxyuridine, EdU)试剂盒, FOXC1 siRNA购自广州市锐博生物科技有限公司, siRNA序列如下: 靶序列: 5'-CCAGTGAACGGGAATAGTA-3'; 正义链: 5'-CCAGUGAACGGGAAUAGUAdTdT-3'反义链: 5'-dTdT GGUCACUUGCCCUUAUCAU-3'; FOXC1、GAPDH一抗购自Abcam公司; Cyclin D1、P53、P21一抗购自武汉博士德生物技术公司; 辣根过氧化物酶标记的山羊抗小鼠IgG购自Santa Cruz公司; 激光扫描共聚焦显微镜LSM710型(德国 ZEISS).
1.2.1 细胞培养: Capan-2、PANC-1细胞常规培养; HPDE, Normal Epithelia细胞培养在含有10%胎牛血清的CS-C培养基中[11,12], 细胞培养在37 ℃、50 mL/L CO2培养箱内, 根据细胞的生长情况2-3 d更换新鲜培养基继续培养或细胞传代培养.
1.2.2 qRT-PCR检测胰腺癌细胞及胰腺癌组织FOXC1 mRNA表达: 参考文献[13]: TRIzol法提取胰腺癌Capan-2和PANC-1细胞及18对原发性胰腺癌组织总RNA, 检测RNA的A260/280值为1.8-2.0之间为RNA样品合格. 按宝生物(大连)工程有限公司提供的PCR反应步骤检查FOXC1 mRNA的表达. PCR检测系统为BIO-RED公司, 采用两步法PCR扩增标准程序上机操作, 反应结束后确认FOXC1的扩增曲线和融解曲线, 采用2-△△Ct方法计算FOXC1 mRNA相对表达量: △Ct = Ct待测基因-CtU6; 2-△△Ct即为各实验组待测基因的相对表达量.
1.2.3 FOXC1 siRNA转染与验证: 取对数生长期的Capan-2与PANC-1细胞, 胰酶消化后, 用10%FBS培养液重悬, 细胞接种于6孔板. 根据说明书建议, 将siRNA浓度稀释为50 nmol/L, 用脂质体转染法转染siRNA, qRT-PCR及Western blot验证胰腺癌细胞FOXC1基因的沉默效率.
1.2.4 EdU细胞增殖检测实验: EdU是一种人工合成的胸腺嘧啶核苷类似物, 能够在DNA复制时期代替胸腺嘧啶渗入正在合成的DNA分子中, 其连有的炔烃基团在天然化合物中极少见, EdU可与荧光染料Apollo产生特异性反应, 根据Apollo染料的荧光数量即可直观地观察到细胞DNA的复制活性, 了解细胞增殖情况[14,15]. 分别将NC siRNA组及FOXC1 siRNA组接种于共聚焦培养皿, 100 μL 50 μmol/L EdU培养液孵育2 h, 4%多聚甲醛固定, 50 μL 2 mg/mL甘氨酸溶液脱色, 分步Apollo、Hoechst染色反应液染色, 激光扫描共聚焦显微镜观察并拍照记录, 图像Merge后, 计算Apollo染色阳性细胞与Hoechst染色阳性细胞的比值, t检验分析组间差异.
1.2.5 流式细胞技术检测细胞周期分布: 胰酶消化对数生长期的各组人胰腺癌细胞, 收集到离心管中, 室温离心(1000 r/min, 5 min)收集细胞, 用4 ℃预冷的PBS重悬洗涤细胞2次后, 加入200 μL PBS重悬细胞, 缓慢滴加1.5 mL预冷处理的无水乙醇, 最后放置在-20 ℃固定过夜. 第2天, 取出存放的固定好的细胞, 离心(1000 r/min, 4 ℃, 5 min)收集细胞, 4 ℃预冷的PBS洗2次, 500 μL PBS重悬细胞, 然后加入终浓度为50 μg/mL的RNA酶及碘化丙啶(propidium iodide, PI), 室温条件下避光反应30 min. 用300目筛网过滤得单细胞悬液, 流式仪检测细胞周期的分布, 采用CellQuest和ModFit软件分析实验结果, 每组细胞设置3个复孔.
1.2.6 Western blot检测FOXC1基因沉默对胰腺癌细胞增殖相关蛋白的影响: Western blot法检腺癌细胞生长相关通路蛋白P21、P53及Cyclin D1的表达, 初步探讨FOXC1基因沉默对胰腺癌细胞增殖抑制的机制, 收集各组细胞, 裂解后提取总蛋白, BCA法检测蛋白浓度后, 蛋白上样量50 μg, 聚丙烯酰胺凝胶(SDS-PAGE)电泳, 电转至硝酸纤维素膜上, 5%脱脂奶粉封闭, 按抗体使用说明用5%脱脂奶粉稀释一抗, 4 ℃孵育过夜, TBST洗3次, 每次15 min. 加入对应的HRP标记的二抗室温孵育1 h. TBST洗3次, 每次15 min, ECL发光, 曝光机上曝光, 根据曝光结果可调整曝光时间, 采集图像后用图像分析软件Image J进行灰度分析, 以GAPDH作为内参.
统计学处理 实验结果数据采用SPSS19.0进行统计分析, 以mean±SD记录, 组间差异采用t检验, P<0.05为差异有统计学意义. 所有实验重复3次.
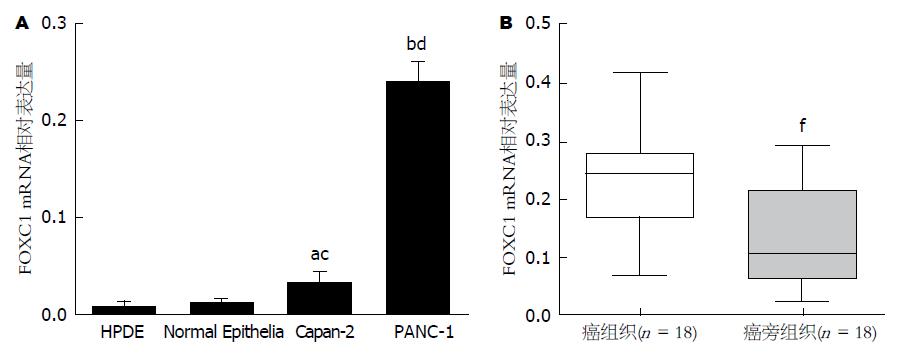
采用qRT-PCR检测目的基因FOXC1 mRNA在2株人胰腺癌Capan-2和PANC-1细胞及2株正常胰腺上皮细胞(HPDE、Normal Epithelia)中表达情况, 结果显示, FOXC1 mRNA在胰腺癌细胞中的相对表达量比正常胰腺上皮细胞低, 两组之间差异有统计学意义(图1A). 同时, 检测18例原发性胰腺癌组织和癌旁对应无瘤组织FOXC1 mRNA表达情况, 结果显示, FOXC1 mRNA在胰腺癌组织中表达比正常胰腺组织中高, 两组之间差异有统计学意义(图1B).
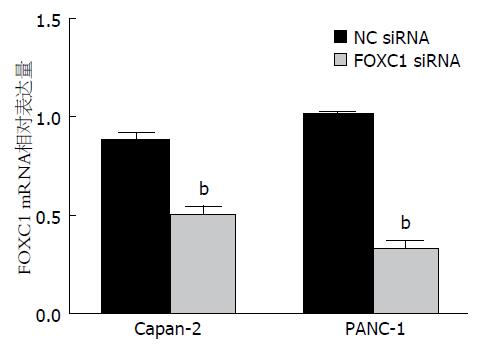
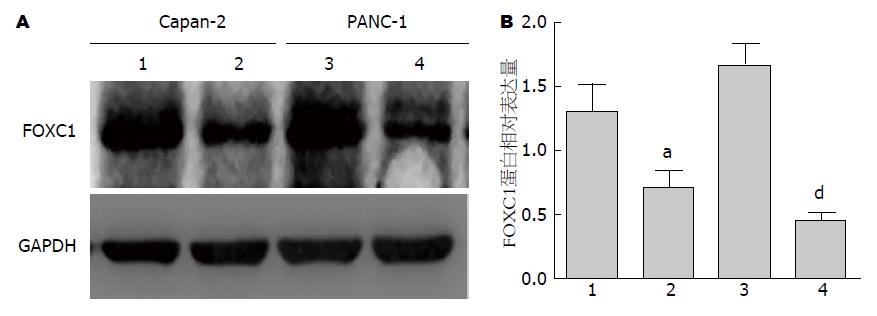
人胰腺癌Capan-2和PANC-1细胞转染FOXC1 siRNA后48 h, 采用qRT-PCR检测目的基因FOXC1 mRNA的表达情况, 判断沉默效果, 实验组较阴性对照组, 2株胰腺癌细胞FOXC1 mRNA的表达量均减少>70%, 差异有统计学意义(P<0.05, 图2). 同时, 采用Western blot进一步检测目的基因FOXC1蛋白的表达情况, 结果与qRT-PCR一致, 2株胰腺癌细胞FOXC1蛋白的表达量减少分别达72.56%和78.23%, 差异有统计学意义(P<0.05, 图3).
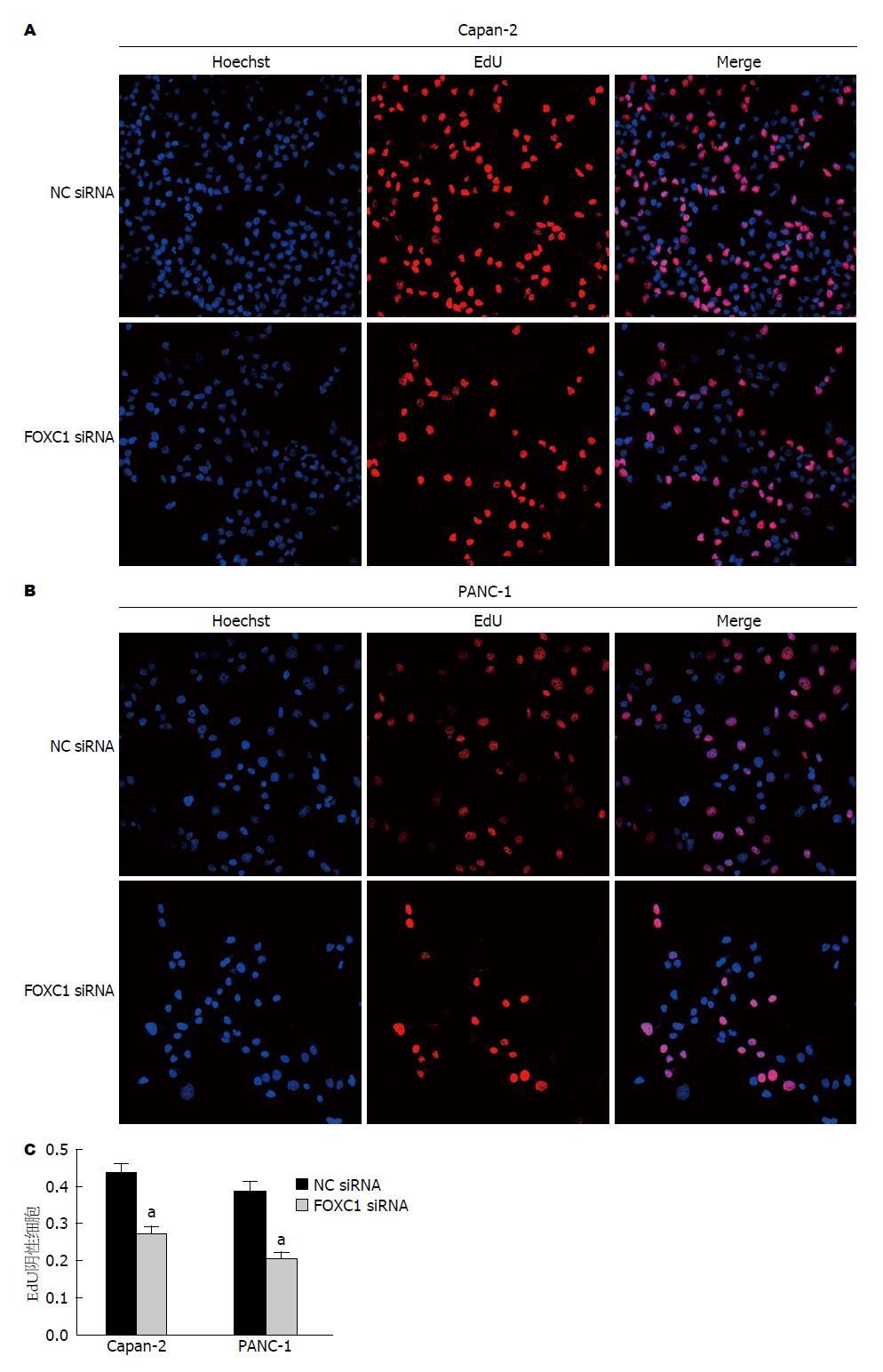
人胰腺癌Capan-2和PANC-1细胞转染FOXC1 siRNA后48 h, 采用EdU检测siRNA沉默胰腺癌细胞增殖情况, FOXC1 siRNA能够明显抑制胰腺癌Capan-2和PANC-1细胞增殖, 实验组较阴性对照组, 胰腺癌增殖细胞分别减少38.23%和42.17%, 差异具有统计学意义(P<0.05, 图4).
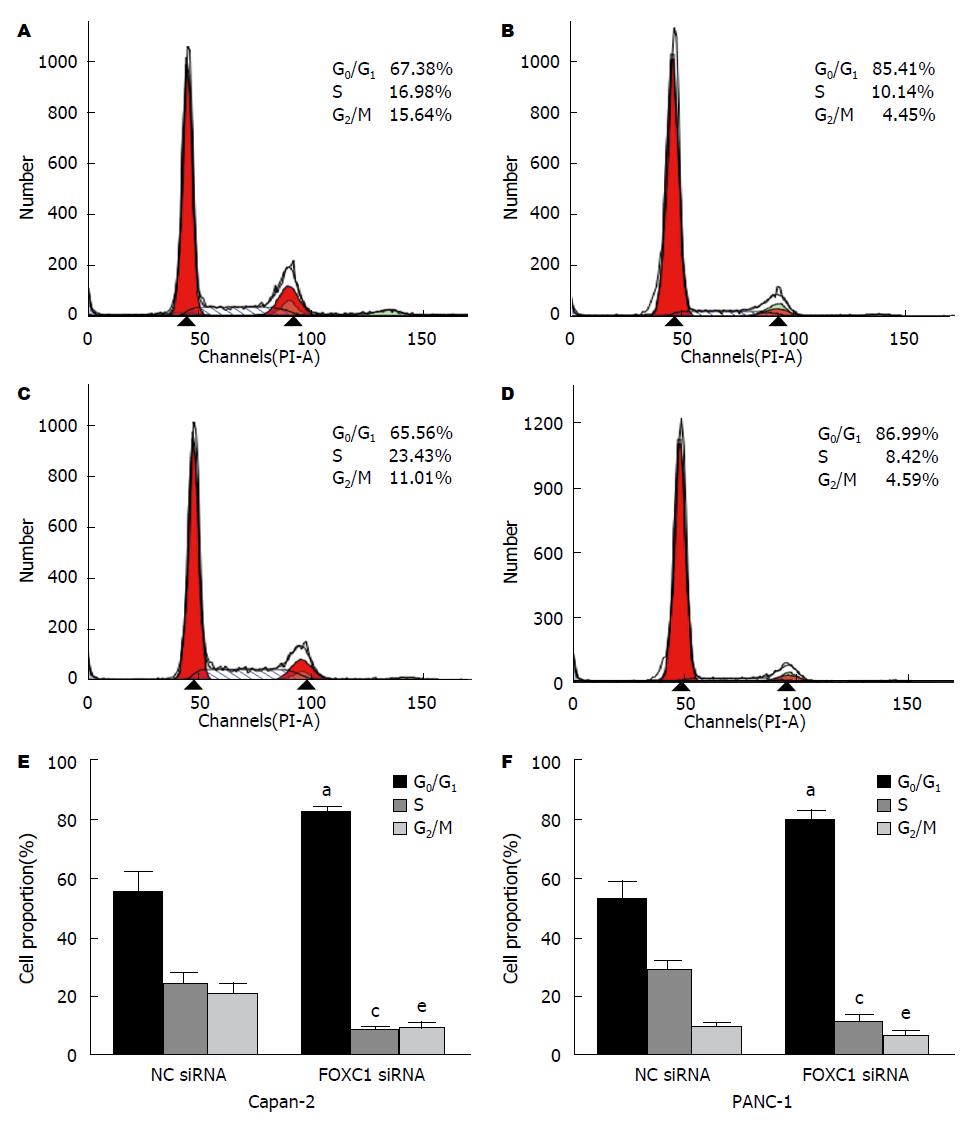
与阴性对照组比较, 两株胰腺癌细胞FOXC1 siRNA组细胞处于G0/G1细胞周期的细胞比例均明显增高, S期、G2/M期所占比例均减少, 差异具有统计学意义(P<0.05)
(图5).
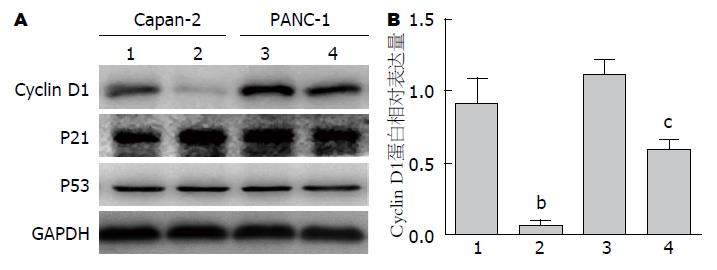
采用Western blot检测胰腺癌细胞生长相关通路蛋白, 内源性P21、P53及Cyclin D1的表达, 初步探讨FOXC1基因沉默对胰腺癌细胞增殖抑制的可能机制, 以GAPDH作为内参, 与阴性对照组比较, 结果显示, FOXC1 siRNA组Cyclin D1的表达明显被抑制, 蛋白表达量分别下降了86.23%和52.71%, 差异具有统计学意义(P<0.05)(图6).
转录因子FOXC1属于FOXC亚族, 该亚族目前发现2个成员, 分别为FOXC1和FOXC2, FOXC1基因位于染色体6p25, 全长3500 bp, 仅包含一个长于1600 bp的外显子, 编码的蛋白含553个氨基酸残基, 蛋白产物FOXC1从N端至C端依次为转录激活区(active domain 1, AD1)、叉头区(forkhead domain, FHD)、抑制区/磷酸化区和转录激活区(active domain 2, AD2)[16], 在叉头区内的两端各有一个核定位信号(nuclear localizationsignal, NLS), 其中C端为经典NLS基序. 早期对FOXC1的研究发现, 其主要与Axenfeld-Rieger综合症和胚胎发育有关[17-20], 近年来的研究表明, FOXC1在肿瘤发生、发展中发挥重要作用. Hayashi等[21]发现FOXC1与Notch信号和血管内皮生长因子相互作用多步骤地调节血管基因的表达, 诱导肿瘤血管生成. Ray等[9]研究发现, FOXC1 mRNA高表达者颅内无瘤生存期显著缩短, 此外, FOXC1的异位表达可诱导肿瘤侵袭性增强, 如促进细胞上皮-间质转化(epithelial-mesenchymal transition, EMT)过程, 而EMT正是肿瘤细胞从原位向远处迁移和侵袭的重要细胞生物学机制[22,23]. 应用shRNA敲除基底细胞样乳腺癌癌细胞中内源性高表达的FOXC1后, 肿瘤丧失侵袭性行为.
然而, FOXC1基因在胰腺癌中的作用却知之甚少, 首先, 我们应用qRT-PCR检测FOXC1基因在原发性胰腺癌组织及胰腺癌细胞中的表达情况, 我们发现, FOXC1在原发性胰腺癌组织及胰腺癌细胞中均高表达, 说明其可能跟胰腺癌的发生、发展相关, 为了进一步探讨FOXC1在胰腺癌中的具体作用, 我们通过siRNA技术, 沉默胰腺癌细胞FOXC1基因表达, 发现胰腺癌细胞的增殖受到明显抑制. 细胞的增殖和细胞周期密切相关[24-26]. 癌细胞和正常细胞一样细胞周期依次包括G0期、G1期、S期、G2期、M期. S期为DNA合成期、M期为细胞分裂期、G1-S期的转换是细胞周期中的关键调节点, 外界因素影响干扰癌细胞G1-S期转换都可能影响到细胞周期的进行, 抑制癌细胞的增殖[27,28], 我们通过流式细胞周期分析进一步发现siRNA沉默胰腺癌细胞FOXC1基因表达后, 细胞被阻滞于G0/G1期, 并通过Western blot验证细胞周期相关的几个蛋白P53、P21、Cyclin D1的表达变化, 我们发现周期蛋白Cyclin D1的表达明显受到抑制. Cyclin D1是细胞周期蛋白中的一员, 主要负责细胞周期的G1-S期的转换, 是公认的癌基因[29-31]. 可见, FOXC1影响胰腺癌细胞的增殖机制可能是部分通过调控细胞周期蛋白Cyclin D1而实现的.
总之, 本研究发现FOXC1在胰腺癌中是作为癌基因形式存在, 并通过调控细胞周期蛋白Cyclin D1的表达影响胰腺癌细胞的增殖, 这为胰腺癌的基因诊断和生物靶向治疗提供新的思路.
胰腺癌高度恶性、预后较差, 5年生存率不到5%; 大多数患者因确诊时伴有局部和/或远处的侵袭转移而丧失手术时机, 即使行根治性切除术, 效果也不理想, 胰腺癌的早期转移、极易复发及化疗耐药是其致死的关键因素. 所以, 研究促进胰腺癌发生发展的关键分子, 对治疗胰腺癌具有重要意义.
刘亮, 副主任医师, 复旦大学胰腺癌研究所, 复旦大学附属肿瘤医院胰腺肝胆外科
叉头框蛋白C1(fork head box C1, FOXC1)基因是一种转录因子, 早期对FOXC1的研究发现, 其主要与Axenfeld-Rieger综合症和胚胎发育有关; 近年来, 越来越多的研究发现FOXC1与肿瘤的发生、发展密切相关. FOXC1基因在胰腺癌中的作用却知之甚少.
有研究报道, FOXC1基因高表达者颅内无瘤生存期显著缩短, 此外, FOXC1的异位表达可增强肿瘤侵袭性, 加快细胞上皮-间质转化(epithelial-mesenchymal transition, EMT)过程, 应用shRNA敲除基底细胞样乳腺癌癌细胞中内源性高表达的FOXC1后, 肿瘤丧失侵袭性行为.
文献报道FOXC1基因能促进肿瘤的血管生成和侵袭转移, 加快肿瘤的EMT进程,而本研究发现胰腺癌组织及细胞中FOXC1基因高表达, 与胰腺癌细胞的恶性增殖密切相关.
研究发现FOXC1在胰腺癌中是一种癌基因, 其过表达会促进胰腺癌细胞增殖, 可望为胰腺癌的基因诊断和生物靶向治疗提供新的思路.
FOXC1基因在胰腺癌中的研究甚少, 其上下游调控机制尚未完全明确, 本文在揭示胰腺癌的进展机制方面具有一定的意义, 可能为治疗提供参考靶点和理论依据.
编辑: 郭鹏 电编: 都珍珍
| 1. | Danovi SA, Wong HH, Lemoine NR. Targeted therapies for pancreatic cancer. Br Med Bull. 2008;87:97-130. [PubMed] [DOI] |
| 2. | Chen WQ, Zheng RS, Zhang SW, Li N, Zhao P, Li GL, Wu LY, He J. Report of incidence and mortality in china cancer registries, 2008. Chin J Cancer Res. 2012;24:171-180. [PubMed] [DOI] |
| 3. | Gillen S, Schuster T, Meyer Zum Büschenfelde C, Friess H, Kleeff J. Preoperative/neoadjuvant therapy in pancreatic cancer: a systematic review and meta-analysis of response and resection percentages. PLoS Med. 2010;7:e1000267. [PubMed] [DOI] |
| 4. | Bardeesy N, DePinho RA. Pancreatic cancer biology and genetics. Nat Rev Cancer. 2002;2:897-909. [PubMed] [DOI] |
| 5. | Siegel R, Ma J, Zou Z, Jemal A. Cancer statistics, 2014. CA Cancer J Clin. 2014;64:9-29. [PubMed] [DOI] |
| 6. | Michl P, Gress TM. Current concepts and novel targets in advanced pancreatic cancer. Gut. 2013;62:317-326. [PubMed] [DOI] |
| 7. | Rochefort MM, Ankeny JS, Kadera BE, Donald GW, Isacoff W, Wainberg ZA, Hines OJ, Donahue TR, Reber HA, Tomlinson JS. Impact of tumor grade on pancreatic cancer prognosis: validation of a novel TNMG staging system. Ann Surg Oncol. 2013;20:4322-4329. [PubMed] [DOI] |
| 8. | Ray PS, Bagaria SP, Wang J, Shamonki JM, Ye X, Sim MS, Steen S, Qu Y, Cui X, Giuliano AE. Basal-like breast cancer defined by FOXC1 expression offers superior prognostic value: a retrospective immunohistochemical study. Ann Surg Oncol. 2011;18:3839-3847. [PubMed] [DOI] |
| 9. | Ray PS, Wang J, Qu Y, Sim MS, Shamonki J, Bagaria SP, Ye X, Liu B, Elashoff D, Hoon DS. FOXC1 is a potential prognostic biomarker with functional significance in basal-like breast cancer. Cancer Res. 2010;70:3870-3876. [PubMed] [DOI] |
| 10. | Xu Y, Shao QS, Yao HB, Jin Y, Ma YY, Jia LH. Overexpression of FOXC1 correlates with poor prognosis in gastric cancer patients. Histopathology. 2014;64:963-970. [PubMed] [DOI] |
| 11. | Xiao J, Peng F, Yu C, Wang M, Li X, Li Z, Jiang J, Sun C. microRNA-137 modulates pancreatic cancer cells tumor growth, invasion and sensitivity to chemotherapy. Int J Clin Exp Pathol. 2014;7:7442-7450. [PubMed] |
| 12. | Li Z, Li X, Yu C, Wang M, Peng F, Xiao J, Tian R, Jiang J, Sun C. MicroRNA-100 regulates pancreatic cancer cells growth and sensitivity to chemotherapy through targeting FGFR3. Tumour Biol. 2014;35:11751-11759. [PubMed] [DOI] |
| 13. | Yu C, Yang SL, Fang X, Jiang JX, Sun CY, Huang T. Hypoxia disrupts the expression levels of circadian rhythm genes in hepatocellular carcinoma. Mol Med Rep. 2015;11:4002-4008. [PubMed] [DOI] |
| 14. | Qiu S, Huang D, Yin D, Li F, Li X, Kung HF, Peng Y. Suppression of tumorigenicity by microRNA-138 through inhibition of EZH2-CDK4/6-pRb-E2F1 signal loop in glioblastoma multiforme. Biochim Biophys Acta. 2013;1832:1697-1707. [PubMed] [DOI] |
| 15. | Yang S, Luo A, Hao X, Lai Z, Ding T, Ma X, Mayinuer M, Shen W, Wang X, Lu Y. Peroxiredoxin 2 inhibits granulosa cell apoptosis during follicle atresia through the NFKB pathway in mice. Biol Reprod. 2011;84:1182-1189. [PubMed] [DOI] |
| 16. | Berry FB, Saleem RA, Walter MA. FOXC1 transcriptional regulation is mediated by N- and C-terminal activation domains and contains a phosphorylated transcriptional inhibitory domain. J Biol Chem. 2002;277:10292-10297. [PubMed] [DOI] |
| 17. | Nishimura DY, Searby CC, Alward WL, Walton D, Craig JE, Mackey DA, Kawase K, Kanis AB, Patil SR, Stone EM. A spectrum of FOXC1 mutations suggests gene dosage as a mechanism for developmental defects of the anterior chamber of the eye. Am J Hum Genet. 2001;68:364-372. [PubMed] [DOI] |
| 18. | Tanwar M, Kumar M, Dada T, Sihota R, Dada R. MYOC and FOXC1 gene analysis in primary congenital glaucoma. Mol Vis. 2010;16:1996-2006. [PubMed] |
| 19. | Aldinger KA, Lehmann OJ, Hudgins L, Chizhikov VV, Bassuk AG, Ades LC, Krantz ID, Dobyns WB, Millen KJ. FOXC1 is required for normal cerebellar development and is a major contributor to chromosome 6p25.3 Dandy-Walker malformation. Nat Genet. 2009;41:1037-1042. [PubMed] [DOI] |
| 20. | Kume T. The cooperative roles of Foxc1 and Foxc2 in cardiovascular development. Adv Exp Med Biol. 2009;665:63-77. [PubMed] |
| 21. | Hayashi H, Kume T. Forkhead transcription factors regulate expression of the chemokine receptor CXCR4 in endothelial cells and CXCL12-induced cell migration. Biochem Biophys Res Commun. 2008;367:584-589. [PubMed] [DOI] |
| 22. | Jechlinger M, Grunert S, Tamir IH, Janda E, Lüdemann S, Waerner T, Seither P, Weith A, Beug H, Kraut N. Expression profiling of epithelial plasticity in tumor progression. Oncogene. 2003;22:7155-7169. [PubMed] [DOI] |
| 23. | Strizzi L, Bianco C, Normanno N, Seno M, Wechselberger C, Wallace-Jones B, Khan NI, Hirota M, Sun Y, Sanicola M. Epithelial mesenchymal transition is a characteristic of hyperplasias and tumors in mammary gland from MMTV-Cripto-1 transgenic mice. J Cell Physiol. 2004;201:266-276. [PubMed] [DOI] |
| 24. | Schwartz GK, Shah MA. Targeting the cell cycle: a new approach to cancer therapy. J Clin Oncol. 2005;23:9408-9421. [PubMed] [DOI] |
| 25. | Olivera-Martinez I, Schurch N, Li RA, Song J, Halley PA, Das RM, Burt DW, Barton GJ, Storey KG. Major transcriptome re-organisation and abrupt changes in signalling, cell cycle and chromatin regulation at neural differentiation in vivo. Development. 2014;141:3266-3276. [PubMed] [DOI] |
| 26. | Galderisi U, Jori FP, Giordano A. Cell cycle regulation and neural differentiation. Oncogene. 2003;22:5208-5219. [PubMed] [DOI] |
| 27. | Evan GI, Vousden KH. Proliferation, cell cycle and apoptosis in cancer. Nature. 2001;411:342-348. [PubMed] [DOI] |
| 29. | Motokura T, Bloom T, Kim HG, Jüppner H, Ruderman JV, Kronenberg HM, Arnold A. A novel cyclin encoded by a bcl1-linked candidate oncogene. Nature. 1991;350:512-515. [PubMed] [DOI] |