修回日期: 2014-11-14
接受日期: 2014-11-25
在线出版日期: 2015-01-08
目的: 筛选胃癌(gastric cancer, GC)转移相关的差异表达微小RNA(microRNA, miRNA), 探讨miRNA-210与GC转移的可能机制.
方法: 应用miRNA表达谱芯片筛选GC高转移细胞株RF48及低转移细胞株RF1的差异表达miRNAs, RT-PCR检测miRNA-210在7种GC细胞株中的表达情况, 利用miRWalk软件获得miRNA-210的靶基因, 通过David在线软件(包括GO分析及KEGG通路预测)分析miR-210靶基因的可能生物学功能.
结果: 与RF1相比, miRNA芯片在RF48细胞中共筛选出21个表达上调和15个表达下调的miRNA, 其中miRNA-210在高转移细胞株RF48中的表达增高; RT-PCR结果显示miRNA-210在高转移GC细胞株中表达增高(P<0.05); miRWalk共获得155个miRNA-210靶基因; GO分析及KEGG pathway分析得到miRNA-210靶基因功能, 这些靶基因参与了肿瘤的发生、发展及转移.
结论: 利用miRNA芯片技术获得了GC转移相关miRNA表达谱; miRNA-210的异常表达可能与GC的转移有关, 为GC转移的早期诊断及发现新的治疗靶点奠定了基础.
核心提示: 本文利用微小RNA(microRNA, miRNA)芯片技术获得了胃癌(gastric cancer, GC)转移相关miRNA表达谱; miRNA-210的异常表达可能与GC的转移有关, 为GC转移的早期诊断及发现新的治疗靶点奠定了基础.
引文著录: 张小静, 冯先玲, 黄勇, 高燕, 曹子一, 黄伟玲, 张媛, 简千贺, 钟锦成, 杨梦婷, 范新民, 金哲. miRNA-210在胃癌转移细胞株中的表达及功能预测. 世界华人消化杂志 2015; 23(1): 78-84
Revised: November 14, 2014
Accepted: November 25, 2014
Published online: January 8, 2015
AIM: To screen microRNAs (miRNAs) associated with metastasis of gastric cancer (GC) by miRNA microarray and to explore the possible role of miRNA-210 in GC metastasis by bioinformatics.
METHODS: GC cell lines with low (RF1) or high metastatic potential (RF48) were used for miRNA expression profiling using human miRNA microarray. Expression of miRNA-210 in 7 GC cell lines was detected by RT-PCR. MiRNA-210 targets were obtained using miRWalk, and functions of these targets in GC were predicted with David online.
RESULTS: Compared with RF1 cells, 21 and 15 miRNAs were up-regulated and down-regulated in RF48 cells, respectively. Expression of miRNA-210 was further validated by real-time quantitative RT-PCR in multiple GC cell lines with different metastatic potential, which showed that miRNA-210 was overexpressed in GC cell lines with high metastatic potential. Bioinformatics analysis suggested that miRNA-210 was related with tumorgenesis and metastasis.
CONCLUSION: Screening miRNAs associated with metastasis lays a foundation for identifying early diagnostic markers and new therapeutic targets for GC metastasis. Expression profile of miRNAs associated with metastasis was obtained by miRNA microarray; dysregulated expression of miRNA-210 may be related with GC metastasis, and may serve as an early diagnostic biomarker and new treatment target.
- Citation: Zhang XJ, Feng XL, Huang Y, Gao Y, Cao ZY, Huang WL, Zhang Y, Jian QH, Zhong JC, Yang MT, Fan XM, Jin Z. Expression of miRNA-210 in gastric cancer cell lines and its function prediction. Shijie Huaren Xiaohua Zazhi 2015; 23(1): 78-84
- URL: https://www.wjgnet.com/1009-3079/full/v23/i1/78.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v23.i1.78
微小RNA(microRNA, miRNA)是一类长约18-25个核苷酸, 具有转录后调控作用的非编码RNA, 通过与靶基因的结合, 直接降解或抑制靶基因的mRNA的表达而发挥作用[1,2]. 至从1993年被首次报导, 已有上千种人miRNA相继被发现, 这些miRNA能调节人类的蛋白编码基因的表达. 大量文献[3-5]报道人类肿瘤中存在miRNA的异常表达, 例如, 在乳腺癌、结直肠癌、肝癌等多种癌症中miRNA的异常表达相继被报道. miRNA的异常表达与肿瘤的形成、细胞的增殖、凋亡、侵袭及血管形成等过程密切相关[6,7]. miR-449a能诱导前列腺癌细胞的生长阻滞[8]. miR-24通过抑制靶基因RegⅣ的活性, 限制GC细胞的增殖及侵袭[9]. miR-378能增强恶性胶质瘤细胞存活, 降低Caspase3的活性, 从而促进肿瘤细胞的生长及血管形成[10]. 本课题采用miRNA表达谱芯片技术, 筛选胃癌(gastric cancer, GC)高转移细胞株RF48及低转移细胞株RF1的差异表达miRNA, 以发现可能参与调控GC细胞转移相关的miRNA, 并对差异表达miRNA-210的生物学功能进行生物信息学分析, 为阐明miRNA在GC细胞转移过程中的调控机制奠定基础.
人GC细胞株MGC803、BGC823、KATO Ⅲ、NCI-N87、RF48、MNK28及RF1由本课题组保存. 7株GC细胞株中, RF-1和RF-48分别为同一个患者的原发灶和转移灶, MNK28来自GC原发灶, MGC803、BGC823均来自低分化胃腺癌[11,12], KATO Ⅲ、NCI-N87分别来自GC的淋巴结和肝脏的转移灶, 因此, RF-1和MNK28为低转移细胞株, MGC803、BGC823、KATO Ⅲ、NCI-N87和RF48均为高转移细胞株. 本课题利用具有强转移对比性的RF48及RF1进行miRNA芯片表达谱的筛选, 采用Taqman荧光定量RT-PCR检测miRNA-210在高转移细胞株(MGC803、BGC823、KATO Ⅲ、NCI-N87、RF48)和低转移细胞株(MNK28和RF1)中的表达情况, 且对芯片结果进行验证. BGC823、MNK28用含有10%胎牛血清的1640培养液进行培养, 其余5株细胞株均用含10%胎牛血清的DMED培养液进行培养, 在37 ℃、95%湿度、50 mL/L CO2条件下生长, 取生长状态良好的细胞进行后续实验.
1.2.1 细胞总RNA的提取及质量检测: 取生长状态良好的RF48及RF1细胞株按每毫升1×104个细胞接种于25 cm2培养瓶, 离心后弃上清液, 加TRIzol试剂(Invitrogen), 48 h后利用TRIzol抽取总RNA, 步骤按照说明书进行. 利用1%琼脂糖凝胶电泳检测总RNA的完整性, 测定A260/280比值检测RNA纯度, RNA产物置-80 ℃保存, 备用.
1.2.2 miRNA芯片实验和数据分析: 取约100 ng的总RNA, 利用小分子RNA的分离试剂盒(Ambion's miRNA Isolation Kit)进行小分子RNA的分离, 利用小分子RNA的Cy3荧光标记试剂盒(Agilent's miRNA Complete Labeling and Hyb Kit)将小分子RNA进行3-pCp荧光标记(Agilent, Santa Clara, CA, USA). 将标记好的小分子RNA与miRNA芯片进行杂交反应, 芯片采用美国Agilent公司的Human microRNA Microarray(V2)对样品进行杂交. Human microRNA Microarray (V2)芯片为Human miRNA芯片, 含有723条人microRNA. 每张miRNA芯片中包含有至少1500个特异性探针、对照探针以及无探针的空白对照. 杂交后的芯片应用Agilent Microarray Scanner(Agilent, version A.7.0.1)进行扫描分析. miRNA相对表达量大于或等于对照组2倍的为差异基因筛选标准.
1.2.3 荧光实时定量RT-PCR反应: 取100 ng总RNA, 应用TaqMan miRNA Reverse Transcription Kit逆转录合成cDNA, 再进行定量PCR检测. 25 μL反应体系, PCR条件如下: 95 ℃ 15 s, 60 ℃ 30 s, 72 ℃ 3 s, 共35个循环. U6 RNA作为内参照, 所有样品设3次重复.
1.2.4 差异表达miRNA生物信息学分析: 首先应用miRWalk数据库(http://www.umm.uni-heidelberg.de/apps/zmf/mirwalk/index.html)获得差异表达miRNA-210的靶基因. miRWalk数据库集成了DIANA-mT、miRanda、miRDB、RNAhybrid、PICTAR4、PICTAR5、PITA、RNA22、TargetScan及miRWalk等10大miRNA靶基因的软件预测数据库, 并且涵盖了来源于实验验证后及文献报导的"Experimental Validated"数据, 收录了包括miRNA靶基因以及miRNA与信号通路、遗传性的基因疾病疾病及组织特异性表达相关的数据信息, 是一个miRNA相关的综合数据库. 本研究中利用Experimental Validated数据获得miRNA-210的靶基因, 随后利用David数据库对miRNA-210靶基因进行GO(Gene ontology)分析及KEGG(kyoto encyclopedia of genes and genomes)信号通路分析.
统计学处理 通过2-△△Ct方法分析miRNA的相对基因表达量, △Ct sample = Ct sample - Ct U6 sample, △Ct control = Ct control - Ct U6 control, △△Ct = △Ct sample-△Ct control, 通过2-△△Ct获得相对基因表达差异的倍数. 所有数据均采用统计软件SPSS13.0处理, 以mean±SD表示, 组间两两比较采用t检验. P<0.05为差异具有统计学意义.
miRNA芯片筛选结果显示, 与RF1细胞株相比, RF48细胞株中共有21个miRNA表达上调, 15个miRNA表达下调, 其中miRNA-210在高转移细胞株RF48中的表达水平高于低转移细胞株RF1 3.359倍(表1), 且miRNA-210在GC中表达及机制研究在国内尚未见报道, 因此, 本课题后续验证了miRNA-210的表达水平, 并对其功能进行了生物信息预测.
| MicroRNA | RF48/RF1 | MicroRNA | RF48/RF1 |
| Up regulaiton | Fold change | Down regulaiton | Fold change |
| miRNA-193 | 5.133 | miRNA-455 | 0.065 |
| miRNA-30b | 4.632 | miRNA-196b | 0.187 |
| miRNA-194 | 4.513 | miRNA-744-3p | 0.355 |
| miRNA-551b | 3.866 | miRNA-139-3p | 0.365 |
| miRNA-215 | 3.559 | miRNA-885 | 0.386 |
| miRNA-2101 | 3.359 | miRNA-630 | 0.389 |
| miRNA-301a | 3.305 | miRNA-196a | 0.412 |
| miRNA-33a | 3.205 | miRNA-432 | 0.417 |
| miRNA-192 | 3.038 | miRNA-1227 | 0.447 |
| miRNA-100 | 2.871 | miRNA-223 | 0.451 |
| miRNA-23b | 2.757 | miRNA-30b* | 0.454 |
| miRNA-145 | 2.742 | miRNA-197 | 0.455 |
| miRNA-342 | 2.708 | miRNA-181d | 0.470 |
| hsa-let-7e | 2.405 | miRNA-574 | 0.487 |
| miRNA-192 | 2.189 | hsa-let-7b | 0.499 |
| miRNA-30a | 2.160 | ||
| miRNA-324 | 2.110 | ||
| miRNA-425 | 2.072 | ||
| miRNA-27b | 2.034 | ||
| miRNA-24 | 2.032 | ||
| miRNA-125a | 2.005 |
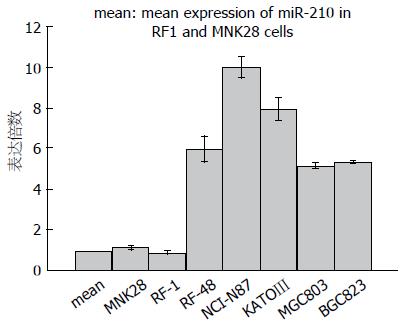
通过RT-PCR检测miRNA-210在GC细胞株MGC803、BGC823、KATO Ⅲ、NCI-N87、RF48、MNK28及RF1中的表达情况, 且对芯片结果进行验证. 结果表明miRNA-210在高转移细胞株(MGC803、BGC823、KATO Ⅲ、NCI-N87、RF48)中的表达明显高于低转移细胞株MNK28及RF1(MNK28和RF1的表达量的均数及各自分别表达量), 差异具有统计学意义(P<0.05)(图1), 与芯片结果一致.
首先应用miRWalk数据库的Experimental Validated数据萃取并整合miRNA-210的靶基因, 结果显示共有155个相关靶基因. 进一步将获得的靶基因输入David数据库, 分别进行靶基因的GO聚类分析及KEGG通路的预测. GO聚类分析结果显示, 相关靶基因的分子功能(molecule function, MF)、生物学过程(biological progress, BP)和细胞组分(cellular component, CC)分别得到51条、407条及31条相关注释描述, 其主要功能与细胞的增殖、凋亡、坏死及有丝分裂等有关, 参与细胞器的膜及胞质内囊泡的形成, 且与膜受体蛋白络氨酸激酶活力、酶的结合及转录因子的结合等密切相关(表2).
| 名称 | 基因数 | P值 | 富集倍数 |
| GOTERM_BP_FAT n = 407 | |||
| GO:0042127-regulation of cell proliferation | 29 | 1.85×10-11 | 4.491 |
| GO:0043066-negative regulation of apoptosis | 17 | 2.58×10-8 | 5.853 |
| GO:0043069-negative regulation of programmed cell death | 17 | 3.13×10-8 | 5.771 |
| GO:0060548-negative regulation of cell death | 17 | 3.26×10-8 | 5.755 |
| GO:0051329-interphase of mitotic cell cycle | 10 | 1.48×10-7 | 11.832 |
| GOTERM_CC_FAT n = 31 | |||
| GO:0031967-organelle envelope | 12 | 0.005 | 2.660 |
| GO:0031975-envelope | 12 | 0.005 | 2.652 |
| GO:0031410-cytoplasmic vesicle | 12 | 0.006 | 2.569 |
| GO:0031982-vesicle | 12 | 0.008 | 2.462 |
| GO:0030141-secretory granule | 6 | 0.009 | 4.581 |
| GOTERM_MF_FAT n = 51 | |||
| GO:0004714-transmembrane receptor protein tyrosine kinase activity | 6 | 1.95×10-4 | 11.073 |
| GO:0019899-enzyme binding | 14 | 2.70×10-4 | 3.310 |
| GO:0008134-transcription factor binding | 13 | 8.06×10-4 | 3.133 |
| GO:0004672-protein kinase activity | 14 | 0.001 | 2.857 |
| GO:0043566-structure-specific DNA binding | 7 | 0.001 | 5.969 |
KEGG信号通路分析[P<0.05, FDR(false discovery rate)<0.05]结果显示, miRNA-210的靶基因可能参与多种肿瘤的发生发展及转移等过程, 例如: 前列腺癌、膀胱癌、胰腺癌、子宫内膜癌及结直肠癌等, 且与ErbB信号通路关系密切(表3).
| 名称 | 百分比(%) | P值 | 富集倍数 | FDR值 |
| hsa05200: Pathways in cancer | 16.40 | 5×10-9 | 5.167 | 5.769×10-6 |
| hsa05215: Prostate cancer | 9.48 | 3×10-8 | 11.026 | 3.263×10-5 |
| hsa05219: Bladder cancer | 6.90 | 3×10-7 | 16.992 | 0.293 |
| hsa04012: ErbB signaling pathway | 8.62 | 3×10-7 | 10.254 | 0.356×10-3 |
| hsa05212: Pancreatic cancer | 7.76 | 9×10-7 | 11.151 | 0.984×10-3 |
| hsa05213: Endometrial cancer | 6.90 | 10-6 | 13.724 | 0.133×10-2 |
| hsa05220: Chronic myeloid leukemia | 7.76 | 10-6 | 10.705 | 0.135×10-2 |
| hsa05210: Colorectal cancer | 6.90 | 3×10-5 | 8.4962 | 0.034 |
肿瘤的侵袭和转移是一个多因素、 多步骤、连续的复杂过程, miRNA芯片技术是一种高通量、集成化、自动化的检测手段, 能在同一时间对多个miRNA进行检测, 可在仅有微量样品的条件下, 实现同时对成百上千条基因的研究, 并且现已应用在基因表达检测、基因功能研究、疾病相关的生物标志等多个研究领域. 本研究中, 我们应用miRNA芯片技术结合生物信息学相关方法探讨GC在发生、进展、转移过程中的miRNA差异表达的变化趋势, 试图寻找出GC转移过程中的关键miRNA及其相互作用的靶基因和相互作用的通路.
在本研究中, 首先对高转移GC细胞株RF48及低转移GC细胞株RF1中差异表达的miRNA进行了表达谱芯片筛选, 得到21个表达上调miRNA, 15个表达下调miRNA, 然而目前, 国内尚未见关于GC中miRNA表达谱的文献报道, 因此本课题为GC的转移相关的细胞表达谱奠定了基础(表1). 差异表达的miRNA中, miRNA-210在RF48中的表达增高3.359倍, 加之目前关于miRNA-210在GC中报道较少, 因此, 本课题将对miRNA-210在GC细胞中的表达情况及生物学功能等方面进行进一步研究.
miRNA-210在实体瘤中研究较多, 在多种恶性实体瘤中的表达上调, 例如乳腺癌、非小细胞肺癌、头颈部癌、胰腺癌、口腔癌、肝细胞癌(hepatocellular carcinoma, HCC)、肾上腺皮质癌(adrenal cortical carcinoma, ACC)、结肠癌、卵巢癌、恶性胶质瘤及肾细胞癌等[13,14], 但在食管癌鳞癌尤其在低分化食管癌中, 表达明显下调[15]. 通过研究miRNA-210在乳腺癌中的表达情况, 结合患者的生存随访的调查, 结果显示miRNA-210在肿瘤中的表达是一个独立的危险因素, 表达越高患者的预后越差, 生存率越低[16]. 同样在乳腺癌中, Camps等[17]也发现, miRNA-210的表达与无疾病存活率及总生存率呈现负相关, 随着miRNA-210表达的增高, 患者的无疾病存活率及总生存率则越低. 本研究利用TagMan法RT-PCR对miRNA-210在7种GC细胞株中的表达, 结果表明, 在高转移细胞株MGC803、BGC823、KATO Ⅲ、NCI-N87、RF48中miR-210呈高表达, 差异具有统计学意义(P<0.05), 验证了其在芯片中的表达情况, 与芯片结果一致. 目前, 关于GC中miRNA-210的文献报道国内仅1篇, 刘福囝等[18]采用实时定量RT-PCR检测miRNA-210在60例GC组织中的表达, 结果显示miRNA-210在GC组织中表达明显增高, 且miRNA-210的表达与淋巴结转移、远处转移等密切相关, 提示miRNA-210在GC的发生发展及转移过程中发挥了重要作用. 最新研究[19]表明, miRNA-210在结直肠癌组织中的表达显著增高, 在与临床病理指标相关性分析中发现, 其表达与淋巴结转移等密切相关, 即miRNA-210表达越高, 预示预后越差, 且在结直肠癌的高转移细胞株中表达明显高于低转移细胞株, 从而促进结直肠癌细胞的侵袭和转移. 综合本研究结果及相关文献报道, 我们提出miRNA-210与GC的转移关系密切, 可能成为GC转移的早期诊断标志物.
由于miRNA通过对靶基因的表达调控而发挥作用, 因此我们对miRNA-210的155个靶基因进行GO分析, 从而探讨miRNA-210在GC转移中的作用机制. 利用Daivd数据库分析得知, miRNA-210靶基因的主要功能集中在细胞的增殖、凋亡、坏死及有丝分裂等方面. 有文献报道, miRNA-210通过诱导细胞的死亡及细胞周期捕获, 在G1/G0及G2/M期来抑制肿瘤细胞的增殖及存活[20]. 此外, 这些靶基因参与了细胞器膜及胞质内囊泡的形成, 且与膜受体蛋白络氨酸激酶活力、酶的结合及转录因子的结合等相关方面的功能密切相关(表2). 同时通过David数据库预测miRNA-210相关靶基因可能参与的KEGG信号通路, 由于本研究中选取P<0.05、FDR<0.05的信号通路, 因此预测信号通路具有较强的可信性.
KEGG通路结果显示, miRNA-210的靶基因参与了众多的肿瘤相关通路, 例如, miRNA与前列腺癌或结直肠癌相关的通路等, 提示miRNA-210通过其靶基因参与了多种肿瘤的发生发展及侵袭转移. KEGG通路结果还显示miRNA-210的靶基因参与ErbB信号通路, ErbB通路通过ErbB3与受体酪氨酸激酶(receptor tyrosine kinases, RTKs)相结合, 并且被ErbB2等磷酸化, 从而激活包括AKT, MEK/MAPK及Jak/Stat通路等一系列致癌通路. 在细胞增殖、分化、凋亡等过程中, 信号通路中任何组分的变化都会导致细胞的恶性增生, 发生癌变, 尤其是RTKs系统, 调控着其下游多个信号通路[21]. 因此, miRNA-210的靶基因可能通过ErbB信号通路促进GC等多种肿瘤的发生及转移.
总之, 本研究通过miRNA表达谱芯片筛选了GC高转移和低转移细胞株之间的差异表达miRNA表达谱, 共筛选出36个表达失调的miRNA, 为GC转移相关的miRNA表达谱的建立奠定了基础. 此外, 本课题通过对miRNA-210的表达检测及生物学功能的信息学分析, 提出miRNA-210在肿瘤的发生、发展、侵袭及转移中起到一定的作用, 且有望成为GC转移相关的特异性miRNA, 从而为GC转移的早期临床诊断及治疗提供了新的研究靶点, 具有潜在的应用前景.
胃癌是一种严重威胁人类健康的恶性肿瘤, 且发病率逐年上升, 目前以手术为主, 但总体疗效不佳. 本课题利用微小RNA(microRNA, miRNA)表达谱芯片筛选胃癌(gastric cancer, GC)转移相关的差异表达miRNA, 探讨差异表的miRNA miRNA-210与GC转移的可能机制, 为胃癌的基础研究提供新的研究靶点.
黄颖秋, 教授, 本溪钢铁(集团)总医院消化内科
miRNA具有转录后调控作用的非编码RNA, 通过与靶基因的结合, 直接降解或抑制靶基因的mRNA的表达而发挥作用, 越来越多的研究表明miRNA参与基因的表达调控, 与肿瘤的发生发展密切相关而成为当前的研究热点.
国外研究发现多种miRNA在胃癌组织中异常表达, 且在胃癌的发生过程中多种的miRNA通过不同途径发挥着各自的重要作用, 但关于胃癌中差异表的miRNA生物信息预测报道鲜见.
本文作者针对胃癌的miRNA表达谱进行研究, 发现胃癌有特异miRNA表达谱, 且利用生物信息技术队差异miRNA进行功能预测, 具有重要的创新价值.
对miRNA的深入研究, 将有助于对胃癌发病机制的理解, 对差异表达miRNA miRNA-210的生物信息的预测将为其作为肿瘤治疗的新的靶位点提供理论依据, 为胃癌的诊治提供了新的思路.
Gene ontology: GO注释分析, 即基因本体论分析, 也就是gene ontology annotation, 按照了三个大的标准(相当于前面所说的属性)将每个基因聚类(分别是根据基因的功能, 参与代谢的过程, 以及这个基因产物的定位)进行分析;
KEGG(kyoto encyclopedia of genes and genomes): 是系统分析基因功能、基因组信息数据库, 他有助于研究者把基因及表达信息作为一个整体网络进行研究.
本文内容全面, 可读性较好, 有利于读者对miRNA在胃癌中的作用全面了解.
编辑: 郭鹏 电编:都珍珍
| 1. | Hutvágner G, Zamore PD. A microRNA in a multiple-turnover RNAi enzyme complex. Science. 2002;297:2056-2060. [PubMed] |
| 3. | Corcoran C, Friel AM, Duffy MJ, Crown J, O'Driscoll L. Intracellular and extracellular microRNAs in breast cancer. Clin Chem. 2011;57:18-32. [PubMed] [DOI] |
| 4. | Murakami Y, Yasuda T, Saigo K, Urashima T, Toyoda H, Okanoue T, Shimotohno K. Comprehensive analysis of microRNA expression patterns in hepatocellular carcinoma and non-tumorous tissues. Oncogene. 2006;25:2537-2545. [PubMed] |
| 5. | Wang YX, Zhang XY, Zhang BF, Yang CQ, Chen XM, Gao HJ. Initial study of microRNA expression profiles of colonic cancer without lymph node metastasis. J Dig Dis. 2010;11:50-54. [PubMed] [DOI] |
| 6. | Hong L, Han Y, Li S, Yang J, Zheng J, Zhang H, Zhao Q, Wu K, Fan D. The malignant phenotype-associated microRNA in gastroenteric, hepatobiliary and pancreatic carcinomas. Expert Opin Biol Ther. 2010;10:1693-1701. [PubMed] [DOI] |
| 7. | 苏 娟, 张 庆瑜, 康 春生, 张 安玲, 王 涛, 张 洁. miR-200a通过β-catenin/TCF-4 信号通路抑制胃癌细胞系生长侵袭能力的体外研究. 中国肿瘤临床. 2012;39:65-69. |
| 8. | Noonan EJ, Place RF, Pookot D, Basak S, Whitson JM, Hirata H, Giardina C, Dahiya R. miR-449a targets HDAC-1 and induces growth arrest in prostate cancer. Oncogene. 2009;28:1714-1724. [PubMed] [DOI] |
| 9. | Duan Y, Hu L, Liu B, Yu B, Li J, Yan M, Yu Y, Li C, Su L, Zhu Z. Tumor suppressor miR-24 restrains gastric cancer progression by downregulating RegIV. Mol Cancer. 2014;13:127. [PubMed] [DOI] |
| 10. | Lee DY, Deng Z, Wang CH, Yang BB. MicroRNA-378 promotes cell survival, tumor growth, and angiogenesis by targeting SuFu and Fus-1 expression. Proc Natl Acad Sci U S A. 2007;104:20350-20355. [PubMed] |
| 13. | Devlin C, Greco S, Martelli F, Ivan M. miR-210: More than a silent player in hypoxia. IUBMB Life. 2011;63:94-100. [PubMed] [DOI] |
| 14. | McCormick R, Buffa FM, Ragoussis J, Harris AL. The role of hypoxia regulated microRNAs in cancer. Curr Top Microbiol Immunol. 2010;345:47-70. [PubMed] [DOI] |
| 15. | Tsuchiya S, Fujiwara T, Sato F, Shimada Y, Tanaka E, Sakai Y, Shimizu K, Tsujimoto G. MicroRNA-210 regulates cancer cell proliferation through targeting fibroblast growth factor receptor-like 1 (FGFRL1). J Biol Chem. 2011;286:420-428. [PubMed] [DOI] |
| 16. | Toyama T, Kondo N, Endo Y, Sugiura H, Yoshimoto N, Iwasa M, Takahashi S, Fujii Y, Yamashita H. High expression of microRNA-210 is an independent factor indicating a poor prognosis in Japanese triple-negative breast cancer patients. Jpn J Clin Oncol. 2012;42:256-263. [PubMed] [DOI] |
| 17. | Camps C, Buffa FM, Colella S, Moore J, Sotiriou C, Sheldon H, Harris AL, Gleadle JM, Ragoussis J. hsa-miR-210 Is induced by hypoxia and is an independent prognostic factor in breast cancer. Clin Cancer Res. 2008;14:1340-1348. [PubMed] [DOI] |
| 19. | Qu A, Du L, Yang Y, Liu H, Li J, Wang L, Liu Y, Dong Z, Zhang X, Jiang X. Hypoxia-inducible MiR-210 is an independent prognostic factor and contributes to metastasis in colorectal cancer. PLoS One. 2014;9:e90952. [PubMed] [DOI] |
| 20. | Kim HW, Haider HK, Jiang S, Ashraf M. Ischemic preconditioning augments survival of stem cells via miR-210 expression by targeting caspase-8-associated protein 2. J Biol Chem. 2009;284:33161-33168. [PubMed] [DOI] |
| 21. | Emre H, Tekin SB, Doğan H, Çayır K, Bilici M, Sipal S, Pirim I. Investigation of ErbB-2 overexpression on patients with gastric cancer in Eastern Anatolia of Turkey. Turk J Gastroenterol. 2013;24:211-217. [PubMed] |