修回日期: 2014-04-14
接受日期: 2014-04-24
在线出版日期: 2014-06-08
目的: 将来自中国不同地区不同民族乙型肝炎检测"大三阳"学生的外周血乙型肝炎病毒(hepatitis B virus, HBV)进行基因分型并探讨不同方法对HBV基因分型的效果进行比较, 建立较为客观精确的HBV基因分子分型方法和标准.
方法: 反映HBV-S区的型特异性的聚合酶链式反应-限制性酶切片断长度多态性(polymerase chain reaction- restriction fragment length polymorphism, PCR-RFLP)方法进行基因分型; 依据HBV-S区的型特异性的PCR扩增片段大小直接进行分型; 通过对NCBI数据库中共计220个条目已确认B、C、D型的HBV-S基因DNA序列的同源性比对, 构建一个以HBV的S区DNA序列分子进化树为基础的基因分型标准. 对待检样品S区的PCR产物进行DNA测序并与标准进化树的DNA序列进行比对并重新构建进化树而进行HBV基因分型.
结果: 在共计216例乙型肝炎检测"大三阳"(乙型肝炎两对半检查中, HBsAg、HBeAg和HBcAb均为阳性)样品中, 第1种分型方法鉴定出: B型155例、C型41例、D型7例, 尚有13例不能确定是C型还是D型. 进一步经第2种分型方法确认其中10例未分型的均为C型, 余3例不能确定是C型还是D型. 经测序、比对及构建分子进化树, 测序的30例中与PCR-RFLP分型特别是亚型的结论多不相同. 并且首次在国内发现3例HBV-G型(为广西壮族和瑶族男性).
结论: 3种方法比较各有优劣. PCR-RFLP法较为精细可以分到亚型, 但该法的缺陷是不能区分C2型和D2型. 根据PCR片段直接分型法较为快捷简便, 但引物特异性不高, 分型不准确. 将待检HBV-S基因DNA序列导入标准进化树的分型方法较为准确客观, 是HBV基因分型的"金标准". 但针对HBV的普遍变异和种群异质性, 需要建立HBV-S的蛋白分型标准.
核心提示: 为选择合适乙型肝炎病毒(hepatitis B virus, HBV)基因分型方法, 分别用针对HBV-S区基因的聚合酶链式反应-限制性酶切片断长度多态性(polymerase chain reaction-restriction fragment length polymorphism)法、型特异性PCR片段直接法和DNA测序结合标准进化树法进行基因分型. 因HBV的普遍变异和种群异质性, 需建立HBV-S的蛋白分型标准. 以期确立客观、精确、简便的HBV分型方法.
引文著录: 李可可, 刘莹, 姚品芳, 陈真, 王红, 李劲. 中国人群乙型肝炎病毒基因分型不同方法和标准的比较. 世界华人消化杂志 2014; 22(16): 2306-2316
Revised: April 14, 2014
Accepted: April 24, 2014
Published online: June 8, 2014
AIM: To genotype hepatitis B virus (HBV) in college students who tested positive HBsAg, HBeAg, and HBcAb using three different methods and compare the advantages and disadvantages of these genotyping methods.
METHODS: According to the specificity of HBV-S, polymerase chain reaction-restriction fragment length polymorphism (PCR-RFLP) or direct genotyping based on the length of PCR amplified fragment were used for HBV genotyping. By means of homology alignment of 220 items of HBV-S gene sequences with HBV-B/C/D type identified in NCBI database, a molecular evolutionary tree as a genotyping standard has been constructed. The test samples were genotyped by carrying out HBV-S gene PCR amplification, sequencing, alignment and constructing an HBV-S region evolutionary tree together with the standard phylogenic tree.
RESULTS: In a total of 216 HBV positive samples, the genotyping result of PCR-RFLP was: genotype B, 155; genotype C, 41; genotype D, 7; Unknown, 13. Of these unknown cases, 10 were identified as genotype C by the direct genotyping method based on the length of PCR amplified fragment, and 3 were still determined. The genotyping results for 30 selected samples were substantially different between the method of the sequencing-evolutionary tree and PCR-RFLP. Three cases of HBV genotype G, which come from the men of Zhuang and Yao nationalities in Guangxi, were first discovered in China using the sequencing-evolutionary tree method.
CONCLUSION: The three genotyping methods have different advantages and disadvantages: PCR-RFLP can distinguish between sub-genotypes, but can not distinguish C2 and D2; direct genotyping method based on PCR amplified fragment size is simple and direct, but its accuracy and specificity are not high; the sequencing-evolutionary tree method is accurate and specific, representing a "golden" standard for HBV genotyping.
- Citation: Li KK, Liu Y, Yao PF, Chen Z, Wang H, Li J. Comparison of different genotyping methods for hepatitis B virus in a Chinese population. Shijie Huaren Xiaohua Zazhi 2014; 22(16): 2306-2316
- URL: https://www.wjgnet.com/1009-3079/full/v22/i16/2306.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v22.i16.2306
病毒性乙型肝炎是由乙型肝炎病毒(hepatitis B virus, HBV)的感染引起的, 是一种流行范围广、危害程度大的世界性传染病. 据WHO报道, 全球60亿人口中, 约20亿人曾感染过HBV, 其中3.5亿例是慢性HBV感染者, 在感染乙型肝炎的患者中约1/4数量的患者将产生慢性肝炎、肝硬化和原发性肝细胞癌, 全球层面上统计, 每年大约有100万例死于感染HBV后继发导致的肝脏衰竭、肝硬化和原发性肝癌及其并发症, 如肝源性糖尿病、心血管病变等[1-5]. 与世界上其他国家相比, 在我国乙型肝炎感染率属全球第1位, 根据1992年乙型肝炎血清流行病调查数据显示, 一般人群乙型肝炎病毒表面抗原携带率曾高达9.75%, 2006年乙型肝炎血清流行病学的数据降为7.18%[6]. 在传统血清免疫学检测中, HBV的分型主要取决于外膜蛋白HBsAg的抗原性不同. 1988年Okamoto等[7]通过对18株不同亚型的HBV DNA全序列比较后发现, 血清亚型的区分并不能真正反映HBV基因组的差异. 随后的研究证明, HBV基因型比血清型更具有更广泛的应用价值. Norder等[8]通过对HBV全序列分析发现, 在不同基因型之间S区段异质性最大, 而相同基因型内S区段的异质性最小, 从而可以用S区代替全序列进行基因分型. 因此依据HBV的全基因组序列或者S区同源性将HBV分为了A-I 9种基因型. HBV的基因型是由漫长持续的病毒突变和宿主的不断筛选累积形成的结果. HBV基因型的分布与地域和种族呈明显的正相关性[9,10]. 我国的乙型肝炎主要有B、C、D 3种型别[11]. 南方地区B型占优势[12], 北方则以C型为主[13], 西藏地区以D型为主[14]. 目前的研究显示, 不同基因型HBV在流行病学特征、病毒变异、疾病表现和治疗应答等方面均存在着差异, HBV的基因分型研究对进一步明确乙型肝炎发病机制、提高检测效率、寻找治疗对策和实现流行病学控制具有非常重要的意义[15,16]. 为比较和选择合适的HBV基因分型方法, 我们分别采用了针对HBV-S区基因的聚合酶链式反应-限制性酶切片断长度多态性(polymerase chain reaction-restriction fragment length polymorphism, PCR-RFLP)法、型特异性PCR片段直接分型法和DNA测序结合标准进化树法进行基因分型. 由于HBV的普遍变异和种群异质性, 需要建立HBV-S的蛋白分型标准. 通过对上述分型方法的全面比较, 以期确立客观、精确、简便的HBV分型方法.
血样来源: 某高校来自广西、湖南、广东、湖北、云南、福建、山西、山东、重庆、四川、江西、吉林、宁夏、黑龙江、浙江、贵州、江苏17个省市的不同民族大学生检测为乙型肝炎大三阳的血样共216例. 其中汉族107例、壮族36例、瑶族13例、畲族11例、回族10例、土家族8例、苗族8例、黎族4例、蒙古族2例、布依族2例、侗族2例、仡佬族2例、彝族2例、满族2例、仫佬族1例、朝鲜族1例、白族1例、土族2例、东乡族1例、毛南族1例. 男女比例为114:102(1.117:1).
1.2.1 HBV的DNA提取: 直接煮沸法[17], 50 µL血清直接煮沸10 min, 12000 r/min离心5 min, 取上清备用.
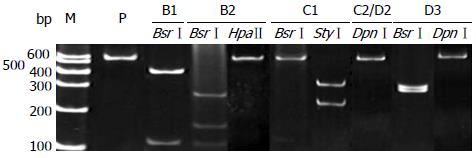
1.2.2 PCR-RFLP: 经巢式PCR[18]扩增出585 bp的DNA片段, 然后按照表1中BsrⅠ→StyⅠ→DpnⅠ→HpaⅡ的顺序进行酶切. Outer primer: PrsS2(sense, 2820-2837 nt, 5'-GGGACACCATATTCTTGG-3'), S1R(antisense, 842-821 nt, 5'-TTAGGGTTTAAATGTATACCCA-3'); Inner primer: YS1(sense, 203-221 nt, 5'-GCGGGGTTTTTCTTGTTGA-3'), YS2(antisense, 787-767 nt, 5'-GGGACTCAAGATGTTGTACAG-3'). 第1次PCR: PCR反应混合液Ⅰ: 15.5 µL ddH2O, 2.5 µL 10×Taq buffer, 1.5 µL dNTP(1 mmol/L), 0.5 µL PrsS2-S(1 nmol)和0.5 µL S1R-AS(1 nmol), 1.5 µL Taq酶(0.25 U), 3 µL HBV DNA提取液. 循环参数为: 94 ℃预变性300 s, 94 ℃变性45 s、58 ℃退火60 s、72 ℃延伸45 s, 扩增30个循环, 72 ℃终延伸300 s; 第2次PCR: PCR反应混合液Ⅱ: 15.5 µL ddH2O, 2.5 µL 10×Taq buffer, 1.5 µL dNTP(1 mmol/L), 0.5 µL YS1-S(1 nmol)和0.5 µL YS2-AS(1 nmol), 1.5 µL Taq酶(0.25 U). DNA: 取第1次PCR产物3 µL加入到上述PCR混合液Ⅱ中, 配成25 µLPCR体系, 混匀, 10000 r/min瞬时离心. PCR循环参数为: 94 ℃预变性300 s, 94 ℃变性45s、58 ℃退火60 s、72 ℃延伸45 s, 扩增30个循环, 72 ℃终延伸300 s.
| HBV基因型 | HBV亚型 | 酶切片段大小(bp) | |||
| BsrⅠ | StyⅠ | DpnⅠ | HpaⅡ | ||
| B | B1 | 126, 459 | 585 | 289, 296 | 585 |
| B2 | 126, 184, 275 | 585 | 289, 296 | 585 | |
| C | C1 | 585 | 253, 332 | 585 | 585 |
| C2 | 585 | 585 | 585 | 585 | |
| D | D1 | 585 | 585 | 289, 296 | 585 |
| D2 | 585 | 585 | 585 | 585 | |
| D3 | 300, 285 | 585 | 585 | 585 | |
1.2.3 依据HBV-S区的型特异性PCR片段直接分型法进行分型: 利用3'端的差异来设计相对应的引物利用扩增片段的长度来区分B、C、D型[19]. 其中B型引物为N1(S): 5'-GCCTCCAAGCTGTGCCTTG-3'(1868-1886 nt), N1(A): 5'-GGGGCCCCACAAATTGCTT-3'(2601-2583 nt); C、D型引物: N2(S): 5'-CCTGCTGGTGGCTCCAGT-3'(56-73 nt), C: N2(A): 5'-CGAATTTTGGCCAGGACAAGT-3'(313-293 nt), D: N2(A): 5'-TGTGATCTTGTGGCAATGACCCAT-3'(922-899 nt). B型产物为734 bp, C型产物为258 bp, D型产物为867 bp. PCR反应体系: 17.5 µL ddH2O, 2.5 µL 10×Taq buffer, 1.5 µL dNTP(1 mmol/L), 0.5 µL正向引物(10 µmol/L)和0.5 µL反向引物(10 µmol/L), 1.5 µL Taq酶(0.25 U), 1 µL HBV DNA模板. PCR反应程序: 94 ℃预变性300 s; 94 ℃变性60 s; (B型)68 ℃退火60 s, (C型)68 ℃退火60 s, (D型)55 ℃退火60 s; 72 ℃ 45 s; 扩增30个循环; 72 ℃终延伸300 s.
1.2.4 改良的型特异PCR片段直接分型法: 依据序列比对结果寻找HBV-S基因保守区的B、C、D碱基差异设计区分B、C、D型的引物, 利用嵌套PCR设计的引物: 长片段引物: sense: 5'-ACCGAATTCCCCTATCTTATCAACACTTCC-3' (2302-2331 nt), antisense: (969-998 nt)5'-ATTCGTCGACATACTTTCCAATCAATAGG-3', 全长1912 bp. B引物: sense: (2925-2946 nt)5'-CGATCATCAGTTGGACCCTGCA-3', antisense: (321-345 nt)5'-CAGGTTGGTGAGTGACTGGAGATTT-3', 总长: 635 bp; C引物: sense: (2926-2946 nt)5'-GATCACCAGTTGGACCCTGCG-3', antisense: (321-345 nt)5'-GAGGTTGGTGAGTGATTGGAGGTTG-3', 总长: 635 bp; D引物: sense: (2743-2767 nt)5'-ATTATTTACACACTCTATGGAAGGC-3', antisense: (3054-3071 nt)5'-CTGAGGGCTCCACCCCAA-3', D型总长311 bp, 而B、C型则为344 bp.
1.2.5 DNA测序-构建进化树法: (1)构建进化树标准: 在NCBI数据库中查询和下载已经确定分型及亚型的HBV的B、C、D型DNA全序列, 截取HBV-S区序列(31-1200 nt)利用Clustal-X软件进行序列比对, 再利用MEGA-5软件构建进化树, 并作为HBV基因分型的标准. 本实验室在NCBI数据库中下载已确定分型的HBV-B、C、D型各亚型的共计220条基因序列(表2). 其中B型(B1、B2、B3、B4、B6、B7); C型(C1、C2、C3、C4、C5、C6、C11、C12); D型(D1、D2、D3、D4、D5、D7). 还包括美国国家生物技术信息中心(NCBI)网页提供的HBV基因分型工具(http://www.ncbi.nlm.nih.gov/projects/genotyping/formpagex.cgi)中的B、C、D、G四型共12条标准基因序列; (2)测序采用S区的PCR产物, 测序引物为5'-GCAACGGGGTAAAGGTTCA-3'(1136-1154 nt), 其中共测序30例, 将其DNA序列或氨基酸序列和B、C、D、G型标准DNA序列或氨基酸序列分别用Clustal-X软件进行序列比对, 并用MEGA-5软件构建进化树. 检查其分型情况; (3)血清型: 根据S基因编码第122位氨基酸(d/y亚型决定簇): 赖氨酸(AAA, d)或精氨酸(AGA, y); 及第160位氨基酸(w/r亚型决定簇): 赖氨酸(w)或精氨酸(r)[20].
| 基因型 | 全基因组ID号 |
| B1(5) | AB602818; AB642091; AB642093; AB642098; AB642101 |
| B2(9) | GQ924653; GQ924610; GQ924608; GQ924603; GQ377569; GQ377558; GQ377547; AB642094; AB555498 |
| B3(10) | GQ358136; GQ924617; GQ924621; GQ924625; GQ924628; GQ924637; GQ924638; GQ924639; GQ924654; GQ924656 |
| B4(7) | AB368295; FJ023632; FJ023633; FJ023634; FJ023635; FJ023636; GQ924626 |
| B6(7) | AB287314; AB287316; AB287318; AB287320; AB287323; DQ463787; DQ463789 |
| B7(13) | AP011088; AP011090; AP011091; AP011092; EF473976; EF473977; GQ358137; GQ358138; GQ358139; GQ358140; GQ358141; GQ358142; GQ358143 |
| C1(15) | GQ377536; GQ924604; GQ924609; GQ924612; GQ924613; GQ924615; GQ924616; GQ924618; GQ924629; GQ924636; GQ924642; GQ924643; GQ924649; GQ924650; GQ924655 |
| C2(42) | EU939538; EU939540; EU939616; EU939652: FJ386575; FJ386576; FJ386587; FJ386588; Fj386609; FJ386639; FJ562218; Fj562232; FJ562233; FJ562244; FJ562282; FJ562318; FJ562332; FJ787464; FJ787465; FJ899761; GQ377514; GQ377517; GQ377523; GQ377527; GQ377528; GQ377531; GQ377533; GQ377535; GQ377540; GQ377551; GQ377553; GQ377554; GQ377559; GQ377563; GQ377572; GQ377576; GQ377598; GQ377600; GQ377611; GQ377616; GQ377617; GQ924633 |
| C3(4) | FJ621762; FJ622627; X75656; X75665 |
| C4(2) | AB048704; AB048705 |
| C5(12) | AB241110; AB241111; AB241113; EU410079; EU410080; EU410081; GQ924620; GQ924622; GQ924657; AP011099; AP011100; AP011101 |
| C6(4) | AB554014; AB554021; AP011102; AP011103 |
| C11(2) | AB554019; AB554020 |
| C12(4) | AB554018; AB554025; AB560661; AB560662 |
| D1(11) | AF280817; EU594396; EU594397; FJ904443; GQ377589; GU456648; JF754591; JF754596; JF754601; JF754624; JF754628 |
| D2(29) | AB330370; EU594399; EU594400; EU594401; EU594402; EU594403; EU594405; EU594406; EU594407; EU594408; EU594409; EU594410; EU594415; EU594416; EU594421; EU594422; EU594423; EU594424; EU594425; EU594426; EU594427; EU594428; EU594430; EU594431; EU594432; EU594433; GU456635; JF754597; JF754621 |
| D3(13) | AB583679; DQ315776; EU594382; EU594434; EU594435; EU594436; FJ692506; FJ692507; GQ922001; GQ922002; HQ236014; HQ236015; HQ236016 |
| D4(6) | FJ692532; FJ692533; FJ692536; GQ922003; GQ922004; GQ922005 |
| D5(9) | GQ205377; GQ205378; GQ205379; GQ205380; GQ205382; GQ205384; GQ205385; GQ205387; GQ205389 |
| D7(16) | FJ904447; FJ904441; FJ904440; FJ904438; FJ904435; FJ904433; FJ904419; FJ904413; FJ904408; FJ904407; FJ904406; FJ904404; FJ904403; FJ904398; FJ904396; FJ904394; |
| NCBI标准型 | B: AF100309; D00329; AB033554 |
| C: AB014381; M12906; X04615 | |
| D: X65259; M32138; X85254 | |
| G: AF160501; AF405706; AB064310 |
按照表1酶切顺序进行酶切: 如图1所示的待分型样品PCR产物首先用BsrⅠ酶切, 若可以切成126、459 bp, 则为B1型; 若可以切成300, 285 bp, 且用HpaⅡ不能切开则为D3型; 若切成126、184、275 bp; 且用HpaⅡ不能切开, 则为B2型. 若不能被BsrⅠ酶切开的, 用StyⅠ酶进行酶切, 可以切成253、332 bp, 则为C1型. 若用4种酶都不能切开的则认为是C2或D2型. 分型结果: B1型151例(汉族78例、壮族17例、瑶族11例、畲族10例、苗族6例、回族例6、土家族8例、布依族2例、东乡族1例、毛南族1例、仡佬族1例、彝族2例、黎族1例、侗族2例、蒙古族1例、白族1例、满族2例、仫佬族1例); B2型4例(汉族3例、壮族1例); C1型41例(汉族20例、壮族12例、黎族2例、回族2例、蒙古族1例、畲族1例、朝鲜族1例、仡佬族1例、土族1例); D3型7例(汉族4例、壮族2例、黎族1例); C2或D2型13例(汉族2例、壮族4例、瑶族2例、回族2例、土族1例、苗族2例), 其中13例是该方法自身的缺陷即C2和D2酶切结果相同, 故不能区分C2和D2.
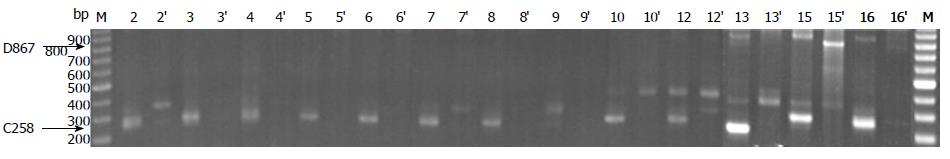
第1种方法未确定C2或是D2基因型的13例, 通过分别用C型和D型引物进行扩增, 用C引物能扩增出258 bp目的片段为C型; 能用D引物扩增出867 bp目的片段的为D型. 如图2所示, 虽然样品3、4、5、6、8、16号可以判断是C型, 样品2、7、10、11、12号按照C引物PCR结果可以判断是C型, 但D引物PCR结果没有其特异带型, 但是有杂带, 也可确定是C型. 样品9、15号不能判断是哪种基因型.
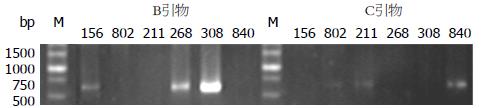
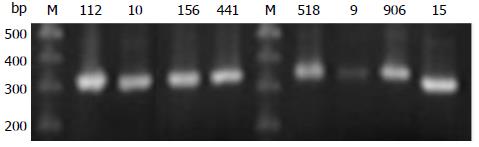
同样的HBV DNA提取液分别用B引物、C引物扩增. 在相对应的引物下可以扩增出来635 bp, 并且在另一种引物扩增下不能扩增出635 bp的便是相应引物的基因型. 如图3所示, 样品156、268、308号是在B引物的扩增下可以扩增出635 bp, 样品802、211、842号未扩增出目的产物, 故样品156、268、308号是B型; 而在C引物的扩增下样品802、211、842号可以扩增出635 bp, 样品156、268、308号未扩增出目的产物, 故样品802、211、842号是C型. HBV DNA提取液可利用C型和D型引物扩增得到不同大小的片段: C型扩增产物为344 bp, D型扩增产物为311 bp. 如图4所示, 样品号为112、10、156、441、518、9、906的DNA提取液的D引物PCR扩增片段为344 bp, 故他们为C型; 样品号为15 的DNA提取液的D引物PCR扩增片段为311 bp, 样品15号为D型.
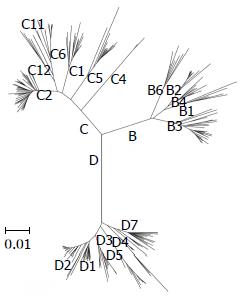
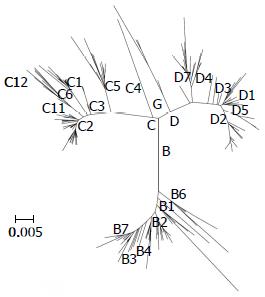
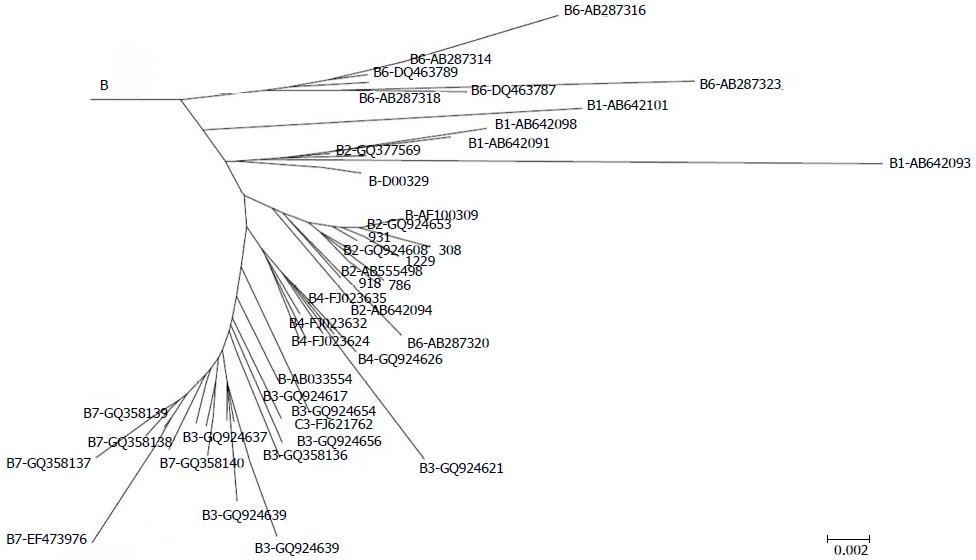
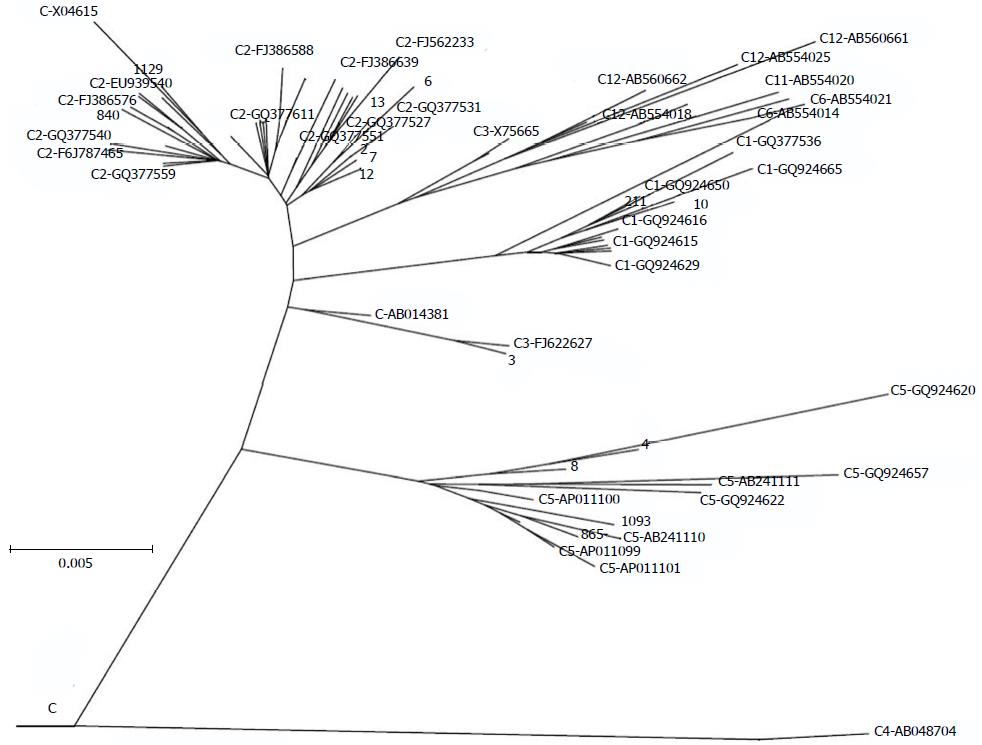
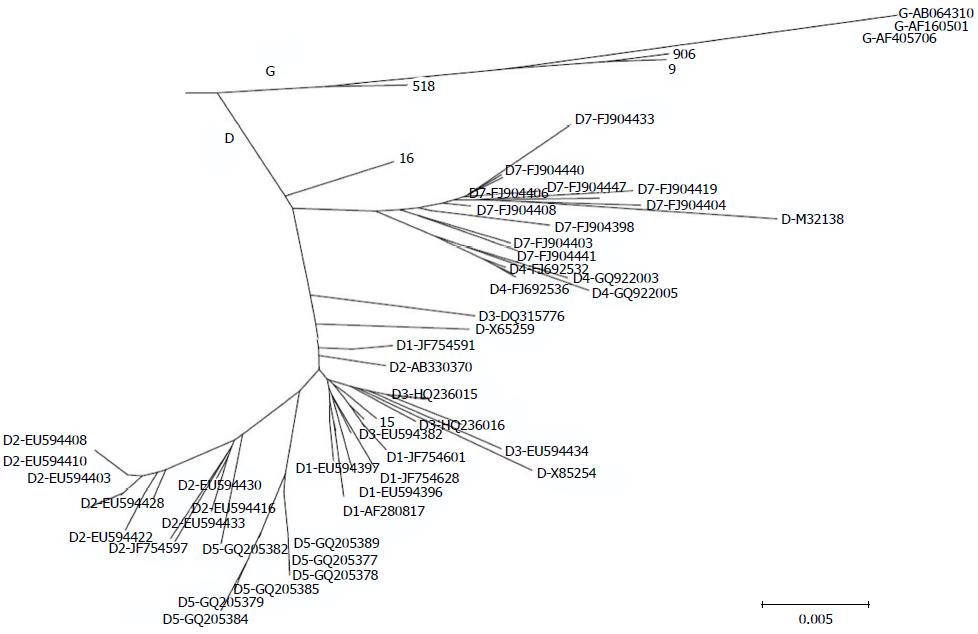
标准进化树如图5所示. 测序产物和标准序列进行比对后构建进化树, 测序产物依照碱基序列的相似度进行聚类(图6), 依据归类到相应的标准基因型或亚型判断测序产物的分型. 如图7所示, 样品441、156、918、931、268、308、1229、786号为DNA测序后代入标准进化树, 被分为B2型. 如图8所示, 样品211、10号为C1型; 样品2、5、6、7、12、13、836、840、1129、802号为C2型; 样品8、865、4、1093号为C5型; 样品3号为C型. 如图9所示, 样品15号为D3型, 样品518、9、906号为G型. 由表3和表4可以看出, PCR-RFLP法和型特异PCR片段直接法的HBV分型(特别是亚型分类)的准确性与视为"金标准"的HBV-S测序法基因分型的差异很大; 基于DNA序列比对而构建进化树的HBV-S-Ag的基因分型与依据同样HBV-S基因序列推定的S-Ag氨基酸序列进行比对而构建进化树的HBV-S-Ag蛋白分型(分型进化树未列出)相比较, 两者在大型分类上大体一致, 而在亚型分类上区别明显. 其中, 通过与NCBI提供的网上分型工具中的B、C、G标准DNA序列(B-D00329、C-AB014381和G-AB064310)比对发现: 样品号为802的病例因为S基因编码区第4位的碱基缺失G导致三联密码移框和第9位的氨基酸密码转变为TAG终止码, 引起S-Ag缺失, 其基因分型仍为C1. 样品号为211的病例(基因分型为C1)的S基因编码区的第13位碱基缺失A, 引发移框突变和第9位氨基酸密码变为TAG终止码, 致使S-Ag缺失. 同样, 基因分型为G的样品号为9的病例的S基因编码区第7位的碱基缺失A, 导致移框突变和第22位氨基酸密码改变为TGA终止码, 致S-Ag缺失. 样品号为918的病例基因分型为B1, 但其S基因编码区第23位插入的碱基A导致移框突变和第23位氨基酸密码改变为TGA终止码, 致S-Ag缺失.
| 病例号 | PCR-RFLP | 型特异PCR | 改良型特异 | S-DNA测序 | S-Ag氨基酸 | 血清型 | 分型结果比较 |
| 2 | C2或D2 | C | B或C | C2 | C | adr | 酶切不确定, PCR定C型, 测序定C2型, 氨基酸序列定C型; 大型一致 |
| 3 | C2或D2 | C | B或C | C | C | adr | 酶切不确定, PCR, S-DNA测序及氨基酸序列均仅定C型; 大型一致 |
| 4 | C2或D2 | C | - | C5 | C51 | adw | 酶切和PCR定C型, S-DNA测序及氨基酸序列均定C5型; 大型一致 |
| 5 | C2或D2 | C | B或C | C2 | C | adr | 酶切不确定, PCR定C型, 测序定C2型, 氨基酸序列定C型; 大型一致 |
| 6 | C2或D2 | C | - | C2 | C | adr | 酶切不确定, PCR定C型, 测序定C2型, 氨基酸序列定C型; 大型一致 |
| 7 | C2或D2 | C | B或C | C2 | C | adr | 酶切不确定, PCR定C型, 测序定C2型, 氨基酸序列定C型; 大型一致 |
| 8 | C2或D2 | C | B或C | C5 | C51 | adw | 酶切不确定, PCR定C型, DNA测序及氨基酸序列均定C5型; 大型一致 |
| 9 | C2或D2 | C2或D2 | - | G | 突变(缺失) | adN2 | 酶切和PCR不确定, 测序定G型, 氨基酸序列突变, S缺失; 分型错误 |
| 10 | C2或D2 | C | B或C | C1 | C | adr | 酶切不确定, PCR定C型, 测序定C1型, 氨基酸序列定C型; 大型一致 |
| 12 | C2或D2 | C | - | C2 | C | adr | 酶切不确定, PCR定C型, 测序定C2型, 氨基酸序列定C型; 大型一致 |
| 13 | C2或D2 | C | B或C | C2 | C | adr | 酶切不确定, PCR定C型, 测序定C2型, 氨基酸序列定C型; 大型一致 |
| 15 | C2或D2 | C2或D2 | D | D3 | D | ayw | 酶切不确定, PCR定D型, 测序定D3型, 氨基酸序列定D型; 大型一致 |
| 16 | C2或D2 | C | B或C | D | D | adw | 酶切不确定, PCR定C型, 测序及氨基酸序列仅定D型; 测序大型一致 |
| 156 | B1 | - | B | B2 | B | adw | 酶切定B1型, PCR定B型, 测序定B2型, 氨基酸序列定B型; 大型一致 |
| 211 | C1 | - | C | C1 | 突变(缺失) | ayD2 | 酶切和测序定C1型, PCR定C型, 氨基酸序列突变, S缺失; 分型错误 |
| 268 | B1 | - | B | B2 | B | adw | 酶切定B1型, PCR定B型, 测序定B2型, 氨基酸序列定B型; 大型一致 |
| 308 | B1 | - | B | B2 | B | adw | 酶切定B1型, 测序定B2型, 氨基酸序列定B型; 大型一致 |
| 441 | D3 | - | - | B2 | B | adw | 酶切定D3型, 测序定B2型, 氨基酸序列定B型; 测序大型一致 |
| 518 | D3 | - | - | G | G | adw | 酶切定D3型, 测序和氨基酸序列定G型; 测序大型一致 |
| 786 | B1 | - | B | B2 | B | adw | 酶切定B1型, PCR定B型, 测序定B2型, 氨基酸序列定B型; 大型一致 |
| 802 | C1 | - | C | C2 | 突变(缺失) | ED2 | 酶切定C1型, PCR定C型, 测序定C2型, 氨基酸序列突变, S缺失; 分型错误 |
| 836 | D3 | - | - | C2 | C | adr | 酶切定D3型, 测序定C2型, 氨基酸序列定C型; 测序大型一致 |
| 840 | C1 | - | C | C2 | C | adr | 酶切定C1型, PCR定C型, 测序定C2型, 氨基酸序列定C型; 大型一致 |
| 865 | C1 | - | - | C5 | C51 | adw | 酶切定C1型, S-DNA测序及氨基酸序列均定C5型; 大型一致 |
| 906 | D3 | - | B或C | G | G | adw | 酶切定D3型, PCR不确定, 测序和氨基酸序列定G型; 测序大型一致 |
| 918 | B1 | - | B或C | B2 | 突变(缺失) | AV2 | 酶切定B1型, PCR不确定, 测序定B2型, 氨基酸序列突变, S缺失; 分型错误 |
| 931 | B1 | - | B或C | B2 | B | adw | 酶切定B1型, PCR不确定, 测序定B2型, 氨基酸序列定B型; 大型一致 |
| 1093 | C1 | - | - | C5 | C51 | adw | 酶切定C1型, S-DNA测序及氨基酸序列均定C5型; 大型一致 |
| 1129 | C1 | - | - | C2 | C | ayr | 酶切定C1型, 测序定C2型, 氨基酸序列仅定C型; 大型一致 |
| 1229 | B2 | - | - | B2 | B | ayr | 酶切定B2型, 测序定B2型, 氨基酸序列仅定B型; 大型一致 |
| 病例号 | 性别 | 籍贯 | 民族 | 基因型 | S-Ag氨基酸序列 | 碱基缺失 | |
| 802 | 男 | 吉林 | 朝鲜族 | C1 | MRAQHQDS-DPCSCYRRGFSC- | ATG(缺失G)AGAGCACAACATCAGGATTCC TAG | |
| 211 | 女 | 广西 | 壮族 | C1 | MESTHQDS-DPCSCYRRGFSC- | ATGGAGAGCACA(缺失A)CATCAGGATTCC TAG | |
| 9 | 男 | 广西 | 瑶族 | G | METSHQDSSDPCSCYRRGFSC- | ATGGAG(缺失A) | |
| ACATCACATCAGGATTCCTCGGACCCCTGCTCGTGTTACAGGCGGGGTTTTTCTTGT TGA | |||||||
| 918 | 男 | 广西 | 汉族 | B1 | MENLASGTPRTPARVTGGGFSC- | ATGGAGAACCTCGCATCAGGAA(插入A) | |
| CTCCTAGAACCCCTGCTCGTGTTACAGGCGGGGGTTTTTCTTGT TGA | |||||||
HBV基因型不仅有流行病学的意义外, 与乙型肝炎的表现和转归也具有相关性. 相关研究表明HBV中C型和B型相比, 前者引起的炎性坏死和纤维化程度更高, 且C型与肝硬化和肝癌明显相关[21]. 不同的基因型, 甚至是同一基因型的不同亚型在毒力和临床表现上都有明显的区别, 是影响病情的进展、预后及抗病毒治疗应答的重要因素[22].
随着HBV基因分型研究的不断深入, 基因分型的方法也有了很大的发展. 目前的乙型肝炎分型方法有很多. 乙型肝炎分型方法主要发展为以下几种方法: 全基因测序法、表面抗原基因(S基因)序列分析法、PCR-RFLP基因分型法、单克隆抗体酶联免疫吸附试验基因分型法、型特异性引物-PCR法、PCR微板核酸杂交-ELISA法、线性探针分析基因分型法、实时定量PCR-解链曲线分析法(real-time PCR and melting curve analysis)等[23]. 但是这些方法对于医疗系统在医疗实践中都存在不同程度的缺陷, 如有的因HBV基因组的变异而导致分型错误甚至无法确定型别, 有的成本过于昂贵、难以普及, 有的完成所需周期过长等, 尤其是在单核苷酸差异上区分只能依赖于机器, 不能达到定性判断[24].
本实验室对216例乙型肝炎检测为"大三阳"的患者血样以3种方法进行基因分型. 3种方法比较各有优缺点, 在总共216例乙型肝炎检测为"大三阳"的患者中, 第1种PCR-RFLP分型方法鉴定出: B型155例, C型41例, D型7例. 因为针对C2和D2型的酶切结果完全相同, 尚有13例不能确定是C2型还是D2型. 其方法简单, 易于操作, 但因HBV分子进化速率很快, 其基因组的变异常导致分型错误甚至无法确定型别.
第2种型特异性PCR方法如图2中所示, 13例中大部分可以被分型, 样品9、15号不能被分型, 本实验室改良的型特异PCR片段法的结果如图3, 图4所示, B、C型引物可以得到分型, 但特异性不高, D型引物中样品15号的分型正确, 但样品518、906号不能正确分型. 方法2和本实验室改良的型特异PCR片段分型方法法虽然特异性较高, 结果较可靠, 但均不能分到亚型. 该法的局限性较大, PCR的非特异性扩增问题和HBV基因的不断变异, 均影响结果判断, 使该法在某些情况下无法准确分型.
基于HBV基因组或其S-Ag基因的DNA序列的基因分型方法[即, 基于序列的系统发生分析(sequence-based phylogenetic analysis)]是HBV基因分型的金标准, 该法获取信息最多, 最为直接、客观、可靠, 是分析和确定新基因型或不同基因型重组的唯一准确的方法, 但其对混合基因型的病例无法快速正确分型或检出. 虽然基于DNA测序的基因分型方法有美国NCBI提供的网上分型工具, 但其仅能分大型, 无法分到亚型. 本研究的第3种基因分型方法通过搜索NCBI数据库, 选择适合系统发生分析的已确定B、C、D亚型的HBV全基因组DNA序列(如表2)作为B、C、D基准亚型构建的HBV标准. 通过对表2中HBV-B、C、D型(共计220条)和NCBI网上HBV分型工具中的A-H型标准基因组的全序列和S基因序列分别进行序列比对和构建进化树发现, 全序列进化树和S基因进化树分布几乎完全一致. 将样品HBV-S区DNA序列测序的结果分别代入全序列标准进化树和S区序列进化树的分型结果一致. 故选用将样品测序结果代入HBV-S区标准进化树进行基因分型, 分型结果准确一致. 我们利用此法在30例的DNA测序分型中发现样品号9、518和906为HBV-G型, 其来源为国内乙型肝炎高发地区广西, 病例为壮族和瑶族男性, 这在国内尚未见报道. 基于DNA测序和系统发生分析软件的基因分型方法, 其临床推广应用就现有正规医院而言已具实际可行性, 即医院检验科室的样品PCR加第三方公司化的DNA测序及简单分析服务, 无论时间(3-7天)还是价格(成本60-100元)及操作的简单快捷(PCR)均具实用性和竞争性, 应大力推广临床应用.
依据HBV-S基因序列推测的S-Ag氨基酸序列进行比对而构建进化树的HBV-S-Ag蛋白分型更能反映HBV的特性, 如在S-Ag基因上因碱基缺失或插入产生的点突变和氨基酸密码移框而提前出现终止码并最终导致的HBV S-Ag缺失(对应的是HBV血清型无法判定); 基于HBV-S DNA序列的基因分型和依据同样序列推定的HBV-S-Ag氨基酸序列的蛋白分型, 两者在HBV大型分类上大体一致, 但在亚型分类上分歧严重. 表现为S-Ag蛋白分型与S-Ag基因分型的亚型分类中型别交错和混乱, 如表3中"C51"型位于HBV-S-Ag蛋白分型进化树的D大型(按基因分型系统标识)的范围内. 总之, 在HBV亚型分类上, 基于DNA测序的HBV-S-Ag的基因分型和基于同样DNA测序的蛋白分型的结果完全不同.
PCR-RFLP法和型特异PCR法的HBV分型(特别是分亚型)的准确性和灵敏性与DNA测序法分型相比是较低的; 而依据HBV-S基因测序推定的S-Ag氨基酸序列进行HBV蛋白分型更能反映HBV的特性, 其与HBV的活性和感染力、乙型肝炎病程、预后及治疗应答的关联应该更直接. 基于对HBV在人体内病毒基因组的广泛变异和病毒种群普遍异质性的认识, 以及众多HBV病毒蛋白和细胞相关蛋白对HBV繁殖和致病性反式调节作用的更多了解[25], 急需重新建立一套既基于HBV基因组构成又反映病毒蛋白(特别是具反式调节作用蛋白)差异的新型HBV分型系统和快速检测方法, 才能进一步明确乙型肝炎发病机制、提高检测效率、寻找治疗对策和实现乙型肝炎流行病学控制.
总之, 酶切方法较为精细可以分到亚型, 但本身缺陷不能区分C2型和D2型. 根据PCR扩增片段大小直接分型方法较为直接简单, 但是引物自身特异性不高, 分型不准确. 本实验室设计的碱基型特异性引物对于区分B、C型有较好的效果, 但对于区分D型效果不佳. 需要进一步完善. HBV-S基因测序后序列比对并构建进化树的方法较为准确客观, 此方法是较为精准的方法, 但需要以蛋白分型方法对其进行补充和校对. 由于HBV基因组成的不断变异和人体内病毒种群的平衡改变, 还需要不断地改进和完善HBV的分型系统、分型标准和分型方法.
依据乙型肝炎病毒(hepatitis B virus, HBV)的全基因组序列或者S区同源性可将HBV分为了A-I九种基因型. 我国的乙型肝炎主要有B、C、D 3种基因型. 并且基因分型还可进一步分到亚型.
吴君, 主任医师, 贵阳医学院附属医院感染科
目前的研究显示, 不同基因型HBV在流行病学特征、病毒变异、疾病表现和治疗应答等方面均存在着差异, 故HBV的基因分型有着重要的意义, 探索更为精确的HBV基因分型的方法也就至关重要.
Norder等通过对HBV全序列分析发现, 在不同基因型之间S区段异质性最大, 而相同基因型内S区段的异质性最小, 从而可以用S区代替全序列进行基因分型.
样品HBV-S基因测序后与一定数量的HBV标准型序列进行序列比对并一同构建进化树的HBV分型方法较为准确和客观, 但在HBV亚型的区分上需要以更精确的HBV蛋白分型方法对其进行校对核准和修改补充. 由于HBV基因组成的不断变异和个体内病毒种群的平衡改变, 还需要不断地完善HBV的分型系统、标准和方法. 首次在国内发现3例HBV-G基因型(来源为广西壮族和瑶族
男性).
本研究构建的HBV标准进化树可作为HBV基因分型和亚型的标准. 其分型方法可在正规医院推广临床应用. 依据HBV-S基因测序推定的S-Ag氨基酸序列进行HBV蛋白分型更能反映HBV的特性, 其与HBV的活性和感染力、乙型肝炎病程、预后及治疗应答的关联应该更直接.
作者提出的DNA测序结合标准进化树的HBV基因分型方法有较好的临床实用价值; HBV蛋白分型方法可发展为更精确的HBV分型方法, 特别是对亚型分类上; 研究结果具一定学术价值.
编辑: 郭鹏 电编:都珍珍
| 1. | La Torre G, Nicolotti N, de Waure C, Chiaradia G, Specchia ML, Mannocci A, Ricciardi W. An assessment of the effect of hepatitis B vaccine in decreasing the amount of hepatitis B disease in Italy. Virol J. 2008;5:84. [PubMed] [DOI] |
| 2. | Ahmad N, Alam S, Mustafa G, Adnan AB, Baig RH, Khan M. e-antigen-negative chronic hepatitis B in Bangladesh. Hepatobiliary Pancreat Dis Int. 2008;7:379-382. [PubMed] |
| 3. | Zhou YH, Wu C, Zhuang H. Vaccination against hepatitis B: the Chinese experience. Chin Med J (Engl. ). 2009;122:98-102. [PubMed] |
| 4. | Lok AS, McMahon BJ. Chronic hepatitis B: update of recommendations. Hepatology. 2004;39:857-861. [PubMed] [DOI] |
| 5. | Van Damme P, Van Herck K. A review of the efficacy, immunogenicity and tolerability of a combined hepatitis A and B vaccine. Expert Rev Vaccines. 2004;3:249-267. [PubMed] [DOI] |
| 6. | Liang X, Bi S, Yang W, Wang L, Cui G, Cui F, Zhang Y, Liu J, Gong X, Chen Y. Epidemiological serosurvey of hepatitis B in China--declining HBV prevalence due to hepatitis B vaccination. Vaccine. 2009;27:6550-6557. [PubMed] [DOI] |
| 7. | Okamoto H, Tsuda F, Sakugawa H, Sastrosoewignjo RI, Imai M, Miyakawa Y, Mayumi M. Typing hepatitis B virus by homology in nucleotide sequence: comparison of surface antigen subtypes. J Gen Virol. 1988;69:2575-2583. [PubMed] [DOI] |
| 8. | Norder H, Couroucé AM, Magnius LO. Complete genomes, phylogenetic relatedness, and structural proteins of six strains of the hepatitis B virus, four of which represent two new genotypes. Virology. 1994;198:489-503. [PubMed] [DOI] |
| 11. | 范 金水, 庄 辉, 李 远贵, 朱 晓洁, 徐 德忠, 马 为民, 王 跃民, 陈 雅洁, 娄 国强, 马 廷贤. 我国8城市HBsAg阳性和阴性乙肝思者的病毐血淌型和基因翌分析. 中华微生物学和免疫学杂志. 1998;18:88-92. |
| 13. | 李 劲, 刘 莹, 邓 志芳, 胡 文娟, 高 奎, 赵 瑞杰, 王 红, 宋 发军. 不同民族大学生乙肝病毒基因型分布状况调查. 中南民族大学学报(自然科学版). 2012;31:38-41. |
| 15. | Wang Z, Tanaka Y, Huang Y, Kurbanov F, Chen J, Zeng G, Zhou B, Mizokami M, Hou J. Clinical and virological characteristics of hepatitis B virus subgenotypes Ba, C1, and C2 in China. J Clin Microbiol. 2007;45:1491-1496. [PubMed] [DOI] |
| 17. | Wang Z, Hou J, Zeng G, Wen S, Tanaka Y, Cheng J, Kurbanov F, Wang L, Jiang J, Naoumov NV. Distribution and characteristics of hepatitis B virus genotype C subgenotypes in China. J Viral Hepat. 2007;14:426-434. [PubMed] [DOI] |
| 18. | Zeng GB, Wen SJ, Wang ZH, Yan L, Sun J, Hou JL. A novel hepatitis B virus genotyping system by using restriction fragment length polymorphism patterns of S gene amplicons. World J Gastroenterol. 2004;10:3132-3136. [PubMed] |
| 19. | Liu WC, Lindh M, Buti M, Phiet PH, Mizokami M, Li HH, Sun KT, Young KC, Cheng PN, Wu IC. Genotyping of hepatitis B virus--genotypes a to g by multiplex polymerase chain reaction. Intervirology. 2008;51:247-252. [PubMed] [DOI] |
| 20. | 郭 亚兵, 侯 金林, 戴 炜, 骆 抗先. 乙型肝炎病毒中国株全基因参照序列的初步建立. 中华微生物学和免疫学杂. 1999;19:197-200. |
| 21. | Magnius LO, Norder H. Subtypes, genotypes and molecular epidemiology of the hepatitis B virus as reflected by sequence variability of the S-gene. Intervirology. 1995;38:24-34. [PubMed] |
| 22. | Schaefer S, Magnius L, Norder H. Under construction: classification of hepatitis B virus genotypes and subgenotypes. Intervirology. 2009;52:323-325. [PubMed] [DOI] |
| 23. | Gandhe SS, Chadha MS, Arankalle VA. Hepatitis B virus genotypes and serotypes in western India: lack of clinical significance. J Med Virol. 2003;69:324-330. [PubMed] [DOI] |
| 24. | Wei Y, Neuveut C, Tiollais P, Buendia MA. Molecular biology of the hepatitis B virus and role of the X gene. Pathol Biol (Paris). 2010;58:267-272. [PubMed] |