修回日期: 2013-10-23
接受日期: 2013-11-06
在线出版日期: 2013-12-28
目的: 筛选胃癌血清差异表达蛋白, 寻找胃癌血清标志物.
方法: 采集胃癌患者血清和正常对照人群血清各45例, 所有血清样本去除高丰度血清蛋白质后, 采用相对和绝对定量同位素标志(isobaric tags for relative and absolute quantitation, iTRAQ)技术联合二维液相色谱-串联质谱(two-dimensional liquid chromatography/tandem mass spectrometry, 2D-LC-MS/MS)分析和鉴定两组间的差异表达蛋白; 采用生物信息学对差异蛋白进行分析; 用Western blot对蛋白组中的代表性血清差异表达蛋白进行表达验证.
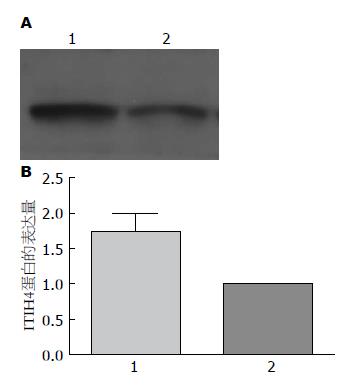
结果: iTRAQ标记联合2D-LC-MS/MS分析一共鉴定出10540条独特肽段, 对应199个非冗余蛋白, 其中符合条件的差异蛋白17个, 其中12个蛋白在胃癌患者中表达上调, 5个蛋白在胃癌患者中表达下调. 17种差异表达蛋白参与10种生物学过程、9种分子作用和2种细胞组分, 涉及的5个生物代谢途径. Western blot结果显示, 与正常对照人群相比, 代表性差异表达蛋白ITIH4在胃癌患者血清中表达量上调, 差异具有统计学意义(1.7517±0.247 vs 1±0, P<0.05).
结论: ITIH4可能是诊断胃癌新的潜在生物标志物.
核心提示: 胃癌(gastric cancer, GC)是严重危害人类健康的常见病和多发病, 可见早期诊断的意义尤为重要. 本研究通过iTRAQ结合2D-LC-MS/MS筛选GC血清差异表达蛋白并通过Western blot验证, 初步筛选得到ITIH4为GC差异表达蛋白, ITIH4可能是诊断GC的潜在生物标志物. 表明该技术用于GC生物标志物的血清蛋白质组学研究具有良好的前景.
引文著录: 韦春回, 赖铭裕, 莫喜晶. 基于iTRAQ结合2D-LC-MS/MS筛选胃癌血清差异表达蛋白. 世界华人消化杂志 2013; 21(36): 4146-4152
Revised: October 23, 2013
Accepted: November 6, 2013
Published online: December 28, 2013
AIM: To screen serum differentially expressed proteins in gastric cancer (GC) patients and search biomarkers for GC diagnosis.
METHODS: Forty-five serum samples from GC patients and 45 from normal controls were collected. High-abundance proteins in serum were removed by immune-chromatography assay. Isobaric tags for relative and absolute quantitation (iTRAQ) coupled with two-dimensional liquid chromatography/tandem mass spectrometry (2D-LC-MS/MS) were used to analyze and identify differentially expressed serum proteins between the two groups. Bioinformatics was used to analyze the identified differentially expressed proteins, and the expression of representative differential proteins was verified by Western blot.
RESULTS: The iTRAQ coupled with 2D-LC-MS/MS analysis led to the identification of a total of 199 proteins, of which 17 were differentially expressed proteins between the two groups, including 12 up-regulated ones and 5 down-regulated ones in GC patients.These differentially expressed proteins are involved in 10 biological processes, 9 molecular functions and 2 cellular components. Western blot results showed that compared with the normal control controls, the expression of representative differential protein ITIH4 in serum of GC patients was significantly up-regulated (1.7517 ± 0.247 vs 1 ± 0, P < 0.05).
CONCLUSION: ITIH4 may be used as a new potential biomarker for diagnosis of GC.
- Citation: Wei CH, Lai MY, Mo XJ. Screening differentially expressed serum proteins in gastric cancer by iTRAQ labeling coupled with 2D-LC-MS/MS. Shijie Huaren Xiaohua Zazhi 2013; 21(36): 4146-4152
- URL: https://www.wjgnet.com/1009-3079/full/v21/i36/4146.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i36.4146
胃癌(gastric cancer, GC)是人类常见的恶性肿瘤之一, 居全球肿瘤发病率的第4位和肿瘤死亡率的第2位[1], 在中国, 其发病率和死亡率分别为第2和第3位[2]. 虽然近年来对胃癌的治疗取得了进展, 其方法有手术、放化疗等, 但多数患者出现临床症状时已处于中晚期并出现转移, 生存率低, 有研究显示[3,4], 如果胃癌在早期就被诊断, 患者的5年生存率可达约90%, 相反, 胃癌中晚期患者的5年生存率不足10%. 因此早期发现并治疗是提高胃癌疗效的关键因素. 近年来已发现一些潜在标志物用于临床诊断肿瘤, 但CEA、CA-199、CA72-4等用于诊断胃癌的特异性和敏感性不高, 研究显示他们的敏感性仅为18%-57%[5]. 因此目前诊断胃癌主要依靠对有症状患者行影像学、胃镜及手术病理活检等方法, 尚缺乏特异性标志物对胃癌高危人群进行筛查.
蛋白质组学(proteomics)是以全部蛋白质为对象, 研究其组成及变化规律, 以了解机体生理病理过程的科学[6]. 目前用于研究肿瘤的蛋白质组学, 采用蛋白质组学技术为手段, 揭示肿瘤发生的可能分子机制, 筛选出能用于临床的肿瘤生物标志物及肿瘤生物治疗的靶标. 随着蛋白质组学技术的迅速发展, 已有可能同时筛选和疾病发生机制、临床诊断、治疗及预后相关的差异表达蛋白[7]. 相对和绝对定量同位素标志(isobaric tags for relative and absolute quantitation, iTRAQ)技术是美国ABI公司于2004年研发的一种新的多肽体外同位素标记相对和绝对定量技术[8]. iTRAQ试剂由平衡基团、报告基团及肽段反应基团等三部分构成. iTRAQ试剂由等量异位标签构成, 他们的分子均量为145 kDa, 其中报告基团的质量为114、115、116、117、118、119、12和121 kDa. 自有学者[9]于2004年首次应用iTRAQ标记技术筛选及鉴定野生型酵母菌株及其同源突变株差异表达蛋白, 其结果显示iTRAQ标记对蛋白质定量分析有较高的覆盖率后, 该技术在生命科学领域特别是肿瘤研究方面已得到广泛应用, 如在肺癌[10]、肝癌[11]、口腔癌[12]、卵巢癌[13]、子宫癌[14]、结肠癌[15]等的研究中均有采用iTRAQ标记技术筛选肿瘤标志物和治疗靶标方面的报道, 但用于筛选胃癌血清差异表达蛋白的研究报道较少.
本研究采用iTRAQ标记联合二维液相色谱-串联质谱(two-dimensional liquid chromatography/tandem mass spectrometry, 2D-LC-MS/MS)技术对胃癌患者和正常人群血清进行蛋白谱表达研究, 筛选胃癌血清差异表达蛋白, 采用生物信息学对血清差异蛋白进行分析, 并对差异蛋白进行表达验证, 旨在寻找与胃癌相关的新的潜在生物标志物.
选取2011-02/2012-02广西医科大学第一附属医院就诊的胃癌患者血清45例, 入选标准为: (1) 经手术或胃镜活检病理确诊为胃癌患者; (2)在本实验抽取血液前均未接受包括治疗性手术、化放疗以及其他任何相关治疗; (3)排除肝肾等其他脏器肿瘤以及急慢性感染性疾病. 最终符合条件的男27例, 女18例; 年龄52-70岁, 平均年龄59岁±10.53岁; 按胃癌第七版TNM分期, Ⅰ期12例, Ⅱ期18例, Ⅲ期15例; 45例均为低分化腺癌; 20例无淋巴结转移, 25例已有淋巴结转移. 正常对照人群来自同时期广西医科大学第一附属医院的健康体检者血清45例, 入选标准为: (1)近半年内曾行胃镜检查, 报告显示无明显异常; (2)无胃胀胃痛、反酸、嗳气等不适主诉; (3)经血常规、尿常规、肝肾功能、肿瘤生物标志物、心电图及腹部B超等检查排除肝肾等其他脏器疾病以及急慢性感染性疾病. 最终符合条件的男26例, 女19例; 年龄41-59岁, 平均年龄47.53岁±5.69岁; 所有受试者均知情同意, 实验经过获得医院伦理委员会审批. 多重亲和MARS human 14色谱柱购自Aglient公司; iTRAQ试剂盒、QSTAR XL质谱仪、Protein Pilot 4.2软件均购自美国应用生物系统公司; 乙腈(ACN)、氯化钾、丙酮、甲酸等购自荷兰Valkenswaard公司; 一抗: 山羊抗人ITIH4, 购自美国Santa Cruz公司、二抗: 辣根酶标记兔抗山羊IgG, 购自北京中杉金桥生物技术有限公司、PVDF膜, 购自美国Milipore公司、液相色谱仪RPCL(岛津20AD), 购自日本岛津公司; 强离子交换柱, 2.1 mm×100 mm, 5 μm, 300Å, 购自美国Nest Group, Inc.公司; 反相离子交换柱, ZORBAX 300SB-C18 column(5 μm, 300Å, 0.1 mm ×150mm)购自美国Microm公司; ECL化学发光底物系统, 购自上海碧云天生物技术有限公司; 垂直电泳槽, 购自美国Bio-Rad公司; 凝胶成像仪, 购自上海梅特勒-托利多公司.
1.2.1 样本采集与制备: 抽取4 mL空腹静脉血, 置于采用血清分离管(serum separation tubes, SST)中, 冰上凝结30 min后, 4 ℃离心10 min(2000 r/min), 取上清液, 以200 μL分装入1.5 mL EP管中, 编号后保存于-80 ℃超低温冰箱以备用.
1.2.2 去除高丰度蛋白: 按照多重亲和去除系统human14色谱柱操作说明, 去除高丰度蛋白, 收集到的馏分用冻干机浓缩.
1.2.3 脱盐和蛋白含量检测: 用3000 MWCO超滤离心管, 加入50 mmol/L pH 8.5三乙胺碳酸氢缓冲液, 反复3次, 脱盐和收集蛋白片段; 通过bradford法进行蛋白定量, 每组低丰度蛋白取100 μg/管, 冻干.
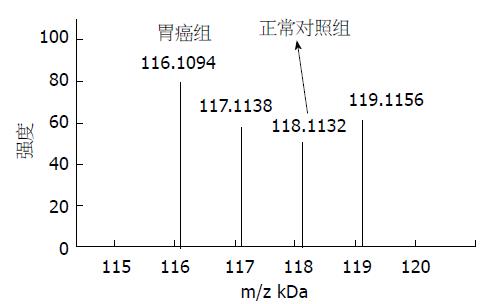
1.2.4 iTRAQ标记: 取各组样品各100 μg加入还原试剂, 60 ℃反应1 h, 加入半胱氨酸封闭试剂室温下放置10 min. 加入胰蛋白酶, 37 ℃酶解过夜, 离心使样品沉至管底. 各管iTRAQ标记试剂分别加入各管样品中, 室温下反应1 h. 加入三倍体积超纯水, 使标记试剂分解, 标记和样品对应关系为: iTRAQ 116: 胃癌组, iTRAQ 118: 正常对照组. 将iTRAQ标记好的各组样品混合至低吸附管, 真空离心蒸干, -80 ℃储存备用.
1.2.5 2D-LC-MS/MS: 不同标记的iTRAQ标记蛋白样品混合, 经第一维强阳离子柱(SCX)分离, 共收取20个馏分进行第二维分析; 第二维反相色谱-质谱联用(RPLC-MS), 色谱分离90 min后行质谱鉴定, MS扫描范围m/z 400-1800, MS/MS扫描范围m/z 100-2000.
1.2.6 质谱数据分析: 采用Protein Pilot 4.2软件检索质谱分析所得数据, 设置蛋白鉴定置信度为95%, 即Prot Score>1.3, 且至少有一个肽段和库中的肽段95%以上相匹配. 同时进行蛋白反库搜索, 设置假阳性率(FDR)<5%, Ratio>1.6或Ratio< 0.625即认为该蛋白存在表达差异.
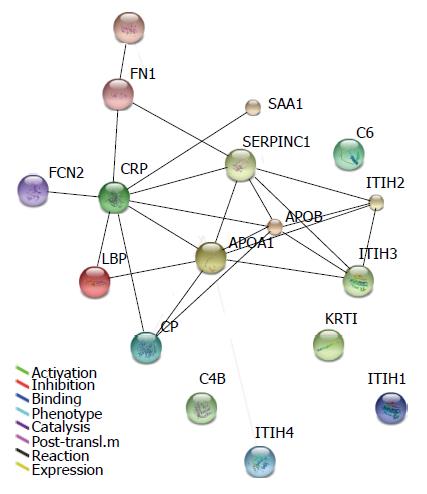
1.2.7 生物信息学分析: 采用Uniprot数据库中的注释信息分析iTRAQ筛选出来的血清差异表达蛋白, 进行GO分析, 用Reactome数据库中的生物代谢途径数据分析血清差异蛋白涉及生物代谢途径. STRING9.0数据库构建参胃癌血清差异表达蛋白相互作用网络图.
1.2.8 Western blot验证ITIH4蛋白的表达: 将20 μL血清样品加入l×还原型上样缓冲液混匀, 变性, 将样品加入凝胶孔道中进行电泳, 100 mA稳流状态下转膜, 将蛋白转至PVDF膜, 用5%脱脂牛奶封闭, 再把膜置于一抗(1:500)稀释液中摇床冰上孵育过夜, 次日用TBST洗膜后置于二抗(1:10000)稀释液中室温孵育1 h, 洗膜, 显影, 定影, 经凝胶分析软件分析其灰度值, 计算ITIH4蛋白在各组表达条带的灰度值, 两者灰度值的比值反映各组蛋白的表达.
统计学处理 应用SPSS16.0软件进行统计学分析. 计量资料以mean±SD表示, 两样本均数间的比较采用t检验. P<0.05为差异有统计学意义.
依据设定的ProteinPiot4.2软件搜库条件要求, 共检测到10540个条独特肽段, 199个非冗余蛋白质. 按Ratio>1.6(上调的差异蛋白)或者Ratio<0.625(下调的差异蛋白)的标准进一步筛选所鉴定的蛋白. 胃癌组与正常对照组血清差异表达蛋白筛选, 共筛选出17个差异蛋白, 其中补体C4-B(C4B)、载脂蛋白B-100(APOB)、抗凝血酶III(SERPINC1)、α胰蛋白酶抑制剂重链H4(ITIH4)、α-1-抗胰蛋白酶(SERPINA1)等12个蛋白在胃癌患者中表达上调, 纤连蛋白(FN1)、α胰蛋白酶抑制剂重链H1(ITIH1)、α胰蛋白酶抑制剂重链H2(ITIH2)、角蛋白, Ⅱ型细胞骨架1(KRT1)和纤胶凝蛋白2(FCN2), 共5个蛋白在胃癌患者中表达下调(表1), 其中代表性差异蛋白ITIH4的肽段相对定量信息(图1).
| 蛋白名称 | 116:118 | 生物学过程 | 分子作用 | 细胞组分 |
| 补体C4-B | 1.94089 | 补体激活 | 肽链内切酶抑制 | 细胞外区 |
| 载脂蛋白B | 1.6904 | 脂质代谢过程 | 脂质转运 | 细胞外区 |
| 纤连蛋白 | 0.4285 | 细胞黏附 | 蛋白结合 | 细胞外区 |
| 抗凝血酶III | 2.8576 | 血液凝固 | 丝氨酸型肽链内切酶抑制剂 | 细胞外区 |
| 载脂蛋白A1 | 1.8197 | 脂质代谢过程 | 脂质转运 | 细胞外区 |
| α胰蛋白酶抑制剂重链H1 | 0.4447 | 肽酶活性的负调控 | 丝氨酸型肽链内切酶抑制剂 | 细胞外区 |
| α胰蛋白酶抑制剂重链H2 | 0.4699 | 肽酶活性的负调控 | 丝氨酸型肽链内切酶抑制剂 | 细胞外区 |
| α胰蛋白酶抑制剂重链H3 | 3.0479 | 肽酶活性的负调控 | 丝氨酸型肽链内切酶抑制剂 | 细胞外区 |
| α胰蛋白酶抑制剂重链H4 | 2.5822 | 肽酶活性的负调控 | 丝氨酸型肽链内切酶抑制剂 | 细胞外区 |
| α抗胰蛋白酶 | 3.1915 | 血液凝固 | 蛋白结合 | 细胞外区 |
| 补体C6 | 1.6596 | 免疫反应 | 蛋白结合 | 细胞外区 |
| 纤胶凝蛋白2 | 0.4285 | 补体激活 | 钙依赖蛋白结合 | 细胞外区 |
| 脂多糖结合蛋白 | 1.8879 | 非特异性免疫反应 | 蛋白结合 | 细胞外区 |
| 血清淀粉样蛋白A | 17.2187 | 急性期反应 | g蛋白耦合受体结合 | 细胞外区 |
| 角蛋白, II型细胞骨架1 | 0.0787 | 补体激活 | 受体激活 | 中间纤维 |
| C反应蛋白 | 5.8076 | 炎症反应 | 蛋白结合 | 细胞外区 |
| 血浆铜蓝蛋白 | 3.4040 | 离子转运 | 铁氧化酶 | 细胞外区 |
通过Uniprot数据库中的注释信息对本实验获得的17种差异表达蛋白进行GO分析, 这些蛋白主要参与10种生物学过程、9种分子作用和2种细胞组分(表1). 使用Reactome数据库中的生物代谢途径数据分析血清差异蛋白涉及的5个生物代谢途径: 免疫系统代谢途径、止血代谢途径、脂质与脂蛋白代谢途径、信号转导代谢途径以及跨膜转运小分子代谢途径. 蛋白质相互作用网络图可见大部分差异蛋白存在相互作用(图2).
经条带灰度分析, 与正常对照组相比, 胃癌组ITIH4蛋白条带明显增强, 蛋白表达量上调, 差异具有统计学意义(1.7517±0.247 vs 1±0, P<0.05)(图3), 蛋白表达趋势与质谱鉴定结果一致.
iTRAQ标记技术是近年来新开发的定量蛋白质组学研究方法, 对差异蛋白有良好的定量鉴定效果, 且操作重复性高, 可同时对四种或八种复杂样品、细胞裂解液、细胞器、组织及血清等不同样品进行相对和绝对定量分析, 可标记样本中所用的蛋白质且其报告离子的分子量小(113-121 kDa), 既提高了蛋白的覆盖率也提高了质谱检测的灵敏度, 适合用于筛选和鉴定生物标志物. 近年来已有学者采用iTRAQ技术筛选出一些肿瘤生物标志物, 如筛选出真核翻译延长因子1A1(eEF1A1)、溶酶体相关膜蛋白1(LAMP-1)及前梯度同源蛋白2(AGR2), 分别可作为前列腺癌[16]、卵巢癌[17]及肺腺癌[10]的生物标志物, 也有iTRAQ标记技术用于胃癌差异蛋白的研究, 而其中对于胃癌细胞的研究占大多数. 例如, 有学者[18]采用iTRAQ联合2D-LC-MALDI-TOF/TOF MS技术对胃癌细胞株进行研究认为钙结合蛋白(caldesmon)可作为一个潜在胃癌转移相关蛋白; Loei等[19]iTRAQ联合2D-LC-MS/MS技术检测胃癌细胞株蛋白谱, 共鉴定出47中差异表达蛋白; Yang等[20]采用iTRAQ联合串联质谱技术对胃癌细胞株进行研究发现组织蛋白酶s(cathepsin s)在胃癌细胞转移和浸润方面发挥重要作用; Deng等[21]采用iTRAQ联合ESI-Q-TOF-MS技术发现可溶抗药性相关钙结合蛋白(sorcin)在胃癌细胞中表达上调. 而血液样品采集方便, 对受试者损伤小, 较易接受, 且由于机体代谢时, 血液作为载体为组织及器官供输营养物质并带走代谢产物, 可较真实的反应机体的生理病理状态. 因此, 对血液标本的研究在探索肿瘤发病机制及寻找肿瘤生物标志物等方面存在巨大潜能.
本研究采用iTRAQ标记联合2D-LC-MS/MS技术筛选出胃癌患者和正常人群血清中的17种差异表达蛋白, 其中12种蛋白表达上调, 5种蛋白表达下调, 采用GO分析显示这些蛋白主要参与10种生物学过程、9种分子作用和2种细胞组分; 生物代谢途径数据分析显示涉及5个生物代谢途径: 免疫系统代谢途径、止血代谢途径、脂质与脂蛋白代谢途径、信号转导代谢途径以及跨膜转运小分子代谢途径, 提示胃癌的发生发展可能与上述5种代谢途径密切相关; 蛋白质相互作用网络图显示大部分表达蛋白存在相互联系; Western blot验证代表性差异蛋白ITIH4的表达, 结果显示, 其蛋白表达趋势与质谱鉴定结果一致.
ITIH4又称血浆激肽释放酶敏感糖蛋白, 是新发现的间α胰蛋白酶抑制剂重链家族成员, 其相对分子量为120 kDa[22,23], 和其他家族成员(ITIH1-3, ITIH5-7)不同, ITIH4以重链亚型的形式存在血液中[24], 易被血浆激肽释放酶系统裂解为25和85 kDa的片段, 后者可进一步裂解为57和28 kDa的片段[25], 而至今尚未有检测到28 kDa的片段的研究报道, 提示其可能裂解为无法检测到的更小片段.
目前, ITIH4蛋白的确切作用仍然不清楚, Piñeiro等[26]认为ITIH4是一个急性期反应蛋白, 在手术后患者的血清样本中明显升高. 报道显示ITIH4与急性反应期发展中的细胞增殖和迁移有关, 在感染和炎症发展过程中发挥重要作用[27]. Saroha等[25]研究结果显示ITIH4蛋白在类风湿性关节炎患者血浆中表达升高. 也有研究结果证实ITIH4蛋白在路易体痴呆患者脑脊液中表达升高[28]. 大量研究显示, ITIH4还和肿瘤有密切关系. Song等[29]采用MALDI-TOF-MS技术发现ITIH4片段在乳腺癌患者和前列腺癌患者血清中表达上调, 对ITIH4完整蛋白和肿瘤关系的研究也有报道, 有学者[30]采用iTRAQ联合2D-LC-MS/MS技术对乳腺癌患者血清进行检测, 发现ITIH4表达升高. 目前, 研究ITIH4和胃癌的关系报道较少, Hamm等[23]采用RT-PCR技术检测ITIH4 mRNA在胃部肿瘤组织表达的研究显示ITIH4在63%的胃部肿瘤组织中表达下调, 在其余的胃癌组织中表达上调. 而本研究应用Western blot分析胃癌患者血清发现ITIH4蛋白表达比正常对照人群升高, 提示ITIH4和胃癌的发生发展有着密切关系, 其结果与蛋白质谱鉴定结果一致, ITIH4可能是诊断胃癌的潜在生物标志物.
总之, 本研究通过iTRAQ结合2D-LC-MS/MS筛选胃癌血清差异表达蛋白并通过Western blot验证, 初步筛选得到ITIH4为胃癌差异表达蛋白, 表明该技术用于胃癌生物标志物的血清蛋白质组学研究具有良好的前景. 但这些蛋白在胃癌发生、发展中的作用尚未完全清楚, 需要进一步评价该血清差异蛋白作为诊断胃癌的候选标志物的可靠性.
目前诊断胃癌主要依靠对有症状患者行胃镜及手术病理活检等方法, 但因其为有创检查,不能对胃癌高危人群进行筛查. 其他辅助诊断如血液生化及影像学等检查由于准确性等原因, 不能如实反映胃癌实际情况. 临床上迫切需要找到特异性及敏感性更高的胃癌诊断指标.
高泽立, 副教授, 上海交大医学院九院周浦分院
相对和绝对定量同位素标志(isobaric tags for relative and absolute quantitation, iTRAQ)联合二维液相色谱-串联质谱(two-dimensional liquid chromatography/tandem mass spectrometry, 2D-LC-MS/MS)技术是近年来最新开发的一种定量蛋白质组学技术, 用于检测样品中低丰度蛋白, 有灵敏度高、蛋白覆盖率高、重复性好等优势. 通过iTRAQ技术可能为无创诊断胃癌提供更多有价值的标志物.
Hendrick等采用iTRAQ联合2D-LC-MS/MS技术研究胃癌AGS和MKN7细胞株及正常胃黏膜上皮细胞蛋白时, 发现43个差异表达蛋白, 其中颗粒蛋白GRN在胃癌细胞株中高表达.
本研究运用血清蛋白质组学技术对胃癌患者和正常人群血清进行蛋白谱表达研究,筛选胃癌血清差异表达蛋白, 采用生物信息学对血清差异蛋白进行分析, 并对差异蛋白进行表达验证, 发现ITIH4可以作为胃癌血清候选标志物.
深入研究胃癌的候选血清标志物在胃癌发生发展过程的作用, 将为胃癌的无创诊断提供新的思路.
本文紧扣当前胃癌防治研究热点, 对研究对象的选择较科学, 结果有可比性, 对胃癌临床研究提供一定理论依据.
编辑:田滢 电编:闫晋利
| 1. | Jemal A, Bray F, Center MM, Ferlay J, Ward E, Forman D. Global cancer statistics. CA Cancer J Clin. 2011;61:69-90. [PubMed] [DOI] |
| 3. | Ohta H, Noguchi Y, Takagi K, Nishi M, Kajitani T, Kato Y. Early gastric carcinoma with special reference to macroscopic classification. Cancer. 1987;60:1099-1106. [PubMed] |
| 4. | Peddanna N, Holt S, Verma RS. Genetics of gastric cancer. Anticancer Res. 1995;15:2055-2064. [PubMed] |
| 5. | Ebert MP, Röcken C. Molecular screening of gastric cancer by proteome analysis. Eur J Gastroenterol Hepatol. 2006;18:847-853. [PubMed] |
| 6. | Blackstock WP, Weir MP. Proteomics: quantitative and physical mapping of cellular proteins. Trends Biotechnol. 1999;17:121-127. [PubMed] |
| 7. | Aebersold R, Mann M. Mass spectrometry-based proteomics. Nature. 2003;422:198-207. [PubMed] [DOI] |
| 8. | Ross PL, Huang YN, Marchese JN, Williamson B, Parker K, Hattan S, Khainovski N, Pillai S, Dey S, Daniels S. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents. Mol Cell Proteomics. 2004;3:1154-1169. [PubMed] [DOI] |
| 9. | Wu WW, Wang G, Baek SJ, Shen RF. Comparative study of three proteomic quantitative methods, DIGE, cICAT, and iTRAQ, using 2D gel- or LC-MALDI TOF/TOF. J Proteome Res. 2006;5:651-658. [PubMed] [DOI] |
| 10. | Chung K, Nishiyama N, Wanibuchi H, Yamano S, Hanada S, Wei M, Suehiro S, Kakehashi A. AGR2 as a potential biomarker of human lung adenocarcinoma. Osaka City Med J. 2012;58:13-24. [PubMed] |
| 11. | Wang C, Jiang K, Kang X, Gao D, Sun C, Li Y, Sun L, Zhang S, Liu X, Wu W. Tumor-derived secretory clusterin induces epithelial-mesenchymal transition and facilitates hepatocellular carcinoma metastasis. Int J Biochem Cell Biol. 2012;44:2308-2320. [PubMed] [DOI] |
| 12. | Lo WY, Wang HJ, Chiu CW, Chen SF. miR-27b-regulated TCTP as a novel plasma biomarker for oral cancer: from quantitative proteomics to post-transcriptional study. J Proteomics. 2012;77:154-166. [PubMed] [DOI] |
| 13. | Shetty V, Hafner J, Shah P, Nickens Z, Philip R. Investigation of ovarian cancer associated sialylation changes in N-linked glycopeptides by quantitative proteomics. Clin Proteomics. 2012;9:10. [PubMed] [DOI] |
| 14. | Yokoyama T, Enomoto T, Serada S, Morimoto A, Matsuzaki S, Ueda Y, Yoshino K, Fujita M, Kyo S, Iwahori K. Plasma membrane proteomics identifies bone marrow stromal antigen 2 as a potential therapeutic target in endometrial cancer. Int J Cancer. 2013;132:472-484. [PubMed] [DOI] |
| 15. | Hammoudi A, Song F, Reed KR, Jenkins RE, Meniel VS, Watson AJ, Pritchard DM, Clarke AR, Jenkins JR. Proteomic profiling of a mouse model of acute intestinal Apc deletion leads to identification of potential novel biomarkers of human colorectal cancer (CRC). Biochem Biophys Res Commun. 2013;440:364-370. [PubMed] [DOI] |
| 16. | Rehman I, Evans CA, Glen A, Cross SS, Eaton CL, Down J, Pesce G, Phillips JT, Yen OS, Thalmann GN. iTRAQ identification of candidate serum biomarkers associated with metastatic progression of human prostate cancer. PLoS One. 2012;7:e30885. [PubMed] [DOI] |
| 17. | Marzinke MA, Choi CH, Chen L, Shih IeM, Chan DW, Zhang H. Proteomic analysis of temporally stimulated ovarian cancer cells for biomarker discovery. Mol Cell Proteomics. 2013;12:356-368. [PubMed] [DOI] |
| 18. | Hou Q, Tan HT, Lim KH, Lim TK, Khoo A, Tan IB, Yeoh KG, Chung MC. Identification and functional validation of caldesmon as a potential gastric cancer metastasis-associated protein. J Proteome Res. 2013;12:980-990. [PubMed] [DOI] |
| 19. | Loei H, Tan HT, Lim TK, Lim KH, So JB, Yeoh KG, Chung MC. Mining the gastric cancer secretome: identification of GRN as a potential diagnostic marker for early gastric cancer. J Proteome Res. 2012;11:1759-1772. [PubMed] [DOI] |
| 20. | Yang Y, Lim SK, Choong LY, Lee H, Chen Y, Chong PK, Ashktorab H, Wang TT, Salto-Tellez M, Yeoh KG. Cathepsin S mediates gastric cancer cell migration and invasion via a putative network of metastasis-associated proteins. J Proteome Res. 2010;9:4767-4778. [PubMed] [DOI] |
| 21. | Deng L, Su T, Leng A, Zhang X, Xu M, Yan L, Gu H, Zhang G. Upregulation of soluble resistance-related calcium-binding protein (sorcin) in gastric cancer. Med Oncol. 2010;27:1102-1108. [PubMed] [DOI] |
| 22. | Cai T, Yu P, Monga SP, Mishra B, Mishra L. Identification of mouse itih-4 encoding a glycoprotein with two EF-hand motifs from early embryonic liver. Biochim Biophys Acta. 1998;1398:32-37. [PubMed] |
| 23. | Hamm A, Veeck J, Bektas N, Wild PJ, Hartmann A, Heindrichs U, Kristiansen G, Werbowetski-Ogilvie T, Del Maestro R, Knuechel R. Frequent expression loss of Inter-alpha-trypsin inhibitor heavy chain (ITIH) genes in multiple human solid tumors: a systematic expression analysis. BMC Cancer. 2008;8:25. [PubMed] [DOI] |
| 24. | Zhuo L, Kimata K. Structure and function of inter-alpha-trypsin inhibitor heavy chains. Connect Tissue Res. 2008;49:311-320. [PubMed] [DOI] |
| 25. | Saroha A, Kumar S, Chatterjee BP, Das HR. Jacalin bound plasma O-glycoproteome and reduced sialylation of alpha 2-HS glycoprotein (A2HSG) in rheumatoid arthritis patients. PLoS One. 2012;7:e46374. [PubMed] [DOI] |
| 26. | Piñeiro M, Andrés M, Iturralde M, Carmona S, Hirvonen J, Pyörälä S, Heegaard PM, Tjørnehøj K, Lampreave F, Piñeiro A. ITIH4 (inter-alpha-trypsin inhibitor heavy chain 4) is a new acute-phase protein isolated from cattle during experimental infection. Infect Immun. 2004;72:3777-3782. [PubMed] [DOI] |
| 27. | Yang MH, Yang YH, Lu CY, Jong SB, Chen LJ, Lin YF, Wu SJ, Chu PY, Chung TW, Tyan YC. Activity-dependent neuroprotector homeobox protein: A candidate protein identified in serum as diagnostic biomarker for Alzheimer's disease. J Proteomics. 2012;75:3617-3629. [PubMed] [DOI] |
| 28. | Dieks JK, Gawinecka J, Asif AR, Varges D, Gmitterova K, Streich JH, Dihazi H, Heinemann U, Zerr I. Low-abundant cerebrospinal fluid proteome alterations in dementia with Lewy bodies. J Alzheimers Dis. 2013;34:387-397. [PubMed] [DOI] |
| 29. | Song J, Patel M, Rosenzweig CN, Chan-Li Y, Sokoll LJ, Fung ET, Choi-Miura NH, Goggins M, Chan DW, Zhang Z. Quantification of fragments of human serum inter-alpha-trypsin inhibitor heavy chain 4 by a surface-enhanced laser desorption/ionization-based immunoassay. Clin Chem. 2006;52:1045-1053. [PubMed] [DOI] |
| 30. | Opstal-van Winden AW, Krop EJ, Kåredal MH, Gast MC, Lindh CH, Jeppsson MC, Jönsson BA, Grobbee DE, Peeters PH, Beijnen JH. Searching for early breast cancer biomarkers by serum protein profiling of pre-diagnostic serum; a nested case-control study. BMC Cancer. 2011;11:381. [PubMed] [DOI] |