修回日期: 2013-10-28
接受日期: 2013-11-06
在线出版日期: 2013-12-08
目的: 采用质谱成像技术比较大肠高分化腺癌组织与腺瘤组织的蛋白质组表达差异, 寻找大肠腺癌相关的蛋白质分子标志物.
方法: 应用质谱成像技术对人体大肠腺癌组织和大肠腺瘤组织冰冻切片进行质谱扫描, 得到蛋白质、多肽的原位分布信息, 初步建立了以质谱成像技术寻找大肠高分化腺癌与腺瘤中差异蛋白的方法. 质谱成像切片制备: 常规10 μm冰冻切片, 用含0.1%三氟乙酸的50%乙腈水溶液配制成15 g/L的α-腈基-4-羟基肉桂酸(α-cyano-4-hydroxycinnamic-acid, CHCA)基质均匀喷洒. 质谱分析条件: 线性正离子模式, 激光能量范围30%-50%, 照射100次, 采样点距200 μm. 获取到的质谱图采用遗传算法计算峰面积. 用峰面积来确定两组之间的差异分子. 采用t检验比较两组间峰差异的显著性.
结果: 与大肠腺瘤比较, 大肠高分化腺癌有显著差异蛋白分子共16个, 其中上调7个, 下调9个.
结论: 质谱成像技术用于大肠高分化腺癌与大肠腺瘤蛋白质组对照研究能够直观反映出大肠高分化腺癌与大肠腺瘤间的蛋白质分子差异, 这些差异分子可能与大肠腺癌的发生、发展相关.
核心提示: 本文采用质谱成像技术对人组织切片进行研究, 探索样品制备方法、质谱检测技术条件, 初步建立了以质谱成像技术比较研究大肠高分化腺癌与大肠腺瘤差异蛋白的方法. 在实验中,与大肠腺瘤比较, 大肠高分化腺癌有显著差异蛋白多肽分子共16个, 其中上调7个, 下调9个. 从大肠腺瘤演变、发展到高分化腺癌 癌这一阶段蛋白质组发生了改变, 出现了差异蛋白标志物. 而这些标志物, 可能与大肠腺癌的发生、发展相关, 可能具有重要的生物学意义.
引文著录: 何滔, 郭赟, 何轩, 胡骁, 于婷婷, 郑淑芳. 大肠高分化腺癌与大肠腺瘤的质谱成像. 世界华人消化杂志 2013; 21(34): 3806-3812
Revised: October 28, 2013
Accepted: November 6, 2013
Published online: December 8, 2013
AIM: To identify colorectal adenocarcinoma associated protein molecular markers by comparing well-differentiated colorectal adenocarcinoma with adenoma using mass spectrometry imaging technology.
METHODS: Frozen tissue sections of human colorectal adenocarcinoma and colon adenoma was scanned by mass spectrometry imaging for polypeptide distribution information in situ. Mass spectrum analysis conditions were linear positive ion mode, laser energy range of 30%-50%, irradiated 100 times, and spot range of 200 μm. Genetic algorithm was used to calculate peak area. Significantly differential peaks between the two groups were identified by using the t-test.
RESULTS: Compared with colorectal adenoma, 16 protein molecules were significantly differentially expressed in well-differentiated colorectal adenocarcinoma, including 7 up-regulated and 9 down-regulated.
CONCLUSION: Mass spectrometry imaging allows to identify significantly differentially expressed proteins between colorectal adenocarcinoma and colon adenoma, and these identified molecules may have important roles in the development of colorectal adenocarcinoma.
- Citation: He T, Guo Y, He X, Hu X, Yu TT, Zheng SF. Mass spectrometry imaging for identification of differentially expressed proteins between colorectal adenocarcinoma and colon adenoma. Shijie Huaren Xiaohua Zazhi 2013; 21(34): 3806-3812
- URL: https://www.wjgnet.com/1009-3079/full/v21/i34/3806.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i34.3806
大肠腺癌是胃肠道常见的恶性肿瘤, 据美国统计, 大肠癌占所有恶性肿瘤发病率和死亡率的第2位[1]. 大肠腺癌的发生、发展是多基因参与、多阶段、多步骤的过程, 涉及分子遗传学和组织形态学的改变. 与大肠腺癌相关的癌前病变包括腺瘤、非腺瘤性息肉病和炎症性肠病, 目前发现95%的早期大肠癌与腺瘤有关.
生物组织质谱成像是基于最新的生物质谱和蛋白质组学、代谢组学技术发展起来的分子成像技术. 他能够在组织水平、细胞及亚细胞水平对特定分子的分布情况进行扫描分析进而获得该分子的分布图[2-5]不需要特殊标记和复杂的样本处理, 不需将样本中的多肽、蛋白质等提取出来, 可以直接得到蛋白质、多肽的原位分布信息, 目前已被广泛用于蛋白质组[6-8]、脂组[9,10]和药物代谢[11,12]等研究领域.
本研究采用质谱成像技术比较大肠高分化腺癌与大肠腺瘤的蛋白质组表达差异, 寻找与大肠腺癌相关的蛋白质, 为进一步研究、筛选大肠腺癌相对特异、敏感的蛋白质和肿瘤蛋白质组学研究提供新的方法和思路.
基质α-腈基-4-羟基肉桂酸(α-cyano-4-hydroxycinnamic-acid, CHCA)纯度98.5%、乙腈(acetonitrile, ACN)、三氟乙酸(trifluoroacetic acid, TFA)、无水乙醇(C2H5OH)均为色谱纯, 以上均购自美国Sigma公司. HM525冰冻组织切片机: 德国美康公司; AutoFlex MALDI-TOF-MS: 德国布鲁克公司; 基质喷雾器: 德国布鲁克公司; -80 ℃低温冰箱: 日本三洋公司. 质谱成像软件Fleximaging、Flexcontrol质谱操控软件和差异分析软件ClinProtools2.0: 德国布鲁克公司.
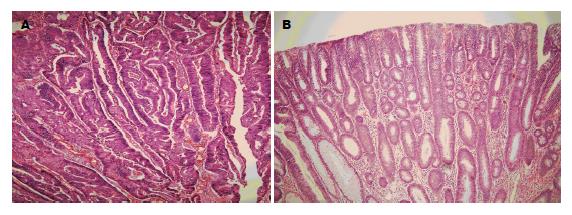
1.2.1 常规冰冻切片、HE染色: (1)手术送检大肠癌与腺瘤标本, 不经固定, 用消毒手术刀片切取组织, 厚度为3 mm, 消毒干纱布拭去血水, 将组织轻轻包入铝箔, 用液氮预冷后转至-80 ℃低温冰箱储存; (2)预冷组织支承器, 在其表面滴少许去离子水, 将-80 ℃低温冰箱内储存的大肠高分化腺癌与大肠腺瘤标本黏附于组织支承器上, 不使用包埋剂, 切片厚度5 μm, 将制备好的切片黏附至普通的载玻片上; (3)染色方法: 切片固定1 min, 水洗5 min, 染苏木素3 min, 盐酸酒精分化10 s, 氨水中返蓝20 s, 伊红染色10-20 s, 梯度乙醇脱水, 二甲苯透明, 中性树胶封固. 制备完成的切片由2名高年资的病理医生审核, 确诊为大肠高分化腺癌与大肠腺瘤的标本入组进行下面实验, 每组5例. 诊断标准[13]: (1)高分化腺癌: 肿瘤呈浸润性生长, 突破黏膜肌层侵及黏膜下层和肌层, 腺样结构超过肿瘤的95%; (2)腺瘤: 呈息肉状生长, 组织富于细胞、核大且深染、不同程度的核复层并缺乏极向, 无组织浸润.
1.2.2 质谱成像组织切片样品制备: 上述确诊大肠高分化腺癌组织与大肠腺瘤组织分别于-20 ℃在冰冻切片机上制作冰冻切片, 组织不使用包埋剂, 切片厚度10 μm. 将组织切片直接从切片刀黏附到专用导电载玻片的导电面上, 然后将载玻片放入真空干燥器内室温干燥5 min. 取出已干燥切片, 将其缓慢放入700 mL/L乙醇中泡洗1-2次, 每次15-30 s, 再缓慢放入950 mL/L乙醇泡洗15 s[14,15].
1.2.3 质谱成像切片基质覆盖: 用含0.1%TFA的50%ACN水溶液配制成15 g/L的CHCA基质溶液. 取上述制备好的载玻片, 放置于基质喷雾器中, 调整好喷雾距离, 将基质溶液均匀喷洒覆盖于组织表面. 结束后, 将载玻片置于室温下自然干燥. 干燥后的载玻片用导电胶带贴在MALDI不锈钢靶板上, 送入质谱仪分析.
1.2.4 质谱扫描和数据分析: 使用Bluker公司Flexcontrol质谱操控软件, 设置质谱扫描条件: 线性正离子模式, 激光能量范围30%-50%, 每个采样点照射100次, 采样点距200 μm, 质量扫描范围m/z 1000-20000.
统计学处理 Clinprotools2.0分析软件中, 采用遗传算法, 分别计算出腺癌组和腺瘤组的平均质谱峰值, 自动计算峰面积. 用峰面积来确定两组之间的差异分子. 采用t检验确定两组之间P<0.01的质谱峰为显著差异峰.
制备完成的切片经由2名高年资的病理医生审核, 选取确诊为大肠高分化腺癌和大肠腺瘤的标本各5例(图1), 进行质谱成像实验.
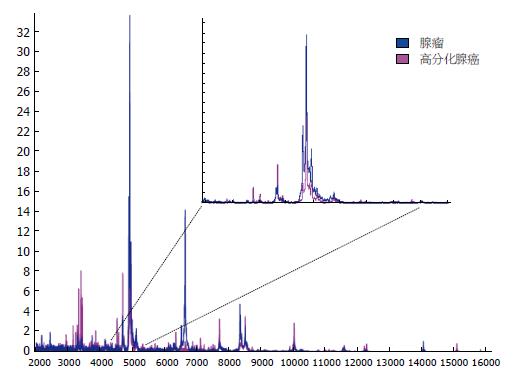
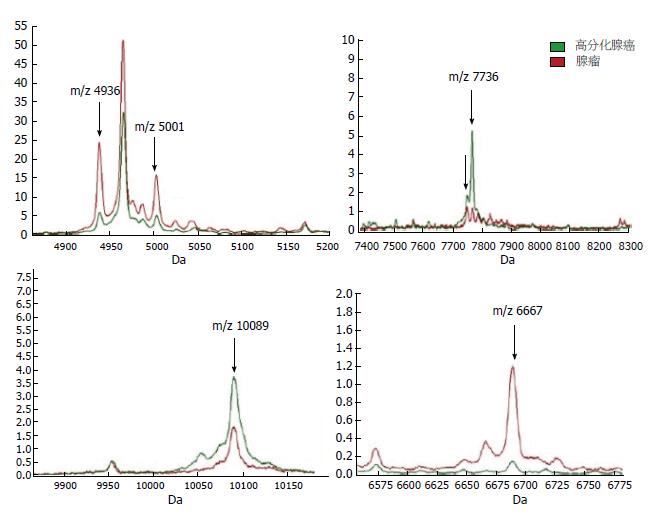
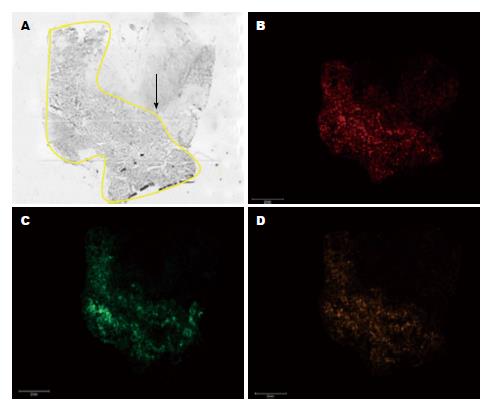
质谱扫描得到大肠高分化腺癌和大肠腺瘤质谱峰图(图2), 可见, 大肠高分化腺癌和大肠腺瘤质谱峰图簇峰基本集中于在m/z 1500-11000之间. 差异分析结果为: 与大肠腺瘤比较, 大肠高分化腺癌有显著差异蛋白多肽共16个, 其中上调7个, 下调9个(表1, 图3). 部分多肽分子显著高表达于高分化腺癌组织中(图4).
质谱成像技术是一种全新的分子成像技术, 他可在组织切片中同时找到多种蛋白多肽分子, 与双向凝胶电泳、毛细血管电泳、蛋白芯片等蛋白质组学技术相比, 其最大的优势是能够提供蛋白多肽分子在组织中空间分布的精确信息, 准确定位, 并对蛋白含量进行相对定量.
医学影像学技术已经成为临床诊断的必备手段, 但是绝大部分不能在分子水平上提供组织成分的分布信息, 而质谱成像技术能在分子水平上发现病变组织的标志物. 质谱成像技术在疾病机制的研究中已经有着广泛的应用, 目前研究涉及的疾病包括帕金森病[16]、阿尔茨海默氏症[17]、法布里病[18]、乳腺癌[19]、黑色素瘤[20]、卵巢癌[21]、结肠癌[22]等.
目前, 在临床病理诊断中, 对大肠腺瘤与腺癌的鉴别, 基本都依赖于病理医生的经验, 当送检材料不足时, 往往会造成诊断的困难, 导致临床处置进退两难. 目前为止, 大肠肿瘤仍缺乏肿瘤特异性标志物[23], 临床常用的大肠肿瘤诊断标志物(如CEA)特异性低、敏感性差, 不能用于早期诊断. 目前发现95%的早期大肠癌与腺瘤有关. 很多学者认为由正常上皮发展为大肠癌一般需经过上皮过度增生、腺瘤期(早、中、晚期)、癌变及癌浸润和转移等阶段[24], 这是一个多阶段、多基因改变的、复杂的积累过程, 而这些改变最终会体现在蛋白表达的差异, 此进程需10-15年, 而从腺瘤发展到癌又是其中最关键的一步. de Wit等[25]采取凝胶电泳、纳米液相色谱和串联质谱技术对结肠癌与腺瘤细胞膜表面蛋白研究中发现44种差异蛋白. Luo等[26]采用2-D双向凝胶电泳技术在对腺瘤与结肠癌和正常黏膜的蛋白质组比较研究中发现24种差异蛋白. Choi等[27]采用2-D双向凝胶电泳技术在对结肠癌和结肠腺瘤患者血清蛋白质组比较研究中发现结肠癌患者血清有11种蛋白表达增高, 13种表达下降.
本工作采用质谱成像技术对人组织切片进行研究, 探索样品制备方法、质谱检测技术条件, 初步建立了以质谱成像技术比较研究大肠高分化腺癌与大肠腺瘤差异蛋白的方法. 在实验中, 与大肠腺瘤比较, 大肠高分化腺癌有显著差异蛋白多肽分子共16个, 其中上调7个, 下调9个. 从一个方面证实从大肠腺瘤演变、发展到高分化腺癌这一阶段蛋白质组发生了改变, 出现了差异蛋白标志物. 而这些标志物, 可能与大肠腺癌的发生、发展相关, 可能具有重要的生物学意义. 但目前对组织中蛋白质和多肽直接鉴定, 还是研究的难点问题[28-31], 原位酶切技术的应用可以提高鉴定的成功率.
目前为止, 质谱成像技术还处于起步阶段, 样品制备、质谱条件、数据处理等方面还需要进一步探索、改进和完善. 随着该技术的日趋成熟, 鉴定组织中特异性分布的蛋白质和多肽, 以及对其功能进行验证, 也将是质谱成像技术的一个重要发展方向.
生物组织质谱成像是基于最新的生物质谱技术. 他能够在组织水平、细胞及亚细胞水平对特定分子的分布情况进行扫描分析进而获得该分子的分布图. 不需要特殊标记和复杂的样本处理, 不需将样本中的多肽、蛋白质等提取出来, 可以直接得到蛋白质、多肽的原位分布信息, 目前已被广泛用于蛋白质组、脂组和药物代谢等研究领域.
白雪巍, 副主任医师, 哈尔滨医科大学
目前采用蛋白质组学技术研究和探索大肠癌诊断标志物、进行早期筛查是研究的热点. 质谱成像技术能够提供蛋白多肽分子在组织中空间分布的精确信息, 准确定位, 并对蛋白含量进行相对定量. 但目前该技术出峰率较低、蛋白后续鉴定困难、价格昂贵都限制了其应用.
该实验采用质谱成像技术对人体组织切片进行研究, 探索样品制备方法、质谱检测等技术条件, 初步建立了以质谱成像技术比较研究大肠高分化腺癌与大肠腺瘤差异蛋白的方法.
在临床病理诊断中, 对大肠腺瘤与腺癌的鉴别, 基本都依赖于病理医生的经验,当送检材料不足时, 往往会造成诊断的困难, 导致临床处置进退两难. 以质谱成像技术比较研究大肠高分化腺癌与大肠腺瘤差异蛋白, 为临床病理诊断提供了新的思路. 在常规光镜难以明确诊断及判断预后的疾病, 如甲状腺滤泡癌、消化系神经内分泌肿瘤、胰腺实性-假乳头状肿瘤、子宫增生活跃的平滑肌瘤、微小浸润和转移癌等将有良好的应用前景.
本文具有一定指导意义.
编辑: 田滢 电编:闫晋利
| 1. | Rozen P. Report of the World Organization for Digestive Endoscopy Colorectal Cancer Screening Committee Meeting, Chicago, 2005. Eur J Cancer Prev. 2006;15:95-102. [PubMed] [DOI] |
| 2. | Setou M, Shrivas K, Sroyraya M, Yang H, Sugiura Y, Moribe J, Kondo A, Tsutsumi K, Kimura Y, Kurabe N. Developments and applications of mass microscopy. Med Mol Morphol. 2010;43:1-5. [PubMed] [DOI] |
| 3. | Ruotolo BT, Gillig KJ, Woods AS, Egan TF, Ugarov MV, Schultz JA, Russell DH. Analysis of phosphorylated peptides by ion mobility-mass spectrometry. Anal Chem. 2004;76:6727-6733. [PubMed] [DOI] |
| 4. | Luxembourg SL, Mize TH, McDonnell LA, Heeren RM. High-spatial resolution mass spectrometric imaging of peptide and protein distributions on a surface. Anal Chem. 2004;76:5339-5344. [PubMed] [DOI] |
| 5. | Rubakhin SS, Jurchen JC, Monroe EB, Sweedler JV. Imaging mass spectrometry: fundamentals and applications to drug discovery. Drug Discov Today. 2005;10:823-837. [PubMed] [DOI] |
| 6. | Chughtai K, Heeren RM. Mass spectrometric imaging for biomedical tissue analysis. Chem Rev. 2010;110:3237-3277. [PubMed] [DOI] |
| 7. | Shimma S, Sugiura Y, Hayasaka T, Zaima N, Matsumoto M, Setou M. Mass imaging and identification of biomolecules with MALDI-QIT-TOF-based system. Anal Chem. 2008;80:878-885. [PubMed] [DOI] |
| 8. | Sugiura Y, Konishi Y, Zaima N, Kajihara S, Nakanishi H, Taguchi R, Setou M. Visualization of the cell-selective distribution of PUFA-containing phosphatidylcholines in mouse brain by imaging mass spectrometry. J Lipid Res. 2009;50:1776-1788. [PubMed] [DOI] |
| 9. | Moritake S, Taira S, Sugiura Y, Setou M, Ichiyanagi Y. Magnetic nanoparticle-based mass spectrometry for the detection of biomolecules in cultured cells. J Nanosci Nanotechnol. 2009;9:169-176. [PubMed] |
| 10. | Ageta H, Asai S, Sugiura Y, Goto-Inoue N, Zaima N, Setou M. Layer-specific sulfatide localization in rat hippocampus middle molecular layer is revealed by nanoparticle-assisted laser desorption/ionization imaging mass spectrometry. Med Mol Morphol. 2009;42:16-23. [PubMed] [DOI] |
| 11. | Zaima N, Matsuyama Y, Setou M. Principal component analysis of direct matrix-assisted laser desorption/ionization mass spectrometric data related to metabolites of fatty liver. J Oleo Sci. 2009;58:267-273. [PubMed] [DOI] |
| 12. | Végvári A, Fehniger TE, Gustavsson L, Nilsson A, Andrén PE, Kenne K, Nilsson J, Laurell T, Marko-Varga G. Essential tactics of tissue preparation and matrix nano-spotting for successful compound imaging mass spectrometry. J Proteomics. 2010;73:1270-1278. [PubMed] [DOI] |
| 13. | Hamilton SR, Aalton LA. Pathology and genetics tumours of digestive system. Lyon: IARC Press 2000; 105-143. |
| 14. | 刘 念, 刘 峰, 许 彬, 高 亚兵, 李 向红, 魏 开华, 张 学敏, 杨 松成. 生物组织质谱成像方法的建立及其在微波辐射后大鼠海马组织的蛋白组分析中的应用. 分析化学. 2008;36:421-425. |
| 15. | Chaurand P, Cornett DS, Caprioli RM. Molecular imaging of thin mammalian tissue sections by mass spectrometry. Curr Opin Biotechnol. 2006;17:431-436. [PubMed] [DOI] |
| 16. | Pierson J, Norris JL, Aerni HR, Svenningsson P, Caprioli RM, Andrén PE. Molecular profiling of experimental Parkinson's disease: direct analysis of peptides and proteins on brain tissue sections by MALDI mass spectrometry. J Proteome Res. 2004;3:289-295. [PubMed] |
| 17. | Grossi C, Francese S, Casini A, Rosi MC, Luccarini I, Fiorentini A, Gabbiani C, Messori L, Moneti G, Casamenti F. Clioquinol decreases amyloid-beta burden and reduces working memory impairment in a transgenic mouse model of Alzheimer's disease. J Alzheimers Dis. 2009;17:423-440. [PubMed] [DOI] |
| 18. | Roy S, Touboul D, Brunelle A, Germain DP, Prognon P, Laprévote O, Chaminade P. [Imaging mass spectrometry: a new tool for the analysis of skin biopsy. Application in Fabry's disease]. Ann Pharm Fr. 2006;64:328-334. [PubMed] [DOI] |
| 19. | Rauser S, Marquardt C, Balluff B, Deininger SO, Albers C, Belau E, Hartmer R, Suckau D, Specht K, Ebert MP. Classification of HER2 receptor status in breast cancer tissues by MALDI imaging mass spectrometry. J Proteome Res. 2010;9:1854-1863. [PubMed] [DOI] |
| 20. | Thiery G, Shchepinov MS, Southern EM, Audebourg A, Audard V, Terris B, Gut IG. Multiplex target protein imaging in tissue sections by mass spectrometry--TAMSIM. Rapid Commun Mass Spectrom. 2007;21:823-829. [PubMed] [DOI] |
| 21. | Lemaire R, Menguellet SA, Stauber J, Marchaudon V, Lucot JP, Collinet P, Farine MO, Vinatier D, Day R, Ducoroy P. Specific MALDI imaging and profiling for biomarker hunting and validation: fragment of the 11S proteasome activator complex, Reg alpha fragment, is a new potential ovary cancer biomarker. J Proteome Res. 2007;6:4127-4134. [PubMed] [DOI] |
| 22. | Shimma S, Sugiura Y, Hayasaka T, Hoshikawa Y, Noda T, Setou M. MALDI-based imaging mass spectrometry revealed abnormal distribution of phospholipids in colon cancer liver metastasis. J Chromatogr B Analyt Technol Biomed Life Sci. 2007;855:98-103. [PubMed] [DOI] |
| 25. | de Wit M, Jimenez CR, Carvalho B, Belien JA, Delis-van Diemen PM, Mongera S, Piersma SR, Vikas M, Navani S, Pontén F. Cell surface proteomics identifies glucose transporter type 1 and prion protein as candidate biomarkers for colorectal adenoma-to-carcinoma progression. Gut. 2012;61:855-864. [PubMed] [DOI] |
| 26. | Luo YX, Cui J, Wang L, Chen DK, Peng JS, Lan P, Huang MJ, Huang YH, Cai SR, Hu KH. Identification of cancer-associated proteins by proteomics and downregulation of β-tropomyosin expression in colorectal adenoma and cancer. Proteomics Clin Appl. 2009;3:1397-1406. [PubMed] [DOI] |
| 27. | Choi JW, Liu H, Shin DH, Yu GI, Hwang JS, Kim ES, Yun JW. Proteomic and cytokine plasma biomarkers for predicting progression from colorectal adenoma to carcinoma in human patients. Proteomics. 2013;13:2361-2374. [PubMed] [DOI] |
| 28. | Cazares LH, Troyer D, Mendrinos S, Lance RA, Nyalwidhe JO, Beydoun HA, Clements MA, Drake RR, Semmes OJ. Imaging mass spectrometry of a specific fragment of mitogen-activated protein kinase/extracellular signal-regulated kinase kinase kinase 2 discriminates cancer from uninvolved prostate tissue. Clin Cancer Res. 2009;15:5541-5551. [PubMed] |
| 29. | Hanselmann M, Kirchner M, Renard BY, Amstalden ER, Glunde K, Heeren RM, Hamprecht FA. Concise representation of mass spectrometry images by probabilistic latent semantic analysis. Anal Chem. 2008;80:9649-9658. [PubMed] [DOI] |
| 30. | Deininger SO, Ebert MP, Fütterer A, Gerhard M, Röcken C. MALDI imaging combined with hierarchical clustering as a new tool for the interpretation of complex human cancers. J Proteome Res. 2008;7:5230-5236. [PubMed] [DOI] |
| 31. | Hanselmann M, Köthe U, Kirchner M, Renard BY, Amstalden ER, Glunde K, Heeren RM, Hamprecht FA. Toward digital staining using imaging mass spectrometry and random forests. J Proteome Res. 2009;8:3558-3567. [PubMed] [DOI] |