修回日期: 2013-04-20
接受日期: 2013-04-27
在线出版日期: 2013-06-08
目的: 研究食管鳞癌(esophageal squamous cell carcinoma, ESCC)中转录因子的调控作用.
方法: 从GEO下载两个基因数据集, 利用BRB-ArrayTool分析食管癌细胞和正常细胞之间差异基因表达. 从差异表达基因中富集出转录因子, 根据转录因子目的基因、结合蛋白和差异基因的相互作用来分析其调控作用.
结果: 根据463个差异表达基因富集了8个转录因子, 差异基因中含此8个转录因子的110个调控基因, 包括靶基因、和靶基因相互作用基因及其上游的酶基因. 这些基因参与了许多的生物过程, 如细胞周期、有丝分裂、细胞分离、细胞转移、DNA的损伤反应和DNA的修复. 其中37个基因参与了KEGG信号通路, 包括癌化途径、黏着斑、细胞周期、细胞外基质受体相互作用、小细胞肺癌、卵母细胞减数分裂、P53信号通路和脂肪酸代谢通路等. 这110个基因和463个差异表达基因参与的信号通路没有明显的差异. 这110个基因相互作用, 形成有110个节点和316条边组成分子网路.
结论: 在ESCC中, 8个转录因子通过调控其靶基因和相互作用基因而发挥重要作用, 其调控基因组成一个复杂的相互作用分子网络.
核心提示: 分析食管鳞癌(esophageal squamous cell carcinoma)细胞和正常细胞之间463个差异基因表达, 富集了8个转录因子及110个调控基因, 这些基因参与了许多的重要生物过程, 形成有110个节点和316条边组成分子网路.
引文著录: 吴耀松, 陈玉龙, 尹素改, 王慧慧. 转录因子在食管鳞癌基因表达中的调控作用. 世界华人消化杂志 2013; 21(16): 1533-1538
Revised: April 20, 2013
Accepted: April 27, 2013
Published online: June 8, 2013
AIM: To study the role of transcription factors (TFs) in regulation of gene expression in esophageal squamous cell cancer.
METHODS: Two gene microarray data sets downloaded from the GEO BRB-ArrayTool were used to analyze differentially expressed genes between esophageal squamous cell cancer and normal esophageal tissue. TFs were enriched from differentially expressed genes, and the regulatory role of TFs was assessed by analyzing interactions among target genes, binding proteins and differentially expressed genes.
RESULTS: Eight TFs were obtained from 463 differentially expressed genes. There were 110 genes that may be regulated by TFs, including target genes, genes encoding interaction proteins, and upstream enzyme genes. These genes participated in multiple biological processes such as cell cycle, mitosis, cell division, cell migration, response to DNA damage, and DNA repair. Of these 110 genes, 37 were involved in KEGG pathways, including cancer pathways, focal adhesion, cell cycle, ECM-receptor interaction, small cell lung cancer, oocyte meiosis, p53 signaling pathway, and fatty acid metabolism. The pathways involving these 110 genes showed no significant difference with those involving the 463 differentially expressed genes. These 110 genes interact and form a molecular network which is composed of 110 nodes and 316 edges.
CONCLUSION: Eight TFs identify in this study constitute a complex molecular interaction network and play an important role in ESCC by regulating their target and interactive genes.
- Citation: Wu YS, Chen YL, Yin SG, Wang HH. Role of transcription factors in regulation of gene expression in esophageal squamous cell carcinoma. Shijie Huaren Xiaohua Zazhi 2013; 21(16): 1533-1538
- URL: https://www.wjgnet.com/1009-3079/full/v21/i16/1533.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i16.1533
食管癌是世界范围内第6大最常见而致命的肿瘤, 其中95%的患者属食管鳞癌[1,2]. 食管鳞癌(esophageal squamous cell carcinoma, ESCC)是一种恶性肿瘤, 只有在出现症状以后才易诊断, 预后性很差. 为了减少ESCC的死亡率, 许多科学家通过各种技术手段包括cDNA芯片分析技术试图找到一些能进行早期诊断的生物标志物. 利用基因芯片技术能够成功地判断出和生存率、组织学分级、浸润状态及对化疗反应等临床参数相关的食管癌亚型[3,4], 但这些研究大部分都只关注不同ESCC亚型中基因表达的改变或单个差异基因功能, 很少探究这些基因表达改变的原因和差异表达基因之间的相互联系.
许多因素影响基因的表达, 转录因子是其中一种主要的调控因子, 他与基因启动子结合, 调控基因的表达. 不少像基因探针富集分析(gene set enrichment analysis, GSEA)这样的工具为从共表达基因中富集转录因子提供了可能[5-7], 由此, 我们可以通过差异表达基因列表非直接鉴定出激活的转录因子, 从而阐述基因表达发生改变的机理. 本文目的是寻找调控ESCC基因表达的转录因子, 并构建转录因子调控的分子网络.
从GEO下载GSE20347和GSE17351数据集. GSE20347包括34人样本, GSE17351包括了10人样本, 皆为ESCC组织和正常组织配对, 为Affymetrix Human Genome U133 Plus 2.0 Array平台, 该平台包含47000多个转录子和变异体, 涵盖了38500个已经定性清楚的人类基因.
BRB-ArrayTools 4.2软件有Dr. Richard Simon和Amy Peng Lam所建立, 本研究利用此软件对数据进行挖掘. 首先排除相对表达基因中位数任一方向(高于、低于)>1.5倍不到整个芯片数20%的基因数据, 或缺失值超过50%样本的基因数据. 从GSE20347鉴定出肿瘤和正常组织间差异mRNA, 他们必须满足3个统计条件: (1)P<0.001; (2)P<0.001, FDR<0.05; (3)2<正常/癌组织的比值<0.5. 然后, 以GSE2034中筛选出的差异基因作为背景, 应用BRB-Array Tools 4.2软件预测工具中Support Vector Machine and Bayesian Compound Covariate预测器从GSE17351数据集筛选出差异基因, 这样被筛选出的基因认为是ESCC真正相关基因.
利用网上在线的工具(http://www.tfacts.org), Essaghir A法[8]分析这些基因的转录子. 从HPRD、IntAct、DIP数据库中[9-11]寻找转录子相互作用蛋白. 同时, 从HPRD中寻找转录子上游酶类分子. 在下面的分析中, 主要对真正相关基因和其相互作用蛋白进行分析.
利用在线的DAVID工具http://david.abcc.ncifcrf.gov/, 分析转录子靶基因和相互作用的蛋白的本体功能和信号通路. 利用STRINGhttp://string.embl.de和 PSTINGhttp://pstiing.licr.org/search/a_start_batch.jsp在线分析目标基因的相互作用. 用Cytoscape[12]做出相应的网络图形.
在GSE20347数据库中, 有1552个差异探针代表1195基因. 以1195个基因为背景, 通过Support Vector Machine and Bayesian Compound Covariate(P<0.01)预测器, 从GSE17351筛选出463个ESCC相关基因, 其中包括了259个上调基因和204个下调基因, 这些基因能够成功地将癌组织和正常组织区分开来, 其敏感性、特异性、阳性及阴性预测值都等于1.
463个ESCC相关基因主要参与了下面的生物活动: 细胞周期、有丝分裂、细胞分离、DNA复制、转录调控、应对DNA损伤刺激、氧化损伤、发育、细胞增殖、DNA修复、细胞黏附.
有68个基因包括在KEGG信号通路数据中, 其中21个基因参与细胞周期, 10个基因参与DNA复制, 13个基因参与有丝分裂, 9个基因参与ECM受体相互作用, 19个基因参与癌变通路, 14个基因和黏附有关, 8个基因参与p53通路, 5个基因和脂肪酸代谢有关, 5个基因和错配修复有关, 8个基因参与小细胞肺癌. 有7个基因包括于BioCarta通路库中, 其中, 4个基因参与Role of Ran的纺锤体的调节, 3个基因参与应对DNA损伤信号的RB肿瘤抑制/检查点.
应用TFactS富集出8个转录因子, 并符合下列条件的: P<0.05, E<0.05, Q<0.05和FDR<0.05. 在ESCC相关基因中, Myc、GLI2、TCF/BETA-CATENIN、FOXO3、SP、ETV4、FOXO和GLI对应的靶基因数分别为30、14、23、9、13、4、12和13. 只有Myc, SP1和FOXO3分别有19、2、1个相互作用的蛋白, SP1有2个上游酶分子.
共有110个基因, 包括了转录因子靶基因、相互作用蛋白基因和上游酶基因. 我们称这些基因为转录因子调控基因. 通过DAVID在线工具分析这些基因的本体功能和通路. 参与的生物过程主要是细胞周期、有丝分裂、细胞移动、应对DNA损伤刺激、DNA修复.
在KEGG通路上包括了37个基因, 其中19个基因参与癌通路, 10个基因与黏着斑有关, 8个基因与细胞周期有关, 5个基因与ECM受体相互作用有关, 6个基因与小细胞肺癌有关, 5个基因与卵母细胞减数分裂有关, 4个基因与p53信号通路相联系, 3个基因参与脂肪酸代谢, 5个基因与T细胞受体信号通路相关. 有5个基因与BioCarta通路相联系, 包括Erk1/Erk2 Mapk信号通路和BAD磷酸化的调控.
为了探索转录因子的调控作用, 我们用χ2检验比较了463个ESCC相关基因(genuine genes involved in ESCC, GGE)和110个转录因子调控基因(transcription factors regulating genes, TFG)参与KEGG信号通路的差异, 发现虽然单个通路上有明显差别, 但从总体性来说, TFG和GGE没有明显区别, 换而言之, 从KEGG通路来角度, TFG可以代表GGE(表1).
| 癌化 | 黏着斑 | 细胞周期 | ECM受体 | 卵母细胞细胞分裂 | p53信号通路 | 肺癌 | 脂肪酸 | |
| GGE | 19 | 14 | 21 | 9 | 13 | 8 | 8 | 5 |
| TFGs | 19 | 10 | 8 | 5 | 5 | 4 | 6 | 3 |
首先, 110个基因放入http://string.embl.de上的对话框中, 种属选为人类, 所需信度为高信度(0.7), 发现78个相互作用基因. 其中, 37个基因构成71对被其他研究组证实的共表达基因, 这些基因中, 22个基因是Myc的靶基因. 为了证明他们基因是否共表达, 应用Spearman法把22基因中的一个CDC6表达值与其他基因进行相关分析, 发现CDC6表达和其他基因的密切相关(P<0.001, r>0.7). 这22个基因主要参与了如细胞周期和DNA复制等生物过程. 总之, 在78个基因中, 他们相互联系, 或激活或相互抑制或共表达.
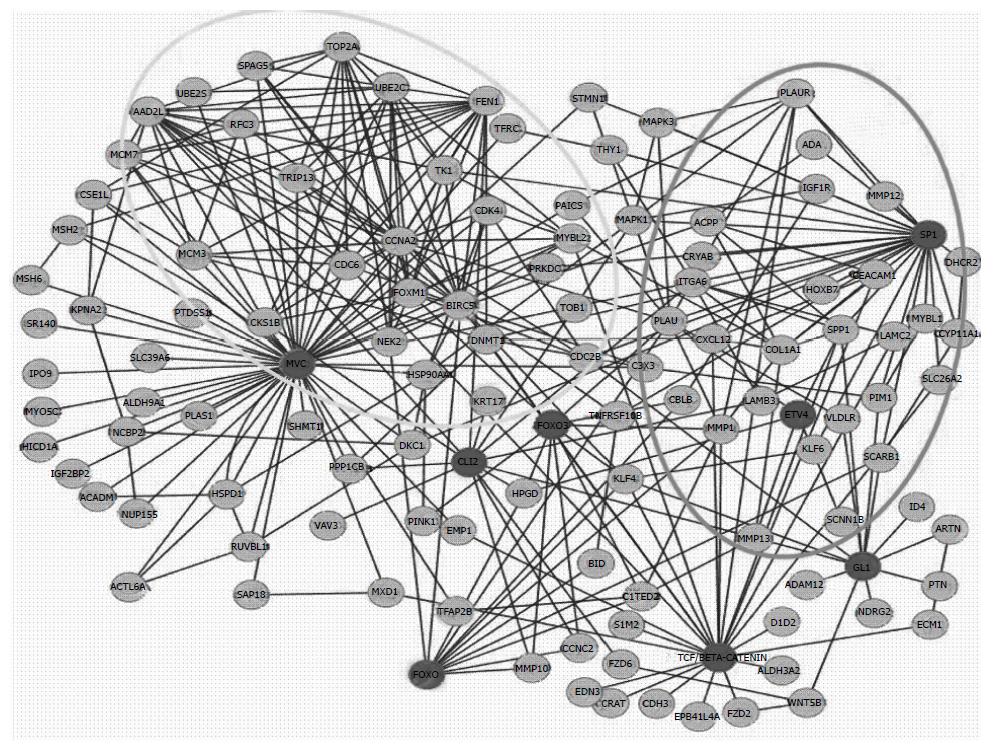
其次, 110个基因行成了316组相互作用的基因, 包括上面提到的78个基因中的156对. 这316组输入到cytoscape工具中, 构建了一个由转录因子调控的分子网络(图1).
图中显示围绕8个转录因子的分子分布, 第1、2部分的联系比其他部分更加紧密. 第1部分的分子主要参与细胞周期, 第2部分分子参与细胞基质和细胞间的信号联系.
基因芯片技术具有高通量和高灵敏度的特点, 此技术可以实现全基因组基因变化检测. 有不少全基因组表达谱研究ESCC的报道[13,14], 但是大部分前人的研究只报道所选择的少数几个基因, 没有显示总体的数据. 不同的研究中, 所报道的差异基因很少有重复. 许多因素, 如样品、平台、分析等等会导致芯片结果的重复性差. 许多科学家做了很大的努力去利用Meta分析从不同芯片中得到可靠结果, 但不能克服一些关键性的问题, 如注解、跨平台的比较等[15]. 在本文中, 我们选择了同一平台的两个数据集分析. 首先, 利用较大的数据集常规分析, 寻找差异基因. 然后, 以这些差异基因为背景, 对另一个数据库进行分类, 确定此数据集中的差异基因. 为了避免假阳性, 我们采用了很多的算法进行分类, 如支持向量机、贝叶斯复合协变量和对角线性判别分析等, 且差异基因必须满足严格的条件, 使肿瘤组织和正常组织100%分离, 且P<0.01. 这个方法不仅避免了通常以计算两个研究的差异基因交叉部分为结果所引起的假阳性, 而且通过整合两项研究的结果得到了真正的肿瘤相关基因.
转录子是调控基因表达的主要因子. 几种方法可以用来整合基因表达数据和其他类型基因组数据以鉴定转录因子靶基因, 其中GRAM、ReMO Discovery和COGRIM就是整合基因表达、ChIP-chip和motif数据寻找基因调控模块的方法[16-18]. ChIP-chip可全基因组检测, 但是他费力, 费时, 并且价格昂贵, 至今为止尚缺乏有关ESCC的ChIP-chip的数据. 位置权矩阵常用来确定已知转录因子结合位点, 但易产生假阳性和假阴性. 我们采用Ahmed Essaghir's的方法, 所利用的转录因子靶基因数据库是经过高质量实验验证, 用Hypergeometric公式进行Fisher's test计算P值[8]. 我们筛选出了8个重要的转录因子. 研究表明这些转录因子和肿瘤发生、发展有关. 其中Myc是一个癌基因, 他特异地影响肿瘤浸润和迁移调节肿瘤转移, 这个基因也被Catenin/TCF调节[19]. GL1与ESCC细胞生存与放化疗敏感性密切相关, 是hedgehog通路组份[20]. SP1通过调节细胞周期相关基因和血管内皮生长因子在多肿瘤生长和转移过程中起到了非常关键作用[21]. Foxo是直接影响到细胞增殖、氧化应激和肿瘤发生的转录因子家族, 其中, Foxo3是肿瘤抑制因子, 研究显示在肿瘤细胞核中Foxo3的积累与化疗敏感性增加及食管癌患者存活率提高密切相关[22].
为了研究食管癌中转录因子的作用, 除了靶基因外, 我们查找了其上游分子, 并通过PPI数据库, 从差异基因中获得与之相互作用的蛋白分子基因. 这些分子和转录因子一起共有110个, 他们参与了一系列重要的与肿瘤的发生有关的生物过程和信号通路, 如25个基因参与了细胞生物周期, 19个基因参与了癌通路. 进一步研究发现, 从KEGG的角度, 这110个基因与463个食管癌相关基因所起作用没有明显的区别, 这意味着这110个基因在通路中的作用能够替代463个食管癌相关基因.
为了研究转录因子之间的关系, 构建了110个基因的分子网络. 首先, 转录因子和他们的靶基因、结合因子以及上游酶类相互联系成154对. 然后我们研究了110个基因(包括8个转录因子)相互关系, 发现有162对, 他或共表达, 或结合、或催化、或激活或抑制, 如FOXM1结合并抑制FOXO3, 调节CDC25B、NEK2、PLU的表达. 最后, 我们研究了转录因子之间的相互关系, 发现: ETV4与SP1形成一个复合物, SP1和β-蛋白/TCF介导c-Myc的表达; Myc抑制FOXO3的活性[23-25]. 这样我们构建了一个由110个点和316条边组成的分子网络.
总之, 我们从基因表达的数据中富集了8个重要的转录因子, 这些因子通过靶基因和相互作用的基因在食管鳞癌中起到非常重要的作用, 并组成了一个相互作用的复杂网络. 本研究为理解食管癌基因变化及转录因子作用提供了有用信息. 今后, 我们将通过实验证明这些结论.
利用基因芯片技术能够成功地判断出和生存率、组织学分级、浸润状态及对化疗反应等临床参数相关的食管癌亚型, 但目前很少有探究差异基因表达改变的原因和差异表达基因之间的相互联系, 转录因子调控基因转录的重要因子, 不少像GSEA这样的工具为从共表达基因中富集转录因子提供了可能.
张志坚, 教授, 福州总医院消化内科
几种方法可以用来整合基因表达数据和其他类型基因组数据以鉴定转录因子靶基因, 其中GRAM、ReMO Discovery和COGRIM就是整合基因表达、ChIP-chip和motif数据寻找基因调控模块的方法.
Ahmed等建立了转录因子靶基因数据库是经过高质量实验验证, 用Hypergeometric公式进行Fisher's test计算P值, 富集表达基因的转录因子, Ma等报道系统挖掘了基因表达谱的顺贩子调控原件以揭示转录条调控机制.
利用生物信息学方法从食管鳞癌差异表达基因中富集出转录因子, 根据转录因子目的基因、结合蛋白和差异基因的相互作用来分析其调控作用, 并建立了相互作用分子网络.
所富集的8个转录因子及其相互作用的基因在食管鳞癌中起到非常重要的作用, 有助于揭示食管癌发生、发展机制, 并可作为药物开发的靶点.
本文技术路线先进, 成熟, 结果可信, 结论客观, 对后续的研究有一定的指导作用.
编辑: 田滢 电编: 闫晋利
| 1. | Parkin DM, Bray F, Ferlay J, Pisani P. Global cancer statistics, 2002. CA Cancer J Clin. 2005;55:74-108. [PubMed] |
| 2. | Enzinger PC, Mayer RJ. Esophageal Cancer. N Engl J Med. 2003;349:2241-2252. [DOI] |
| 3. | Ogawa R, Ishiguro H, Kuwabara Y, Kimura M, Mitsui A, Mori Y, Mori R, Tomoda K, Katada T, Harada K. Identification of candidate genes involved in the radiosensitivity of esophageal cancer cells by microarray analysis. Dis Esophagus. 2008;21:288-297. [PubMed] [DOI] |
| 4. | Sato T, Iizuka N, Hamamoto Y, Yoshino S, Abe T, Takeda S, Uchimura S, Miyamoto T, Sei F, Hamada K. Esophageal squamous cell carcinomas with distinct invasive depth show different gene expression profiles associated with lymph node metastasis. Int J Oncol. 2006;28:1043-1055. [PubMed] |
| 5. | Defrance M, Touzet H. Predicting transcription factor binding sites using local over-representation and comparative genomics. BMC Bioinformatics. 2006;7:396. [PubMed] |
| 6. | Hestand MS, van Galen M, Villerius MP, van Ommen GJ, den Dunnen JT, 't Hoen PA. CORE_TF: a user-friendly interface to identify evolutionary conserved transcription factor binding sites in sets of co-regulated genes. BMC Bioinformatics. 2008;9:495. [PubMed] [DOI] |
| 7. | Ma Q, Chirn GW, Szustakowski JD, Bakhtiarova A, Kosinski PA, Kemp D, Nirmala N. Uncovering mechanisms of transcriptional regulations by systematic mining of cis regulatory elements with gene expression profiles. BioData Min. 2008;1:4. [PubMed] [DOI] |
| 8. | Essaghir A, Toffalini F, Knoops L. et al.Transcription factor regulation can be accurately predicted from the presence of target gene signatures in microarray gene expression data. Nucleic Acids Res. 2010;38:e120. [DOI] |
| 9. | Keshava Prasad TS, Goel R, Kandasamy K, Keerthikumar S, Kumar S, Mathivanan S, Telikicherla D, Raju R, Shafreen B, Venugopal A. Human Protein Reference Database--2009 update. Nucleic Acids Res. 2009;37:D767-D772. [PubMed] [DOI] |
| 10. | Aranda B, Achuthan P, Alam-Faruque Y, Armean I, Bridge A, Derow C, Feuermann M, Ghanbarian AT, Kerrien S, Khadake J. The IntAct molecular interaction database in 2010. Nucleic Acids Res. 2010;38:D525-D531. [PubMed] [DOI] |
| 11. | Xenarios I, Salwínski L, Duan XJ, Higney P, Kim SM, Eisenberg D. DIP, the Database of Interacting Proteins: a research tool for studying cellular networks of protein interactions. Nucleic Acids Res. 2002;30:303-305. [PubMed] |
| 12. | Smoot ME, Ono K, Ruscheinski J, Wang PL, Ideker T. Cytoscape 2.8: new features for data integration and network visualization. Bioinformatics. 2011;27:431-432. [PubMed] [DOI] |
| 13. | Su H, Hu N, Yang HH, Wang C, Takikita M, Wang QH, Giffen C, Clifford R, Hewitt SM, Shou JZ. Global gene expression profiling and validation in esophageal squamous cell carcinoma and its association with clinical phenotypes. Clin Cancer Res. 2011;17:2955-2966. [PubMed] [DOI] |
| 14. | Kashyap MK, Marimuthu A, Kishore CJ, Peri S, Keerthikumar S, Prasad TS, Mahmood R, Rao S, Ranganathan P, Sanjeeviah RC. Genomewide mRNA profiling of esophageal squamous cell carcinoma for identification of cancer biomarkers. Cancer Biol Ther. 2009;8:36-46. [PubMed] |
| 15. | Hong F, Breitling R. A comparison of meta-analysis methods for detecting differentially expressed genes in microarray experiments. Bioinformatics. 2008;24:374-382. [PubMed] [DOI] |
| 16. | Bar-Joseph Z, Gerber GK, Lee TI, Rinaldi NJ, Yoo JY, Robert F, Gordon DB, Fraenkel E, Jaakkola TS, Young RA. Computational discovery of gene modules and regulatory networks. Nat Biotechnol. 2003;21:1337-1342. [PubMed] |
| 17. | Chen G, Jensen ST, Stoeckert CJ. Clustering of genes into regulons using integrated modeling-COGRIM. Genome Biol. 2007;8:R4. [PubMed] |
| 18. | Lemmens K, Dhollander T, De Bie T, Monsieurs P, Engelen K, Smets B, Winderickx J, De Moor B, Marchal K. Inferring transcriptional modules from ChIP-chip, motif and microarray data. Genome Biol. 2006;7:R37. [PubMed] |
| 19. | Zhao L, Shan B, Du Y, Wang M, Liu L, Ren FZ. Periplocin from Cortex periplocae inhibits cell growth and down-regulates survivin and c-myc expression in colon cancer in vitro and in vivo via beta-catenin/TCF signaling. Oncol Rep. 2010;24:375-383. [PubMed] |
| 20. | Zhu W, You Z, Li T, Yu C, Tao G, Hu M, Chen X. Correlation of hedgehog signal activation with chemoradiotherapy sensitivity and survival in esophageal squamous cell carcinomas. Jpn J Clin Oncol. 2011;41:386-393. [PubMed] [DOI] |
| 21. | Li L, Davie JR. The role of Sp1 and Sp3 in normal and cancer cell biology. Ann Anat. 2010;192:275-283. [PubMed] |
| 22. | Chen MF, Fang FM, Lu CH, Lu MS, Chen WC, Lee KD, Lin PY. Significance of nuclear accumulation of Foxo3a in esophageal squamous cell carcinoma. Int J Radiat Oncol Biol Phys. 2008;71:1220-1229. [PubMed] [DOI] |
| 23. | Jiang J, Wei Y, Shen J, Liu D, Chen X, Zhou J, Zong H, Yun X, Kong X, Zhang S. Functional interaction of E1AF and Sp1 in glioma invasion. Mol Cell Biol. 2007;27:8770-8782. [PubMed] |
| 24. | Modak SP, Principaud E, Spohr G. Regulation of Xenopus c-myc promoter activity in oocytes and embryos. Oncogene. 1993;8:645-654. [PubMed] |
| 25. | Chandramohan V, Mineva ND, Burke B, Jeay S, Wu M, Shen J, Yang W, Hann SR, Sonenshein GE. c-Myc represses FOXO3a-mediated transcription of the gene encoding the p27(Kip1) cyclin dependent kinase inhibitor. J Cell Biochem. 2008;104:2091-2106. [PubMed] [DOI] |