修回日期: 2012-06-30
接受日期: 2012-07-20
在线出版日期: 2012-08-08
目的: 探讨针对乙型肝炎病毒(hepatitis B virus, HBV) preS1 dsDNA同聚嘌呤区设计反基因锁核酸分子, 并观察其在HepG2 2.2.15细胞内抑制病毒复制的效果.
方法: 针对HBV preS1 dsDNA的2 941-2 962 nt、3 015-3 036 nt和3 089-3 110 nt三个同聚嘌呤区, 利用RNA structure软件分别设计合成锁核酸、硫代寡核苷酸、未修饰寡核苷酸及无关对照序列, 以阳离子脂质体介导转染HepG2 2.2.15细胞, 采用荧光定量聚合酶链反应技术(FQ-PCR)和时间分辨免疫荧光技术(TRFIA)分别监测1、3、5和7 d细胞培养上清液中HBV DNA和HBsAg的含量; 四甲基偶氮唑蓝(MTT)法检测锁核酸对细胞代谢的影响.
结果: 反基因锁核酸对细胞内的HBV DNA复制与HBsAg表达有明显的抑制作用, 且抑制率随时间呈增高趋势, 7 d后抑制率分别为64.32%和67.51%. 各实验组与对照组比较差异均具有统计学意义(均P<0.05), 而封闭2 941-2 962 nt同聚嘌呤靶区的LNA抑制作用最强, 且最适序列长度为20-30 bp. LNA对细胞代谢无明显影响.
结论: 针对preS1 dsDNA同聚嘌呤区的反基因锁核酸分子, 体外能有效抑制HBV的复制, 以封闭2 941-2 962 nt靶位效果最强, 且合适序列长度为20-30 bp.
引文著录: 邓益斌, 温旺荣. HBV preS1反基因锁核酸的设计及体外对病毒复制的抑制. 世界华人消化杂志 2012; 20(22): 2024-2029
Revised: June 30, 2012
Accepted: July 20, 2012
Published online: August 8, 2012
AIM: To investigate the inhibitory effects of locked nucleic acid (LNA) antisense oligonucleotides targeting the purine region of the hepatitis B virus (HBV) preS1 gene in HepG2 2.2.15 cells, and to screen effective LNA anti-gene oligonucleotides.
METHODS: LNA anti-gene oligonucleotides of different lengths that were complementary to the purine-abundant regions (2 941-2 962 nt, 3 015-3 036 nt and 3 089-3 110 nt) of the HBV preS1 gene were designed, synthesized, and introduced into HepG2 2.2.15 cells by cationic liposome-mediated transfection. Hepatitis B surface antigen (HBsAg) and HBV DNA levels in cell supernatants were tested by time-resolved fluorescence immune assay (TRFIA) and fluorescent quantitative polymerase chain reaction (FQ-PCR) 1, 3, 5 and 7 d after transfection. The cell toxicity of LNA anti-gene oligonucleotides was detected by methyl thiazolyl tetrazolium (MTT) assay.
RESULTS: LNA anti-gene oligonucleotides targeting the HBV preS1 gene showed strong inhibitory effects on HBV DNA replication and HBsAg expression in vitro, and the effects were time-dependent. Seven days after transfection, the reduced rates of HBV DNA and HBsAg levels were 64.32% and 67.51%, respectively. The inhibitory effects were significantly different between each experimental group and control group (all P < 0.05). The inhibitory effect of the LNA anti-gene oligonucleotide targeting the region 2 941-2 962 nt was most strong. The optimal length of LNA anti-gene oligonucleotides ranges from 20 to 30 bases. No obvious cell toxicity was observed with LNA anti-gene oligonucleotides.
CONCLUSION: LNA anti-gene oligonucleotides targeting the HBV preS1 gene showed strong inhibitory effects on HBV replication in vitro. The inhibitory effect of the LNA anti-gene oligonucleotide targeting the region 2 941-2 962 nt was most strong, and the optimal length of LNA anti-gene oligonucleotides ranges from 20 to 30 bases.
- Citation: Deng YB, Wen WR. Antiviral effects of locked nucleic acid antisense oligonucleotides targeting the HBV preS1 gene in HepG2 2.2.15 cells. Shijie Huaren Xiaohua Zazhi 2012; 20(22): 2024-2029
- URL: https://www.wjgnet.com/1009-3079/full/v20/i22/2024.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v20.i22.2024
反基因治疗(anti-gene therapy)是由特定寡聚脱氧核苷酸与靶双链DNA同聚嘧啶或同聚嘌呤区专一性结合形成局部三螺旋结构, 阻止靶DNA与聚合酶、转录因子等蛋白结合, 抑制靶基因的复制与表达, 实现"反基因"抑制之目的. 由于遗传信息从DNA流向RNA是一个遗传信息放大的过程, 而反基因治疗是在转录水平发挥作用, 因而比在翻译水平发挥作用的反义核酸疗法更为有效. 锁核酸(locked nucleic acid, LNA)是新发现的一种带环状结构的核苷酸衍生物[1-4], 与其他寡核苷酸相比, 具有更高的热稳定性[5-8]、更好的分子杂交能力[9-12]、更强的抗核酸酶降解能力[13-16]、更好的脂溶性[17-20]和低细胞毒性[21-25]等优势. 前期研究结果表明, 针对乙型肝炎病毒(hepatitis B virus, HBV) S[26-29]和preS2[30]mRNA单链的反义锁核酸片段可到达肝细胞质内, 发挥抑制HBV复制的作用. 为了进一步观察锁核酸能否进入肝细胞核内, 抑制病毒cccDNA的复制与表达, 本研究针对HBV preS1 dsDNA三个同聚嘌呤区分别设计合成"反基因"锁核酸序列, 脂质体介导转染HepG2 2.2.15细胞株, 并观察其抗病毒效果, 旨在寻找新型抗HBV药物分子.
HepG2 2.2.15细胞为HBV DNA(ayr亚型)全基因转染肝癌细胞株, 能稳定分泌HBV表面抗原(HBsAg), 由中国人民解放军广州军区空军医院刘光泽博士惠赠, 右江民族医学院临床生化与分生教研室常规培养于含G418(380ITI1)、100 mL/L胎牛血清的DMEM培养基中, 37 ℃、50 mL/L CO2条件下每5-6 d传代1次; DMEM培养基、G418等购自GIBCO公司; 胎牛血清购自杭州四季清公司; LipofectamineTM2000购自美国invitrogen公司; 荧光定量PCR检测HBV DNA试剂盒购自深圳匹基公司; HBV表面抗原(HBsAg)定量检测试剂盒购自苏州新波生物技术有限公司; 时间分辨免疫荧光测定仪购自美国Wallac公司; 实时定量PCR仪购自美国ABI公司.
1.2.1 反基因LNA与反义LNA的合成与修饰: 从NCBI基因库中搜索到HBV ayr亚型全基因组序列(3.2 kb DNA, NC: 003977.1, GI: 21326584), 利用RNA structure软件针对编码链的2 941-2 962 nt、3 015-3 036 nt和3 089-3 110 nt三个同聚嘌呤区分别设计5条长度不等的反基因寡核苷酸序列: (1)2 941-2 962 nt同聚嘌呤区: 5'-gAAcgcAgggAccaA-3'(15 bp, 定义为SQⅠ1), 5'-gAAcgcAgggtccaActggA-3'(20 bp, 定义为SQⅠ2), 5'-gAAcgc Agggt ccaA ctgg tgAcgg-3'(25 bp, 定义为SQⅠ3), 5'-gaAcgcAgggtccaActggtgAcggtAagc-3'(30 bp, 定义为SQⅠ4), 5'-gaAcgcAgggtccaActggtgAcggtaAgcttacc-3'(35 bp, 定义为SQⅠ5); (2)3015-3036 nt同聚嘌呤区: 5'-AaaTgcTccgcTccT-3'(15 bp, 定义为SQⅡ1), 5'-aaaTgcTccgcTccTtaccT-3'(20 bp, 定义为SQⅡ2), 5'-aaaTgcTccgcTccTtaccTgaacg-3'(25 bp, 定义为SQⅡ3), 5'-aaaTgcTccgcTccttacc TgaacgAggca-3'(30 bp, 定义为SQⅡ4), 5'-aaaTgcTccgcTccttaccTgaacgAggcaggcat-3'(35 bp, 定义为SQⅡ5); (3)3 089-3 110 nt同聚嘌呤区: 5'-AgTgtTgtcaAtaTg-3'(15 bp, 定义为SQⅢ1), 5'-AgTgtTgtcaataTgcccTg-3'(20 bp, 定义为SQⅢ2), 5'-AgTgtTgtcaataTgcccTgagcct-3'(25 bp, 定义为SQⅢ3), 5'-agTgtTgtcaataTgcccTgagccTtgggc-3'(30 bp, 定义为SQⅢ4), 5'-agTgttgtca ataTgcccTgagccTtgggccAatg-3'(35 bp, 定义为SQⅢ5); (4)结合非同聚嘌呤区序列: 5'-AttgggA acagaAagaTtcgTcggc-3'(25 bp, 定义为SQⅣ); (5)无关序列: 5'-actaattctgggtatccctaggccg-3'(25 bp, 定义为SQⅤ). 以上各序列中, 5个大写字母代表LNA或硫代修饰, 其余小写字母代表DNA, 下划线部分互补于dsDNA的同聚嘌呤区. 各序列经BLAST排除与人同源后由美国Genelink公司合成修饰并纯化.
1.2.2 实验分组与脂质体转染: 本实验设对照组和实验组. 对照组包括空白对照组、无关序列组、未修饰寡核苷酸组和结合非同聚嘌呤区序列组. 实验组包括: (1)互补2 941-2 962 nt同聚嘌呤区序列(SQⅠ1-5): 脂质体-LNA组、脂质体-硫代修饰寡核苷酸组; (2)互补3 015-3 036 nt同聚嘌呤区序列(SQⅡ1-5): 脂质体-LNA组、脂质体-硫代修饰寡核苷酸组; (3)互补3 089-3 110 nt同聚嘌呤区序列(SQⅢ1-5): 脂质体-LNA组、脂质体-硫代修饰寡核苷酸组. 将HepG2 2.2.15细胞按1×105个/mL接种于96孔培养板, 每孔100 μL, 共设定34个组, 每组各设6个复孔, 待细胞贴壁后吸取上清液(-20 ℃保存备用), 分别在各组每孔中一次性加入含LNA或DNA量为10 μmol/L的DMEM混合液1 mL, 分别于1、3、5、7 d收集各孔培养上清液500 μL, 保存于-20 ℃待测. 转染按脂质体说明书操作.
1.2.3 培养上清液HBsAg含量测定: 采用时间分辨免疫荧光技术检测, 每个样品重复3次, 取均值, 严格按试剂盒说明书和仪器说明书操作, HBsAg浓度以μg/L表示.
1.2.4 培养上清液HBV DNA含量测定: 采用实时荧光定量聚合酶链反应(FQ-PCR)法检测. 取培养上清液50 μL, 加等量体积DNA提取液, 充分混匀, 100 ℃恒温处理10 min, 1 200 r/min离心5 min, 取2 μL上清于PCR反应管中并加入反应液, 总体积共25 μL. 各反应管放入荧光定量PCR仪, 扩增条件: 37 ℃保温2 min, 94 ℃预变性3 min, 94 ℃ 5 s, 60 ℃ 40 s, 共35个循环. 由计算机软件自动分析所收集的荧光信号并计算出定量结果, 采用计算对数平均值的方法计算HBV DNA平均拷贝数.
1.2.5 LNA对细胞的毒性检测: 采用四甲基偶氮唑盐(MTT)比色法检测LNA对细胞代谢活性的影响.
统计学处理 所有数据均采用mean±SD表示, 应用SPSS13.0统计软件处理, 组间比较采用重复测量设计方差分析, P<0.05为差异有统计学意义. 抑制率(%) = (用药前P/N-用药后P/N)/(用药前P/N-2.1)×100%. 其中P/N表示阳性孔数值/阴性孔数值.
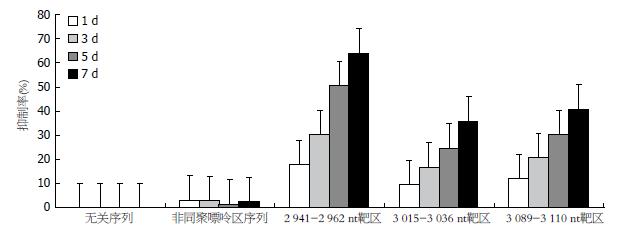
LNA加入1 d后, HBV DNA的复制即开始受抑制, 且抑制率随时间呈增高趋势, 7 d后抑制率最高可达64.32%. 各实验组与对照组比较差异均有统计学意义(均P<0.05), 而封闭2 941-2 962 nt同聚嘌呤区的反基因LNA对HBV DNA复制的抑制效果最明显(表1, 图1).
| 分组 | 0 d | 1 d | 3 d | 5 d | 7 d |
| 空白对照 | 6.102±0.321 | 6.105±0.325 | 6.110±0.316 | 6.108±0.320 | 6.112±0.322 |
| 无关序列对照 | 6.110±0.332 | 6.058±0.360 | 6.120±0.352 | 6.118±0.348 | 6.201±0.355 |
| 非同聚嘌呤区序列 | 6.106±0.318 | 5.915±0.405 | 5.923±0.418 | 6.012±0.401 | 5.951±0.422 |
| 2941-2962 nt靶区 | 6.113±0.325b | 5.024±0.423b | 4.251±0.434b | 3.025±0.402b | 2.181±0.388b |
| 3015-3036 nt靶区 | 6.120±0.344c | 5.524±0.431c | 5.102±0.435c | 4.612±0.429c | 3.910±0.441c |
| 3089-3110 nt靶区 | 6.118±0.335e | 5.382±0.415e | 4.850±0.421e | 4.258±0.418e | 3.621±0.425e |
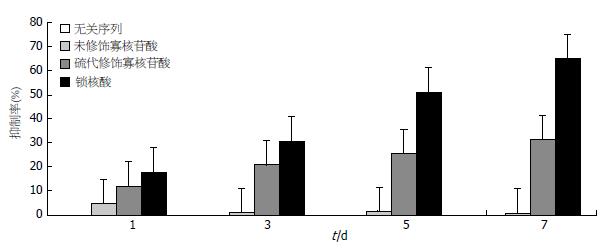
封闭2 941-2 962 nt同一靶位的不同反基因寡核苷酸加入1 d后, 硫代修饰寡核苷酸、锁核酸对HBV DNA的复制开始出现抑制作用, 且抑制率均随时间呈增高趋势. 各实验组与对照组比较差异均有统计学意义(均P<0.05), 其中, 锁核酸对HBV DNA复制的抑制能力最强(表2, 图2).
| 分组 | 0 d | 1 d | 3 d | 5 d | 7 d |
| 空白对照 | 6.102±0.321 | 6.105±0.325 | 6.110±0.316 | 6.108±0.320 | 6.112±0.322 |
| 无关序列 | 6.110±0.332 | 6.058±0.360 | 6.120±0.352 | 6.118±0.348 | 6.201±0.355 |
| 未修饰寡核苷酸 | 6.106±0.318 | 5.815±0.405 | 6.023±0.418 | 6.013±0.401 | 6.051±0.422 |
| 硫代修饰寡核苷酸 | 6.118±0.335a | 5.382±0.415a | 4.850±0.421a | 4.558±0.418a | 4.221±0.425a |
| 锁核酸 | 6.113±0.325c | 5.024±0.423c | 4.251±0.434c | 3.025±0.402c | 2.181±0.388c |
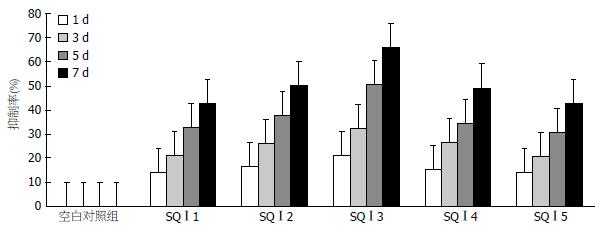
封闭2 941-2 962 nt同一靶位的不同序列长度反基因锁核酸加入1 d后, 开始对HBV DNA的复制发挥抑制作用, 且抑制率均随时间呈增高趋势, 7 d后抑制率最高可达65.91%. 各实验组与对照组比较差异均有统计学意义(均P<0.05), 而对HBV DNA复制的抑制作用最强在SQⅠ2-SQⅠ4组, 即锁核酸序列长度在20-30 bp之间时有最好的抑制效果(表3, 图3).
| 分组 | 0 d | 1 d | 3 d | 5 d | 7 d |
| 空白对照 | 6.102±0.321 | 6.105±0.325 | 6.110±0.316 | 6.108±0.320 | 6.112±0.322 |
| SQⅠ1 | 6.110±0.332a | 5.258±0.360a | 4.820±0.352a | 4.118±0.348a | 3.501±0.355a |
| SQⅠ2 | 6.106±0.318c | 5.115±0.405c | 4.523±0.418c | 3.812±0.401c | 3.051±0.422c |
| SQⅠ3 | 6.113±0.325e | 4.824±0.423e | 4.151±0.434e | 3.025±0.402e | 2.081±0.388e |
| SQⅠ4 | 6.120±0.344g | 5.180±0.431g | 4.502±0.435g | 4.012±0.429g | 3.110±0.441g |
| SQⅠ5 | 6.118±0.335i | 5.282±0.415i | 4.850±0.421i | 4.258±0.418i | 3.521±0.425i |
封闭2 941-2 962 nt同一靶位的不同反基因寡核苷酸加入3 d后, 锁核酸对HBsAg的表达开始发挥抑制作用, 且抑制率均随时间呈增高趋势, 7 d后抑制率达67.51%. 各实验组与对照组比较差异均有统计学意义(均P<0.05), 而锁核酸对HBsAg表达的抑制作用最强(表4).
| 分组 | 0 d | 1 d | 3 d | 5 d | 7 d |
| 空白对照 | 223.45±2.321 | 222.38±2.325 | 223.64±2.316 | 227.41±2.320 | 224.31±2.322 |
| 无关序列 | 226.51±2.332 | 225.48±2.360 | 224.71±2.352 | 223.87±2.348 | 224.55±2.355 |
| 未修饰寡核苷酸 | 225.45±2.318 | 221.32±2.405 | 230.15±2.418 | 226.78±2.401 | 224.55±2.422 |
| 硫代修饰寡核苷酸 | 224.46±2.335a | 201.11±2.415a | 169.41±2.421a | 154.33±2.418a | 125.28±2.425a |
| 锁核酸 | 226.5±2.325c | 186.30±2.423c | 154.50±2.434c | 110.40±2.402c | 73.62±2.388c |
用药7 d后, 采用MTT比色法检测锁核酸组A值为1.184±0.043, 与对照组(A值为1.205±0.038)比较差异无统计学意义(P>0.05), 表明实验范围内锁核酸脂质体混合物对细胞代谢基本无影响.
S区是HBV基因的一个重要开放读码框, 包括preS1、preS2和S三个基因, 共同编码的大蛋白质分子是HBV表面抗原(hepatitis B virus surface antigen, HBsAg)的主要组成部分, 不仅参与病毒的复制、转录、装配和分泌过程, 还与病毒感染引起的细胞及体液免疫应答反应有密切关系.
本研究针对HBV preS1 dsDNA编码链的2 941-2 962 nt、3 015-3 036 nt和3 089-3 110 nt三个同聚嘌呤区设计合成5条序列长度不等的反基因锁核酸分子, 由阳离子脂质体介导转染HepG2 2.2.15细胞株, 通过检测细胞培养上清液中的HBV DNA和HBsAg含量等指标来评价其疗效. 结果显示, 封闭2 941-2 962 nt同聚嘌呤区的反基因锁核酸分子, 体外抑制preS1基因复制与表达的作用最强, 抑制率分别为HBV DNA 65.91%、HBsAg 67.51%, 且抑制作用随时间呈递增趋势. 不同序列长度锁核酸对HBV DNA复制的抑制效果实验结果显示, 锁核酸分子的序列长度在20-30 bp时有最好的抑制效果, 这说明只有序列长度合适的锁核酸分子, 才能有效通过核孔进入细胞核内, 识别并结合到HBV dsDNA的同聚嘌呤或同聚嘧啶靶区形成三链杂交分子, 从而"封闭"病毒靶基因的复制和表达, 这为HBV反基因治疗研究提供了理论与实验依据. 然而, 由于HBV DNA聚合酶缺乏校对功能, 在病毒复制过程中容易产生变异株而导致病毒对药物治疗敏感性下降或耐药现象, 因此, 尚需要研究多基因靶位抗HBV药物或疗法. 此外, MTT检测结果证实锁核酸对细胞的代谢活性无明显毒性作用.
总之, 针对preS1 dsDNA同聚嘌呤区的反基因锁核酸分子, 体外能有效抑制HBV的复制, 以封闭2 941-2 962 nt靶位效果最强, 且合适序列长度在20-30 bp之间, 这既为HBV治疗提供有效靶位, 也为反基因治疗提供理论和实验依据.
锁核酸(LNA)是近几年来新发现的带环状结构的核苷酸衍生物, 具有稳定性好, 分子杂交能力强, 抗核酸酶降解能力高, 脂溶性好和低细胞毒性等特点, 因而具有广阔的研究与应用前景.
朱传武, 主任医师, 苏州市第五人民医院
反基因技术是在反义核酸技术基础上发展起来的一种新型基因治疗技术, 因其能在复制和转录水平发挥阻断作用, 从而成为目前基因治疗研究的热点. 但由于外源性寡核苷酸分子在细胞内易被核酸酶降解, 因而寻找增强外源性核酸药物分子稳定性的修饰方法成为目前亟待解决的问题.
本研究尝试将LNA分子引入针对dsDNA的反基因寡核苷酸分子结构内, 以期通过增强外源核酸分子的稳定性以及寻找有效抑制HBV复制的反基因治疗靶位, 为HBV反基因治疗研究提供理论与实验依据.
本研究针对HBV preS1 dsDNA同聚嘌呤区设计合成的反基因锁核酸序列, 体外能有效抑制HBV preS1基因的复制与表达, 并筛选出有效治疗靶位和适宜序列长度范围, 可为抗HBV反基因治疗的进一步研究提供理论和实验依据.
反基因治疗(anti-gene therapy): 由特定寡聚脱氧核苷酸与靶dsDNA同聚嘧啶或同聚嘌呤区专一性结合形成局部三螺旋结构, 阻止靶DNA与聚合酶、转录因子等蛋白结合, 从而实现抑制靶基因复制与表达的目的. 为了跟以mRNA单链分子为作用靶位的反义核酸技术相区别, 故称之为反基因技术(或疗法).
本研究发现反基因LNA分子能够有效抑制HepG2.2.15细胞的HBV复制和病毒蛋白表达, 对开展HBV的反基因治疗具有一定的意义, 具有较好的学术价值.
编辑: 张姗姗 电编:闫晋利
| 1. | Crouzier L, Dubois C, Edwards SL, Lauridsen LH, Wengel J, Veedu RN. Efficient reverse transcription using locked nucleic acid nucleotides towards the evolution of nuclease resistant RNA aptamers. PLoS One. 2012;7:e35990. [PubMed] [DOI] |
| 2. | Hildebrandt-Eriksen ES, Aarup V, Persson R, Hansen HF, Munk ME, Orum H. A Locked Nucleic Acid Oligonucleotide Targeting MicroRNA 122 Is Well-Tolerated in Cynomolgus Monkeys. Nucleic Acid Ther. 2012;22:152-161. [PubMed] |
| 3. | Mishra S, Ghosh S, Mukhopadhyay R. Ordered self-assembled locked nucleic acid (LNA) structures on gold(111) surface with enhanced single base mismatch recognition capability. Langmuir. 2012;28:4325-4333. [PubMed] [DOI] |
| 4. | Robertson KL, Vora GJ. Locked nucleic acid flow cytometry-fluorescence in situ hybridization (LNA flow-FISH): a method for bacterial small RNA detection. J Vis Exp. 2012;e3655. [PubMed] |
| 5. | Hull C, Szewcyk C, St John PM. Effects of locked nucleic acid substitutions on the stability of oligonucleotide hairpins. Nucleosides Nucleotides Nucleic Acids. 2012;31:28-41. [PubMed] [DOI] |
| 6. | Owczarzy R, You Y, Groth CL, Tataurov AV. Stability and mismatch discrimination of locked nucleic acid-DNA duplexes. Biochemistry. 2011;50:9352-9367. [PubMed] [DOI] |
| 7. | Bhattacharyya J, Maiti S, Muhuri S, Nakano S, Miyoshi D, Sugimoto N. Effect of locked nucleic acid modifications on the thermal stability of noncanonical DNA structure. Biochemistry. 2011;50:7414-7425. [PubMed] [DOI] |
| 8. | Doessing H, Vester B. Locked and unlocked nucleosides in functional nucleic acids. Molecules. 2011;16:4511-4526. [PubMed] [DOI] |
| 9. | Zhang Y, Qu Z, Kim S, Shi V, Liao B, Kraft P, Bandaru R, Wu Y, Greenberger LM, Horak ID. Down-modulation of cancer targets using locked nucleic acid (LNA)-based antisense oligonucleotides without transfection. Gene Ther. 2011;18:326-333. [PubMed] [DOI] |
| 10. | Chabot S, Orio J, Castanier R, Bellard E, Nielsen SJ, Golzio M, Teissié J. LNA-based Oligonucleotide Electrotransfer for miRNA Inhibition. Mol Ther. 2012; May 22. [Epub ahead of print]. [PubMed] [DOI] |
| 11. | Johannsen MW, Veedu RN, Madsen AS, Wengel J. Enzymatic polymerisation involving 2'-amino-LNA nucleotides. Bioorg Med Chem Lett. 2012;22:3522-3526. [PubMed] [DOI] |
| 12. | Chaubey AK, Dubey KD, Ojha RP. Stability and free energy calculation of LNA modified quadruplex: a molecular dynamics study. J Comput Aided Mol Des. 2012;26:289-299. [PubMed] [DOI] |
| 13. | Sun Z, Xiang W, Guo Y, Chen Z, Liu W, Lu D. Inhibition of hepatitis B virus (HBV) by LNA-mediated nuclear interference with HBV DNA transcription. Biochem Biophys Res Commun. 2011;409:430-435. [PubMed] [DOI] |
| 14. | Moschos SA, Frick M, Taylor B, Turnpenny P, Graves H, Spink KG, Brady K, Lamb D, Collins D, Rockel TD. Uptake, efficacy, and systemic distribution of naked, inhaled short interfering RNA (siRNA) and locked nucleic acid (LNA) antisense. Mol Ther. 2011;19:2163-2168. [PubMed] [DOI] |
| 15. | Ivanova MI, Shivarov VS, Hadjiev EA, Naumova EJ. Novel multiplex bead-based assay with LNA-modified probes for detection of MPL exon 10 mutations. Leuk Res. 2011;35:1120-1123. [PubMed] [DOI] |
| 16. | Seth PP, Allerson CR, Siwkowski A, Vasquez G, Berdeja A, Migawa MT, Gaus H, Prakash TP, Bhat B, Swayze EE. Configuration of the 5'-Methyl Group Modulates the Biophysical and Biological Properties of Locked Nucleic Acid (LNA) Oligonucleotides. J Med Chem. 2010; Nov 8. [Epub ahead of print]. [PubMed] [DOI] |
| 17. | Diercks S, Gescher C, Metfies K, Medlin LK. Evaluation of locked nucleic acids for signal enhancement of oligonucleotide probes for microalgae immobilized on solid surfaces. J Appl Phycol. 2009;21:657-668. [DOI] |
| 18. | Chlebowski R. Lifestyle change including dietary fat reduction and breast cancer outcome. J Nutr. 2007;137:233S-235S. [PubMed] |
| 19. | Yang CJ, Wang L, Wu Y, Kim Y, Medley CD, Lin H, Tan W. Synthesis and investigation of deoxyribonucleic acid/locked nucleic acid chimeric molecular beacons. Nucleic Acids Res. 2007;35:4030-4041. [PubMed] [DOI] |
| 20. | Brunet E, Corgnali M, Cannata F, Perrouault L, Giovannangeli C. Targeting chromosomal sites with locked nucleic acid-modified triplex-forming oligonucleotides: study of efficiency dependence on DNA nuclear environment. Nucleic Acids Res. 2006;34:4546-4553. [PubMed] [DOI] |
| 21. | Levin JD, Fiala D, Samala MF, Kahn JD, Peterson RJ. Position-dependent effects of locked nucleic acid (LNA) on DNA sequencing and PCR primers. Nucleic Acids Res. 2006;34:e142. [PubMed] [DOI] |
| 22. | Johnson MP, Haupt LM, Griffiths LR. Locked nucleic acid (LNA) single nucleotide polymorphism (SNP) genotype analysis and validation using real-time PCR. Nucleic Acids Res. 2004;32:e55. [PubMed] [DOI] |
| 23. | Crinelli R, Bianchi M, Gentilini L, Palma L, Sørensen MD, Bryld T, Babu RB, Arar K, Wengel J, Magnani M. Transcription factor decoy oligonucleotides modified with locked nucleic acids: an in vitro study to reconcile biostability with binding affinity. Nucleic Acids Res. 2004;32:1874-1885. [PubMed] [DOI] |
| 24. | Sørensen JJ, Nielsen JT, Petersen M. Solution structure of a dsDNA: LNA triplex. Nucleic Acids Res. 2004;32:6078-6085. [PubMed] [DOI] |
| 25. | Hertoghs KM, Ellis JH, Catchpole IR. Use of locked nucleic acid oligonucleotides to add functionality to plasmid DNA. Nucleic Acids Res. 2003;31:5817-5830. [PubMed] [DOI] |