修回日期: 2012-05-03
接受日期: 2012-05-13
在线出版日期: 2012-06-08
目的: 研究直肠癌microRNA(miRNA)表达谱, 以期发现直肠癌发生过程和正常直肠黏膜组织的miRNA差异表达谱, 并理论上预测可能受其调控的靶基因.
方法: 选择2011-01/03于中国人民解放军第306医院肠镜检查的进展期直肠癌患者, 对直肠癌及正常组织进行miRNA芯片检测, 筛选直肠癌、正常直肠组织差异表达的miRNA, 并运用Gomir分析软件筛选TargetScan、PicTar、miRanda 3个预测软件同时预测出的靶基因.
结果: 选择直肠癌组织、正常直肠黏膜组织标本各2例, 通过Affymetrix miRNAs芯片检测, 发现直肠腺癌与正常直肠黏膜相比, 上调的miRNA为miR-15a、miR-18a、miR-18a*、miR-21、miR-24-2*、miR-27a等37种, 下调的为miR-139-5p、miR-139-3p、miR-215、miR-200b、miR-133a、miR-150等18种. 依据Ratio值, 选择其中最高者miR-31和最低者miR-375进行预测其靶基因. 预测ZFHX4可能为miR-375的靶基因, 预测BACH2、FZD3等可能为miR-31的靶基因.
结论: 直肠癌和正常直肠黏膜相比有其特异的miRNA表达谱, miRNA在直肠癌的发生中可能起到重要作用, 受其调控的靶基因及作用方式值得进一步研究.
引文著录: 屈昌民, 梁淑文, 李连勇, 曹艳菊, 王晓英, 翟静妤, 宫淑娟, 刘庆森. MicroRNA在直肠癌中的表达谱. 世界华人消化杂志 2012; 20(16): 1407-1414
Revised: May 3, 2012
Accepted: May 13, 2012
Published online: June 8, 2012
AIM: To compare the expression profile of microRNA (miRNA) between rectal adenocarcinoma and normal rectal mucosal tissue to identify genes possibly associated with the development of rectal cancer.
METHODS: MiRNA expression profiling of two rectal adenocarcinoma samples and two normal rectal tissue samples was performed using miRNA microarray. The possible targets of miRNAs were predicated using bioinformatic tools.
RESULTS: Fifty-five miRNAs were found to be differentially expressed between rectal adenocarcinoma and normal tissues, with thirty-seven miRNAs up-regulated while eighteen down-regulated. Bioinformatic analyses indicated that ZFHX4 may be the target of miR-375, and that BACH2 and FZD3 may be the targets of miR-31.
CONCLUSION: There is a specific miRNA expression profile in rectal adenocarcinoma. MiRNAs play an important role in the development of rectal adenocarcinoma. Target mRNAs were primarily identified and worthy of further study.
- Citation: Qu CM, Liang SW, Li LY, Cao YJ, Wang XY, Zhai JY, Gong SJ, Liu QS. MiRNA expression profiling of rectal adenocarcinoma. Shijie Huaren Xiaohua Zazhi 2012; 20(16): 1407-1414
- URL: https://www.wjgnet.com/1009-3079/full/v20/i16/1407.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v20.i16.1407
MicroRNA(miRNA)是新发现的一类含有21-25个核苷酸的非编码小分子单链RNA, 通过碱基互补配对的方式与靶基因的3'UTR区部分互补, 抑制转录产物的翻译, 从而调控靶基因的表达, 影响细胞的增殖、分化和凋亡等过程[1-3]. 目前, 国外研究发现多种miRNA在结直肠癌组织中异常表达, 且在结直肠癌的发生过程中不同的miRNA通过不同途径发挥着不同的重要作用[4-6]. 在我国对miRNA研究尚少, 尚无报道直肠癌miRNA的表达谱. 对miRNA的深入研究, 将有助于我们对直肠癌发病机制的理解, 并为其诊治提供理论依据, 具有重大的生物学意义. 为此我们对直肠癌miRNA的表达谱进行初步研究, 以期发现直肠癌的miRNA表达谱, 并对部分差异miRNA进行分析, 运用生物信息学, 预测可能受其调控的靶基因, 以期加深对直肠癌在发生、发展机制的理解, 希望能为今后运用miRNA诊断、乃至治疗直肠癌提供实验依据.
实验所需组织均来自2011-01/03中国人民解放军第306医院消化内镜中心检查的直肠活检组织, 共选择4例, 包括直肠癌组织、正常直肠黏膜组织, 分别编为C1、C2、N1、N2. 分离总RNA: TRIzol Reagent, Invitrogen Life Technologies, P/N 15596-018(美国), MirVana® miRNA isolation Kit, Ambion, AM1560(日本); RNA标签: FlashTagTM Biotin RNA Labeling Kit for Affymetrix® GeneChip® miRNA Arrays(Genisphere, FT30AFYB)(美国), Hybridization Control Kit (Affymetrix, P/N 900454)(美国). 主要实验设备: Affymetrix® Hybridization Oven 640(美国), Affymetrix® Fluidics Station 450(美国), Affymetrix® GeneChip® Scanner 3000(美国). 主要应用软件: Affymetrix® GeneChip® Command ConsoleTM1.1.
1.2.1 组织标本获取: 肠镜检查时发现直肠癌, 应用一次性活检钳对肿瘤组织进行活检. 取8块肿瘤组织立即放置于液氮浸没迅速降温, 6 h后放置于-80 ℃冰箱中储存; 取8块肿瘤组织放置于甲醛溶液中固定, 待HE染色病理检查. 正常直肠黏膜组织取8块立即放置于液氮浸没迅速降温, 6 h后放置于-80 ℃冰箱中储存; 取同部位直肠正常黏膜2块组织放置于甲醛溶液中固定, 待HE染色病理检查.
1.2.2 组织总RNA的抽提及质量检测: 使用TRIzol试剂进行组织总RNA的提取, 后用55 ℃-60 ℃ 50 μL DEPC水溶解RNA样品, 5-10 min. 测吸光度(A)值, 定量RNA浓度, 并行RNA甲醛变性凝胶电泳(5-10 V/cm, 30 min).
1.2.3 miRNA芯片检测: 根据总RNA的起始量选择相应的稀释比例, 依据说明书制备poly(A) Tailing Mix; 然后进行生物素标记miRNA; 取出芯片将其平衡至室温, 杂交液加入到标记好的miRNA体系中, 然后将100 µL杂交液注入至芯片中. 将芯片平衡放置于杂交炉中, 48℃, 60 r/min旋转杂交16 h. 杂交完成后进行芯片清洗染色、扫描. 组织总RNA的抽提及质量检测、miRNA芯片检测步骤由北京博奥生物有限公司完成. 芯片数据显著性分析应用SAM&R软件; 芯片聚类分析应用Cluster3.0软件.
1.2.4 靶基因预测: (1)预测软件. TargetScan(Bartel实验室): http://www.targetscan.org/; PicTar(Raje-
wsky实验室): http://pictar.mdc-berlin.de/; miRanda(Memorial Sloan-Kettering癌症研究中心): http://www.microrna.org/microrna/home.do. 运用Gomir分析软件筛选出3个预测软件同时预测出的靶基因.
1.2.5 直肠癌差异表达miRNA靶基因筛选: 运用该基因名称及直肠癌的主题词于PUBMED网站检索, 筛选出与直肠癌或结肠癌相关的基因.
4例中男女各2例, 年龄60-71岁. C1肠镜表现: 直肠距肛门口1-5 cm可见环半周不规则溃疡, 周边不规则隆起, 表面破溃, 质脆, 触之易出血. C2肠镜表现: 直肠距肛门9-13 cm环周肿物, 肠腔狭窄, 内镜无法通过, 组织较脆, 触之渗血. C1、C2病理诊断均为腺癌.
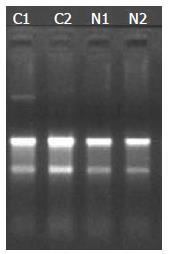
昂飞miRNA表达谱芯片实验要求评价标准为: RNA纯度: A260/280在1.7-2.1之间; RNA总量: 总量≥3 μg; 总RNA完整性: 经甲醛变性凝胶电泳检测, RNA样品电泳28S、18S rRNA条带清晰, 且28S:18S rRNA条带亮度大于或接近2:1. 结果显示, 全部标本RNA完整性及浓度符合下一步实验要求. 组织总RNA提取的完整性及浓度详见表1, 甲醛变性胶电凝泳检测总RNA完整性结果详见图1.
| 组织编号 | A260/280 | A260/230 | 浓度(μg/μL) | 总量(μg) | 样品质量描述 |
| C1 | 2.09 | 2.12 | 0.848 | 39.3 | RNA完好 |
| C2 | 2.06 | 2.16 | 0.606 | 28.0 | RNA完好 |
| N1 | 2.07 | 2.07 | 0.257 | 11.6 | RNA完好 |
| N2 | 2.06 | 2.00 | 0.225 | 10.1 | RNA完好 |
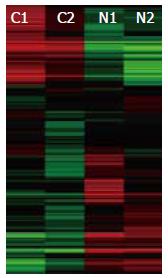
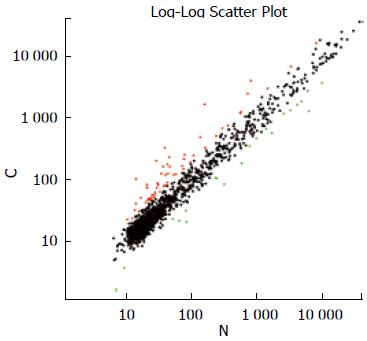
直肠癌患者腺癌组织miRNA表达谱与正常直肠黏膜有明显差异. 聚类分析可以将直肠癌与正常黏膜组织明确区分开(图2). 为了找出正常直肠黏膜、直肠腺癌组织miRNA表达的差异, 将芯片中的数据经归一化后用SAM&R进行统计分析, 按照2倍标准挑选出差异miRNA, 然后对不同组织中表达值有交叉者进行剔除. 直肠腺癌与正常直肠黏膜相比, 上调的miRNA为miR-15a、miR-18a、miR-18a*、miR-21、miR-24-2*、miR-27a等37种, 下调的为miR-139-5p、miR-139-3p、miR-215、miR-200b、miR-133a、miR-150等18种; 直肠腺癌与正常直肠黏膜组织表达差异的miRNA详见表2, 其表达值分布散点图见图3.
| 名称 | C1 | C2 | N1 | N2 | 比值 | 序列 |
| 上调 | ||||||
| miR-15a | 21 | 28 | 13 | 10 | 2.051 | CAGGCCAUAUUGUGCUGCCUCA |
| miR-18a | 2732 | 5333 | 705 | 896 | 5.039 | UAAGGUGCAUCUAGUGCAGAUAG |
| miR-18a* | 116 | 215 | 43 | 28 | 4.658 | ACUGCCCUAAGUGCUCCUUCUGG |
| miR-21 | 207 | 230 | 70 | 80 | 2.903 | CAACACCAGUCGAUGGGCUGU |
| miR-24-2* | 138 | 247 | 132 | 54 | 2.069 | UGCCUACUGAGCUGAAACACAG |
| miR-27a | 171 | 333 | 128 | 82 | 2.406 | AGGGCUUAGCUGCUUGUGAGCA |
| miR-31 | 2774 | 610 | 116 | 196 | 10.845 | AGGCAAGAUGCUGGCAUAGCU |
| miR-92a-1* | 137 | 113 | 41 | 71 | 2.230 | AGGUUGGGAUCGGUUGCAAUGCU |
| miR-29b-1* | 132 | 198 | 55 | 35 | 3.649 | GCUGGUUUCAUAUGGUGGUUUAGA |
| miR-106a | 12612 | 20247 | 7824 | 8532 | 2.009 | AAAAGUGCUUACAGUGCAGGUAG |
| miR-7 | 67 | 35 | 20 | 23 | 2.379 | UGGAAGACUAGUGAUUUUGUUGU |
| miR-182 | 3253 | 1700 | 652 | 771 | 3.480 | UUUGGCAAUGGUAGAACUCACACU |
| miR-183 | 150 | 104 | 44 | 41 | 2.980 | UAUGGCACUGGUAGAAUUCACU |
| miR-221 | 6920 | 6875 | 3409 | 3241 | 2.074 | AGCUACAUUGUCUGCUGGGUUUC |
| miR-223 | 123 | 129 | 23 | 44 | 3.759 | UGUCAGUUUGUCAAAUACCCCA |
| miR-224 | 115 | 246 | 53 | 29 | 4.365 | CAAGUCACUAGUGGUUCCGUU |
| miR-224* | 251 | 420 | 169 | 149 | 2.116 | AAAAUGGUGCCCUAGUGACUACA |
| miR-125b-2* | 55 | 62 | 26 | 22 | 2.417 | UCACAAGUCAGGCUCUUGGGAC |
| miR-188 | 76 | 166 | 63 | 41 | 2.309 | CAUCCCUUGCAUGGUGGAGGG |
| miR-200c | 83 | 79 | 43 | 33 | 2.118 | CGUCUUACCCAGCAGUGUUUGG |
| miR-135b | 20 | 46 | 13 | 13 | 2.532 | AUGUAGGGCUAAAAGCCAUGGG |
| miR-424 | 197 | 268 | 40 | 49 | 5.261 | CAAAACGUGAGGCGCUGCUAU |
| miR-18b | 174 | 336 | 72 | 83 | 3.294 | UAAGGUGCAUCUAGUGCAGUUAG |
| miR-431 | 40 | 55 | 25 | 16 | 2.302 | UGUCUUGCAGGCCGUCAUGCA |
| miR-452 | 59 | 125 | 52 | 31 | 2.228 | AACUGUUUGCAGAGGAAACUGA |
| miR-181d | 154 | 187 | 88 | 52 | 2.434 | AACAUUCAUUGUUGUCGGUGGGU |
| miR-503 | 257 | 404 | 21 | 56 | 8.595 | UAGCAGCGGGAACAGUUCUGCAG |
| miR-509-3p | 39 | 44 | 15 | 12 | 3.035 | UGAUUGGUACGUCUGUGGGUAG |
| miR-552 | 157 | 147 | 86 | 60 | 2.077 | AACAGGUGACUGGUUAGACAA |
| miR-584 | 53 | 109 | 46 | 27 | 2.219 | UUAUGGUUUGCCUGGGACUGAG |
| miR-550* | 153 | 92 | 45 | 34 | 3.118 | UGUCUUACUCCCUCAGGCACAU |
| miR-622 | 97 | 70 | 37 | 22 | 2.814 | ACAGUCUGCUGAGGUUGGAGC |
| miR-421 | 467 | 513 | 148 | 119 | 3.666 | AUCAACAGACAUUAAUUGGGCGC |
| miR-542 | 39 | 57 | 21 | 25 | 2.076 | UCGGGGAUCAUCAUGUCACGAGA |
| miR-1244 | 43 | 74 | 34 | 19 | 2.206 | AAGUAGUUGGUUUGUAUGAGAUGGUU |
| miR-3178 | 4584 | 1515 | 1072 | 1792 | 2.130 | GGGGCGCGGCCGGAUCG |
| miR-3200 | 38 | 62 | 24 | 21 | 2.226 | CACCUUGCGCUACUCAGGUCUG |
| 下调 | ||||||
| miR-139-5p | 75 | 95 | 288 | 331 | 0.276 | UCUACAGUGCACGUGUCUCCAG |
| miR-139-3p | 26 | 18 | 43 | 82 | 0.352 | GGAGACGCGGCCCUGUUGGAGU |
| miR-215 | 393 | 323 | 565 | 955 | 0.471 | AUGACCUAUGAAUUGACAGAC |
| miR-200b | 1279 | 1071 | 2271 | 2827 | 0.461 | UAAUACUGCCUGGUAAUGAUGA |
| miR-133a | 28 | 36 | 61 | 99 | 0.405 | UUUGGUCCCCUUCAACCAGCUG |
| miR-150 | 1892 | 820 | 2894 | 3659 | 0.414 | UCUCCCAACCCUUGUACCAGUG |
| miR-195 | 1669 | 2106 | 5301 | 4240 | 0.396 | UAGCAGCACAGAAAUAUUGGC |
| miR-194* | 565 | 390 | 573 | 1364 | 0.493 | CCAGUGGGGCUGCUGUUAUCUG |
| miR-375 | 1490 | 1099 | 6325 | 6195 | 0.207 | UUUGUUCGUUCGGCUCGCGUGA |
| miR-378* | 207 | 171 | 613 | 546 | 0.326 | CUCCUGACUCCAGGUCCUGUGU |
| miR-378 | 4194 | 3168 | 9494 | 10281 | 0.372 | ACUGGACUUGGAGUCAGAAGG |
| miR-342 | 124 | 96 | 264 | 199 | 0.477 | AGGGGUGCUAUCUGUGAUUGA |
| miR-133b | 22 | 25 | 42 | 59 | 0.469 | UUUGGUCCCCUUCAACCAGCUA |
| miR-422a | 583 | 573 | 2067 | 1291 | 0.344 | ACUGGACUUAGGGUCAGAAGGC |
| miR-574 | 3042 | 2515 | 4567 | 7373 | 0.465 | CACGCUCAUGCACACACCCACA |
| miR-650 | 91 | 121 | 179 | 306 | 0.438 | AGGAGGCAGCGCUCUCAGGAC |
| miR-378c | 1855 | 1442 | 4579 | 3743 | 0.396 | ACUGGACUUGGAGUCAGAAGAGUGG |
| miR-4281 | 642 | 700 | 996 | 1766 | 0.486 | GGGUCCCGGGGAGGGGGG |
依据Ratio值, 选择最高者miR-31和最低者miR-375进行预测其靶基因. miR-31通过筛选发现Targetscan预测miR-31的靶基因为308个, PicTar为165个, miRanda为1 291个, 而3个软件同时预测出的靶基因数目为61个. 之后, 通过PubMed数据库, 检索出其中5个基因可能与直肠癌相关. miR-375通过筛选发现Targetscan预测的靶基因为164个, PicTar为166个, miRanda为278个, 而3个软件同时预测出的靶基因数目为2个, 为ELAVL4和ZFHX4, 其中ZFHX4可能和直肠癌有关(表3). miRNA与预测的靶基因结合位点见表4-9.
| 靶基因 | 基因全称 | 文献数目 | 报道时间 | 预测结合位点 |
| miR-375 | ||||
| ZFHX4 | ZINC FINGER HOMEODOMAIN 4 | 1 | 2010[7] | 表4 |
| miR-31 | ||||
| BACH2 | BTB AND CNC HOMOLOGY 1, BASIC LEUCINE ZIPPER TRANSCRIPTION FACTOR 2 | 1 | 2006[8] | 表5 |
| FZD3 | FRIZZLED HOMOLOG 3 | 1 | 2010[9] | 表6 |
| PEX5 | PEROXISOMAL BIOGENESIS FACTOR 5 | 1 | 1999[10] | 表7 |
| PPP6C | PROTEIN PHOSPHATASE 6, CATALYTIC SUBUNIT | 1 | 2004[11] | 表8 |
| RGS4 | REGULATOR OF G-PROTEIN SIGNALLING 4 | 1 | 2008[12] | 表9 |
| Has-miR-375/ZFHX4 Alignment | Scores |
| 3'agugcgcUCGGCUU--GCUUGUUu 5'hsa-miR-375 | mirSVR score: -0.1512 |
| 1065: 5'cuguuuaAGUACAAGUUGAACAAa 3'ZFHX4 | PhastCons score: 0.7441 |
| Has-miR-31/BACH2 Alignment | Scores |
| 3'ucgaUACGGUCGUAGAACGGa 5'hsa-miR-31 | mirSVR score: -0.0003 |
| 191: 5'aaugGUGCUA-CCCCUUGCCc 3'BACH2 | PhastCons score: 0.6757 |
| Has-miR-31/FZD3 Alignment | Scores |
| 3'ucgAUACGGUCGUAGAACGGa 5'hsa-miR-31 | mirSVR score: -0.9748 |
| 927: 5'cagUGAGAUGUGAUCUUGCCa 3'FZD3 | PhastCons score: 0.7053 |
| Has-miR-31/PEX5 Alignment | Scores |
| 3'ucgauACGG-UCGUAGAACGGa 5'hsa-miR-31 | mirSVR score: -0.4073 |
| 1075: 5'augacUGGUAAGCCUCUUGCCu 3'PPP6C | PhastCons score: 0.6264 |
| Has-miR-31/PPP6C Alignment | Scores |
| 3'ucgaUACGGUCGUAGAACGGa 5'hsa-miR-31 | mirSVR score: -0.6540 |
| 101: 5'ugaaAUGCU-GCCUCUUGCCu 3'PPP6C | PhastCons score: 0.7319 |
| Has-miR-31/RGS4 Alignment | Scores |
| 3'ucGAUACGGUCGUAGAACGGa 5'hsa-miR-31 | mirSVR score: -0.6450 |
| 1906: 5'aaCUUUAUUGG--UCUUGCCc 3'RGS4 | PhastCons score: 0.6315 |
直肠癌是我国常见的恶性肿瘤, 全国范围内其发病率高于结肠癌, 且目前仍以每年1%-2%的速度增长. 早期直肠癌预后较好, 而进展期直肠癌预后较差[13,14]. 依据我们曾经研究的数据, 直肠癌的5年生存率为55.07%. 直肠癌的发病是一个多因素、多步骤的演进过程, 近年一些研究认为, miRNA与包括结直肠癌在内的肿瘤关系密切, 可通过不同信号通路参与调控肿瘤的发生、发展和转归过程[6,15-18].
miRNA是一类含有21-25个核苷酸的非编码小分子单链RNA, 调控生物体生长发育和疾病发生等过程中相关基因表达. miRNA由Lee等[3]首先发现. 经一系列酶和转运蛋白等加工为成熟的miRNA, 在RNA诱导沉默复合物引导下与互补mRNA的3'UTR区互补配对, 指导miRNP复合体对靶mRNA进行阻遏, 抑制其翻译为蛋白质, 从而在转录后水平调控基因表达, 影响细胞的增殖、分化和凋亡等过程[1,19,20]. 单个miRNA表达异常可能导致数百个靶基因表达改变, 进而引起相应蛋白质的差异表达, 这对肿瘤形成具有重要意义[21]. 目前研究发现miRNA具有广泛的基因调节功能, 通过调控细胞凋亡、细胞增殖参与肿瘤以及其他疾病的发生和发展[19]. 肿瘤组织中存在不同miRNA表达量的上调或者下调, 类似于抑癌基因和癌基因的功能. 由于不同肿瘤组织中存在特定的miRNA表达谱, 这就极易令人联想到他在肿瘤的诊断及治疗方面的潜在作用. 寻找与直肠癌相关的miRNA可丰富直肠癌的病因学理论.
本实验用Affymetrix芯片来分析直肠正常黏膜、直肠腺癌组织中差异表达的miRNAs. 该芯片含有1 105条成熟的miRNA探针, 与成熟miRNA完全互补. 能特异性地检测出成熟的miRNA并能很好地区分高度同源的miRNA分子, 该平台还具有特异性高、灵敏度高等特点. 经研究我们发现直肠腺癌与正常直肠黏膜相比, 上调的miRNA 37种, 下调18种; 通过聚类分析发现直肠腺癌与正常黏膜miRNA差异明显, 可以通过聚类分析图进行区分.
我们分别对目前报道的肝癌[22]、胃癌[23]、结肠癌[24]等表达谱进行对照, 发现与直肠癌表达谱存在明显差异. 通过本研究我们认为, 直肠癌有其特异表达谱. Lu等[25]对包含多个人类恶性肿瘤的样本进行217个miRNA检测, 亦通过聚类分析发现不同肿瘤组织具有各自相似的miRNA差异表达谱, 理论上可以作为区分和检测肿瘤的重要手段. 但是因为miRNA检测所受影响因素较多, 重复测定完全一致性较差, 尚待进一步增大样本量, 筛选出特异性更强的差异表达谱.
关于miRNA的研究以结肠癌最多, 研究多认为miRNA与结肠癌的发生发展、预后及治疗疗效均有关, miRNA多方位参与结肠癌进展过程. 近年关于结肠或直肠癌miRNA与我们研究相吻合表达上调的miRNAs有miR-21、miR-27a、miR-31、miR-125b、miR-182, 相吻合表达下调到miRNAs有miR-133b、miR-200b.
上述表达差异的miRNAs中, 目前研究最多的为miR-21, 文献报道多认为miR-21与包括结直肠癌在内的恶性肿瘤的发生与发展有关. Yamamichi等[26]采用原位杂交技术分析了39例结直肠癌和34结直肠腺瘤中miR-21的表达情况, 研究发现miR-21在结直肠癌发展的不同时期中的表达存在差异, miR-21的表达从癌前病变到晚期癌逐渐增加. miR-21表达过量具有促进肿瘤发生和发展的作用, Asangani等[27]研究发现在结肠癌细胞中, miR-21的表达量与程序性细胞死亡因子4(PDCD4)蛋白的表达量呈负相关, 去除miR-21可使PDCD4表达增加, 肿瘤侵袭能力降低. 而miR-21过量表达时, 肿瘤的侵袭和转移能力提高. 目前的研究均提示miR-21在诊断、预测预后及治疗结直肠方面均具有潜在价值.
本研究发现在直肠癌中miR-21表达值较正常直肠黏膜增加2.903倍. 我们亦初步得出相同结论, 认为miR-21与直肠癌的发生发展过程可能有关, 值得进一步深入研究.
miR-224在结直肠癌中表达情况文献报道不一. Wang等[28]对无淋巴结转移的结肠癌进行研究发现, miR-224等14种miRNA上调. 而Rossi等[29]研究发现在HT-29、HCT-116结肠癌细胞系中miR-224表达下调. 我们研究发现与直肠正常黏膜相比, 直肠腺癌中miR-224的表达值上调4.362倍. 分析目前文献报道差异的原因可能为研究者采用样本不同或结肠癌与直肠癌在发病机制上可能存在差异. 包括本研究在内的3个研究分别采用了结肠癌组织、结肠癌细胞系、直肠癌组织进行研究.
对比文献报道, 本研究发现在结直癌中部分miRNA表达变化与既往报道相吻合. 上调的miRNAs包括miR-21[26,29-31]、miR-27a[29]、miR-31[32,33]、miR-125b[34]、miR-182[29], 下调的miRNA包括为miR-133b[32]、miR-200b[29].
由于miRNA在人类与靶基因的作用方式为不完全结合, 对靶基因发挥负性调控作用, 且分子只有22 nt左右, 理论上miRNA的靶基因很多, 准确预测特定的miRNA仍较困难[35]. 研究者通过各种算法, 开发了基于计算机、生物信息学的靶基因预测软件. 应用较广泛的为miRanda、TargetScan、PicTar等. Gomir软件[36]常将上述几种预测软件整合在一起对不同数据库间的靶基因预测进行比较、整合. 本研究通过Gomir软件进行检索发现预测到的靶基因数目仍较多, miR-31的靶基因为61个. 为此我们通过PubMed数据库检索出其中5个基因可能与直肠癌相关, 使下一步研究更加有针对性. 需要指出的是, 上述靶基因仅为预测软件所得, 受其算法的影响预测数目太多, 容易产生假阳性. 最终确定某一mRNA是否为特定miRNA特定的靶基因, 以及其对靶基因功能影响仍然需要用实验方法来验证[35].
总之, 我们应用芯片技术在直肠癌及正常直肠组织中筛选出表达上调或下调的miRNA, 直肠癌在发生过程中有特异miRNA改变. 但其靶基因仍需要试验方法验证, miRNA在直肠癌中的作用机制尚待阐明. 相信随着各方面研究的深入和技术的不断进步, miRNA将在直肠癌的早期有效诊治方面发挥重要作用.
目前, 国外研究发现多种miRNA在结直肠癌组织中异常表达, 且在结直肠癌的发生过程中, 不同的miRNA通过不同途径发挥着不同的重要作用. 在我国对miRNA研究尚少, 尚无报道直肠癌miRNA的表达谱.
刘宝林, 教授, 中国医科大学附属盛京医院
miRNA与包括结直肠癌在内的肿瘤关系密切, 可通过不同信号通路参与调控肿瘤的发生、发展和转归过程.
国外研究发现多种miRNA在结直肠癌组织中异常表达, 且在结直肠癌的发生过程中不同的miRNA通过不同途径发挥着不同的重要作用.
本文作者针对直肠癌基因表达谱进行研究, 发现直肠癌有特异miRNA表达谱, 且miRNA对直肠癌发生有重要作用.
对miRNA的深入研究, 将有助于对直肠癌发病机制的理解, 并为其诊治提供理论依据, 具有重大的生物学意义.
本课题设计合理, 手段先进, 统计分析可靠, 结论可信, 有临床应用价值.
编辑: 曹丽鸥 电编:闫晋利
| 1. | Lund E, Güttinger S, Calado A, Dahlberg JE, Kutay U. Nuclear export of microRNA precursors. Science. 2004;303:95-98. [PubMed] [DOI] |
| 2. | Nana-Sinkam SP, Fabbri M, Croce CM. MicroRNAs in cancer: personalizing diagnosis and therapy. Ann N Y Acad Sci. 2010;1210:25-33. [PubMed] [DOI] |
| 3. | Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 1993;75:843-854. [PubMed] [DOI] |
| 4. | Liu M, Chen H. The role of microRNAs in colorectal cancer. J Genet Genomics. 2010;37:347-358. [PubMed] [DOI] |
| 5. | Schee K, Fodstad Ø, Flatmark K. MicroRNAs as biomarkers in colorectal cancer. Am J Pathol. 2010;177:1592-1599. [PubMed] [DOI] |
| 6. | Xi Y, Formentini A, Chien M, Weir DB, Russo JJ, Ju J, Kornmann M, Ju J. Prognostic Values of microRNAs in Colorectal Cancer. Biomark Insights. 2006;2:113-121. [PubMed] |
| 7. | 邹 健, 于 晓峰, 虞 阳, 张 颖, 史 冬梅, 董 洁, 李 天奇, 刘 雪静. MicroRNA在人结肠癌干细胞中的表达谱. 世界华人消化杂志. 2010;18:173-178. [DOI] |
| 8. | Ruginis T, Taglia L, Matusiak D, Lee BS, Benya RV. Consequence of gastrin-releasing peptide receptor activation in a human colon cancer cell line: a proteomic approach. J Proteome Res. 2006;5:1460-1468. [PubMed] [DOI] |
| 9. | Caldwell GM, Jones CE, Ashley AM, Wei W, Hejmadi RK, Morton DG, Matthews GM. Wnt signalling in adenomas of familial adenomatous polyposis patients. Br J Cancer. 2010;103:910-917. [PubMed] [DOI] |
| 10. | Lauer C, Völkl A, Riedl S, Fahimi HD, Beier K. Impairment of peroxisomal biogenesis in human colon carcinoma. Carcinogenesis. 1999;20:985-989. [PubMed] [DOI] |
| 11. | Hirai A, Tada M, Furuuchi K, Ishikawa S, Makiyama K, Hamada J, Okada F, Kobayashi I, Fukuda H, Moriuchi T. Expression of AIE-75 PDZ-domain protein induces G2/M cell cycle arrest in human colorectal adenocarcinoma SW480 cells. Cancer Lett. 2004;211:209-218. [PubMed] [DOI] |
| 12. | Lu J, Gosslau A, Liu AY, Chen KY. PCR differential display-based identification of regulator of G protein signaling 10 as the target gene in human colon cancer cells induced by black tea polyphenol theaflavin monogallate. Eur J Pharmacol. 2008;601:66-72. [PubMed] [DOI] |
| 13. | Jass JR. Prognostic factors in rectal cancer. Eur J Cancer. 1995;31A:862-863. [PubMed] [DOI] |
| 14. | Liu YL, Zhao FL, Wang XS. [Analysis of clinicopathologic features and prognosis in 2414 patients with rectal cancer]. Zhonghua Weichang Waike Zazhi. 2010;13:913-916. [PubMed] |
| 15. | Akao Y, Nakagawa Y, Naoe T. MicroRNA-143 and -145 in colon cancer. DNA Cell Biol. 2007;26:311-320. [PubMed] [DOI] |
| 16. | Akao Y, Nakagawa Y, Naoe T. let-7 microRNA functions as a potential growth suppressor in human colon cancer cells. Biol Pharm Bull. 2006;29:903-906. [PubMed] [DOI] |
| 17. | Schepeler T, Reinert JT, Ostenfeld MS, Christensen LL, Silahtaroglu AN, Dyrskjøt L, Wiuf C, Sørensen FJ, Kruhøffer M, Laurberg S. Diagnostic and prognostic microRNAs in stage II colon cancer. Cancer Res. 2008;68:6416-6424. [PubMed] [DOI] |
| 18. | Kida Y, Han YP. MicroRNA expression in colon adenocarcinoma. JAMA. 2008;299:2628; author reply 2628-2629. [PubMed] [DOI] |
| 19. | Esquela-Kerscher A, Slack FJ. Oncomirs - microRNAs with a role in cancer. Nat Rev Cancer. 2006;6:259-269. [PubMed] [DOI] |
| 20. | Lee Y, Jeon K, Lee JT, Kim S, Kim VN. MicroRNA maturation: stepwise processing and subcellular localization. EMBO J. 2002;21:4663-4670. [PubMed] [DOI] |
| 21. | Lea MA. Recently identified and potential targets for colon cancer treatment. Future Oncol. 2010;6:993-1002. [PubMed] [DOI] |
| 22. | Ladeiro Y, Couchy G, Balabaud C, Bioulac-Sage P, Pelletier L, Rebouissou S, Zucman-Rossi J. MicroRNA profiling in hepatocellular tumors is associated with clinical features and oncogene/tumor suppressor gene mutations. Hepatology. 2008;47:1955-1963. [PubMed] [DOI] |
| 23. | Katada T, Ishiguro H, Kuwabara Y, Kimura M, Mitui A, Mori Y, Ogawa R, Harata K, Fujii Y. microRNA expression profile in undifferentiated gastric cancer. Int J Oncol. 2009;34:537-542. [PubMed] |
| 24. | Zhang MX, Cai H, Wang JS, Huang C, Wu WA, Ma RQ. [Expression profiles of microRNA in colon adenocarcinoma]. Xibao Yu Fenzi Mianyixue Zazhi. 2010;26:959-961. [PubMed] |
| 25. | Lu J, Getz G, Miska EA, Alvarez-Saavedra E, Lamb J, Peck D, Sweet-Cordero A, Ebert BL, Mak RH, Ferrando AA. MicroRNA expression profiles classify human cancers. Nature. 2005;435:834-838. [PubMed] [DOI] |
| 26. | Yamamichi N, Shimomura R, Inada K, Sakurai K, Haraguchi T, Ozaki Y, Fujita S, Mizutani T, Furukawa C, Fujishiro M. Locked nucleic acid in situ hybridization analysis of miR-21 expression during colorectal cancer development. Clin Cancer Res. 2009;15:4009-4016. [PubMed] [DOI] |
| 27. | Asangani IA, Rasheed SA, Nikolova DA, Leupold JH, Colburn NH, Post S, Allgayer H. MicroRNA-21 (miR-21) post-transcriptionally downregulates tumor suppressor Pdcd4 and stimulates invasion, intravasation and metastasis in colorectal cancer. Oncogene. 2008;27:2128-2136. [PubMed] [DOI] |
| 28. | Wang YX, Zhang XY, Zhang BF, Yang CQ, Chen XM, Gao HJ. Initial study of microRNA expression profiles of colonic cancer without lymph node metastasis. J Dig Dis. 2010;11:50-54. [PubMed] [DOI] |
| 29. | Rossi L, Bonmassar E, Faraoni I. Modification of miR gene expression pattern in human colon cancer cells following exposure to 5-fluorouracil in vitro. Pharmacol Res. 2007;56:248-253. [PubMed] [DOI] |
| 30. | Schetter AJ, Leung SY, Sohn JJ, Zanetti KA, Bowman ED, Yanaihara N, Yuen ST, Chan TL, Kwong DL, Au GK. MicroRNA expression profiles associated with prognosis and therapeutic outcome in colon adenocarcinoma. JAMA. 2008;299:425-436. [PubMed] [DOI] |
| 31. | Slaby O, Svoboda M, Fabian P, Smerdova T, Knoflickova D, Bednarikova M, Nenutil R, Vyzula R. Altered expression of miR-21, miR-31, miR-143 and miR-145 is related to clinicopathologic features of colorectal cancer. Oncology. 2007;72:397-402. [PubMed] [DOI] |
| 32. | Bandrés E, Cubedo E, Agirre X, Malumbres R, Zárate R, Ramirez N, Abajo A, Navarro A, Moreno I, Monzó M. Identification by Real-time PCR of 13 mature microRNAs differentially expressed in colorectal cancer and non-tumoral tissues. Mol Cancer. 2006;5:29. [PubMed] [DOI] |
| 33. | Cottonham CL, Kaneko S, Xu L. miR-21 and miR-31 converge on TIAM1 to regulate migration and invasion of colon carcinoma cells. J Biol Chem. 2010;285:35293-35302. [PubMed] [DOI] |
| 34. | Svoboda M, Izakovicova Holla L, Sefr R, Vrtkova I, Kocakova I, Tichy B, Dvorak J. Micro-RNAs miR125b and miR137 are frequently upregulated in response to capecitabine chemoradiotherapy of rectal cancer. Int J Oncol. 2008;33:541-547. [PubMed] |
| 35. | Kim S, Choi M, Cho KH. Identifying the target mRNAs of microRNAs in colorectal cancer. Comput Biol Chem. 2009;33:94-99. [PubMed] [DOI] |
| 36. | Roubelakis MG, Zotos P, Papachristoudis G, Michalopoulos I, Pappa KI, Anagnou NP, Kossida S. Human microRNA target analysis and gene ontology clustering by GOmir, a novel stand-alone application. BMC Bioinformatics. 2009;10 Suppl 6:S20. [PubMed] [DOI] |