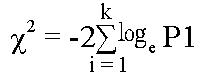修回日期: 2012-01-29
接受日期: 2012-03-15
在线出版日期: 2012-04-28
目的: 研究应用生物信息学技术, 对基因芯片产生的大量数据进行系统地挖掘和分析, 筛选出人类和小鼠不同种属间在肝癌发生发展过程中起关键作用的共同基因.
方法: 首先通过文献检索和收集, 在GEO数据库中下载符合纳入标准的9套样本基因芯片数据, 运用bioconductor和Rversion的2.10.1版本对已下载数据进行标准化处理, 运用软件包affy中的RMA算法对affymetrix平台的原始数据进行背景校正、标准化和log2转换; 其次使用excel的TTEST函数计算每一个基因的显著性, DAVID进行探针基因名称转换, 建立基因名称与样本对应的表达数据表; 再进行Meta分析计算人类及小鼠的共同差显基因, 并通过DAVID中的KEGG库富集调控通路.
结果: 人类与小鼠在肝癌发生发展过程中的共同表达基因52个, 其中5个为上调基因, 4个为下调基因; 富集出7条通路, 其中甘氨酸、丝氨酸、苏氨酸代谢通路和轴突引导通路是文献已报道的与肝癌发生发展密切相关通路.
结论: 通过生物信息学可快速筛选出人类与小鼠在肝癌发生发展过程中的共同关键基因及与肝癌发生发展密切相关通路.
引文著录: 王云, 胡艳玲, 曹骥, 何敏. 生物信息学方法对人类和小鼠共同肝癌相关基因的筛选. 世界华人消化杂志 2012; 20(12): 1012-1017
Revised: January 29, 2012
Accepted: March 15, 2012
Published online: April 28, 2012
AIM: To mine and analyze large amounts of data generated by gene chips and screen key genes expressed in both human and mouse hepatocellular carcinoma using bioinformatic methods.
METHODS: Through literature search and collection, nine sets of gene chip data that meet the criteria were downloaded from GEO database. The data were standardized by using Bioconductor and R version 2.10.1. The original Affymetrix data were normalized, background corrected, standardized and log2 transformed using the RMA algorithm. Excel's TTEST function was used to calculate the significance of each gene. DAVID was used for gene ID conversion and a table was established for samples and the corresponding gene expression data. A meta-analysis was then performed to screen genes expressed in both human and mouse hepatocellular carcinoma. KEGG pathways were then enriched.
RESULTS: A total of 52 genes were found to be expressed in both human and mouse hepatocellular carcinoma. Five of them were up-regulated, while four of them down-regulated. Seven KEGG pathways were enriched, of which glycine, serine, threonine metabolic pathways and axon guidance pathway have been previously reported to associated with the development of hepatocellular carcinoma.
CONCLUSION: Bioinformatic tools allow us to identify key genes and pathways that are closely related to the development of hepatocellular carcinoma in both human and mice.
- Citation: Wang Y, Hu YL, Cao J, He M. Bioinformatic screening of key genes expressed in both human and mouse hepatocellular carcinoma. Shijie Huaren Xiaohua Zazhi 2012; 20(12): 1012-1017
- URL: https://www.wjgnet.com/1009-3079/full/v20/i12/1012.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v20.i12.1012
肝癌(hepatocellular carcinoma, HCC), 是世界第5大高发的恶性肿瘤, 在全球恶性肿瘤死因中名列第3[1]. 至今为止在由世界各地不同实验室报道的巨量的肝癌相关分子中, 被一致认可者不多. 随着人类基因组和大鼠、小鼠等动物基因组测序工程的逐步完成, 基于生物信息学数据库的筛选方法也日益得到科研工作者的应用[2,3]. 该方法可利用已有的数据信息, 进行大规模高通量筛选, 花费少、信息量大. 目前已有不少研究者利用这种方法进行差异基因的筛选[4,5], 但是对于全基因组以及跨种属的肝癌相关基因的筛选研究却鲜有报道. 本研究采用计算机分析和统计学方法找出隐藏在全基因组表达芯片数据下的生物信息, 筛选出人类和小鼠共同肝癌相关差显基因, 为肝癌标志物的筛选策略提供新的思路.
所有HCC有关的全基因组表达芯片数据均来源互联网开放的免费数据库, 从GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)和ONCOMINE数据库(http://www.oncomine.org)下载. 小鼠文献收集在GEO数据库GEO Datasets中以"hepatocellular carcinoma, mus musculus"为关键词检索所有公开发表的文献; 人类文献收集在ONCOMINE数据库中以"hepatocellular carcinoma, homo sapiens "为关键词检索所有公开发表的文献.
1.2.1 文献纳入标准: 符合以下标准的数据集将纳入研究对象: (1)数据集必须是全基因组的表达RNA芯片数据; (2)数据是关于HCC癌组织和正常组织对照; (3)均考虑经标准化或者原始数据集; (4)数据集必须包括超过3个样本以上; (5)物种包括人类和小鼠.
1.2.2 数据处理: 在GEO的supplementary file中下载基因芯片的CEL数据压缩包, 并解压至一个文件夹中待用, 同时下载TXT格式的样本原始数据; 若下载数据处显示无CEL数据压缩包提供, 则在scope、format、amount中分别选择samples、soft、full下载该样本TXT格式的原始数据, 将每套样本数据的基本信息记录在提供的excel里. 通过bioconductor和Rversion的2.10.1版本[6]对已下载数据进行标准化处理, 运用软件包affy中的RMA算法[7,8]对affymetrix平台的原始数据进行背景校正、标准化和log2转换.
1.2.3 基因差异表达的计算分析: 首先通过使用excel的TTEST函数计算每一个基因的显著性; 其次将得到的探针号通过DAVID(http://david.abcc.ncifcrf.gov/)中的gene id conversion tool进行探针基因名称转换, 得到这些探针号的官方基因名称; 最后利用VLOOKUP函数替代原始数据里的探针号, 建立基因名称与样本对应的表达数据表.
1.2.4 Meta分析计算人类的共同差显基因、小鼠的共同差显基因: 为了能有效地解决样本太小问题, 把2套人类和7套小鼠数据集分别通过Meta进行分析, 在Meta分析中先用t检验计算每套数据的每个探针的P值, 再通过下列公式算出每个基因的卡方值:
Math 1
自由度为数据集K的2倍, 最后保留P<0.05的基因, 得到人类的共同差显基因、小鼠的共同差显基因.
1.2.5 人类和小鼠共有的差显基因表达水平: 人类与小鼠Meta分析出的P<0.05的基因结果进行比较得到人类与小鼠HCC共同差显基因, 对以上基因的癌组织和正常对照2类样本的基因芯片数据进行整理, 分类计算出样本数据的平均值, 将HCC癌组织样本数据平均值高于正常对照平均值1.2倍的基因定为HCC上调基因; 将HCC癌组织样本数据平均值<正常对照平均值0.9倍的基因定为HCC下调基因; 同时, 上调基因或者下调基因需出现在5套数据集以上(含5套数据集), 才可最终确定该基因功能为上调或者下调.
1.2.6 人类和小鼠共同表达基因的通路富集: 对人类与小鼠HCC共同表达基因通过DAVID(http://david.abcc.ncifcrf.gov/)中的KEGG库进行富集分析.
根据纳入标准最终9套样本作为研究对象(表1).
| 类别 | GSE14323 | GSE6764 | GSE9809 | GSE9012 | GSE19004 | GSE2127 | GSE23680 | GSE15251 | GSE8642 |
| 物种 | homo sapiens | homo sapiens | mus musculus | mus musculus | mus musculus | mus musculus | mus musculus | mus musculus | mus musculus |
| 文献 | Mas等[9] | Wurmb-ach等[10] | Liao等[11] | Khetcho-umian等[12] | 仅提供芯片信息 | Sheth等[13] | Li等[14] | 仅提供芯片信息 | 仅提供芯片信息 |
| 芯片 | GPL96, GPL571 | GPL570 | GPL1261 | GPL1261 | GPL1261 | GPL339 | GPL6246 | GPL8308 | GPL339 |
| 实验设计 | unpaired tissues | unpaired tissues | unpaired+paired, tissues | unpaired tissues | unpairedtissues | unpaired+paired tissues | paired, tissues | unpairedtissues | unpaired tissues |
| 探针数 | 22k | 55K | 45k | 45k | 45k | 22k | 48k | 49k | 41k |
| HCC组织 | 47 | 35 | 3 | 5 | 5 | 9 | 4 | 2 | 6 |
| 对照 | 19 | 10 | 7 | 5 | 4 | 6 | 4 | 5 | 9 |
对2套人类数据单独进行t检验得出每个基因的P值后, 通过选择的Meta公式进行整合分析, 共筛出4 951个基因(P<0.05).
对7套小鼠数据单独进行t检验得出每个基因的P值后, 通过选择的Meta公式进行整合分析, 共筛出116个基因(P<0.05).
对人类与小鼠Meta分析出的P<0.05的基因结果进行比较分析后发现人类与小鼠HCC共同表达基因有52个, 其中表达水平上调的基因5个, 表达水平下调的基因4个(表2).
| 基因 | 基因名称 | 表达水平 |
| itm2b | integral membrane protein 2B | |
| abcc5 | ATP-binding cassette, sub-family C (CFTR/MRP), member 5 | |
| arf3 | ADP-ribosylation factor 3 | |
| aspm | asp (abnormal spindle)-like, microcephaly associated (Drosophila) | |
| cdc20 | cell division cycle 20 homolog (S.cerevisiae) | 上调 |
| cttn | cortactin; predicted gene 8786 | |
| ddx39 | DEAD (Asp-Glu-Ala-Asp) box polypeptide 39 | |
| dnm1l | dynamin 1-like | |
| fabp4 | fatty acid binding protein 4, adipocyte | 上调 |
| gnb1 | guanine nucleotide binding protein (G protein), beta 1 | |
| golm1 | golgi membrane protein 1 | 上调 |
| gpr56 | G protein-coupled receptor 56 | |
| ier5 | immediate early response 5 | |
| itga3 | integrin alpha 3 | |
| itgb1 | integrin beta 1 (fibronectin receptor beta) | |
| itm2c | integral membrane protein 2C | |
| itpr3 | inositol 1,4,5-triphosphate receptor 3 | |
| klf6 | Kruppel-like factor 6 | |
| lmna | lamin A | |
| lrrc59 | leucine rich repeat containing 59 | |
| ly6d | lymphocyte antigen 6 complex, locus D | 上调 |
| map4k4 | mitogen-activated protein kinase kinase kinase kinase 4 | |
| meox1 | mesenchyme homeobox 1 | |
| nek2 | NIMA (never in mitosis gene a)-related expressed kinase 2 | |
| rac1 | RAS-related C3 botulinum substrate 1 | |
| scn8a | sodium channel, voltage-gated, type VIII, alpha | |
| slc1a4 | solute carrier family 1 (glutamate/neutral amino acid transporter), member 4 | |
| tagln2 | transgelin 2 | |
| tgfbr2 | transforming growth factor, beta receptor II | |
| tmc6 | transmembrane channel-like gene family 6 | |
| tpm1 | tropomyosin 1, alpha | |
| trib3 | tribbles homolog 3 (Drosophila) | |
| tspan8 | tetraspanin 8 | 上调 |
| wsb2 | WD repeat and SOCS box-containing 2 | |
| ywhaz | tyrosine 3-monooxygenase/tryptophan 5-monooxygenase activation protein, zeta polypeptide; predicted gene 4202 | |
| crbn | cereblon | |
| abat | 4-aminobutyrate aminotransferase | |
| acsl1 | acyl-CoA synthetase long-chain family member 1 | |
| akap1 | A kinase (PRKA) anchor protein 1 | |
| cbs | cystathionine beta-synthase | |
| cxcl12 | chemokine (C-X-C motif) ligand 12 | 下调 |
| gas1 | growth arrest specific 1 | 下调 |
| ghr | growth hormone receptor | |
| gnmt | glycine N-methyltransferase | 下调 |
| igfals | insulin-like growth factor binding protein, acid labile subunit | 下调 |
| masp1 | mannan-binding lectin serine peptidase 1 | |
| paics | phosphoribosylaminoimidazole carboxylase, phosphoribosylaminoribosylaminoimidazole, succinocarboxamide synthetase; predicted gene 8445 | |
| pbld | phenazine biosynthesis-like protein domain containing | |
| plxnc1 | plexin C1; similar to plexin C1 | |
| sardh | sarcosine dehydrogenase | |
| scnn1a | sodium channel, nonvoltage-gated 1 alpha | |
| sirt3 | sirtuin 3 (silent mating type information regulation 2, homolog) 3 (S. cerevisiae) |
通过DAVID中的KEGG库, 对人类与小鼠共同表达的52个基因进行功能基因富集, 分别富集出影响这些差显基因的主要通路7条, 其中的2条通路甘氨酸、丝氨酸、苏氨酸代谢通路和轴突引导通路是文献已报道与肝癌发生发展密切相关通路(表3).
| 通路名称 | 基因 | P值 | 与肝癌关系的文献报道 |
| 肥厚型心肌病 | lmna, itga3, tpm1, itgb1 | 0.0088804 | 无 |
| 甘氨酸、丝氨酸、苏氨酸代谢通路 | gnmt, sardh, cbs | 0.0106734 | 有[15,16] |
| 扩张型心肌病 | lmna, itga3, tpm1, itgb1 | 0.0110271 | 无 |
| 轴突引导通路 | plxnc1, rac1, itgb1, cxcl12 | 0.0270935 | 有[17-20] |
| 味觉转导 | gnb1, itpr3, scnn1a | 0.0285055 | 无 |
| 致病性大肠杆菌感染 | cttn, ywhaz, itgb1 | 0.0337804 | 无 |
| 致心律失常型右室心肌病 | lmna, itga3, itgb1 | 0.0568722 | 无 |
20世纪90年代初以来, 生物芯片技术取得了迅猛发展, 由此带来了海量微阵列数据, 对这些数据进行挖掘和处理, 找出隐藏在生物芯片数据下的生物信息, 是生物信息学的重要任务之一. 生物信息学基因芯片数据分析方法主要分为如下几大类: (1)基因差异表达分析; (2)聚类分析; (3)判别分析; (4)构建基因调控网络; (5)荟萃分析, 即Meta分析.
已有的研究工作大都开始并集中于运用基因差异表达分析方法对肿瘤基因表达数据的特征提取和类别分类上, 其主要目标就是发现与肿瘤病变有关的特征基因. 如应用基因芯片筛选人肝癌、肝硬化及正常肝组织中差异表达的基因[4], 利用基因表达差异鉴别正常细胞与白血病细胞[21], Rubin等[22]利用基因芯片技术发现基因amacr在前列腺组织中明显升高, 然后利用含342个标本的组织芯片进行检测, 证实其在前列腺癌组织中高表达, 并用94个细针穿刺标本进一步检测, 发现其用于诊断前列腺癌的敏感性为97%, 特异性为100%, 提示amacr可能是前列腺癌良好的诊断指标.
而多癌症分子共性的Meta分析则是显著性差异表达研究的补充和发展[23-25]. 自2002年Rhodes等[26]首先将Meta分析引入基因芯片数据分析领域以来, 已经有非常多的关于Meta分析处理多试验基因芯片数据的报道[26-29]. Rhodes等利用来自不同独立研究得到的单基因P值合并成一个综合P值, 对4套来自不同实验室采用不同芯片平台产生的前列腺癌表达数据进行Meta分析, 得到了一批在多个独立实验之间表现一致, 与前列腺癌有关的基因.
因此, 本研究主要也运用了上述的基因差异表达分析方法、Meta分析方法以及差异表达基因的功能富集分析方法进行基因芯片数据的生物信息学分析. 通过对标准化以后的2套人类及7套小鼠HCC全基因组表达芯片数据, 应用基因差异表达分析t检验、Meta分析及代谢通路富集分析, 在人类与小鼠全基因组范围内比较正常肝组织与肝癌组织之间基因表达谱的差异, 在P<0.05水平, 发现了52个在两种种属间共同表达的基因, 其中5个为上调基因, 4个为下调基因, 可能为肝癌的发生发展密切相关的关键基因. 美国冷泉港实验室最近以"加速发现肿瘤基因的整体肿瘤基因组学策略(integrative oncogenomic approaches for accelerated cancer-gene discovery)"[30]为题撰文讨论跨种属的比较基因组学研究策略, 认为这种高性价比的研究策略极有希望加速发现人类肿瘤基因的进程.
上述52个基因富集分析出7条代谢通路, 其中已有文献报道[15-20]甘氨酸、丝氨酸、苏氨酸代谢通路和轴突引导通路与肝癌发生发展密切相关. 富集于这两条通路上的GNMT, CBS, RAC1基因, Avila等[15,16]在研究人类肝硬化和肝细胞癌的氨基酸代谢途径中发现GNMT, CBS的mRNA含量在人类肝硬化和肝细胞癌组织中显著减少, 是HCC的肿瘤抑制基因, 他们可通过与其他蛋白的直接作用来调节肿瘤的细胞生长、蛋白合成、核糖体生成及自我吞噬等; 而Rac1本身以及 Rac1的多种上下游分子如GEF、TRE(顺式作用元件)17等都是癌基因, 他的激活能导致成纤维细胞的恶性转化, 在肿瘤组织中表达较正常组织中明显升高, 通过参与肿瘤的侵袭和转移、肿瘤细胞转录的调节、肿瘤新生血管的形成诸多环节促进肿瘤的发生、进展[17-20].
由此可见生物信息学在处理基因芯片产生的海量数据中是必不可少的工具. 生物信息学筛选是一种快速有效的筛选方法. 随着芯片应用的推进, 芯片数据分析的新理论和新算法也将不断地被研究出来, 本研究所得结果对今后筛选跨种属肝癌标志物的鉴定奠定了基础, 也为肿瘤标志物的筛选策略提供了新的思路.
至今已有不少研究者利用生物信息学方法进行差异基因的筛选, 但对于在全基因组以及跨种属的肝癌相关基因的筛选研究却鲜有报道.
荚卫东, 教授, 安徽省立院肝脏外科
随着人类基因组和大鼠、小鼠等动物基因组测序工程的逐步完成, 带来了海量微阵列数据, 应用跨种属的比较基因组学研究策略, 对跨种属的生物芯片数据下的生物信息进行挖掘, 极有希望加速发现人类肿瘤基因的进程.
本研究所得结果对今后筛选跨种属肝癌标志物的鉴定奠定了基础, 也为肿瘤标志物的筛选策略提供了新的思路.
本文研究方法创新性好, 内容较新颖, 数据可靠, 结果有新意, 具有较好的科学意义.
编辑: 曹丽鸥 电编: 闫晋利
| 1. | Thun MJ, DeLancey JO, Center MM, Jemal A, Ward EM. The global burden of cancer: priorities for prevention. Carcinogenesis. 2010;31:100-110. [PubMed] [DOI] |
| 2. | Rajkovic A, Yan M S C, Klysik M, Matzuk M. Discovery of germ cell-specific transcripts by expressed sequence tag database analysis. Fertil Steril. 2001;76:550-554. [PubMed] [DOI] |
| 3. | Wang J, Liang P. DigiNorthern, digital expression analysis of query genes based on ESTs. Bioinformatics. 2003;19:653-654. [PubMed] [DOI] |
| 6. | Gentleman RC, Carey VJ, Bates DM, Bolstad B, Dettling M, Dudoit S, Ellis B, Gautier L, Ge Y, Gentry J. Bioconductor: open software development for computational biology and bioinformatics. Genome Biol. 2004;5:R80. [PubMed] [DOI] |
| 7. | Irizarry RA, Hobbs B, Collin F, Beazer-Barclay YD, Antonellis KJ, Scherf U, Speed TP. Exploration, normalization, and summaries of high density oligonucleotide array probe level data. Biostatistics. 2003;4:249-264. [PubMed] [DOI] |
| 8. | Gautier L, Cope L, Bolstad BM, Irizarry RA. affy--analysis of Affymetrix GeneChip data at the probe level. Bioinformatics. 2004;20:307-315. [PubMed] [DOI] |
| 9. | Mas VR, Maluf DG, Archer KJ, Yanek K, Kong X, Kulik L, Freise CE, Olthoff KM, Ghobrial RM, McIver P. Genes involved in viral carcinogenesis and tumor initiation in hepatitis C virus-induced hepatocellular carcinoma. Mol Med. 2009;15:85-94. [PubMed] [DOI] |
| 10. | Wurmbach E, Chen YB, Khitrov G, Zhang W, Roayaie S, Schwartz M, Fiel I, Thung S, Mazzaferro V, Bruix J. Genome-wide molecular profiles of HCV-induced dysplasia and hepatocellular carcinoma. Hepatology. 2007;45:938-947. [PubMed] [DOI] |
| 11. | Liao YJ, Liu SP, Lee CM, Yen CH, Chuang PC, Chen CY, Tsai TF, Huang SF, Lee YH, Chen YM. Characterization of a glycine N-methyltransferase gene knockout mouse model for hepatocellular carcinoma: Implications of the gender disparity in liver cancer susceptibility. Int J Cancer. 2009;124:816-826. [PubMed] [DOI] |
| 12. | Khetchoumian K, Teletin M, Tisserand J, Mark M, Herquel B, Ignat M, Zucman-Rossi J, Cammas F, Lerouge T, Thibault C. Loss of Trim24 (Tif1alpha) gene function confers oncogenic activity to retinoic acid receptor alpha. Nat Genet. 2007;39:1500-1506. [PubMed] [DOI] |
| 13. | Sheth SS, Bodnar JS, Ghazalpour A, Thipphavong CK, Tsutsumi S, Tward AD, Demant P, Kodama T, Aburatani H, Lusis AJ. Hepatocellular carcinoma in Txnip-deficient mice. Oncogene. 2006;25:3528-3536. [PubMed] [DOI] |
| 14. | Li H, Malani N, Hamilton SR, Schlachterman A, Bussadori G, Edmonson SE, Shah R, Arruda VR, Mingozzi F, Wright JF. Assessing the potential for AAV vector genotoxicity in a murine model. Blood. 2011;117:3311-3319. [PubMed] [DOI] |
| 15. | Avila MA, Berasain C, Torres L, Martín-Duce A, Corrales FJ, Yang H, Prieto J, Lu SC, Caballería J, Rodés J. Reduced mRNA abundance of the main enzymes involved in methionine metabolism in human liver cirrhosis and hepatocellular carcinoma. J Hepatol. 2000;33:907-914. [PubMed] [DOI] |
| 16. | Yen CH, Lu YC, Li CH, Lee CM, Chen CY, Cheng MY, Huang SF, Chen KF, Cheng AL, Liao LY. Functional characterization of glycine N-methyltransferase and its interactive protein DEPDC6/DEPTOR in hepatocellular carcinoma. Mol Med. 2012;18:286-296. [PubMed] |
| 17. | 陈 保东, 高 永中, 蒋 太鹏, 丁 建军, 姜 晓丹, 徐 如祥. Rac1在垂体腺瘤组织中的表达及意义. 中国微侵袭神经外科杂志. 2010;15:268-270. |
| 19. | 董 朝富, 周 安立, 陈 刚, 朱 智, 颜 廷启, 刘 顺方, 刘 锋, 沈 文状, 杨 志芳, 易 继林. 抑制Rac1的活性对人肝癌细胞生长及侵袭能力的影响. 肝胆胰外科杂志. 2009;21:288-291. |
| 20. | Wu L, Cai C, Wang X, Liu M, Li X, Tang H. MicroRNA-142-3p, a new regulator of RAC1, suppresses the migration and invasion of hepatocellular carcinoma cells. FEBS Lett. 2011;585:1322-1330. [PubMed] [DOI] |
| 21. | Golub TR, Slonim DK, Tamayo P, Huard C, Gaasenbeek M, Mesirov JP, Coller H, Loh ML, Downing JR, Caligiuri MA. Molecular classification of cancer: class discovery and class prediction by gene expression monitoring. Science. 1999;286:531-537. [PubMed] [DOI] |
| 22. | Rubin MA, Zhou M, Dhanasekaran SM, Varambally S, Barrette TR, Sanda MG, Pienta KJ, Ghosh D, Chinnaiyan AM. alpha-Methylacyl coenzyme A racemase as a tissue biomarker for prostate cancer. JAMA. 2002;287:1662-1670. [PubMed] [DOI] |
| 23. | Smid M, Dorssers LC, Jenster G. Venn Mapping: clustering of heterologous microarray data based on the number of co-occurring differentially expressed genes. Bioinformatics. 2003;19:2065-2071. [PubMed] [DOI] |
| 24. | Rhodes DR, Yu J, Shanker K, Deshpande N, Varambally R, Ghosh D, Barrette T, Pandey A, Chinnaiyan AM. Large-scale meta-analysis of cancer microarray data identifies common transcriptional profiles of neoplastic transformation and progression. Proc Natl Acad Sci USA. 2004;101:9309-9314. [PubMed] [DOI] |
| 25. | Segal E, Friedman N, Koller D, Regev A. A module map showing conditional activity of expression modules in cancer. Nat Genet. 2004;36:1090-1098. [PubMed] [DOI] |
| 26. | Rhodes DR, Barrette TR, Rubin MA, Ghosh D, Chinnaiyan AM. Meta-analysis of microarrays: interstudy validation of gene expression profiles reveals pathway dysregulation in prostate cancer. Cancer Res. 2002;62:4427-4433. [PubMed] |
| 27. | Conlon EM, Song JJ, Liu A. Bayesian meta-analysis models for microarray data: a comparative study. BMC Bioinformatics. 2007;8:80. [PubMed] [DOI] |
| 28. | Conlon EM. A Bayesian mixture model for metaanalysis of microarray studies. Funct Integr Genomics. 2008;8:43-53. [PubMed] [DOI] |
| 29. | Liang Y, Kelemen A. Bayesian models and meta analysis for multiple tissue gene expression data following corticosteroid administration. BMC Bioinformatics. 2008;9:354. [PubMed] [DOI] |
| 30. | Zender L, Lowe SW. Integrative oncogenomic approaches for accelerated cancer-gene discovery. Curr Opin Oncol. 2008;20:72-76. [PubMed] [DOI] |