修回日期: 2011-10-28
接受日期: 2011-11-04
在线出版日期: 2011-12-18
目的: 探讨5-Aza-dC及TSA对人胃癌细胞系SGC-7901中抑癌基因Runx3启动子区甲基化、mRNA及蛋白表达水平的影响.
方法: 单独或联合应用5-Aza-dC及TSA处理体外培养的SGC-7901细胞, 提取各组细胞的DNA、RNA及蛋白质, 应用甲基化特异性定量PCR法(QMSP)检测Runx3基因启动子区甲基化状态, 逆转录PCR法(RT-PCR)检测Runx3 mRNA的表达, 免疫印迹法(Western blotting)法检测Runx3蛋白表达水平.
结果: 5-Aza-dC和TSA均能降低Runx3基因启动子区的甲基化水平(5-Aza-dC组及TSA组分别为对照组的0.70倍、0.63倍), 提高mRNA表达水平(0.29±0.01、0.28±0.03 vs 0.14±0.03, P<0.05)及蛋白表达水平(0.35±0.02、0.37±0.02 vs 0.09±0.01, P<0.05); 与单独使用5-Aza-dC和TSA相比, 两药联合组Runx3基因启动子区甲基化水平(对照组的0.37倍)及mRNA表达水平(0.45±0.02)和蛋白表达水平(0.50±0.01)均较单药组效果更明显(P<0.05).
结论: 5-Aza-dC和TSA均能逆转胃癌细胞SGC-7901 Runx3基因的甲基化水平, 恢复其mRNA和蛋白表达, 且具有协同作用, 为5-Aza-dC和TSA应用于胃癌的临床治疗提供了试验依据.
引文著录: 方中良, 沈干, 胡世莲, 孙玉蓓, 徐维平, 黄大兵, 姜晓东, 王海, 黄毕林. 5-Aza-dC及TSA对人胃癌细胞株SGC-7901 Runx3基因甲基化及表达水平的影响. 世界华人消化杂志 2011; 19(35): 3562-3567
Revised: October 28, 2011
Accepted: November 4, 2011
Published online: December 18, 2011
AIM: To evaluate the effect of 5-Aza-2'-deoxy-citydine (5-Aza-dC) and trichostatin A (TSA) on the methylation and expression of the Runx3 gene in human gastric cancer cell line SGC-7901.
METHODS: After cultured SGC-7901 cells were treated with 5-Aza-dC and TSA, the methylation levels of the promoter region of the Runx3 gene were detected by quantitative real-time methylation-specific polymerase chain reaction (QMSP), and Runx3 mRNA and protein expression was detected by reverse transcription-polymerase chain reaction (RT-PCR) and Western blotting, respectively.
RESULTS: Treatment with 5-Aza-dC or TSA alone reduced the methylation levels of the promoter region of the Runx3 gene (70%, 63% vs 100%) and increased Runx3 mRNA (0.29 ± 0.01, 0.28 ± 0.03 vs 0.14 ± 0.03, both P < 0.05) and protein expression levels (0.35 ± 0.02, 0.37 ± 0.02 vs 0.09 ± 0.01, P < 0.05) compared to control cells. Treatment with 5-Aza-dC in combination with TSA could more significantly reduce Runx3 gene promoter methylation levels (37%) and increase Runx3 mRNA (0.45 ± 0.02) and protein expression levels (0.50 ± 0.01) compared to cells treated with 5-Aza-dC or TSA alone (all P < 0.05).
CONCLUSION: 5-Aza-dC and TSA can synergistically reverse Runx3 gene methylation and recover Runx3 mRNA and protein expression in SGC-7901 cells.
- Citation: Fang ZL, Shen G, Hu SL, Sun YB, Xu WP, Huang DB, Jiang XD, Wang H, Huang BL. Effect of 5-Aza-2'-deoxycytidine and trichostatin A on expression and methylation of the Runx3 gene in human gastric carcinoma line SGC-7901. Shijie Huaren Xiaohua Zazhi 2011; 19(35): 3562-3567
- URL: https://www.wjgnet.com/1009-3079/full/v19/i35/3562.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v19.i35.3562
流行病学调查显示胃癌是全球发病率最高的恶性肿瘤之一, 且是第2大癌症致死病因[1,2]. 在我国每年有40万新发胃癌患者, 每3 min就有1人死于胃癌, 其患病率和死亡率均高于世界平均水平, 严重危害我国人民的身体健康[3]. 目前针对胃癌患者的治疗手段主要有手术治疗和化疗药物治疗: 大部分患者发现时已处于胃癌中晚期, 手术效果欠佳; 常规化疗药物也只对部分患者有效, 仍有相当比例患者对其不敏感, 且具有严重的不良反应. 大量相关研究证明DNA甲基化在胃癌的发生发展过程中起到重要作用, 其中抑癌基因启动子区异常甲基化导致的抑癌基因失活参与了胃癌的发生发展[4-8], 由于抑癌基因的异常甲基化具有可逆性[9-12], 因此应用相关药物逆转抑癌基因的异常甲基化状态, 使其重新表达从而发挥抑癌基因的作用具有重要的临床意义. 我们通过研究药物干预对胃癌细胞系SGC-7901中Runx3基因启动子区甲基化状态及其mRNA和蛋白表达水平的影响, 探讨去甲基化药物5-氮杂-2'-脱氧胞苷(5-Aza-2'-deoxy-citydine, 5-Aza-dC)和组蛋白去乙酰化酶抑制剂曲古抑菌素A(Trichostatin A, TSA)在胃癌细胞系SGC7901中对Runx3基因甲基化、mRNA及蛋白表达水平的影响, 为5-Aza-dC和TSA应用于胃癌的临床治疗提供试验依据.
胃癌细胞系SGC-7901由安徽医科大学分子生物学实验室赠予, RPMI1640培养基和胎牛血清为GIBCO公司产品, 5-Aza-dC及TSA为Sigma产品, DNA提取试剂盒购自TIANGEN公司, DNA修饰试剂盒(BisulFlash DNA Modification Kit)为GEPIGENTEK公司产品, QMSP所需的Real Time PCR试剂为TAKARA公司产品, RNA抽提试剂TRIzol为Invitrogen公司产品, RT-PCR试剂盒为Promega公司产品, Taq酶及dNTP均为Fermentas公司产品, 引物及探针的合成由TAKARA公司完成, Runx3兔抗人多克隆抗体购自博奥森生物技术有限公司, β-Actin一抗、二抗为北京中杉金桥公司产品.
1.2.1 细胞培养及药物干预: 将胃癌细胞SGC-7901接种于含100 mL/L的胎牛血清的RPMI 1640培养基中, 在37 ℃, 50 mL/L CO2的培养箱中培养. 取对数生长期细胞分为以下4组: (1)对照组: 不加药; (2)5-Aza-dC组: 在5-Aza-dC浓度为5 µmol/L的培养基中培养72 h; (3)TSA组: 在TSA浓度为300 nmol/L的培养基中培养24 h; (4)5-Aza-dC+TSA组: 在5-Aza-dC浓度为5 µmol/L的培养基中培养48 h后再加TSA 300 nmol/L培养24 h.
1.2.2 甲基化特异性定量PCR法分析各组细胞中Runx3基因甲基化状态: 按DNA提取试剂盒说明书提取各组细胞的DNA, 用紫外分光光度计检测DNA的纯度和浓度, 并用琼脂糖凝胶电泳检测; 用BisulFlash DNA Modification Kit对DNA进行修饰, 引物和探针(表1), 在ABI 7500定量PCR仪上扩增, 扩增条件为: 95 ℃ 30 s, 然后95 ℃ 5 s、60 ℃ 34 s扩增50个循环, 各组细胞的目的基因和内参基因分别做3个复孔, 用相对定量法中的△△Ct法对各组细胞中Runx3基因甲基化水平进行分析(甲基化水平的平均相对含量 = 加药组/对照组 = 2-△△Ct).
1.2.3 逆转录PCR法分析各组细胞中Runx3基因mRNA的表达: 按TRIzol试剂盒说明书方法提取各组细胞的总RNA, 用PROMEGA试剂盒将其逆转录为cDNA, Runx3及β-Actin基因的引物由TAKARA公司合成(表2), 按反应条件进行扩增: 95 ℃预变性5 min后, 95 ℃ 30 s、52 ℃ 30 s、72 ℃ 30 s扩增35个循环, 再72 ℃延伸5 min, 结束后用琼脂糖凝胶电泳验证试验结果, 并使用Quantity One软件分析Runx3基因与β-Actin基因目的条带灰度值的比值, 重复实验3次并计算各组灰度值比值的mean±SD作为Runx3 mRNA表达水平的结果.
1.2.4 免疫印迹法分析各组细胞中Runx3基因蛋白的表达: 收集各组细胞按方法提取蛋白, 采用BCA蛋白浓度测定试剂盒测定蛋白浓度. 取调节后的蛋白加上样缓冲液煮沸5 min使其充分变性. 用15%SDS-PAGE凝胶电泳, 恒压15 V半干转膜30 min后, 将凝胶上的蛋白转移至PVDF膜上. 用50 g/L脱脂奶粉室温封闭PVDF膜1 h, 加入一抗稀释液(1:500稀释)4 ℃孵育过夜, 第2天用TBST漂洗PVDF膜8 min×4次, 加入二抗稀释液(1:5 000) 37 ℃孵育1 h, 第2天用TBST漂洗PVDF膜8 min×4次. 采用辣根过氧化物酶HRP-ECL发光法曝光. 使用Bio-Rad凝胶图像处理系统分析结果, 以Runx3蛋白与β-Actin蛋白条带灰度值比值作为Runx3蛋白的相对表达水平.
统计学处理 采用SPSS13.0软件进行统计分析, mRNA及蛋白表达结果以mean±SD的形式表示, 2组均数的比较采用t检验, P<0.05为差异有统计学意义.
以对照组的甲基化水平作为1, 其他3组加药细胞的甲基化水平的平均相对含量与对照组比较: 5-Aza-dC组的甲基化水平为对照组的0.70倍, TSA组的甲基化水平为对照组的0.63倍, 5-Aza-dC+TSA组的甲基化水平为对照组的0.37倍(图1, 表3).
| 分组 | Ct(Runx3) | Ct(β-Actin) | △Ct | △△Ct | 2-△△Ct |
| 对照组 | 31.28±0.18 | 30.40±0.20 | 0.88 | 0 | 1 |
| 5-Aza-dC组 | 31.35±0.23 | 29.96±0.14 | 1.39 | 0.51 | 0.70 |
| TSA组 | 31.68±0.12 | 30.13±0.25 | 1.55 | 0.67 | 0.63 |
| 5-Aza-dC + TSA组 | 31.63±0.27 | 29.31±0.19 | 2.32 | 1.44 | 0.37 |
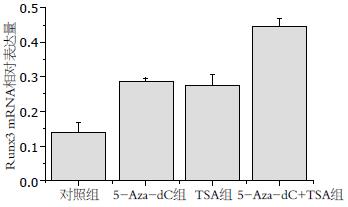
各组细胞中Runx3基因mRNA相对表达量分别为: 对照组: 0.14±0.03; 5-Aza-dC组: 0.29±0.01; TSA组: 0.28±0.03; 5-Aza-dC+TSA组: 0.45±0.02. 5-Aza-dC组、TSA组、5-Aza-dC+TSA组相对表达量与对照组相对表达量比较, 差异有统计学意义(P<0.05); 5-Aza-dC组、TSA组相对表达量与5-Aza-dC+TSA组相对表达量比较差异有统计学意义(P<0.05, 图2, 3).
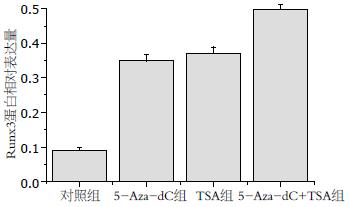
对照组、5-Aza-dC组、TSA组及5-Aza-dC+TSA组细胞的蛋白表达水平分别为0.09±0.01、0.35±0.02、0.37±0.02、0.50±0.01, 对照组蛋白表达水平与其他3组蛋白表达水平比较差异都有统计学意义(P<0.05), 5-Aza-dC+TSA组蛋白表达水平分别与5-Aza-dC组及TSA组蛋白水平比较差异都有统计学意义(P<0.05, 图4, 5).
现代肿瘤学理论认为, 遗传基因缺陷以及基因表观遗传学改变会导致恶性肿瘤的发生. 基因缺陷如突变、缺失会破坏编码区结构和功能; 表观遗传学改变通过DNA甲基化或组蛋白乙酰化/去乙酰化等自身化学修饰方式的改变来影响转录水平, 进而影响基因表达, 从而调控DNA功能[13-15]. 胃癌的发生发展是一个多因素参与的过程, 其中涉及表观遗传学改变, 已有大量研究证据表明胃癌的发生与抑癌基因启动子区异常甲基化有关[16-19]. Runx3是2000年发现的与胃癌等肿瘤密切相关的抑癌基因, 在胃黏膜上皮生长调控过程中起重要作用, 研究显示Runx3基因在胃癌患者中目前发现的杂合性缺失、高甲基化和点突变等机制均参与了Runx3基因在胃癌中的表达缺失或下调, 其中的Runx3基因启动子区域CpG岛的异常甲基化是导致其在胃癌中失活的主要机制[20-22].
本课题组前期研究显示Runx3基因启动子区的异常甲基化及蛋白表达与胃癌的分化程度、大小、浸润深度相关[23,24], 由于抑癌基因的异常甲基化具有可逆性[25-27], 可以通过相关药物逆转其甲基化状态, 使其恢复表达, 重新发挥抑癌基因作用[28,29]. 目前去甲化疗法的药物主要有DNA甲基化转移酶抑制剂5-Aza-dC和组蛋白去乙酰基酶抑制剂TSA, 本研究结果显示经5-Aza-dC和TSA单独或联合干预后的细胞中Runx3的甲基化水平均显著降低, 且联合用药组降低更明显, 表明两药联用较单药去甲基化作用强, 具有协同作用, 可能与DNA去甲基化和组蛋白乙酰化通过甲基结合蛋白相互作用有关[30]. mRNA表达水平结果显示5-Aza-dC和TSA均能增加Runx3基因mRNA表达水平, 两药联合较单药作用明显, 表明Runx3 mRNA表达水平与其启动子区甲基化水平相关, 5-Aza-dC和TSA能逆转Runx3基因启动子区的异常甲基化状态, 使Runx3基因重新表达, 从而使Runx3基因重新起到抑癌基因的作用, 与国内外研究结果一致[30,31]. Runx3蛋白表达水平结果显示各加药组mRNA表达水平越高其蛋白表达水平越高, 进一步验证了实验结果的可靠性, 并且从蛋白表达水平证明了5-Aza-dC和TSA的药效以及两药联合的协调作用.
本研究中QMSP法是目前国际上检测DNA甲基化的较新颖的方法, 能够定量检测出Runx3的甲基化水平, 使各加药组甲基化状态与mRNA和蛋白表达水平的关系得到直观展现; 同时mRNA和蛋白的表达水平随甲基化水平改变而改变, 联合用药组较单药组mRNA和蛋白表达水平高, 甲基化水平较低组的mRNA和蛋白表达水平越高, 表明Runx3基因启动子区甲基化水平与其mRNA和蛋白表达水平相关, 进一步验证了Runx3基因异常甲基化是导致该基因失活的机制[20-22]之一.
总之, 5-Aza-dC和TSA均能逆转胃癌细胞系中Runx3基因启动子区异常甲基化水平, 提高Runx3基因的mRNA和蛋白表达水平, 同时两药联合起到协同作用, 增强了单药药效, 提示抑癌基因Runx3启动子区异常甲基化是导致该基因失活的重要原因之一, 同时为5-Aza-dC和TSA应用于胃癌的临床治疗提供了试验依据.
感谢唐杨琛、刘林青、吴蕾、靳松、沈建军、沈国栋、徐婷娟、杨雯雯及何国平同志在本试验和论文写作中给予的大力帮助.
胃癌是全球发病率最高的癌症之一, 每年新发病例近百万, 其中约2/3的患者来自发展中国家, 仅中国就占到42%, 目前普遍认为表观遗传学改变是继遗传学改变之外, 与胃癌的发生发展和治疗具有密切关系的另一重要机制.
陈国忠, 副主任医师, 广西中医学院第一附属医院消化内科
随着人类对胃癌个体化治疗认识的深入, 抑癌基因甲基化与肿瘤病人对化疗药物敏感性之间的机制研究逐渐成为肿瘤患者个体化治疗领域研究的热点之一, 甲基化水平的监测和逆转为进一步探索靶向及个体化治疗提供了一个新的可能的突破点.
有研究证实decitabine能够逆转胃癌细胞系中RASSF1A 基因的甲基化水平, 并且HDACs抑制剂与decitabine联用能协同逆转RASSF1A基因的甲基化状态和mRNA表达水平, 较单一应用decitabine的作用显著增强.
本研究通过QMSP方法检测基因甲基化状态, 使各组细胞Runx3基因的甲基化水平得到直观的展现, 目前此方法在国内研究中尚较少使用.
通过5-Aza-dC及TSA干预胃癌细胞系SGC-7901, 检测各加药组细胞中Runx3基因甲基化水平及表达水平并对试验结果进行分析, 提示抑癌基因Runx3启动子区甲基化是导致该基因失活的重要原因之一, 同时为5-Aza-dC和TSA应用于胃癌的临床治疗提供了实验依据.
本研究创新性一般, 但实验设计合理, 研究方法可靠, 结果和结论可信, 具有一定的科学意义.
编辑: 曹丽鸥 电编:闫晋利
| 1. | Krejs GJ. Gastric cancer: epidemiology and risk factors. Dig Dis. 2010;28:600-603. [PubMed] [DOI] |
| 2. | Parkin DM, Bray F, Ferlay J, Pisani P. Global cancer statistics, 2002. CA Cancer J Clin. 2005;55:74-108. [DOI] |
| 3. | Yang L. Incidence and mortality of gastric cancer in China. World J Gastroenterol. 2006;12:17-20. [PubMed] |
| 4. | Sato F, Meltzer SJ. CpG island hypermethylation in progression of esophageal and gastric cancer. Cancer. 2006;106:483-493. [PubMed] [DOI] |
| 5. | Esteller M. Epigenetics in cancer. N Engl J Med. 2008;358:1148-1159. [PubMed] [DOI] |
| 6. | Hsu HS, Chen TP, Hung CH, Wen CK, Lin RK, Lee HC, Wang YC. Characterization of a multiple epigenetic marker panel for lung cancer detection and risk assessment in plasma. Cancer. 2007;110:2019-2026. [PubMed] [DOI] |
| 7. | Taberlay PC, Jones PA. DNA methylation and cancer. Prog Drug Res. 2011;67:1-23. [PubMed] |
| 8. | Yamashita K, Sakuramoto S, Watanabe M. Genomic and epigenetic profiles of gastric cancer: potential diagnostic and therapeutic applications. Surg Today. 2011;41:24-38. [PubMed] [DOI] |
| 9. | Cohen Y, Merhavi-Shoham E, Avraham RB, Frenkel S, Pe'er J, Goldenberg-Cohen N. Hypermethylation of CpG island loci of multiple tumor suppressor genes in retinoblastoma. Exp Eye Res. 2008;86:201-206. [PubMed] [DOI] |
| 10. | Eads CA, Lord RV, Wickramasinghe K, Long TI, Kurumboor SK, Bernstein L, Peters JH, DeMeester SR, DeMeester TR, Skinner KA. Epigenetic patterns in the progression of esophageal adenocarcinoma. Cancer Res. 2001;61:3410-3418. [PubMed] |
| 11. | Yamada C, Ozaki T, Ando K, Suenaga Y, Inoue K, Ito Y, Okoshi R, Kageyama H, Kimura H, Miyazaki M. RUNX3 modulates DNA damage-mediated phosphorylation of tumor suppressor p53 at Ser-15 and acts as a co-activator for p53. J Biol Chem. 2010;285:16693-16703. [PubMed] [DOI] |
| 12. | Liu Y, Zhang W, Zhang X, Qi Y, Huang D, Zhang Y. Arsenic trioxide inhibits invasion/migration in SGC-7901 cells by activating the reactive oxygen species-dependent cyclooxygenase-2/matrix metalloproteinase-2 pathway. Exp Biol Med (Maywood). 2011;236:592-597. [PubMed] [DOI] |
| 13. | Baylin SB, Herman JG. DNA hypermethylation in tumorigenesis: epigenetics joins genetics. Trends Genet. 2000;16:168-174. [DOI] |
| 14. | Vogelauer M, Wu J, Suka N, Grunstein M. Global histone acetylation and deacetylation in yeast. Nature. 2000;408:495-498. [PubMed] [DOI] |
| 15. | Rodríguez-Paredes M, Esteller M. Cancer epigenetics reaches mainstream oncology. Nat Med. 2011;17:330-339. [PubMed] [DOI] |
| 16. | Kurkjian C, Kummar S, Murgo AJ. DNA methylation: its role in cancer development and therapy. Curr Probl Cancer. 2008;32:187-235. [PubMed] [DOI] |
| 18. | Tamura G. Alterations of tumor suppressor and tumor-related genes in the development and progression of gastric cancer. World J Gastroenterol. 2006;12:192-198. [PubMed] |
| 19. | Tahara T, Shibata T, Nakamura M, Yamashita H, Yoshioka D, Okubo M, Yonemura J, Maeda Y, Maruyama N, Kamano T. Increased number of CpG island hypermethylation in tumor suppressor genes of non-neoplastic gastric mucosa correlates with higher risk of gastric cancer. Digestion. 2010;82:27-36. [PubMed] [DOI] |
| 20. | Kim TY, Lee HJ, Hwang KS, Lee M, Kim JW, Bang YJ, Kang GH. Methylation of RUNX3 in various types of human cancers and premalignant stages of gastric carcinoma. Lab Invest. 2004;84:479-484. [PubMed] [DOI] |
| 21. | Homma N, Tamura G, Honda T, Matsumoto Y, Nishizuka S, Kawata S, Motoyama T. Spreading of methylation within RUNX3 CpG island in gastric cancer. Cancer Sci. 2006;97:51-56. [PubMed] [DOI] |
| 22. | Fan XY, Hu XL, Han TM, Wang NN, Zhu YM, Hu W, Ma ZH, Zhang CJ, Xu X, Ye ZY. Association between RUNX3 promoter methylation and gastric cancer: a meta-analysis. BMC Gastroenterol. 2011;11:92. [PubMed] [DOI] |
| 23. | Hu SL, Huang DB, Sun YB, Wu L, Xu WP, Yin S, Chen J, Jiang XD, Shen G. Pathobiologic implications of methylation and expression status of Runx3 and CHFR genes in gastric cancer. Med Oncol. 2011;28:447-454. [PubMed] [DOI] |
| 24. | Hu SL, Kong XY, Cheng ZD, Sun YB, Shen G, Xu WP, Wu L, Xu XC, Jiang XD, Huang DB. Promoter methylation of p16, Runx3, DAPK and CHFR genes is frequent in gastric carcinoma. Tumori. 2010;96:726-733. [PubMed] |
| 25. | Luczak MW, Jagodziński PP. The role of DNA methylation in cancer development. Folia Histochem Cytobiol. 2006;44:143-154. [PubMed] |
| 26. | Deaton AM, Bird A. CpG islands and the regulation of transcription. Genes Dev. 2011;25:1010-1022. [DOI] |
| 27. | Ren J, Singh BN, Huang Q, Li Z, Gao Y, Mishra P, Hwa YL, Li J, Dowdy SC, Jiang SW. DNA hypermethylation as a chemotherapy target. Cell Signal. 2011;23:1082-1093. [PubMed] [DOI] |
| 29. | Shen WJ, Dai DQ, Teng Y, Liu HB. Regulation of demethylation and re-expression of RASSF1A gene in gastric cancer cell lines by combined treatment of 5-Aza-CdR and NaB. World J Gastroenterol. 2008;14:595-600. [PubMed] [DOI] |
| 30. | Ou JN, Torrisani J, Unterberger A, Provençal N, Shikimi K, Karimi M, Ekström TJ, Szyf M. Histone deacetylase inhibitor Trichostatin A induces global and gene-specific DNA demethylation in human cancer cell lines. Biochem Pharmacol. 2007;73:1297-1307. [PubMed] [DOI] |