修回日期: 2011-04-13
接受日期: 2011-04-26
在线出版日期: 2011-05-08
目的: 探讨microRNA(miR)-145是否通过调控c-Myc参与肝癌行为的调控.
方法: 实时定量PCR检测肝组织及细胞系miR-145表达水平; 检测不同肝组织c-Myc的mRNA和蛋白表达量; 通过转染上调miR-145在HepG2细胞表达后, 检测转染前后空白组、实验组、阴性对照组HepG2中c-Myc的mRNA和蛋白表达量; 应用流式细胞仪检测转染前后各组HepG2的凋亡情况.
结果: miR-145 mRNA在正常肝组织中表达明显高于癌旁和肝癌组织(0.878±0.146 vs 0.265±0.084, 0.271±0.096, 均P<0.05); 癌旁和肝癌组织中miR-145 mRNA的表达量差别无统计学意义. miR-145 mRNA在LO-2中的表达明显高于HepG2(0.755±0.185 vs 0.471±0.074, P<0.05). c-Myc mRNA和蛋白在癌旁和肝癌中表达均高于正常肝组织(mRNA: 0.136±0.071, 0.451±0.026 vs 0.029±0.023; 蛋白: 0.301±0.022, 0.445±0.018 vs 0.137±0.011, 均P<0.05), 且在肝癌组织中表达量较癌旁组织中明显升高(P<0.05). 转染miR-145 mimics后空白组、实验组、阴性对照组c-Myc mRNA表达差异无统计学意义; 而相对于空白组和阴性对照组, 实验组c-Myc蛋白表达量表达明显下降(0.146±0.011 vs 0.366±0.014, 0.350±0.013, 均P<0.05), 空白组和阴性对照组之间差异无统计学意义. 流式细胞术检测发现, 在HepG2中上调miR-145的表达后, 细胞的凋亡明显增多, 实验组与空白组和阴性对照组之间有明显差异(3.000±0.100 vs 1.167±0.153, 0.933±0.208, 均P<0.05).
结论: miR-145反向调节c-Myc的表达; 表达下调的miR-145丧失对c-Myc的抑制可能是肝癌发生的重要机制; miR-145能够作为抑癌基因促进细胞凋亡.
引文著录: 由法平, 宋孟锜, 陈立波. 低表达的microRNA-145对c-Myc表达的影响. 世界华人消化杂志 2011; 19(13): 1411-1416
Revised: April 13, 2011
Accepted: April 26, 2011
Published online: May 8, 2011
AIM: To investigate whether microRNA-145 (miR-145) controls hepatoma progression through modulating c-Myc activity.
METHODS: Quantitative real-time PCR was used to detect miR-145 expression in different liver lesions and cell lines. Quantitative real-time PCR and Western blot were adopted to detect the expression of c-Myc mRNA and protein in different liver lesions, respectively. After transfection of HepG2 cells with an miR-145 mimic, quantitative real-time PCR, Western blot and flow cytometry were used to detect the expression of c-Myc mRNA and protein and cell apoptosis, respectively.
RESULTS: MiR-145 mRNA was underexpressed in hepatocellular carcinoma (HCC) and tumor-adjacent tissue compared to normal liver tissue (0.878 ± 0.146 vs 0.265 ± 0.084, 0.271 ± 0.096, both P < 0.05). MiR-145 mRNA was also underexpressed in HepG2 cells compared to L-02 cells (0.755 ± 0.185 vs 0.471 ± 0.074, P < 0.05). C-Myc mRNA and protein were overexpressed in HCC and tumor-adjacent tissue compared to normal liver tissue (mRNA: 0.136 ± 0.071, 0.451 ± 0.026 vs 0.029 ± 0.023; protein: 0.301 ± 0.022, 0.445 ± 0.018 vs 0.137 ± 0.011; all P < 0.05). C-Myc protein expression was down-regulated in HepG2 cells transfected with an miR-145 mimic compared to cells transfected with empty vector and untransfected cells (0.146 ± 0.011 vs 0.366 ± 0.014, 0.350 ± 0.013, both P < 0.05). Transfection of an miR-145 mimic promoted the apoptosis of HepG2 cells compared to cells transfected with empty vector and untransfected cells (3.000 ± 0.100 vs 1.167 ± 0.153, 0.933 ± 0.208, both P < 0.05).
CONCLUSION: MiR-145 down-regulation-mediated ablation of inhibition of c-Myc may be a major cause of hepatocarcinogenesis. MiR-145 can promote cell apoptosis.
- Citation: You FP, Song MQ, Chen LB. Down-regulation of microRNA-145 ablates c-Myc inhibition and promotes hepatoma progression. Shijie Huaren Xiaohua Zazhi 2011; 19(13): 1411-1416
- URL: https://www.wjgnet.com/1009-3079/full/v19/i13/1411.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v19.i13.1411
MicroRNA(miRNA)是22 nt左右的微小RNA, 通过RNA诱导的沉默复合物(RNA-induced silencing complex, RISC)与靶基因mRNA的3'UTR区互补结合降解或者抑制靶基因mRNA的翻译从而影响靶基因蛋白的表达[1,2]. 越来越多的证据表明在人类许多肿瘤中都存在miRNA的异常表达. 例如, miR-15a、miR-16-1在慢性淋巴细胞白血病中[3,4], miR-143、miR-145在结直肠癌中[5], let-7在肺癌中[6], miR-155在弥漫性大B细胞淋巴瘤中均有异常表达[7]. miRNA的作用很强大, 研究表明每个miRNA与相当数量的基因有联系(平均每个miRNA家族作用于500个基因)[8-11]. 例如miR-21在胰腺癌、乳腺癌、肺癌、胃癌、CLL、AML的均表达上调[12-17]; miR-34a、miR-34b、miR-34c在结肠癌、肺癌、乳腺癌、肾癌膀胱癌、神经母细胞瘤[18]等肿瘤中表达下调. 异常表达的miRNA[19-21]通过类似于癌基因/抑癌基因功能调控着肝癌的发生发展. 在结肠癌和乳腺癌中有报道P53通过诱导miR-145抑制c-Myc的表达[22], 但miR-145在肝癌中的表达水平以及其与c-Myc之间的关系还未见报道. c-Myc是肝癌发生进展中的关键癌基因, 因而探讨肝癌中miR-145和c-Myc的关系具有重要的价值. 本文通过检测不同肝组织及肝癌细胞系miR-145、c-Myc mRNA及c-Myc蛋白的表达量以及功能学研究, 初步探讨肝癌中miR-145与c-Myc表达异常的关系及miR-145在肝癌发生中的生物学行为.
18例正常肝组织, 53例肝癌组织来源于2009-08-15/2010-11-08在华中科技大学同济医学院附属协和医院门诊肝组织活检, 肝外伤破裂及肝癌切除病例. 所有病例均为35-45岁男性患者, 肝癌患者术前未接受化疗治疗, 最后病理结果诊断明确. 标本切除后30 min以内放入-80 ℃保存备用. LO-2细胞和HepG2细胞均由华中科技大学同济医学院附属协和医院普外实验室冻存, 按常规培养; c-Myc一抗(鼠抗人)购自美国Santa Cruz公司; mRNA实时定量PCR试剂盒购自Invitrogen公司; 引物由Invitrogen公司合成; 凋亡试剂盒Annexin V-FITC Apoptosis Detection Kit(KeyGEN, China)购自凯基公司.
1.2.1 实时定量PCR检测肝脏组织及细胞中miRNA-145 mRNA的表达: 应用TRIzol分别提取组织和细胞的总RNA, 逆转录得到cDNA后进行实时定量PCR. 引物设计资料: hsa-miR-145 MIMAT0000437 GUCCAGUUUUCCCAGGAAUCCCU, 正向特异性引物5'-GTCCAGTTTTCCCAGGA-3'; 内参U6正向特异性引物5'-CTCGCTTCGGCAGCAC A-3'; 反应参数设置: 95 ℃ 5 min; 95 ℃ 10 s, 60 ℃ 20 s, 72 ℃ 10 s, 扩增40个循环. 所得数据用2-ΔΔCt公式校正后再评定个样本之间的差异.
1.2.2 实时定量PCR检测肝脏组织和细胞中c-Myc mRNA的表达量: 应用TRIzol一步法提取各肝组织中总RNA, 逆转录得到cDNA, 然后进行实时定量PCR反应, c-Myc上、下游引物序列分别为5'-CGTCTCCACACATCAGCACAA-3'和5'-TCTTGGCAGCAGGATAGTCCTT-3', 内参β-actin正向引物5'-GAACGGTGAAGGTGACAG-3', 反向引物5'-TAGAGAGAAGTGGGGTGG-3', 反应参数设置: 95 ℃ 2 min; 94 ℃ 30 s, 57 ℃ 30 s; 72 ℃ 30 s, 循环45次; 72 ℃ 5 min[23], 所得数据用2-ΔΔCt公式校正后用来评定各样本间的差异.
1.2.3 Western blot检测肝组织及肝细胞中c-Myc蛋白表达量: 各肝脏组织取0.5 mg, 收集六孔板中融合达80%的细胞提取总蛋白, 采用Bradford蛋白浓度测定试剂盒(碧云天)测定总蛋白浓度, 将定量的蛋白以每孔20-40 µg加样于SDS-PAGE凝胶上样孔中, 200 V电泳至溴酚蓝迁移到距分离胶底部0.5 cm处, 关闭电源; 用300 mA转膜70 min; 将电转膜置于5%的脱脂奶粉(PBS配置)中封闭, 37 ℃振荡2 h; 后加入1:400 c-Myc一抗1 mL, 4 ℃孵育过夜, PBST漂洗3次, 后加入1: 5 000 HRP标记的二抗(用含5%脱脂奶粉的PBS稀释)室温下摇床孵育1 h, PBST漂洗3次, 化学发光显影, 胶片曝光, 结果用UVP扫描仪扫描成像.
1.2.4 转染miR-145 mimics对HepG2细胞中miR-145表达的影响: 取稳定生长的对数生长期的HepG2细胞消化计数, 以平均每孔5.0×105个细胞种植于6孔板中, 分空白组、实验组、阴性对照组, 空白组加无血清无抗生素Opti-MEM培养基2 mL, 实验组加终浓度为50 nmol/L的miR-145 mimics(广东锐博)和5 μL Lipofectamine 2000(Invitrogen), 阴性对照组加终浓度为50 nmol/L的阴性对照试剂(广东锐博)和5 μL Lipofectamine 2000(Invitrogen), 后各组均用Opti-MEM培养基调至2 mL, 转染步骤严格按说明书执行, 转然24 h后检测各组c-Myc的mRNA表达; 48 h后检测各组c-Myc蛋白的表达.
1.2.5 流式检测转染miR-145 mimics后HepG2细胞的凋亡: 用Annexin V-FITC Apoptosis Detection Kit(KeyGEN, China)严格按说明书处理细胞. 将转染48 h后的各组细胞用不含EDTA的0.25%胰酶消化后离心收集细胞, PBS洗涤细胞两次, 离心收集细胞; 加入500 μL的Binding Buffer悬浮细胞, 加入5 μL Annexin V-FITC混匀, 加入5 μL Propidium Iodide, 混匀; 室温, 避光, 反应15 min; 用FACScan进行流式细胞术定量检测, 最后用CELL Quest软件进行数据分析.
统计学处理 各组实验重复3次数据经SPSS18.0软件分析, 计量资料采用mean±SD表示, 组间计量资料采用t检验和方差分析(One-Way ANOVA, LSD method), 以P<0.05为具有统计学意义.
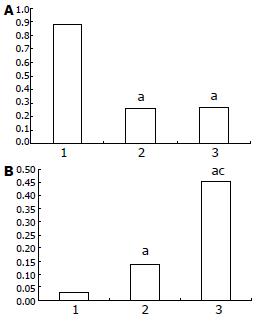
miR-145 mRNA在正常肝脏, 癌旁及肝癌组织中的表达量分别为0.878±0.146, 0.265±0.084, 0.271±0.096(图1A); miR-145 mRNA在LO-2和HepG2细胞系中表达量分别为0.755±0.185, 0.471±0.074; miR-145 mRNA在正常肝组织中表达明显高于癌旁和肝癌组织(均P<0.05); 癌旁和肝癌组织中miR-145 mRNA的表达量差别无统计学意义. miR-145 mRNA在LO-2中明显高于HepG2中的表达(P<0.05).
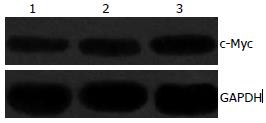
实时定量PCR结果显示正常肝组织, 癌旁组织和肝癌组织中mRNA的表达量分别为: 0.029±0.023, 0.136±0.071, 0.451±0.026(图1B); Western blot显示在正常肝组织, 癌旁组织和肝癌组织中蛋白表达量分别为0.137±0.011, 0.301±0.022, 0.445±0.018(图2). 统计结果显示c-Myc mRNA和蛋白在癌旁和肝癌中表达均高于正常肝组织(P<0.05), 且在肝癌组织中表达量较癌旁组织中明显升高(P<0.05).
根据患者性别、年龄、原发肿瘤大小、分化程度、是否合并肝内子灶、血清AFP及HbsAg检测结果进行分组, 定量比较各组中miR-145的表达状况. 统计结果显示miR-145 mRNA表达量与患者的性别、年龄及肿瘤的大小无关(P>0.05), 但与肝癌分化程度、是否合并肝内转移子灶有关(P<0.05), 与患者血清AFP的浓度及是否合并HBV感染有关(P<0.05, 表1).
| 病理参数 | n | miR-145的表达量 | P值 |
| 性别 | |||
| 男 | 39 | 0.278±0.096 | >0.05 |
| 女 | 14 | 0.252±0.095 | |
| 年龄(岁) | |||
| >50 | 21 | 0.278±0.098 | >0.05 |
| <50 | 32 | 0.267±0.095 | |
| 肿瘤大小(cm) | |||
| <3 | 11 | 0.321±0.089 | >0.05 |
| >3 | 42 | 0.258±0.094 | |
| 分化程度 | |||
| 好 | 16 | 0.315±0.098 | <0.05 |
| 差 | 37 | 0.252±0.089 | |
| 肝内转移子灶 | |||
| 有 | 12 | 0.204±0.048 | <0.05 |
| 无 | 41 | 0.291±0.094 | |
| AFP浓度(μg/L) | |||
| <400 | 18 | 0.310±0.084 | <0.05 |
| >400 | 35 | 0.252±0.096 | |
| HbsAg | |||
| 阳性 | 39 | 0.254±0.095 | <0.05 |
| 阴性 | 14 | 0.318±0.082 |
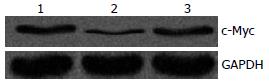
转染miR-145 mimics 24 h后提取总RNA, 进行实时定量PCR检测空白组、实验组、阴性对照组c-Myc mRNA表达量分别为2.614±0.863, 2.382±0.769, 2.771±0.614; 转染48 h后提取总蛋白Western blot检测空白组、实验组、阴性对照组c-Myc蛋白的表达量分别为0.366±0.014, 0.146±0.011, 0.350±0.013(图3). 统计结果显示, 转染miR-145 mimics后空白组、实验组、阴性对照组c-Myc mRNA表达差异无统计学意义; 而相对于空白组和阴性对照组, 实验组c-Myc蛋白表达量明显下降(P<0.05), 空白组和阴性对照组之间差异无统计学意义.
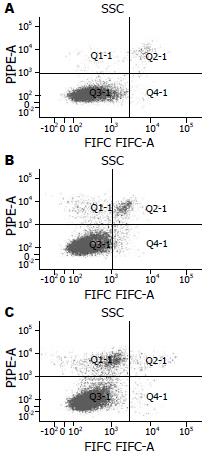
转染miR-145 mimics后48 h行流式细胞术定量检测HepG2细胞的凋亡水平: 在HepG2中上调miR-145的表达后, 细胞的凋亡明显增多(图4). 空白对照组、实验组、阴性对照组细胞凋亡率分别为1.167±0.153, 3.0±0.1, 0.933±0.208, 实验组与空白组和阴性对照组之间有明显差异(P<0.05).
迄今为止, 人类有近15%-20%的基因受c-Myc的直接或间接的调控, 这些基因与细胞的周期调控、蛋白合成、细胞结构的形成、运动性、代谢等等有关[24], 共同调控着细胞的生长、分化和凋亡[25]. 基因分析在病毒相关性肝癌及酒精相关性肝癌中有70%的患者存在c-Myc的高表达[26]; 早已有报道c-Myc能直接上调miR-17-92簇的表达, 促进肿瘤的发生发展[27]; Chang等[28]在此研究的基础上发现在B细胞淋巴瘤中有超过10种miRNA的表达受c-Myc的调控, 染色体免疫共沉淀研究证实c-Myc能与miRNA的上游保守序列或启动子序列特异性结合, 抑制miRNA的表达, 促进肿瘤的发展. 因此c-Myc的异常表达与肿瘤有密切的关系. 但c-Myc的异常表达机制的原因尚不明确, 是否受miRNA的调控仍不明确. 有报道在HeLa细胞中研究发现let-7能特异性结合c-Myc的3'UTR在mRNA和蛋白水平抑制c-Myc的表达[29]; 生理状态下P53能够直接作用于miR-34家族抑制c-Myc的表达诱导细胞周期停止和细胞凋亡[30], 等等. 在结肠癌和乳腺癌中曾有报道c-Myc是miR-145的直接作用靶点[22], 且P53通过诱导miR-145表达能调控c-Myc的表达. 但在肝癌中miR-145与肝细胞癌之间类似的关系尚未见报道, 是否miR-145同样反向调控c-Myc的表达尚不清楚.
我们发现miR-145 mRNA在正常肝组织中表达明显高于癌旁和肝癌组织; c-Myc mRNA和蛋白在癌旁和肝癌中表达均高于正常肝组织, 且在肝癌组织中表达量较癌旁组织中明显升高, 提示c-Myc的异常高表达可能与miR-145异常低表达有关; 临床病理参数显示miR-145的低表达与患者的性别、年龄及肿瘤大小无关, 与肿瘤的分化程度、是否合并HBV感染和肝内转移子灶及患者血清AFP浓度等有关, 提示miR-145低表达的患者, 肿瘤恶性程度高、容易转移, 预后较差; 由于miRNA-145 mRNA在LO-2中明显高于HepG2中的表达, 我们利用miR-145 mimics转染HepG2细胞, 上调miR-145的表达量进一步验证了, miR-145能够在蛋白水平抑制c-Myc的表达, 但在mRNA水平实验组与空白组及阴性对照组无明显差异. 结合在组织中miR-145与c-Myc的异常表达, 提示在肝癌中miR-145能反向调控c-Myc的表达, 进一步研究发现上调miR-145还能促进肝癌细胞凋亡.
总之, 我们的研究证实了在肝癌中miR-145与c-Myc的关系及miR-145能够作为抑癌基因促进癌细胞凋亡, 为肝细胞癌的治疗提供了一条新的途径, 但是在肝细胞肝癌中miR-145异常低表达的机制尚不明确, 是否受DNA甲基化或(和)组蛋白甲基化的调控及HBV的影响将是我们下一步的研究方向.
人类许多肿瘤中都存在miRNA的异常表达, 异常表达的miRNA通过类似于癌基因/抑癌基因功能调控着肝癌的发生发展. 有报道P53通过诱导miR-145抑制c-Myc的表达, 但miR-145在肝癌中的表达水平以及其与c-Myc之间的关系还未见报道. c-Myc是肝癌发生进展中的关键癌基因, 因而探讨肝癌中miR-145和c-Myc的关系具有重要的价值.
荚卫东, 教授, 安徽省立医院肝胆外科
现在关于miRNA的研究主要集中在miRNA在肿瘤的发生发展中与癌基因/抑癌基因的相互作用及miRNA本身变化对其他基因的影响.
Chang等在此研究的基础上发现在B细胞淋巴瘤中有超过10种miRNA的表达受c-Myc的调控, 染色体免疫共沉淀研究证实c-Myc能与miRNA的上游保守序列或启动子序列特异性结合, 抑制miRNA的表达促进肿瘤的发展. 因此c-Myc的异常表达与肿瘤有密切的关系.
在结肠癌和乳腺癌中曾有报道c-Myc是miR-145的直接作用靶点, 且P53通过诱导miR-145表达能调控c-Myc的表达. 但在肝癌中miR-145与肝细胞癌之间类似的关系尚未见报道, 是否miR-145同样反向调控c-Myc的表达尚未见报道.
本文的研究证实了在肝癌中miR-145与c-Myc的关系, miR-145能够作为抑癌基因促进癌细胞凋亡, 为肝细胞癌的治疗提供了一条新的途径.
本文选题新颖, 有一定的科学价值.
编辑: 李薇 电编:李薇
| 1. | Gregory RI, Chendrimada TP, Cooch N, Shiekhattar R. Human RISC couples microRNA biogenesis and posttranscriptional gene silencing. Cell. 2005;123:631-640. [PubMed] [DOI] |
| 2. | Tang F, Hajkova P, O'Carroll D, Lee C, Tarakhovsky A, Lao K, Surani MA. MicroRNAs are tightly associated with RNA-induced gene silencing complexes in vivo. Biochem Biophys Res Commun. 2008;372:24-29. [PubMed] [DOI] |
| 3. | Calin GA, Liu CG, Sevignani C, Ferracin M, Felli N, Dumitru CD, Shimizu M, Cimmino A, Zupo S, Dono M. MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias. Proc Natl Acad Sci U S A. 2004;101:11755-11760. [PubMed] [DOI] |
| 4. | Calin GA, Sevignani C, Dumitru CD, Hyslop T, Noch E, Yendamuri S, Shimizu M, Rattan S, Bullrich F, Negrini M. Human microRNA genes are frequently located at fragile sites and genomic regions involved in cancers. Proc Natl Acad Sci U S A. 2004;101:2999-3004. [PubMed] [DOI] |
| 5. | Michael MZ, O' Connor SM, van Holst Pellekaan NG, Young GP, James RJ. Reduced accumulation of specific microRNAs in colorectal neoplasia. Mol Cancer Res. 2003;1:882-891. [PubMed] |
| 6. | Johnson SM, Grosshans H, Shingara J, Byrom M, Jarvis R, Cheng A, Labourier E, Reinert KL, Brown D, Slack FJ. RAS is regulated by the let-7 microRNA family. Cell. 2005;120:635-647. [PubMed] [DOI] |
| 7. | Eis PS, Tam W, Sun L, Chadburn A, Li Z, Gomez MF, Lund E, Dahlberg JE. Accumulation of miR-155 and BIC RNA in human B cell lymphomas. Proc Natl Acad Sci U S A. 2005;102:3627-3632. [PubMed] [DOI] |
| 8. | Lewis BP, Shih IH, Jones-Rhoades MW, Bartel DP, Burge CB. Prediction of mammalian microRNA targets. Cell. 2003;115:787-798. [PubMed] [DOI] |
| 9. | Krek A, Grün D, Poy MN, Wolf R, Rosenberg L, Epstein EJ, MacMenamin P, da Piedade I, Gunsalus KC, Stoffel M. Combinatorial microRNA target predictions. Nat Genet. 2005;37:495-500. [PubMed] [DOI] |
| 10. | Betel D, Wilson M, Gabow A, Marks DS, Sander C. The microRNA.org resource: targets and expression. Nucleic Acids Res. 2008;36:D149-D153. [PubMed] [DOI] |
| 11. | Friedman RC, Farh KK, Burge CB, Bartel DP. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 2009;19:92-105. [PubMed] [DOI] |
| 12. | Calin GA, Ferracin M, Cimmino A, Di Leva G, Shimizu M, Wojcik SE, Iorio MV, Visone R, Sever NI, Fabbri M. A MicroRNA signature associated with prognosis and progression in chronic lymphocytic leukemia. N Engl J Med. 2005;353:1793-1801. [PubMed] [DOI] |
| 13. | Iorio MV, Ferracin M, Liu CG, Veronese A, Spizzo R, Sabbioni S, Magri E, Pedriali M, Fabbri M, Campiglio M. MicroRNA gene expression deregulation in human breast cancer. Cancer Res. 2005;65:7065-7070. [PubMed] [DOI] |
| 14. | Garzon R, Volinia S, Liu CG, Fernandez-Cymering C, Palumbo T, Pichiorri F, Fabbri M, Coombes K, Alder H, Nakamura T. MicroRNA signatures associated with cytogenetics and prognosis in acute myeloid leukemia. Blood. 2008;111:3183-3189. [PubMed] [DOI] |
| 15. | Schetter AJ, Leung SY, Sohn JJ, Zanetti KA, Bowman ED, Yanaihara N, Yuen ST, Chan TL, Kwong DL, Au GK. MicroRNA expression profiles associated with prognosis and therapeutic outcome in colon adenocarcinoma. JAMA. 2008;299:425-436. [PubMed] [DOI] |
| 16. | Calin GA, Dumitru CD, Shimizu M, Bichi R, Zupo S, Noch E, Aldler H, Rattan S, Keating M, Rai K. Frequent deletions and down-regulation of micro- RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia. Proc Natl Acad Sci U S A. 2002;99:15524-15529. [PubMed] [DOI] |
| 17. | Yanaihara N, Caplen N, Bowman E, Seike M, Kumamoto K, Yi M, Stephens RM, Okamoto A, Yokota J, Tanaka T. Unique microRNA molecular profiles in lung cancer diagnosis and prognosis. Cancer Cell. 2006;9:189-198. [PubMed] [DOI] |
| 18. | Ciafrè SA, Galardi S, Mangiola A, Ferracin M, Liu CG, Sabatino G, Negrini M, Maira G, Croce CM, Farace MG. Extensive modulation of a set of microRNAs in primary glioblastoma. Biochem Biophys Res Commun. 2005;334:1351-1358. [PubMed] [DOI] |
| 19. | Jiang J, Gusev Y, Aderca I, Mettler TA, Nagorney DM, Brackett DJ, Roberts LR, Schmittgen TD. Association of MicroRNA expression in hepatocellular carcinomas with hepatitis infection, cirrhosis, and patient survival. Clin Cancer Res. 2008;14:419-427. [PubMed] [DOI] |
| 20. | Pineau P, Volinia S, McJunkin K, Marchio A, Battiston C, Terris B, Mazzaferro V, Lowe SW, Croce CM, Dejean A. miR-221 overexpression contributes to liver tumorigenesis. Proc Natl Acad Sci U S A. 2010;107:264-269. [PubMed] [DOI] |
| 21. | 马 兆龙, 杨 炼, 陈 立波, 黄 金明, 王 冬冬, 王 国斌. miRNA在HBV从感染经由肝硬化到肝癌进程中表达谱的变化. 世界华人消化杂志. 2009;17:2112-2116. [DOI] |
| 22. | Sachdeva M, Zhu S, Wu F, Wu H, Walia V, Kumar S, Elble R, Watabe K, Mo YY. p53 represses c-Myc through induction of the tumor suppressor miR-145. Proc Natl Acad Sci U S A. 2009;106:3207-3212. [PubMed] [DOI] |
| 24. | Gao P, Tchernyshyov I, Chang TC, Lee YS, Kita K, Ochi T, Zeller KI, De Marzo AM, Van Eyk JE, Mendell JT. c-Myc suppression of miR-23a/b enhances mitochondrial glutaminase expression and glutamine metabolism. Nature. 2009;458:762-765. [PubMed] [DOI] |
| 25. | Dang CV, O'Donnell KA, Zeller KI, Nguyen T, Osthus RC, Li F. The c-Myc target gene network. Semin Cancer Biol. 2006;16:253-264. [PubMed] [DOI] |
| 26. | Schlaeger C, Longerich T, Schiller C, Bewerunge P, Mehrabi A, Toedt G, Kleeff J, Ehemann V, Eils R, Lichter P. Etiology-dependent molecular mechanisms in human hepatocarcinogenesis. Hepatology. 2008;47:511-520. [PubMed] [DOI] |
| 27. | O'Donnell KA, Wentzel EA, Zeller KI, Dang CV, Mendell JT. c-Myc-regulated microRNAs modulate E2F1 expression. Nature. 2005;435:839-843. [PubMed] [DOI] |
| 28. | Chang TC, Yu D, Lee YS, Wentzel EA, Arking DE, West KM, Dang CV, Thomas-Tikhonenko A, Mendell JT. Widespread microRNA repression by Myc contributes to tumorigenesis. Nat Genet. 2008;40:43-50. [PubMed] [DOI] |
| 29. | Corney DC, Flesken-Nikitin A, Godwin AK, Wang W, Nikitin AY. MicroRNA-34b and MicroRNA-34c are targets of p53 and cooperate in control of cell proliferation and adhesion-independent growth. Cancer Res. 2007;67:8433-8438. [PubMed] [DOI] |
| 30. | Koscianska E, Baev V, Skreka K, Oikonomaki K, Rusinov V, Tabler M, Kalantidis K. Prediction and preliminary validation of oncogene regulation by miRNAs. BMC Mol Biol. 2007;8:79. [PubMed] [DOI] |