修回日期: 2009-04-23
接受日期: 2009-05-11
在线出版日期: 2009-06-08
目的: 筛选、鉴定及验证植物乳酸杆菌CGMCC No. 1258表层黏附蛋白.
方法: 用盐酸胍结合超速离心法提取表层膜蛋白, 通过HRP标记的黏蛋白作抗体Western blot找到含有黏附蛋白的条带进行质谱分析, 生物信息学的TPP软件分析获得可能的蛋白, 进行蛋白的分段表达克隆, 并对纯化的蛋白用Western blot进行再次鉴定找到靶蛋白. 同时将黏蛋白连接的sepharose 4B柱提取菌体黏附蛋白, 通过制备的靶蛋白多克隆抗体对菌体黏附蛋白作Western blot进一步验证.
结果: 表层蛋白提取后, 电泳及Western blot结果显示30-33 kDa处可及HRP标记的黏蛋白结合的阳性条带, 随后进行质谱及TPP分析获得的蛋白为: (1)A型GTP连接蛋白; (2)丙酮酸氧化酶; (3)细胞分裂激活蛋白; (4)整合膜蛋白; (5)后期能力蛋白. 并进行克隆表达及分段表达, 并在此后的Western blot鉴定中呈强阳性条带显示靶蛋白是整合膜蛋白的第2段. 在应用该靶蛋白制备的多克隆抗体进一步Western blot验证试验中发现同菌体黏附蛋白相作用的强阳性条带.
结论: 植物乳酸杆菌表层黏附蛋白为整合膜蛋白, 并且该蛋白的黏附区域位于其第2段氨基酸序列中.
引文著录: 沈通一, 张明, 周玉坤, 陈红旗, 张鹏, 杭晓敏, 秦环龙. 基于生物信息学的植物乳酸杆菌表层黏附蛋白的筛选及鉴定. 世界华人消化杂志 2009; 17(16): 1626-1631
Revised: April 23, 2009
Accepted: May 11, 2009
Published online: June 8, 2009
AIM: To identify, screen and verify the surface layer protein of Lactobacillus plantarum CGMCC NO.1258 based on the adhering character of the Lactobacillus.
METHODS: Surface layer protein was extracted by guanidine hydrochloride and ultracentrifugation. The band contained the adhering protein was found by Western blot method using the hrp-labeled mucin as the antibody, and was analyzed by mass spectrometry and TPP software to detect the potential proteins. Fragments of these proteins were cloned and expressed, and these purified proteins were screened by Western blot to find out the target protein. Meanwhile the adhesive protein of the germ was extracted by mucin linked sepharose-4B and was further verified with Western blot using the prepared polyclonal antibody of the target protein.
RESULTS: The surface layer of Lactobacillus was isolated. The electrophoresis and Western blot result showed that the positive band which reacted with the HRP labeled mucin existed in 30 kDa. This band analyzed by LC-MS/MS and the TPP software contained five kind of protein including GTP-binding protein TypA (GBP), pyruvate oxidase (PO), cell division initiation protein FtsQ (CD), integral membrane protein (IMP), late competence protein (LCP). These proteins were cloned and expressed fragmentally. The positive band in the Western blot showed that the second section of the integral membrane protein (IMP2) was the target protein. In the further Western blot verification with the polyclonal antibody prepared by the IMP2 showed the positive band in the adhesive proteins of the germ.
CONCLUSION: The surface layer adhesive protein of Lactobacillus plantarum cgmcc N01258 is an integral membrane protein, and the adhering region is located at the second fragment of the amino acid sequence.
- Citation: Shen TY, Zhang M, Zhou YK, Chen HQ, Zhang P, Hang XM, Qin HL. Identification and screening of Lactobacillus plantarum surface layer adhesive protein based on bioinformatics. Shijie Huaren Xiaohua Zazhi 2009; 17(16): 1626-1631
- URL: https://www.wjgnet.com/1009-3079/full/v17/i16/1626.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v17.i16.1626
肠道内的乳酸杆菌作为一种益生菌, 是肠道生物屏障的重要组成部分, 他可以通过其竞争黏附作用有效抑制条件致病菌对肠上皮的定植, 进而抑制细菌移位起到了屏障功能[1-2]. 近来研究较多的黏附分子包括脂磷壁酸(lipoteichoic acids, LTA)、细胞外多糖(extracellular polysaccharide, EPS)、细胞表层蛋白等, 其中细胞表层蛋白起着关键性的作用[3]. 由于表层的膜蛋白特有的亲疏水性结构所导致的生化纯化及结构功能分析的困难, 使其相比可溶性蛋白, 较少深入研究[4]. 受此影响, 国内外对乳酸杆菌同肠上皮黏附过程中的起作用的表层膜蛋白的报道较少, 为进一步研究乳酸杆菌和肠上皮的黏附机制, 本研究以植物乳酸杆菌CGMCC No. 1258(Lactobacillus plantarum CGMCC No. 1258, L. plantarum CGMCC No. 1258)为对象, 对其表层黏附蛋白进行反复的筛选和鉴定, 进一步验证其作用.
L. plantarum CGMCC No. 1258由交通大学昂立微生物研究所提供. 超速离心机购自Beckman公司. Mucin购自Sigma公司, Fermentas蛋白Marker及Cell Signal生物素标记Marker购自吉泰公司. 表达质粒pET16b购自Novagen司, 在多克隆位点添加了BglⅡ和XhoⅠ酶切位点. 限制性内切酶、连接酶、及TaqDNA聚合酶和IPTG均购自MBI公司. 质粒提取试剂盒、DNA凝胶回收试剂盒AXgen和HRP标记的山羊抗鼠IgG购于北京中杉生物工程公司. DH10B细胞由本室保存. 引物合成及测序工作由生工完成.
1.2.1 L. plantarum的培养和表层蛋白的提取: 将L. plantarum CGMCC No. 1258接种于新鲜配置的MRS液体培养基, 37 ℃ 200 r/min培养24 h. 将菌液3500 g, 4 ℃离心20 min, 去除上清, 再用PBS混匀3500 g, 4 ℃离心20 min, 去除上清后加入2 mol/L盐酸胍室温200 r/min, 3 h. 将菌液6000 g, 4 ℃离心20 min取上清液置半透膜内0.01 mol/L的PBS透析过夜, 加入离心管放进4 ℃超速离心机40000 g, 离心60 min, 小心抽去上清. 沉淀溶于0.1 mol/L的PBS, 放于70 ℃恒温水槽孵育30 min, 16000 g, 4 ℃离心20 min, 上清液移出后即可进行后续Western blot操作.
1.2.2 对黏蛋白作辣根过氧化酶(HRP)标记: 根据Rojas et al[5]的方法, 取黏蛋白溶解于0.1 mol/L碳酸盐缓冲液中(pH 9.5), 配置成4 g/L的黏蛋白溶液. 将辣根过氧化物酶(HRP)8 mg溶解于2 mL蒸馏水中, 配置成HRP溶液, 然后加入至400 μL新鲜配置的0.1 mol/L过碘酸钠溶液中, 混合物室温下搅拌20 min, 然后置于0.001 mol/L(pH 4.4)乙酸盐缓冲液中, 4 ℃透析过夜. 取出透析后的HRP, 加入0.1 mol/L碳酸盐缓冲液中(pH 9.5)20 μL调节pH至9.0-9.5, 取1 mL黏蛋白和1 mL HRP溶液混合, 置于室温下混合搅拌2 h, 然后加入新鲜配置的4 mg/L硼氢化钠溶液100 μL以去除连接反应. 混合液置于0.1 mol/L(pH 7.4)的硼酸缓冲液中4 ℃透析过夜, 标记好的黏蛋白等体积加入80%甘油, -20 ℃保存[6].
1.2.3 表层蛋白的Western blot分析: 表达产物经SDS-PAGE电泳, 采用10%分离胶和5%浓缩胶的不连续浓度梯度, 恒压60V 30 min, 后改120 V 90 min. 电泳完毕后, 部分胶用考马斯亮蓝进行染色, 另一部分胶移至电转膜仪上, 100 V 120 min,用半干转移系统将蛋白转印至PVDF膜上, 根据同细菌表面黏附蛋白可以同黏蛋白特异性结合的原理, 加入1:600稀释的HRP标记的黏蛋白作为抗体, 同PVDF膜上的相关蛋白进行杂交, 4 ℃孵育过夜, TBST洗膜3次, 每次30 min, ECL显色检测目的蛋白.
1.2.4 LC-MS/MS: 根据Western blot结果选取PAGE胶上蛋白条带, 通过液质联用(LC-MS/MS)测定氨基酸序列, 并通过Sequest软件做初步分析. 根据质谱分析的结果, 通过生物信息数据分析软件(trans-proteomics pipeline, TPP)软件包及FASTA软件进一步筛选分析相关蛋白.
1.2.5 初筛蛋白基因的克隆: MS结果发现可能是以下5种蛋白: (1)A型GTP连接蛋白(GTP-binding protein TypA, GBP); (2)丙酮酸氧化酶(pyruvate oxidase, PO); (3)细胞分裂激活蛋白(cell division initiation protein FtsQ, CD); (4)整合膜蛋白(integral menbrane protein, IMP); (5)后期能力蛋白(late competence protein, LCP), 分别对其进行克隆和表达(表1); 应用expasy(http://cn.expasy.org/)中的TMHMM工具, 显示跨膜区. 并根据蛋白质序列分析对Integral menbrane protein的膜外部分作分段表达. 以GenBank中各蛋白的基因序列为模板, 设计引物(表1), 进行PCR反应. 反应程序为基因组DNA经94 ℃变性3 min; 随后按94 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 2 min; 30个循环, 72 ℃ 10 min. PCR结束后10 g/L琼脂糖电泳鉴定.
| 基因名称 | 上游引物 | 下游引物 |
| PO | atcagatctgATGGCAAAGATGATTGAAGC | atcctcgagCTATTCGACGTTGAACTCCT |
| GB | atcagatctgTTGAAATTTAGAGATGATAT | atcctcgagTTACTTGTTCATTTTACGCT |
| CD | atcagatctgGTGGGTGTGATGGCCATTTT | atcctcgagTCAATTATTCTGATTCTTGA |
| LC | atcagatctgGTGCTAGCATGCCTACTGTG | atcctcgagTTAACGTGCTAGCGTTAGCC |
| IMP1 | atcagatctgTTGAAGACAGTACGGAAGAT | atcctcgagAATCGCAGCAAGATCAACGC |
| IMP2 | atcagatctgAATACTAATTCTAATAGTAC | atcctcgagAAGATAGAGTTCCGTATTTC |
| IMP3 | atcagatctgAAGCCAACCGTTACCGGAAA | atcctcgagTTTTAGTCCTGGTGTGTGAT |
PCR产物经过AXgen胶试剂盒回收后, 与原核表达载体pET16b同时用核酸内切酶BglⅡ和XhoⅠ酶切过夜, 以T4连接酶连接, 构建原核重组表达质粒. 然后转化DH10B感受态, 铺板, 挑选单克隆培养, 菌液PCR筛选阳性克隆的质粒进行测序, 测序结果用在线程序BLAST进行比较.
1.2.6 初筛蛋白的表达: 将测序正确的克隆抽质粒, 转化BL21(DE3)感受态, 次日挑取菌落接种于含氨苄和氯霉素TB培养基, 37 ℃ 200 r/min到A值0.6时, 加IPTG, 并调整IPTG, 终浓度为1 mmol/L, 诱导时间3 h, 以期获得重组蛋白的表达. 诱导结束, 经离心收集细菌后, 冰浴下超声裂解细菌. 4 ℃, 14000 r/min离心10 min分离上清. 上清中加入His-Tag Fusion Protein Purification bead, 震荡30 min纯化浓缩上清中的蛋白.
1.2.7 表达蛋白的Weastern blot分析: 方法同前, 纯化的蛋白经SDS-PAGE电泳, 部分胶用考马斯亮蓝进行染色, 另一部分胶转印至PVDF膜上,加入1:600稀释的HRP标记的黏蛋白进行杂交, ECL 显色检测目的蛋白.
1.2.8 表层黏附蛋白多克隆抗体制备: 将确定的靶蛋白纯化, 按常规方法免疫家兔. 首次免疫时, 按每只家兔600 μg的剂量, 将纯化得到的蛋白溶于PBS(终浓度为2 g/L)中, 加入等体积的弗氏完全佐剂, 充分乳化后背部皮下及腹股沟多点注射; 每点200 μg乳化抗原. 加强免疫, 抗原用量为初次免疫的一半, 加弗氏不完全佐剂, 于前次免疫后的2 wk进行. 最后1次加强免疫后的14 d后放血, 取血清. 免疫血清用抗原亲和层析柱进行纯化, -20 ℃保存.
多克隆抗体鉴定: (1)Western blot鉴定: 方法同前, 将1、10、100 pg的纯化的重组蛋白作 SDS-PAGE分离, 转移到PVDF膜上, 加入纯化后的兔抗IMP2多克隆抗体(1:10000稀释), 然后加入辣根过氧化物酶标记的羊抗兔二抗, ECL检测. (2)ELISA检测: 将纯化的重组蛋白用PBS稀释至1 mg/L, 按照100 μL/孔的量包被到聚苯乙烯反应(PVC)板上, 4 ℃过夜包被; 用PBST洗涤3次, 然后按照200 μL/孔的量加入封闭液; 用PBST洗涤3次, 每孔加入100 μL倍比的免疫血清, 同时设正常兔血清作为阴性对照, 每个样品设2个复孔, 37 ℃孵育2 h; 用PBST洗涤3次, 每孔均加入100 μL PBS(含1 g/L BSA)稀释的二抗(辣根过氧化物酶标记的羊抗兔二抗, 1:5000稀释, Santa Cruz), 37 ℃孵育1 h; 用PBST洗涤3次, 加入50 μL OPD显色液, 室温避光显色10-15 min, 用25 μL 2 mol/L的硫酸终止反应; 在490 nm波长下用酶标仪读数.
1.2.9 全菌体黏附蛋白的纯化: 菌液离心后, 先用PBS将细菌沉淀洗2-3遍, 然后按10-50 mL裂解液/1 g湿菌体加入裂解液重悬菌体, 裂解液的成分: 50 mmol/L Tris-HCl, pH 8.0, 2 mmol/L EDTA, 100 mmol/L NaCl, pH值8.5-9.0, 加溶菌酶至100 mg/L, 0.5% Triton X-100, 1 mL/L的蛋白酶抑制剂PMSF. 进行超声粉碎, 超声功率300 W, 时间20 min. 离心后收取上清中的可溶性蛋白. 蛋白溶液过空sepharose 4B柱去除非特异结合的蛋白, 蛋白溶液过黏蛋白连接的sepharose 4B柱, 柱上的黏附蛋白用20 mmol/L的1, 3-二氨基丙烷(1, 3-diaminopropane, DAP)洗脱而得到纯化的全菌体黏附蛋白[7].
1.2.10 Western blot第3次验证: 具体方法同前, 将纯化全菌体黏附蛋白作SDS-PAGE电泳分离, 转移到PVDF膜上, 加入纯化后的兔抗IMP2多克隆抗体(1:10000稀释), 然后加入辣根过氧化物酶标记的羊抗兔二抗, ECL检测.
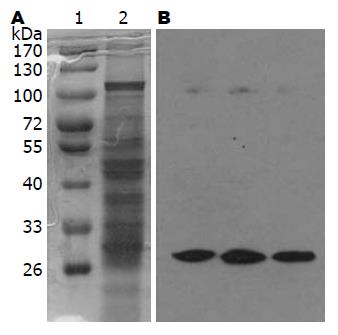
对照Marker可及在100-130、55、35-40、30-33 kDa等位置有多条条带(图1A). 在PVDF膜的30 kDa处有一强阳性条带, 黏附蛋白位于这条带中(图1B).
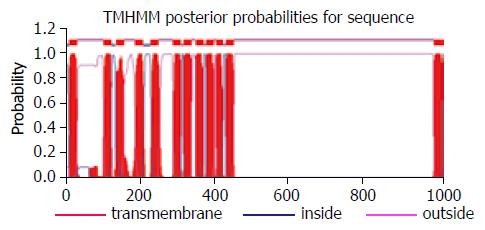
数据库检索分析以及ProteinProphet算法[8]进行结果过滤, 结果显示有5种蛋白(表2)同植物乳酸菌WCFS的蛋白库符合率达到90%. 其中, 整合膜蛋白分为3段蛋白片段(图2), 分别称为IMP1(SEQ ID No: 1中第32-100位序列)、IMP2(SEQ ID No: 1中第455-755位序列)、IMP3(SEQ ID No: 1中第693-993位序列).
| 编号 | 蛋白名称 | PI | GenBank登录号 | 表达途径 |
| No.1 | 丙酮酸氧化酶 | 5.0 | ABC47131.1 | 全长 |
| No.2 | 细胞分裂激活蛋白 | 10.32 | NP_785691.1 | 全长 |
| No.3 | A型GTP-结合蛋白 | 5.12 | NP_785653.1 | 全长 |
| No.4 | 后期能力蛋白 | 9.84 | NP_784489.1 | 全长 |
| No.5 | 整合膜蛋白 | 10.02 | NP_785773.1 | 3段 |
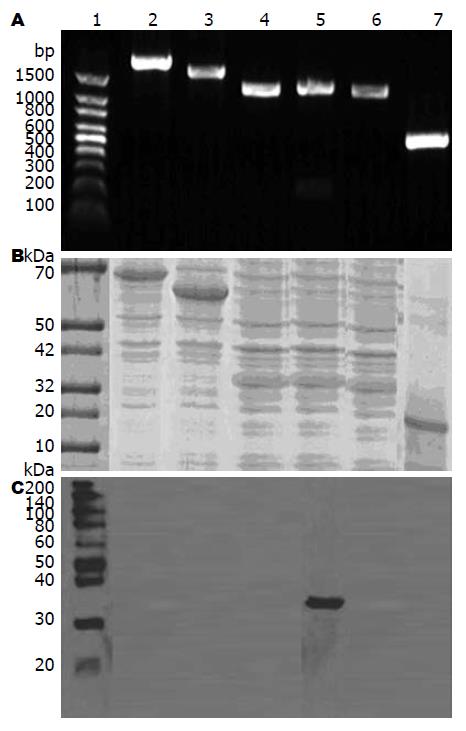
通过PCR对L. plantarum CGMMCC No. 1258的目标基因进行扩增. PCR产物电泳后在10 g/L琼脂凝胶的1500-500 bp位置呈现单根条带, 其结果符合预期(图3). PCR产物经琼脂糖凝胶电泳分离纯化后重组到PET-16B载体中. 质粒转化感受态DH10B并测序. 结果显示L. plantarum CGMMCC No. 1258的扩增基因同GenBank中的L. plantarum WSFC资料一致. 随后含有目的基因的质粒转染入E.coli BL23. isopropyl-b-D-thiogalactopyranoside(IPTG)诱导3 h. 超声裂解细胞, His-tag fusion protein purification beads抽提纯化目的蛋白. 蛋白洗脱后100 g/L SDS-PAGE电泳分离同时用HRP-mucin作抗体Western blot分析(图3C). 结果显示所有目的蛋白表达量均占菌体总蛋白的90%以上. PVDF膜IMP2的30 kDa处有一相同形状的强阳性条带, 即黏附蛋白, 而其他蛋白未见阳性条带. Western blot结果显示纯化的IMP2蛋白就是目的蛋白.
Western blot检测结果显示, 重组蛋白IMP2免疫新西兰兔制备的多克隆抗体能很好地和IMP2特异性结合, 即使是含量为1 pg的IMP2在PVDF膜上也能够很好的分辨出来(图4). 采用间接ELISA法(表3)检测多克隆抗体的效价, 结果表明本实验所获得的兔抗IMP2多克隆抗体用包被IMP2进行质量鉴定, 效价为1:2×106, A值为1.186.
| 名称 | 阴性血清 | 空白对照 | IM2抗体 | IM2抗体 | IM2抗体 | IM2抗体 | IM2抗体 | IM2抗体 |
| 抗体稀释度 | 1/1000 | 1/1000 | 1/16000 | 1/64000 | 1/256000 | 1/512000 | 1/1024000 | 1/2048000 |
| A值 | 0.023 | 0.021 | 2.772 | 2.635 | 2.580 | 2.218 | 1.799 | 1.186 |
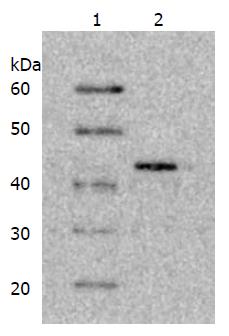
通过mucin-sepharose 4B column亲和纯化的全菌体黏附蛋白电泳后, 用抗IMP2多克隆抗体作Western blot分析, 结果可见40-50 kDa有与理论相符的阳性条带(图5), 这结果反映了IMP2存在于全菌体黏附蛋白中.
本研究将质谱分析结果同L. plantarum WCFS-1的蛋白库比对, 并通过对相关蛋白的表达和筛选, 在国际上首次发现L. plantarum CGMCC No. 1258表层存在着同肠道内黏蛋白黏附的整合膜蛋白(integral membrane protein, IMP, GI: 28378881), 其氨基酸序列和L. plantarum WCFS-1的蛋白序列同源. 在以往的研究中, Rojas et al[5]根据乳酸杆菌同黏蛋白的黏附作用, 以黏蛋白作为抗体, 用HRP标记后与L. fermentum 104R的菌体表层蛋白作Western blot, 通过肽段测序鉴定了菌体表面蛋白中参与黏附的蛋白. Granato et al[9]将L. johnsonii NCC533(La1)用LiCl分离到一种50 kDa的表面蛋白, 能够介导La1对Caco-2细胞及黏蛋白的黏附, 通过测序和比对结果表明, 他与L. plantarum WCFS-1的EF-Tu在氨基酸序列上有83%的同源性, 并通过重组EF-Tu竞争抑制了La1对Caco-2细胞的黏附. 这些报道奠定了对黏附蛋白的研究基础, 我们在此基础上通过生物信息学软件对质谱分析结果检索过滤, 并对多种可能蛋白的表达纯化和鉴定, 是一种基于生物信息学的新的黏附蛋白筛选和鉴定方法. 一方面我们在提取的表层蛋白中找到能与黏蛋白相作用的靶蛋白IMP2, 并制备多克隆抗体; 另一方面通过用黏蛋白sepharose 4B柱获得纯化的全菌体黏附蛋白, 用IMP2多克隆抗体在Western blot中验证该全菌体黏附蛋白同前面获取的靶蛋白IMP2是一致的, 这又进一步验证了前期的结果, 使实验结果更有说服力, 对后续生物学分析和功能验证更有价值.
质谱分析技术(mass spectrometry, MS)已经成为生命科学领域广泛应用和有效的分析方式[10-11]. 他虽然具有高通量, 大规模的特点, 但数据的庞大也为他以后的分析和筛选带来了困难. 利用SEQUEST软件(Thermo Fisher Inc.)进行质谱的数据库检索分析, 得到样本中蛋白质成分, 然后利用TPP软件[8]进行结果过滤, 减小实验假阳性结果[12-13]. 通过该软件我们将原来83个蛋白缩减到了5个同源性达到90%以上的蛋白, 精确了研究对象的范围. 蛋白克隆表达过程中, 我们利用pET16b在多克隆位点添加BglⅡ和XhoⅠ酶切位点以达到各个基因在同一个载体表达, 相互作为参照, 取得很好的效果, 除LCP蛋白的表达量不高外, 其他蛋白的表达量可达500 mg/L左右, 而LCP蛋白没有高表达的原因尚需进一步探讨. 由于IMP蛋白含有1100个氨基酸, 用常规方法作表达存在一定的困难, 我们对其进行跨膜区域的分析(TMHMM软件[14-15]), 发现N端100个左右的氨基酸和C端450个氨基酸为膜位区域, 除去膜位蛋白, 其余序列我们分3段来表达, 每段200-300个左右氨基酸. 并且在克隆表达后每个蛋白增加一段17个氨基酸的HIS tag(MGHHHHHHHHHHSEFAD), 目前我们的技术尚没有能力将他去除. 在随后的Western blot结果显示IMP的第2段(IMP2, 从第455个氨基酸到755个氨基酸)表达蛋白呈强阳性, 表明该段蛋白能与肠上皮表面黏蛋白相识别并杂交, 而且由于其他几个表达蛋白均含有相同的tag而与黏蛋白没有结合, 故该段tag对黏附作用没用影响.
不同菌株的乳酸杆菌黏附蛋白在抑制病原菌的黏附方面作用是高度特异和复杂的, 他需要一步步的分类进行鉴定及反复的验证. 如何准确地找到这些黏附蛋白仍旧是一个问题. 我们的研究不仅找到了黏附蛋白IMP及初步发现该蛋白的黏附区域, 更重要的是他提供了一种基于生物信息学的新方法. 虽然目前存在着通过已知的黏附结构域来找到黏附蛋白的方法, 但是他也存在着已知结构域的数量的限制. 随着生物信息学及系统生物学的广泛应用, 我们相信对乳酸杆菌同肠上皮的黏附及保护机制将不再困难.
肠道内的乳酸杆菌作为一种益生菌, 是肠道生物屏障的重要组成部分, 他可以通过其竞争黏附作用有效抑制条件致病菌对肠上皮的定植, 进而抑制细菌移位起到了屏障功能. 近来研究较多的黏附分子包括脂磷壁酸(LTA)、细胞外多糖(EPS)、细胞表层蛋白等, 其中细胞表层蛋白起着关键性的作用. 由于表层的膜蛋白特有的亲疏水性结构所导致的生化纯化及结构功能分析的困难, 使其相比可溶性蛋白, 较少深入研究.
王健生, 教授, 西安交通大学医学院第一附属医院肿瘤外科
肠道屏障破坏引起的细菌移位是当前研究的热点, 而通过乳酸杆菌改善肠道菌群进而保护肠道屏障功能是今后研究的方向.
Granato et al将L. johnsonii NCC533(La1)用LiCl分离到一种50 kDa的表面蛋白, 能够介导La1对Caco-2细胞及黏蛋白的黏附, 通过测序和比对结果表明, 他与L. plantarum WCFS-1的EF-Tu在氨基酸序列上有83%的同源性, 并通过重组EF-Tu竞争抑制了La1对Caco-2细胞的黏附.
本研究通过使用TPP等生物信息学软件过滤可以减少分析质谱海量数据的难度, 通过对蛋白表达进而验证可以使实验更具说服力.
乳酸杆菌可通过宿主表面的蛋白、糖蛋白和糖脂, 识别其特异受体, 并通过菌体特异性配体进一步与宿主细胞相应的受体之间特异性的结合.
本研究设计合理, 结果可靠, 对于探讨细菌表层黏附蛋白及其黏附机制具有一定的科学意义.
编辑: 李军亮 电编: 何基才
| 1. | Shen TY, Qin HL, Gao ZG, Fan XB, Hang XM, Jiang YQ. Influences of enteral nutrition combined with probiotics on gut microflora and barrier function of rats with abdominal infection. World J Gastroenterol. 2006;12:4352-4358. [PubMed] |
| 2. | Horie M, Ishiyama A, Fujihira-Ueki Y, Sillanpää J, Korhonen TK, Toba T. Inhibition of the adherence of Escherichia coli strains to basement membrane by Lactobacillus crispatus expressing an S-layer. J Appl Microbiol. 2002;92:396-403. [PubMed] [DOI] |
| 3. | Vélez MP, De Keersmaecker SC, Vanderleyden J. Adherence factors of Lactobacillus in the human gastrointestinal tract. FEMS Microbiol Lett. 2007;276:140-148. [PubMed] [DOI] |
| 4. | Wells JM, Mercenier A. Mucosal delivery of therapeutic and prophylactic molecules using lactic acid bacteria. Nat Rev Microbiol. 2008;6:349-362. [PubMed] [DOI] |
| 5. | Rojas M, Ascencio F, Conway PL. Purification and characterization of a surface protein from Lactobacillus fermentum 104R that binds to porcine small intestinal mucus and gastric mucin. Appl Environ Microbiol. 2002;68:2330-2336. [PubMed] [DOI] |
| 6. | Wang B, Wei H, Yuan J, Li Q, Li Y, Li N, Li J. Identification of a surface protein from Lactobacillus reuteri JCM1081 that adheres to porcine gastric mucin and human enterocyte-like HT-29 cells. Curr Microbiol. 2008;57:33-38. [PubMed] [DOI] |
| 7. | Moncada D, Keller K, Chadee K. Entamoeba histolytica cysteine proteinases disrupt the polymeric structure of colonic mucin and alter its protective function. Infect Immun. 2003;71:838-844. [PubMed] [DOI] |
| 8. | Nesvizhskii AI, Keller A, Kolker E, Aebersold R. A statistical model for identifying proteins by tandem mass spectrometry. Anal Chem. 2003;75:4646-4658. [PubMed] [DOI] |
| 9. | Granato D, Bergonzelli GE, Pridmore RD, Marvin L, Rouvet M, Corthésy-Theulaz IE. Cell surface-associated elongation factor Tu mediates the attachment of Lactobacillus johnsonii NCC533 (La1) to human intestinal cells and mucins. Infect Immun. 2004;72:2160-2169. [PubMed] [DOI] |
| 10. | Principe S, Maras B, Schininà ME, Pocchiari M, Cardone F. Unraveling the details of prion (con)formation(s): recent advances by mass spectrometry. Curr Opin Drug Discov Devel. 2008;11:697-707. [PubMed] |
| 11. | Chen CH. Review of a current role of mass spectrometry for proteome research. Anal Chim Acta. 2008;624:16-36. [PubMed] [DOI] |
| 12. | Palmblad M, Bindschedler LV, Cramer R. Quantitative proteomics using uniform (15)N-labeling, MASCOT, and the trans-proteomic pipeline. Proteomics. 2007;7:3462-3469. [PubMed] [DOI] |
| 13. | Keller A, Eng J, Zhang N, Li XJ, Aebersold R. A uniform proteomics MS/MS analysis platform utilizing open XML file formats. Mol Syst Biol. 2005;1:2005.0017. [PubMed] |
| 14. | Schneider G, Fechner U. Advances in the prediction of protein targeting signals. Proteomics. 2004;4:1571-1580. [PubMed] [DOI] |
| 15. | Do JH, Choi DK. Computational approaches to gene prediction. J Microbiol. 2006;44:137-144. [PubMed] |