修回日期: 2007-12-23
接受日期: 2008-01-15
在线出版日期: 2008-01-28
目的: 探讨蛋白指纹图谱技术筛选原发性胆汁性肝硬化(PBC)患者血清中可用于诊断的特异性标志物.
方法: 采用弱阳离子纳米磁性微球捕获血清中的蛋白, ProteinChip PBSII-C型蛋白质芯片阅读仪检测绘制成蛋白指纹图谱. 所有蛋白指纹图谱采用Biomarker Wizard 3.1分析之后用Biomarker Patterns Software 5.0识别最终可能用于PBC诊断的蛋白标志物并优化组合建立诊断模型.
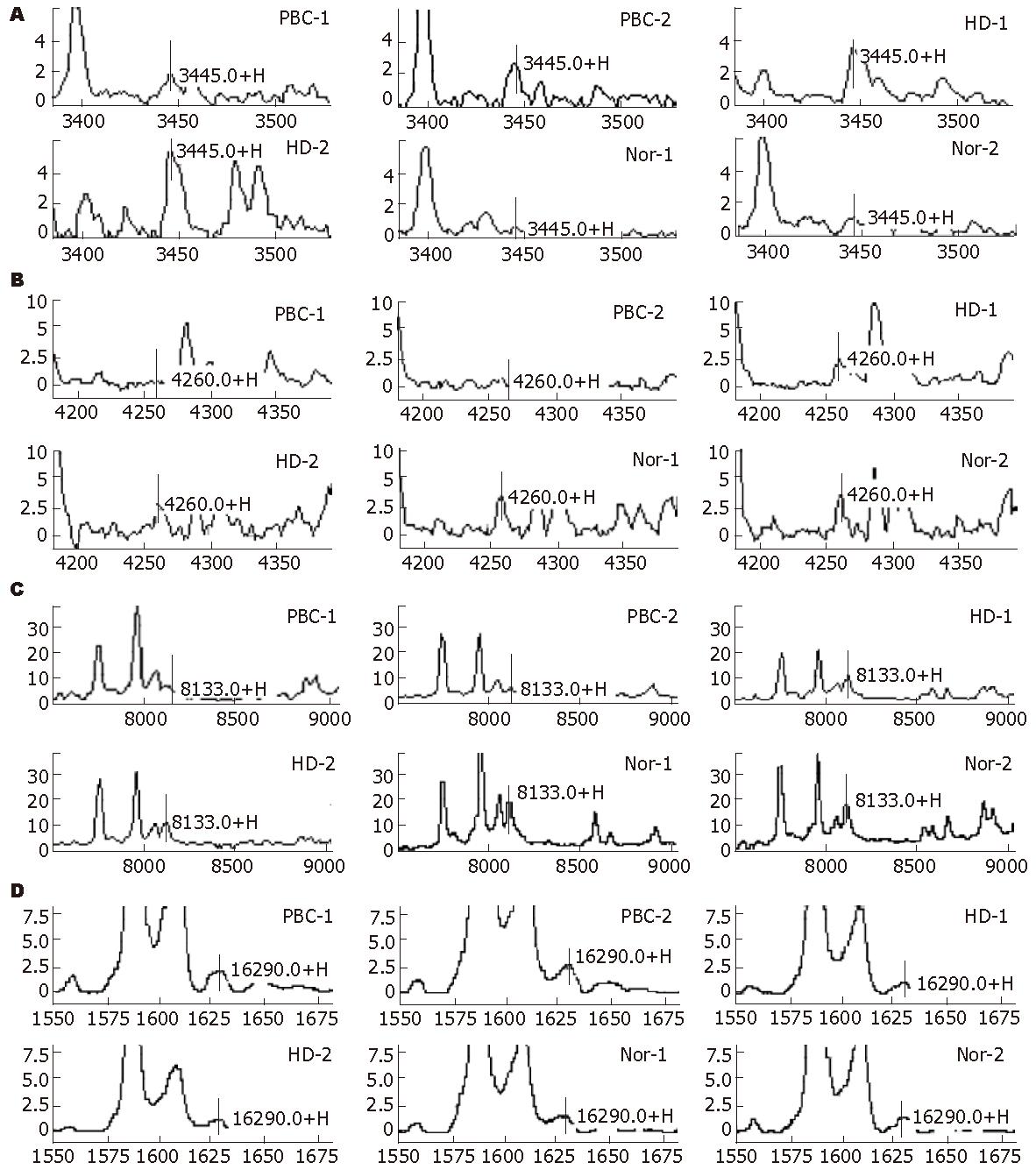
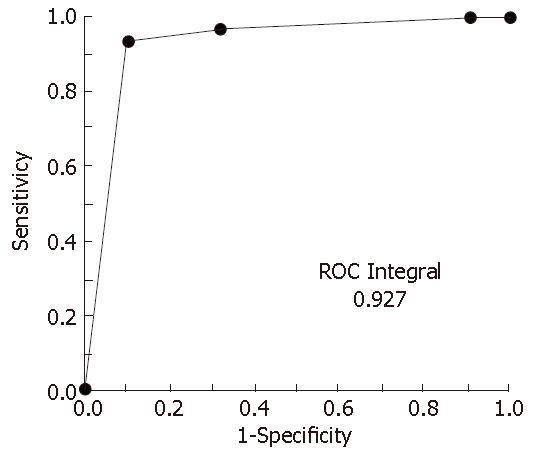
结果: 在PBC患者和对照组之间找到69个差异蛋白峰(P<0.05). 其中质荷比(m/z)为3445, 4260, 8133和16290的蛋白峰建立PBC诊断模型. 该诊断模型能很好地把PBC患者从其他肝脏疾病患者和正常人群中区分出来, 其敏感性为93.3%, 特异性为95.1%. 经双盲实验验证, 该模型对PBC诊断的敏感性为92.9%, 特异性为82.4%.
结论: 采用纳米磁性微球与蛋白质芯片阅读仪联用的蛋白指纹图谱技术可以检测PBC患者血清中的特异性蛋白标志物, 并建立敏感性和特异性均较高的PBC诊断模型.
引文著录: 胡朝军, 李永哲, 赵冠飞, 李宁, 许洋, 佟大伟, 张蜀澜. 应用蛋白指纹图谱技术筛选原发性胆汁性肝硬化患者血清特异性标志物. 世界华人消化杂志 2008; 16(3): 277-283
Revised: December 23, 2007
Accepted: January 15, 2008
Published online: January 28, 2008
AIM: To screen for the potential protein biomarkers in serum for the diagnosis of primary biliary cirrhosis (PBC) using proteomic fingerprint technology.
METHODS: Proteomic fingerprint technology combining magnetic beads with MALDI-TOF-MS was used to profile and compare the serum proteins from 44 patients with PBC, 32 patients with other hepatic diseases and 43 healthy blood donors. Proteomic patterns associated with PBC were identified by Biomarker Patterns Software. Model of biomarkers was constructed and evaluated using the Biomarker Patterns Software.
RESULTS: A total of 69 discriminating m/z peaks were identified that were related to PBC (P < 0.05). The model of biomarkers constructed by the Biomarker Patterns Software based on the four biomarkers (3445, 4260, 8133 and 16290) generated excellent separation between the PBC and control groups. The sensitivity was 93.3% and the specificity was 95.1%. Blind test data indicated a sensitivity of 92.9% and a specificity of 82.4%.
CONCLUSION: Biomarkers for PBC can be discovered in serum by MALDI-TOF-MS combining the use of magnetic beads. The pattern of combined markers provides a powerful and reliable diagnostic method for PBC with a high sensitivity and specificity.
- Citation: Hu CJ, Li YZ, Zhao GF, Li N, Xu Y, Tong DW, Zhang SL. Screening for specific biomarkers in serum for diagnosis of primary biliary cirrhosis using proteomic fingerprint technology. Shijie Huaren Xiaohua Zazhi 2008; 16(3): 277-283
- URL: https://www.wjgnet.com/1009-3079/full/v16/i3/277.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i3.277
原发性胆汁性肝硬化(PBC)是一种慢性自身免疫性肝脏疾病, 该病以肝内细小胆管非化脓性进行性破坏并伴门脉炎症性改变, 长期持续性肝内胆汁淤积、最终导致肝纤维化和肝硬化的慢性进展性自身免疫性疾病为主要特征[1-2]. 近年的调查研究表明, PBC在世界范围内已经不再是罕见疾病[3], 特别是在东方人群中, 无症状的PBC患者可能远远超过我们现在估计的数量, 而且PBC在中国人群中的发病率和死亡率可能都高于其他地方[4]. 目前, 对于PBC诊断最可靠的方法仍然是肝脏组织病理活检[5]. 然而, 很多PBC患者不愿意接受这种具有创伤性的侵入检查[6], 很多接受检查的PBC患者肝脏组织病理活检并不能找到特征性的病理改变. 除此之外, 血清中高滴度的抗线粒体抗体(AMA)目前也作为诊断PBC的一个重要指标[7-8], 但是该抗体对于PBC的诊断并不特异, 在其他疾病状态, 如感染性肝病等也经常出现[9-10], 这对于像我们这样的肝炎大国来说显得非常不利. 还有研究发现, 10%左右的PBC患者血清中检测不到AMA[11-12]. 由于现有的检测技术限制导致很多PBC患者到了疾病终末期才被诊断, 这一现状已经不能满足临床对于疾病早期诊断、早期治疗的要求, 在临床医疗中仍然迫切需要诊断PBC的新方法. 现有的研究表明蛋白指纹图谱技术可用于很多疾病的早期诊断, 如肿瘤、神经性疾病、结核艾滋病毒等感染性疾病和自身免疫性疾病[13-19]. 因此, 我们采用纳米磁性微球和蛋白芯片阅读仪联用的蛋白指纹图谱技术筛选PBC患者血清中的特异性标志物.
2006-06/2007-02北京协和医院体检、门诊和住院患者119例血清样品, 其中包括PBC患者44例(所有病例符合PBC的诊断标准)[5], 其他肝脏疾病患者32例(包括: 自身免疫性肝炎10例、乙型肝炎10例、肝硬化9例、酒精性肝炎1例、药物性肝炎1例和原发性硬化性胆管炎1例), 健康体检者43例, 详细临床资料及分组信息见表1. 所有标本用4 mL无抗凝剂的BD真空采血管空腹采血后于4 ℃静置1h, 4000 g离心5 min. 血清分装5管, 于-80 ℃冻存备用. 弱阳离子(WCX)纳米磁性微球, 购自北京赛尔迪生物技术有限公司. 乙腈、三氟乙酸、尿素、Hepes、水(HPLC级)、Tris碱、CHAPS、DTT、SPA(Sinapinic acid)购自Sigma公司. PBSII-C型蛋白质芯片阅读仪为美国赛弗吉公司产品.
| 分组 | n | 男/女 | 年龄范围 | 平均年龄 | 建模组 | 盲法验证组 |
| 健康对照组 | 43 | 27/16 | 25-78 | 38.6±10.9 | 23 | 20 |
| PBC组 | 44 | 3/41 | 16-74 | 54.1±11.3 | 30 | 14 |
| 其他肝病组 | 32 | 17/15 | 18-75 | 50.6±13.1 | 18 | 14 |
1.2.1 标本准备: 血清从-80 ℃冰箱内取出, 冰浴融化后4 ℃ 20000 g离心10 min. 取10 μL血清, 加20 μL U9缓冲液(9 mol/L尿素, 20 g/L CHAPS, 10 g/L DTT, 50 mmol/L Tris-HCl, pH9.0), 充分混匀, 4 ℃振摇30 min后加入100 mmol/L NaAc 370 μL(pH4.0), 立即混匀.
1.2.2 弱阳离子纳米磁性微球的活化: 每个PCR管中加入50 g/L WCX磁性微球50 μL, 置于磁性分离板上分离2 min, 弃上清液, 加入100 mmol/L NaAc 100 μL活化2次, 5 min/次.
1.2.3 活化的纳米磁性微球捕获血清蛋白: 每份弃上清液活化的磁性微球中加入100 μL处理好的血清样品, 振荡孵育1 h. 置于磁性分离板上分离2 min, 弃上清液, 加入100 mmol/L NaAc 100 μL洗涤2次, 5 min/次. 用5 mL/L的TFA 10 µL将结合在磁性微球上的蛋白洗脱5 min, 取5 μL蛋白洗脱液加入5 μL饱和SPA(50% CAN+5 mL/L TFA), 充分混匀后吸取2 μL蛋白结晶混合物点样于金芯片(此芯片仅起载体作用, Au-chip, Ciphergen), 自然晾干, 采用PBSII-C型蛋白质芯片阅读仪检测蛋白指纹图谱.
1.2.4 芯片检测: 芯片阅读仪参数设定为最高检范围为50000, 优化范围2000-10000, 激光强度205, 检测灵敏度8. 检测前用All-in-one多肽标准芯片校正仪器, 系统质量偏差≤0.1%. 原始数据先以Proteinchip 3.1软件标化处理, 并采用m/z为4091的蛋白峰(每个人都具有的血清蛋白峰)作为内标校正.
统计学处理 采用Ciphergen ProteinChip软件和BioMarker Wizard软件对芯片检测得到的蛋白指纹图谱进行统计学处理. PBC组与对照组(由其他肝病组与正常对照组组成)之间蛋白质峰的比较采用t检验, P<0.05具有统计学意义. 在此基础上采用Biomarker Patterns Software Version 5.0 (BPS)识别诊断PBC的最佳标志物, 并建立PBC诊断模型.
对建模组的71例样本检测得到的蛋白指纹图谱进行分析发现69个蛋白质峰存在显著性差异(P<0.05, 表2).
| 质荷比m/z | P值 | 质荷比m/z | P值 | 质荷比m/z | P值 |
| 3088 | 2.70E-09 | 16489 | 0.000942 | 3681 | 0.012169 |
| 2543 | 1.31E-08 | 3375 | 0.001028 | 28790 | 0.012373 |
| 81331 | 2.20E-07 | 9280 | 0.001045 | 7761 | 0.012457 |
| 7628 | 2.71E-07 | 2869 | 0.001280 | 34451 | 0.013500 |
| 8600 | 2.95E-07 | 16077 | 0.001831 | 25099 | 0.015117 |
| 5477 | 1.55E-05 | 5855 | 0.002259 | 4528 | 0.018915 |
| 31842 | 2.06E-05 | 3774 | 0.002416 | 43219 | 0.019396 |
| 3935 | 4.21E-05 | 8290 | 0.002421 | 24015 | 0.023501 |
| 2759 | 4.67E-05 | 4382 | 0.002668 | 4676 | 0.023565 |
| 4281 | 7.05E-05 | 28026 | 0.002679 | 7049 | 0.027295 |
| 4300 | 7.93E-05 | 4645 | 0.002708 | 3397 | 0.029999 |
| 7967 | 0.000137 | 9487 | 0.002722 | 3887 | 0.030014 |
| 3955 | 0.000150 | 23386 | 0.003273 | 9606 | 0.033309 |
| 22798 | 0.000151 | 5246 | 0.003700 | 32550 | 0.035143 |
| 15874 | 0.000196 | 14016 | 0.004198 | 5214 | 0.038265 |
| 11485 | 0.000212 | 5634 | 0.005086 | 5384 | 0.041371 |
| 4068 | 0.000224 | 9794 | 0.005553 | 7554 | 0.041867 |
| 8071 | 0.000280 | 14103 | 0.005622 | 42601 | 0.043380 |
| 15608 | 0.000325 | 7405 | 0.006566 | 47633 | 0.044072 |
| 5803 | 0.000379 | 5315 | 0.007519 | 1498 | 0.045245 |
| 11697 | 0.000571 | 43917 | 0.010108 | 9399 | 0.046333 |
| 6846 | 0.000664 | 46614 | 0.011099 | 9116 | 0.047415 |
| 162901 | 0.000780 | 8554 | 0.011442 | 5525 | 0.048693 |
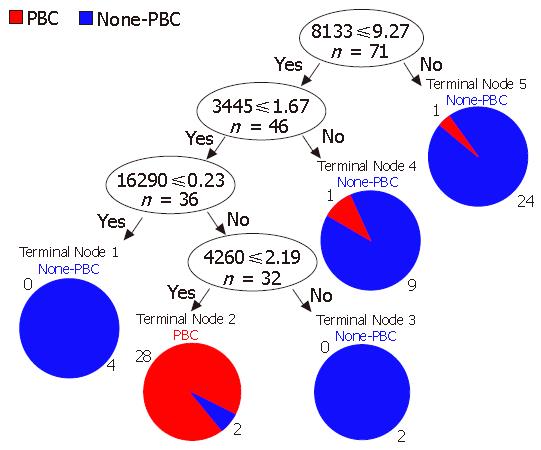
将BioMarker Wizard软件统计分析得到的69个差异蛋白峰导入Biomarker Patterns Software分析软件筛选诊断PBC的最佳标志物. 结果显示m/z为3445、4260、8133和16290的4个蛋白峰为诊断PBC的最佳标志物(图1). 这4个蛋白标志物组成的诊断模型能很好的将PBC患者区分出来, 其诊断模型如图2. 该诊断模型的敏感性为93.3%, 特异性为95.1%(表3), 其诊断ROC曲线如图3.
| 实验分组 | 临床分组 | n | 诊断正确例数 | 诊断正确率 |
| 建模组 | PBC | 30 | 28 | 93.33% |
| 对照 | 41 | 39 | 95.12% | |
| 盲法验证组 | PBC | 14 | 13 | 92.86% |
| 对照 | 34 | 28 | 82.35% |
采用48例样本检测得到的蛋白指纹图谱对已建立的PBC诊断模型进行盲法验证. 验证结果表明该模型对PBC的诊断敏感性为92.9%, 特异性为82.4%(表3).
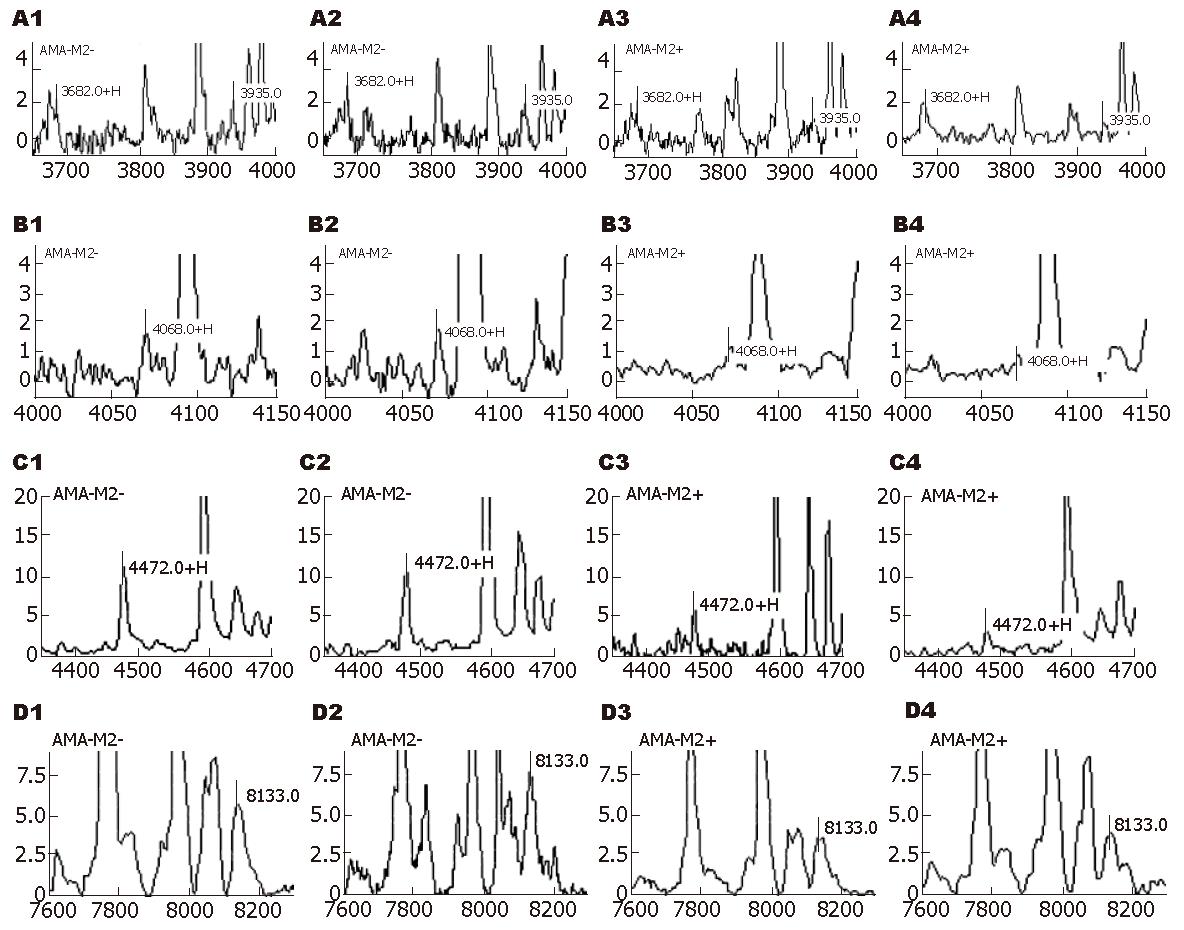
采用Biomarker Patterns Software软件对AMA-M2阳性与AMA-M2阴性PBC患者蛋白指纹图谱进行比较, 发现m/z为3682、3935、4068、4472和8133的5个蛋白质峰在两组患者之间存在显著性差异(P<0.05, 图4).
胆汁淤积性疾病在人群中的发生率非常高, 因此把PBC患者从这类人群中区分出来在临床诊疗中显得非常重要[20]. 目前依靠组织病理活检、生化检查以及血清自身抗体检查仅能发现并诊断50%-60%的PBC患者, 这一现状无法很好的满足临床诊疗需求, 因此, PBC的诊断仍旧显得并不十分完善[21].
近年来, 蛋白质组学的研究得到了空前的发展, 出现了大量的蛋白质组学研究方法并应用于临床研究[22-23]. 蛋白质组学研究领域出现的这些高通量方法使得寻找复杂疾病的生物标志物越来越成功[24-25]. 疾病的生物标志物或者多标志物联合诊断模式在疾病的诊断、鉴别诊断以及治疗方案的制定方面逐渐显示出强大的优越性[26-27]. 通过基质辅助激光解吸电离飞行时间质谱(MALDI-TOF-MS)技术分析血清中的蛋白指纹已经筛选出很多用于疾病诊断的标志物. 与传统蛋白芯片相比, 由于纳米磁性微球具有强大的表面积, 因此他能更好的捕获血清中的小分子多肽或蛋白[28-29]. 结合纳米磁性微球和MALDI-TOF-MS的优势, 采用这两种技术连用的方法能检测到血清中更多的低丰度蛋白. 我们采用WCX纳米磁性微球和MALDI-TOF-MS检测44例PBC患者、32例其他肝病患者和43例健康体检者血清中的蛋白指纹图谱, 研究结果显示m/z为3445、4260、8133 和16290的蛋白峰是诊断PBC的最佳标志物, 4个蛋白组合建立的诊断模型能很好的区分PBC患者, 该诊断模型对PBC的诊断敏感性为93.3%, 特异性为95.1%. 48例标本盲法验证结果显示其敏感性为92.9%, 特异性为82.4%. 对AMA-M2阳性和AMA-M2阴性PBC患者血清蛋白指纹图谱分析比较, 仅发现m/z为3682、3935、4068、4472和8133的5个蛋白质峰在两组患者之间存在显著性差异(P<0.05), 究其原因可能是AMA-M2阳性和AMA-M2阴性PBC患者主要的差异蛋白是大分子蛋白(因AMA为抗体, 分子量较大), 而蛋白指纹图谱技术主要检测的是小分子蛋白, 因此在这两者之间发现的差异蛋白并不多.
由于PBC是一个多因素的复杂疾病, 发现和鉴定PBC相关的标志物不仅可以促进我们在分子层面上对PBC发病机制、疾病过程的理解, 而且多标志物联合诊断模式的建立对于诊断PBC这种复杂疾病也可能更可靠. 目前这些多标志物联合诊断模式的建立都是基于一些神经智能网络软件实现[30], 因此, 应用于临床之前还需要大量的临床验证来证实其可靠性. 由于PBC患者的标本收集较为困难, 本研究中我们建立PBC诊断模型时包括了30例PBC患者, 在48例盲法验证标本中只有14例PBC患者标本. 因此, 本研究中发现的4个PBC诊断标志物更需要扩大样本量来做进一步验证, 以确定其临床实用价值.
总之, 虽然本次研究只包括44例PBC患者, 但是本研究结果的确为采用纳米磁性微球和蛋白芯片阅读仪联用的蛋白指纹图谱技术诊断PBC患者提供了实验依据, 特别是发现联合4个蛋白峰建立的诊断模型对PBC的诊断具有较高的敏感性和特异性, 且在盲法验证结果中也得到了体现.
近年的调查研究表明原发性胆汁性肝硬化(PBC)在世界范围内已经不再是罕见疾病, 但发病机制不清楚且预后差. 目前采用的肝脏组织病理活检和血清抗线粒体抗体检测并不能完全满足临床诊治的要求. 蛋白指纹图谱技术是近年来的热点技术, 已经在临床疾病的研究中得到广泛的应用并展示出较好的应用前景.
张吉翔, 教授, 南昌大学第二附属医院.
蛋白指纹图谱技术出现的同时也提出一种全新的多标志物谱或个性化标志物的诊断模式. 采用该技术筛选PBC患者血清中的特异性标志物, 可以发现一些传统方法不能发现的小分子物质, 对这些小分子物质的鉴定有助于PBC的诊断和对发病机制的探讨. 但目前鉴定采用蛋白指纹图谱技术筛选低丰度小分子难度较大.
血清蛋白指纹的检测一般采用SELDI技术, 本文采用纳米磁性微球技术捕获血清蛋白, 再采用基质辅助激光解析电离飞行时间质谱仪进行蛋白指纹检测.
本研究采用纳米磁性微球和蛋白芯片阅读仪联用的蛋白指纹图谱技术诊断PBC患者, 为读者提供了实验依据.
基质辅助激光解吸电离飞行时间质谱(MALDI-TOF-MS): 是利用激光脉冲辐射使芯池中的分析物解析形成荷电离子, 根据不同质荷比, 这些离子在仪器场中飞行的时间长短不一, 由此绘制出一张质谱图案.
本文研究方法先进, 结果与统计分析可信, 有所创新, 研究结论具有潜在应用价值.
编辑: 程剑侠 电编: 郭海丽
| 1. | Locke GR, Therneau TM, Ludwig J, Dickson ER, Lindor KD. Time course of histological progression in primary biliary cirrhosis. Hepatology. 1996;23:52-56. [PubMed] [DOI] |
| 2. | Kaplan MM, Gershwin ME. Primary biliary cirrhosis. N Engl J Med. 2005;353:1261-1273. [PubMed] [DOI] |
| 3. | Metcalf J, James O. The geoepidemiology of primary biliary cirrhosis. Semin Liver Dis. 1997;17:13-22. [PubMed] [DOI] |
| 4. | Wong GL, Hui AY, Wong VW, Chan FK, Sung JJ, Chan HL. A retrospective study on clinical features and prognostic factors of biopsy-proven primary biliary cirrhosis in Chinese patients. Am J Gastroenterol. 2005;100:2205-2211. [PubMed] [DOI] |
| 5. | Heathcote EJ. Management of primary biliary cirrhosis. The American Association for the Study of Liver Diseases practice guidelines. Hepatology. 2000;31:1005-1013. [PubMed] [DOI] |
| 6. | Zeniya M, Watanabe F, Morizane T, Shibata M, Maeyama S, Kage M, Nakanuma Y, Toda G. Diagnosing clinical subsets of autoimmune liver diseases based on a multivariable model. J Gastroenterol. 2005;40:1148-1154. [PubMed] [DOI] |
| 7. | Leung PS, Coppel RL, Ansari A, Munoz S, Gershwin ME. Antimitochondrial antibodies in primary biliary cirrhosis. Semin Liver Dis. 1997;17:61-69. [PubMed] [DOI] |
| 9. | Toda G, Zeniya M, Watanabe F, Imawari M, Kiyosawa K, Nishioka M, Tsuji T, Omata M. Present status of autoimmune hepatitis in Japan--correlating the characteristics with international criteria in an area with a high rate of HCV infection. Japanese National Study Group of Autoimmune Hepatitis. J Hepatol. 1997;26:1207-1212. [PubMed] [DOI] |
| 10. | Miyakawa H, Tanaka A, Selmi C, Hosoya N, Mataki N, Kikuchi K, Kato T, Arai J, Goto T, Gershwin ME. Serum reactivity against bacterial pyruvate dehydrogenase: increasing the specificity of anti-mitochondrial antibodies for the diagnosis of primary biliary cirrhosis. Clin Dev Immunol. 2006;13:289-294. [PubMed] [DOI] |
| 11. | Kaplan MM. Primary biliary cirrhosis. N Engl J Med. 1996;335:1570-1580. [PubMed] [DOI] |
| 12. | Sakauchi F, Mori M, Zeniya M, Toda G. Antimitochondrial antibody negative primary biliary cirrhosis in Japan: utilization of clinical data when patients applied to receive public financial aid. J Epidemiol. 2006;16:30-34. [PubMed] [DOI] |
| 13. | Dotzlaw H, Schulz M, Eggert M, Neeck G. A pattern of protein expression in peripheral blood mononuclear cells distinguishes rheumatoid arthritis patients from healthy individuals. Biochim Biophys Acta. 2004;1696:121-129. [PubMed] [DOI] |
| 14. | Mei J, Kolbin D, Kao HT, Porton B. Protein expression profiling of postmortem brain in schizophrenia. Schizophr Res. 2006;84:204-213. [PubMed] [DOI] |
| 15. | Shiwa M, Nishimura Y, Wakatabe R, Fukawa A, Arikuni H, Ota H, Kato Y, Yamori T. Rapid discovery and identification of a tissue-specific tumor biomarker from 39 human cancer cell lines using the SELDI ProteinChip platform. Biochem Biophys Res Commun. 2003;309:18-25. [PubMed] [DOI] |
| 16. | Agranoff D, Fernandez-Reyes D, Papadopoulos MC, Rojas SA, Herbster M, Loosemore A, Tarelli E, Sheldon J, Schwenk A, Pollok R. Identification of diagnostic markers for tuberculosis by proteomic fingerprinting of serum. Lancet. 2006;368:1012-1021. [PubMed] [DOI] |
| 17. | Kang X, Xu Y, Wu X, Liang Y, Wang C, Guo J, Wang Y, Chen M, Wu D, Wang Y. Proteomic fingerprints for potential application to early diagnosis of severe acute respiratory syndrome. Clin Chem. 2005;51:56-64. [PubMed] [DOI] |
| 18. | Sun B, Rempel HC, Pulliam L. Loss of macrophage-secreted lysozyme in HIV-1-associated dementia detected by SELDI-TOF mass spectrometry. AIDS. 2004;18:1009-1012. [PubMed] [DOI] |
| 19. | de Seny D, Fillet M, Meuwis MA, Geurts P, Lutteri L, Ribbens C, Bours V, Wehenkel L, Piette J, Malaise M. Discovery of new rheumatoid arthritis biomarkers using the surface-enhanced laser desorption/ionization time-of-flight mass spectrometry ProteinChip approach. Arthritis Rheum. 2005;52:3801-3812. [PubMed] [DOI] |
| 20. | Kim WR, Ludwig J, Lindor KD. Variant forms of cholestatic diseases involving small bile ducts in adults. Am J Gastroenterol. 2000;95:1130-1138. [PubMed] [DOI] |
| 21. | He XS, Ansari AA, Ridgway WM, Coppel RL, Gershwin ME. New insights to the immunopathology and autoimmune responses in primary biliary cirrhosis. Cell Immunol. 2006;239:1-13. [PubMed] [DOI] |
| 22. | Baumeister W. From proteomic inventory to architecture. FEBS Lett. 2005;579:933-937. [PubMed] [DOI] |
| 23. | Chung CH, Levy S, Chaurand P, Carbone DP. Genomics and proteomics: emerging technologies in clinical cancer research. Crit Rev Oncol Hematol. 2007;61:1-25. [PubMed] [DOI] |
| 24. | Choi J, Malakowsky CA, Talent JM, Conrad CC, Gracy RW. Identification of oxidized plasma proteins in Alzheimer's disease. Biochem Biophys Res Commun. 2002;293:1566-1570. [PubMed] [DOI] |
| 25. | Hayman MW, Przyborski SA. Proteomic identification of biomarkers expressed by human pluripotent stem cells. Biochem Biophys Res Commun. 2004;316:918-923. [PubMed] [DOI] |
| 26. | Poon TC, Yip TT, Chan AT, Yip C, Yip V, Mok TS, Lee CC, Leung TW, Ho SK, Johnson PJ. Comprehensive proteomic profiling identifies serum proteomic signatures for detection of hepatocellular carcinoma and its subtypes. Clin Chem. 2003;49:752-760. [PubMed] [DOI] |
| 27. | Hellström M, Lexander H, Franzén B, Egevad L. Proteomics in prostate cancer research. Anal Quant Cytol Histol. 2007;29:32-40. [PubMed] |
| 28. | Villanueva J, Philip J, Entenberg D, Chaparro CA, Tanwar MK, Holland EC, Tempst P. Serum peptide profiling by magnetic particle-assisted, automated sample processing and MALDI-TOF mass spectrometry. Anal Chem. 2004;76:1560-1570. [PubMed] [DOI] |
| 29. | Whiteaker JR, Zhao L, Zhang HY, Feng LC, Piening BD, Anderson L, Paulovich AG. Antibody-based enrichment of peptides on magnetic beads for mass-spectrometry-based quantification of serum biomarkers. Anal Biochem. 2007;362:44-54. [PubMed] [DOI] |
| 30. | Wiesner A. Detection of tumor markers with ProteinChip technology. Curr Pharm Biotechnol. 2004;5:45-67. [PubMed] [DOI] |