修回日期: 2008-04-08
接受日期: 2008-05-16
在线出版日期: 2008-06-08
目的: 研究幽门螺杆菌(H. pylori)分离株的多重耐药机制.
方法: 用氯霉素对临床分离株H. pylori和标准株H. pylori进行体外诱导, 琼脂稀释法测定诱导后菌株对甲硝唑、四环素、琥乙红霉素、环丙沙星和青霉素G的耐药性, 从而筛选出多重耐药株; 实时定量PCR检测多重耐药株和敏感株中编码主动外排泵外膜蛋白的结构基因hefA的mRNA水平. 通过构建hefA基因敲除株, 测定敲除前后菌株对10种抗生素的敏感性. PCR扩增20株H. pylori临床分离株主动外排泵结构基因hefA和hefC.
结果: 经氯霉素诱导后, 筛选出的耐药菌株均表现出相似的多重耐药性, 6株多重耐药株hefA基因mRNA水平显著高于敏感株(5.8466±2.9370 vs 2.6356±1.7245, P = 0.033); 成功构建了hefA基因敲除株(△HpLZ1026), △HpLZ1026对4种抗生素的敏感性明显增加; 所有20株临床分离株中均检测出hefA和hefC基因, 未发现hefABC基因缺失株.
结论: 主动外排系统hefA基因高表达导致体外人工诱导H. pylori多重耐药的产生; hefABC基因在H. pylori中普遍存在, 且在H. pylori多药耐药机制中起重要作用.
引文著录: 刘志强, 郑鹏远. 主动外排泵外膜蛋白编码基因hefA在幽门螺杆菌多重耐药中的重要作用. 世界华人消化杂志 2008; 16(16): 1751-1756
Revised: April 8, 2008
Accepted: May 16, 2008
Published online: June 8, 2008
AIM: To determine whether efflux systems contribute to multidrug resistance(MDR) in Helicobacter pylori (H. pylori).
METHODS: A chloramphenicol-induced MDR model ex vivo was developed in six susceptible H. pylori strains (5 isolates and H. pylori NCTC11637). The minimal inhibitory concentrations (MICs) of erythromycin, metronidazole, penicillin G, tetracycline and ciprofloxacin were determined using agar dilution tests and MDR strains were selected accordingly. The mRNA expression level of hefA was estimated using fluorescence real-time quantitative PCR. A H. pylori LZ1026 knockout mutant (△HpLZ1026) for (putative) efflux protein was constructed through insertion of the Kanamycin resistance cassette from pEGFP-N2 into hefA, and its susceptibility profiles to 10 antibiotics were estimated. Expressions of hefA and hefC genes were detected using PCR in 20 clinically isolated H. pylori strains.
RESULTS: There was similar multidrug-resistance in chloramphenicol-induced screened multidrug-resistant (MDR) strains. Expression level of hefA gene in the 6 MDR strains was significantly higher compared with drug-sensitive strains (5.8466 ± 2.9370 vs 2.6356 ± 1.7245, P = 0.033). △HpLZ1026 was constructed successfully and its sensitivity to four of ten antibiotics was significantly increased. In all the 20 isolated strains, hefA and hefC genes were detected while strains of hefABC gene depletion were not found.
CONCLUSION: Efflux pump gene hefA plays an important role in multidrug resistance of H. pylori in vivo. The hefABC gene exists universally in H. pylori, which plays an essential role in mechanism underlying multidrug resistance.
- Citation: Liu ZQ, Zheng PY. Important role of efflux pump gene hefA in multidrug resistance of Helicobacter pylori. Shijie Huaren Xiaohua Zazhi 2008; 16(16): 1751-1756
- URL: https://www.wjgnet.com/1009-3079/full/v16/i16/1751.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i16.1751
世界50%以上人口存在幽门螺杆菌(Helicobacter pylori, H. pylori)感染[1-4]. 已经证实H. pylori感染是慢性活动性胃炎、消化性溃疡、胃黏膜相关淋巴组织淋巴瘤的主要病因, 与胃癌的发生亦有密切关系[5-6]. 随着抗生素在根除H. pylori治疗中的广泛应用, H. pylori耐药的发生率不断上升, 而且, 已经出现同时对甲硝唑、克拉霉素、阿莫西林耐药的多重耐药株[7]. 研究表明, 能将抗生素主动排出体外的外排泵在细菌的多重耐药机制中起重要作用[8-11]. 与细菌多重抗生素耐药性有关的主动外排泵系统主要归于5个家族/类[12-13], 其中, 耐受-生节-分裂家族(resistance-nodulation-division[RND]family), 在革兰氏阴性菌多重耐药中起重要作用, 能够排出底物相似性很小底物, 包括多类抗菌药物, 也包括用于消毒灭菌的抑菌剂、去污剂、表面活性剂、染料及有机溶剂等[8,10]. 杨海燕 et al[14]研究发现acrAB-RND泵的过度表达与志贺菌的多重耐药产生密切相关. Bina et al[15]鉴定了H. pylori中存在hefABC, hefDEF, hefGHI 3种RND类主动外排系统, 通过基因敲除技术分别使3种外排泵功能失活后, 用纸片扩散法对敲除前后菌株进行药敏试验, 结果显示RND类外排泵在H. pylori产生多重耐药中的作用仍未明了; 而van Amsterdam et al[16]通过敲除tolC同源[17]的外膜通道蛋白编码基因, 显示外排泵在H. pylori对甲硝唑耐药机制中起重要作用, 然而外排泵的编码基因在H. pylori多重耐药株中的表达情况未精确定量. 由于亚洲H. pylori菌株在生物学性状与欧美地区H. pylori菌株存在诸多不同[18], 本文以中国H. pylori菌株为研究对象, 研究主动外排系统hefABC在H. pylori临床分离株中的诱导表达情况, 采用更加精确的琼脂二倍稀释法测定hefA基因敲除株H. pylori对多种抗生素的耐药性, 为克服H. pylori的多重耐药产生提供理论基础. 结果报告如下.
用于体外诱导耐药试验的H. pylori和H. pylori LZ1026株随机选取于消化性溃疡或慢性活动性胃炎患者胃黏膜活检标本(郑州大学第二附属医院消化内科胃镜室, 2006-03/2007-07), 采用含70 mL/L羊血的布氏培养基, 37℃微需氧(850 mL/L N2、100 mL/L CO2、50 mL/L O2)、相对湿度>98%的条件下培养3 d, 菌株经生化反应(尿素酶、触酶、氧化酶)及HE染色、形态学鉴定, 获取临床分离株; H. pylori NCTC11637、质粒pBluescriptⅡSK(-)和pEGFP-N2由河南分子医学重点实验室提供. 阿莫西林、头孢他定、萘啶酸、庆大霉素、多黏霉素B、克拉霉素、氟哌酸、四环素、氯霉素、环丙沙星、甲硝唑、头孢噻肟、琥乙红霉素和青霉素G均购自中国药品生物制品检验所; DNA提取试剂盒、总RNA抽提试剂盒和cDNA第一链合成试剂盒购自北京塞百盛生物公司; pMD19-T vector购自TaKaRa公司; RealMasterMix(SYBR Green)试剂盒均购自宝赛公司; 荧光定量仪: Gene Amp 5700 Sequence Detector定量PCR扩增仪及配套分析软件Gene Amp 5700 SDS(美国Perkin-Elmer公司). PCR引物设计, 参照Bina et al[15]在GenBank上公布的H. pylori hefABC基因序列, 进行同源性序列分析, 在其保守区段内设计, 参照文献[19], 内参选用gyrB基因(DNA旋转酶B亚单位基因), 由上海生物工程技术服务公司合成.
1.2.1 多重耐药H. pylori的体外诱导和筛选: (1)对临床分离敏感菌株进行单菌落传代培养, 测定诱导剂氯霉素对受试菌的最低抑菌浓度(minimal inhibitory concentration, MIC); (2)在含1/2×MIC诱导剂的培养基接种3 μL培养物(108 cfu/L), 37℃培养48 h. 挑选生长良好的菌落, 接种于含1×MIC诱导剂的培养基, 37℃培养48 h. 不断提高诱导剂在培养基中的浓度, 重复传代培养, 直至诱导剂浓度大于或等于128×MIC; (3)上一步最后得到的耐药株, 作4次无诱导剂传代培养后, 接种于含4×MIC诱导剂的培养基培养, 证实耐药菌株为非适应性耐药[20-21]. 将经诱导获得的菌株作琼脂平板培养, 分别接种于含四环素、青霉素G、环丙沙星、甲硝唑、琥乙红霉素药物(4×MIC)的琼脂平板, 37℃培养48 h, 筛选出多重耐药菌株[22].
1.2.2 抗菌药物最低抑菌浓度测定: 用琼脂平皿二倍稀释法测定5种抗菌药物对多重耐药株的最低抑菌浓度. 并确定其多重耐药谱及相应的MIC.
1.2.3 H. pylori hefA、hefC基因的检测: 采用DNA提取试剂盒提取H. pylori基因组DNA. PCR 扩增体系50 μL, 三蒸水24.5 μL, 10×PCR反应缓冲液5 μL, 25 mmol/L MgCl2 3 μL, 1 mmol/L×dNTP 4 μL, 2.5 μmol/L引物各2.5 μL, 模板DNA 8 μL, 5 U Taq酶0.5 μL, 94℃变性1 min, PCR循环参数为: 94℃ 30 s, 55℃ 45 s, 72℃ 120 s共30个循环, 最后72℃延伸600 s. 用含0.5 mg/L溴化乙啶的10 g/L琼脂糖电泳分析PCR产物并拍照, 扩增用引物见表1(hefA, hefC).
| 引物名称 | 序列 | 扩增大小(bp) |
| hefA | F: 5'-ACGCCTCGAGTAAAAGCGCAAGGGAATTTG-3' | 142 |
| R: 5'-ACGCTCTAGATTCGCTAATTGGCCTAGCAT-3' | ||
| hefC | F: 5'-TGGAAATCACAGAAACTAAAAGC -3' | 162 |
| R: 5'-TGGTTGAAGGGGCGAATTA-3' | ||
| gyrB | F: 5'-TTACTACGACTTATCCTGGGGCTAGCGCTG-3' | 267 |
| R: 5'-CCCCATCAATTTCCACATTCTCCGC-3' | ||
| hefAM | F: 5'-GCGCTCGAGGTGGGGCTACCACTAATCTC-3' | 1298 |
| R: 5'-CGTCTAGACAATCCCTGTGCTGTCTA-3' | ||
| kmR | F: 5'-GCAAGCTTAATGATTGAACAAGATGGATTG-3' | 800 |
| R: 5'-GCAAGCTTTCAGAAGAACTCGTCAAGAAG-3' |
1.2.4 H. pylori hefA基因表达水平的检测与分析: 取37℃微需氧(850 mL/L N2、100 mL/L CO2、50 mL/L O2)、相对湿度>98%的条件下培养3 d H. pylori, 从培养基上收集H. pylori重悬于磷酸盐缓冲液中, 紫外分光光度计测定浓度, 当A660nm = 0.1, 取1 mL提取其总RNA. H. pylori总RNA的提取按TRIzol试剂盒说明书步骤操作, 紫外分光光度计测定RNA含量. 反转录体系如下: M-MLV酶200 U, 5×缓冲液4 μL, 0.1 mol/L DTT 2 μL, RNasin 20 U, 5 mmol/L dNTPs 2 μL, RNA 2 μg, 100 mg/L随机引物1 μL, 加DEPC处理水至总体积20 μL, 37℃ 1 h. 将上述反转录产物进行PCR扩增, 引物见表1(hefA, gyrB), 反应体积及参数同1.2.3.
1.2.5 实时定量PCR: 取hefA表达阳性菌株H. pylori 11032的hefA, gyrB反转录PCR产物(cDNA), 纯化后连接于pMD19-T质粒, 重组质粒纯化后进行浓度测定, 梯度稀释作为标准曲线用标准品. 25 μL PCR反应体系中包括: 上下游引物各0.5 μL(10 μmo1), 2×Mix12.5 μL(包括反应缓冲液、dNTP、MgCl2、SYBRGreenⅠ、Taq酶), 双蒸水11 μL, cDNA标本1 μL. 将不同浓度的标准模板同时进行PCR, 扩增条件同1.2.4, 循环35次, 同时进行RNA为模板的阴性对照, 每个样本进行三次重复. 将检测的临界点设定在PCR扩增过程中, 荧光信号由本底进入指数增长阶段的拐点所对应的循环数(threshold cycle, CT)作为模板初始浓度的间接指标, 将不同浓度的标准模板拷贝的对数和相应的CT值作图, 得到标准曲线. 将所扩增的PCR产物同时进行琼脂糖凝胶电泳和溶解曲线分析[23-26].
1.2.6 hefA基因敲除株的构建: DNA扩增、转化、酶切鉴定参照文献[27-28]方法进行, 以临床分离株H. pylori LZ1026(分离于胃溃疡患者)为模板, PCR扩增hefA基因编码区277-1574 bp, 1298 bp大小片段(hefAM)(参照GenBank上公布的H. pylori 11637 hefA序列设计引物), 以质粒pEGFP-N2为模板, 扩增卡那霉素抗性基因(kmR), 引物设计及扩增片段大小见表1(hefAM, kmR), DNA序列测定由上海生物工程技术服务公司完成. hefA, pBluescriptⅡSK(-)经XbaⅠ, XhoⅠ双酶切后, 连接鉴定; 在hefA基因715 bp位点处插入kmR抗性基因, 提取重组质粒电转化经微需氧培养H. pylori: 自培养72 h的固体培养基上刮取H. pylori于1 mL150 mL/L甘油-9%蔗糖溶液, 4℃ 5000 r/min离心10 min(重复3次), 沉淀重悬于50 μL150 mL/L甘油-9%蔗糖溶液中, 应用U-2001紫外可见分光光度计, 测定A660nm吸光度值(A660nm = 1, 细菌浓度约1011 cfu/L), 调整细菌浓度为1013 cfu/L. 4℃放置10 min, 加打靶载体pBSK-hefA-mutant 500 ng, 冰上静置5 min, 移入-20℃预冷的0.1 cm电击池中, 冰浴5 min, 电击池置电穿孔架上, 设定电击条件: 25 F, 1.8 kV, 200 Ω, 4.92 s, 电击后, 立即加SOC缓冲液100 μL与电击池中细菌混匀, 静置5 min, 将电转液均匀涂布于普通H. pylori培养平板上, 37℃微需氧培养48 h后, 刮取平板上所有菌落于无菌生理盐水中混匀, 转涂于含25 mg/L卡那霉素的H. pylori平板上继续培养3-6 d, 挑取单个菌落培养鉴定. 用琼脂平皿二倍稀释法测定10种抗菌药物对hefA基因敲除前后菌株的最低抑菌浓度.
统计学处理 采用配对t检验分析在野生敏感株和多药耐药株之间mRNA表达水平的差异. 数据采用SPSS10.0统计软件包进行统计分析.
在5株野生敏感株和1株标准株(NCTC11637)中, 经氯霉素诱导和筛选出的耐药菌株, 均表现出相似的多重耐药性. 5种受试的抗菌药中, 四环素、青霉素G、环丙沙星和甲硝唑的MIC都程度不同地明显提高, 其中4株菌未诱导出对琥乙红霉素较高浓度的耐药(表2).
| 菌株 | 处理 | 甲硝唑 | 琥乙红霉素 | 环丙沙星 | 四环素 | 青霉素G |
| 03154 | 诱导前MIC | 0.125 | 0.125 | 0.25 | 0.25 | 0.125 |
| 诱导后MIC提高倍数 | 8× | 4× | 4× | 8× | 16× | |
| 12025 | 诱导前MIC | 2.0 | 0.0625 | 0.125 | 0.03 | 0.0625 |
| 诱导后MIC提高倍数 | 16× | 2× | 8× | 4× | 16× | |
| 12021 | 诱导前MIC | 4.0 | 0.125 | 0.25 | 0.125 | 0.0625 |
| 诱导后MIC提高倍数 | 16× | 1× | 16× | 8× | 8× | |
| 11032 | 诱导前MIC | 1.0 | 0.0625 | 0.0625 | 0.0625 | 0.25 |
| 诱导后MIC提高倍数 | 8× | 4× | 4× | 16× | 16× | |
| 03174 | 诱导前MIC | 0.5 | 0.125 | 0.25 | 0.125 | 0.125 |
| 诱导后MIC提高倍数 | 8× | 1× | 8× | 4× | 32× | |
| 11637 | 诱导前MIC | 0.5 | 0.0625 | 0.125 | 0.03 | 0.25 |
| 诱导后MIC提高倍数 | 4× | 2× | 4× | 8× | 16× |
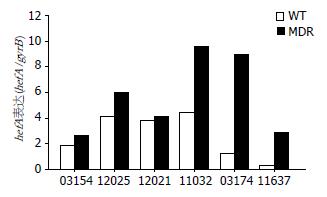
H. pylori RNA提取、反转录PCR结果如图1. 荧光实时定量PCR检测H. pylori hefA相对表达量: 本研究中将hefA基因mRNA的初始浓度与相对应的gyrA基因的初始浓度相除来进行标准化, 从而得出hefA基因产物的相对定量. 5株敏感野生株和H. pylori 11637中均可检测出不同程度的hefA mRNA表达, 相对表达量为2.6356±1.7245; 其相对应的氯霉素诱导后多药耐药株中亦可检测出hefA mRNA的表达, 其相对表达量为5.8466±2.9370(表3). MDR组hefA基因表达水平显著高于敏感野生株(P = 0.033, 图2).
| 菌株 | WT(gyrB) | WT(hefA) | MDR(gyrB) | MDR(hefA) | WT(hefA/gyrB) | MDR(hefA/gyrB) |
| 03154 | 0.379 | 0.71 | 0.33 | 0.86 | 1.87 | 2.67 |
| 12025 | 0.78 | 0.25 | 0.21 | 0.59 | 0.32 | 2.84 |
| 12021 | 0.80 | 0.98 | 0.02 | 0.17 | 1.22 | 8.94 |
| 11032 | 0.32 | 1.26 | 0.03 | 0.18 | 3.84 | 5.04 |
| 03174 | 0.13 | 0.54 | 0.02 | 0.11 | 4.16 | 6.03 |
| 11637 | 0.23 | 1.03 | 0.05 | 0.49 | 4.41 | 9.56 |
成功构建了hefA基因敲除株(△H. pylori LZ1026), hefA基因编码区内1147 bp大小片段被测序并提交GenBank数据库(登录号EU271683). 与基因敲除前野生株H. pylori LZ1026相比, 琼脂稀释法测定MIC显示, 10种抗生素(阿莫西林、头孢他定、萘啶酸、庆大霉素、多粘霉素B、克拉霉素、氟哌酸、环丙沙星、头孢噻肟和氯霉素)中, 有4种抗生素MIC比敲除前野生株下降8倍, 分别为: 克拉霉素降低8倍、头孢噻肟降低32倍、氯霉素降低16倍、庆大霉素降低8倍.
随机筛选20株临床分离幽门螺杆菌均检测出hefA, hefC基因, PCR扩增产物分别为140 bp、162 bp, 未发现hefABC基因缺失株.
研究表明, 能将抗生素主动排出体外的外排系统在细菌的多重耐药机制中起重要作用[8-11], 主动外排系统(外排泵)机制被认为是细菌产生多重耐药的主要机制[8,10]. 革兰氏阴性菌多重耐药中起重要作用RND多重药物主动外排系统, 是由3个蛋白成分形成的蛋白复合体[9,12], 即质膜主动转运体位于细菌质膜(内膜), 并与系于质膜的膜融合蛋白, 及位于细菌外膜但延伸向质膜的外膜因子蛋白一起形成转运复合体, 以达到有效外排药物的功能. RND主动外排系统中3个蛋白成分中任一蛋白的功能缺陷都会导致外排功能的失活. 随着抗生素在根除H. pylori治疗中的广泛应用, H. pylori耐药的发生率不断上升, 而且, 已经出现多重耐药株[7]. RND类外排系统介导的药物外排作用是其可能机制. H. pylori中存在hefABC, hefDEF, hefGHI 3种RND类主动外排系统, 其中hefA, hefD, hefG编码外膜因子蛋白, hefB, hefE, hefH编码膜融合蛋白, hefC, hefF, hefI编码质膜主动转运体; 经过反转录PCR扩增, 显示hefDEF, hefGHI在体外表达量很少或不表达[15]. Apweiler et al[29]报道, 在H. pylori 26695基因组中共有27个外排泵质膜主动转运体基因, 只存在4种编码外膜蛋白基因(H. pylori 0605, H. pylori 0971, H. pylori 1327和H. pylori 1489), 外膜蛋白的缺失将严重影响外排系统的功能[16].
本文以H. pylori的外膜通道蛋白的编码基因hefA(H. pylori 0605)为研究对象, 进行了H. pylori临床分离株在次抑菌浓度下的氯霉素耐药性诱导试验[19], 首次建立了H. pylori的多药耐药性诱导试验模型, 因纸片扩散法敏感性差, 可能导致阴性的药敏结果的产生[15], 我们采用琼脂二倍稀释法测定抗生素对菌株的MIC, 经氯霉素诱导后的6株敏感株H. pylori, 在对氯霉素产生耐药的同时, 也产生了对结构和作用机制不同的四环素、环丙沙星、甲硝唑、琥乙红霉素、青霉素G 5种抗生素不同程度的耐药, 诱导耐药株对5种抗生素的MIC值比诱导前敏感株增加了4倍以上, 提示外排泵的存在[30]. 本实验中4株菌未诱导出对琥乙红霉素较高浓度的耐药, 可能原因为, 与其他革兰氏阴性菌不同[31-33], RND类外排系统在H. pylori中有着不同的底物特异性. 对其外排泵hefABC结构基因中编码外膜通道蛋白基因hefA的mRNA表达量, 用实时定量PCR进行精确定量, 显示在多药耐药株中表达量明显高于野生敏感株(P = 0.033). 对中国临床分离株H. pylori LZ1026通过基因敲除技术, 敲除hefA基因, 使
H. pylori中hefABC-RND外排系统功能失活, 并测定敲除前后对多种抗生素的MIC, 显示敲除后△H. pylori LZ1026对10种抗生素中的4种敏感性明显增加, 进一步证实hefA基因在H. pylori多重耐药产生机制中起重要作用. △H. pylori LZ1026对其他6种抗生素敏感性与敲除前相比未显示增加, 可能因为H. pylori中除了hefABC外排系统, 还有其他外排机制的存在[12].
外排泵在敏感菌株之间不表达或表达量很低, 在接触抗生素之后, 表达量明显增加, 从而对在一定抗生素浓度下的细菌起到保护作用, 为存活下来的细菌进一步获得特异性耐药(如药物靶位突变)提供机会, 产生具有临床意义的多重耐药株. 多重耐药性外排泵和其他耐药机制如基因位点的突变等机制协同作用, 共同提高H. pylori的耐药性, 可能导致高水平耐药株的产生. 本研究发现, 对20株临床分离H. pylori hefABC基因进行扩增显示, 未见hefA, hefC基因缺失株, 证明外排泵在H. pylori中普遍存在. 本研究表明, 外排泵在H. pylori多重耐药中起重要作用, 介导了H. pylori多药耐药株的产生.
本试验在河南省分子重点实验室完成, 在此感谢段广才、黄志刚及许庆党在试验中给予的大力帮助.
在目前广泛应用抗生素的背景下, 细菌耐药已经成为一个世界性的课题, 尤其是多重耐药菌株的出现给临床感染性疾病的治疗带来了新的难题. 对细菌外排泵的研究已有十几年的历史, 其中对大肠杆菌、铜绿假单胞菌和金黄色葡萄球菌等研究比较深入, 存在多种抗生素耐药性有关的主动外排泵系统. 而对H. pylori多重耐药机制的研究较少, 在H. pylori对抗生素耐药机制中外排泵是否起作用还不甚明了. 本文旨在通过分子生物学技术研究H. pylori多重耐药性机制, 为克服H. pylori多重耐药的产生提供理论基础.
赵平, 副教授, 中国人民解放军第二军医大学微生物学教研室
随着H. pylori对抗生素耐药的发生率不断上升, 探讨其发生机制已经成为目前医学界研究的热点. 细菌对抗生素产生耐药的可能内在机制包括药物吸收的减少和外排的增加. 细菌多重耐药机制仍有待于进一步研究.
2000年, Bina et al在H. pylori 11637菌株中鉴定了3种编码RND外排泵系统同源基因, 分别命名为hefABC、hefDEF和hefGHI. 通过敲除相应编码质膜主动转运体hefC、hefF和hefI结构基因及用质子动力解偶联剂氰氯苯腙(CCCP)抑制外排泵, 分别做药敏试验分析, 得出结论认为hef-RND系统在H. pylori对多种抗生素耐药的内在机制中不起作用.
本文以中国H. pylori菌株为研究对象, 研究主动外排系统hefABC在H. pylori临床分离株中的诱导表达情况, 采用更加精确的琼脂二倍稀释法测定hefA基因敲除株H. pylori对多种抗生素的耐药性, 并用实时定量的方法对外排泵的编码基因在H. pylori多重耐药株中的表达情况精确定量.
本研究显示外排泵在H. pylori多重耐药中起重要作用, 从而为H. pylori的耐药机制找到新的依据, 为H. pylori多重耐药外排泵抑制剂的研发提供理论基础.
本研究方法科学, 结果可靠, 结论有价值, 具有较好的学术价值.
编辑: 程剑侠 电编:郭海丽
| 1. | Pounder RE, Ng D. The prevalence of Helicobacter pylori infection in different countries. Aliment Pharmacol Ther. 1995;9 Suppl 2:33-39. [PubMed] |
| 2. | Shi R, Xu S, Zhang H, Ding Y, Sun G, Huang X, Chen X, Li X, Yan Z, Zhang G. Prevalence and risk factors for Helicobacter pylori infection in Chinese populations. Helicobacter. 2008;13:157-165. [PubMed] [DOI] |
| 3. | Raymond J, Nguyen VB, Vidal-Trecan G, Kalach N. Helicobacter pylori infection in children of developing countries. Med Trop (Mars). 2005;65:383-388. [PubMed] |
| 4. | Malaty HM. Epidemiology of Helicobacter pylori infection. Best Pract Res Clin Gastroenterol. 2007;21:205-214. [PubMed] [DOI] |
| 5. | Axon AT. Relationship between Helicobacter pylori gastritis, gastric cancer and gastric acid secretion. Adv Med Sci. 2007;52:55-60. [PubMed] |
| 6. | Makola D, Peura DA, Crowe SE. Helicobacter pylori infection and related gastrointestinal diseases. J Clin Gastroenterol. 2007;41:548-558. [PubMed] [DOI] |
| 8. | Borges-Walmsley MI, Walmsley AR. The structure and function of drug pumps. Trends Microbiol. 2001;9:71-79. [PubMed] [DOI] |
| 9. | Nikaido H. Antibiotic resistance caused by gram-negative multidrug efflux pumps. Clin Infect Dis. 1998;27 Suppl 1:S32-S41. [PubMed] |
| 10. | Brown MH, Paulsen IT, Skurray RA. The multidrug efflux protein NorM is a prototype of a new family of transporters. Mol Microbiol. 1999;31:394-395. [PubMed] [DOI] |
| 11. | De Rossi E, Ainsa JA, Riccardi G. Role of mycobacterial efflux transporters in drug resistance: an unresolved question. FEMS Microbiol Rev. 2006;30:36-52. [PubMed] [DOI] |
| 12. | Putman M, van Veen HW, Konings WN. Molecular properties of bacterial multidrug transporters. Microbiol Mol Biol Rev. 2000;64:672-693. [PubMed] [DOI] |
| 13. | Zgurskaya HI, Krishnamoorthy G, Tikhonova EB, Lau SY, Stratton KL. Mechanism of antibiotic efflux in Gram-negative bacteria. Front Biosci. 2003;8:s862-s873. [PubMed] [DOI] |
| 15. | Bina JE, Alm RA, Uria-Nickelsen M, Thomas SR, Trust TJ, Hancock RE. Helicobacter pylori uptake and efflux: basis for intrinsic susceptibility to antibiotics in vitro. Antimicrob Agents Chemother. 2000;44:248-254. [PubMed] [DOI] |
| 16. | van Amsterdam K, Bart A, van der Ende A. A Helicobacter pylori TolC efflux pump confers resistance to metronidazole. Antimicrob Agents Chemother. 2005;49:1477-1482. [PubMed] [DOI] |
| 17. | Koronakis V, Eswaran J, Hughes C. Structure and function of TolC: the bacterial exit duct for proteins and drugs. Annu Rev Biochem. 2004;73:467-489. [PubMed] [DOI] |
| 18. | Monteiro MA, Zheng P, Ho B, Yokota S, Amano K, Pan Z, Berg DE, Chan KH, MacLean LL, Perry MB. Expression of histo-blood group antigens by lipopolysaccharides of Helicobacter pylori strains from asian hosts: the propensity to express type 1 blood-group antigens. Glycobiology. 2000;10:701-713. [PubMed] [DOI] |
| 23. | Aellen S, Que YA, Guignard B, Haenni M, Moreillon P. Detection of live and antibiotic-killed bacteria by quantitative real-time PCR of specific fragments of rRNA. Antimicrob Agents Chemother. 2006;50:1913-1920. [PubMed] [DOI] |
| 24. | Nygard AB, Jorgensen CB, Cirera S, Fredholm M. Selection of reference genes for gene expression studies in pig tissues using SYBR green qPCR. BMC Mol Biol. 2007;8:67. [PubMed] [DOI] |
| 25. | Romanowski T, Markiewicz A, Bednarz N, Bielawski KP. Housekeeping genes as a reference in quantitative real-time RT-PCR. Postepy Hig Med Dosw (Online). 2007;61:500-510. [PubMed] |
| 26. | Shamanina MIu, Vavilin VA, Mordvinov VA, Katokhin AV, Kuznetsova TN, Liakhovich VV. Real-time polymerase chain reaction and its potential clinical application. Vestn Ross Akad Med Nauk. 2007;37-46. [PubMed] |
| 27. | Yuan JP, Li T, Shi XD, Hu BY, Yang GZ, Tong SQ, Guo XK. Deletion of Helicobacter pylori vacuolating cytotoxin gene by introduction of directed mutagenesis. World J Gastroenterol. 2003;9:2251-2257. [PubMed] |
| 28. | Burns BP, Hazell SL, Mendz GL, Kolesnikow T, Tillet D, Neilan BA. The Helicobacter pylori pyrB gene encoding aspartate carbamoyltransferase is essential for bacterial survival. Arch Biochem Biophys. 2000;380:78-84. [PubMed] [DOI] |
| 29. | Apweiler R, Bairoch A, Wu CH, Barker WC, Boeckmann B, Ferro S, Gasteiger E, Huang H, Lopez R, Magrane M. UniProt: the Universal Protein knowledgebase. Nucleic Acids Res. 2004;32:D115-D119. [PubMed] [DOI] |
| 30. | Wierzbowski AK, Boyd D, Mulvey M, Hoban DJ, Zhanel GG. Expression of the mef(E) gene encoding the macrolide efflux pump protein increases in Streptococcus pneumoniae with increasing resistance to macrolides. Antimicrob Agents Chemother. 2005;49:4635-4640. [PubMed] [DOI] |
| 31. | Burse A, Weingart H, Ullrich MS. NorM, an Erwinia amylovora multidrug efflux pump involved in in vitro competition with other epiphytic bacteria. Appl Environ Microbiol. 2004;70:693-703. [PubMed] [DOI] |
| 32. | Fehlner-Gardiner CC, Valvano MA. Cloning and characterization of the Burkholderia vietnamiensis norM gene encoding a multi-drug efflux protein. FEMS Microbiol Lett. 2002;215:279-283. [PubMed] [DOI] |
| 33. | Morita Y, Kodama K, Shiota S, Mine T, Kataoka A, Mizushima T, Tsuchiya T. NorM, a putative multidrug efflux protein, of Vibrio parahaemolyticus and its homolog in Escherichia coli. Antimicrob Agents Chemother. 1998;42:1778-1782. [PubMed] |