修回日期: 2008-03-19
接受日期: 2008-05-20
在线出版日期: 2008-05-28
目的: 构建和筛选对大鼠肝星状细胞前Ⅰ型胶原α1链(COL1A1)mRNA有抑制作用的COL1A1短发夹RNA (shRNA)的表达质粒.
方法: 从NCBI网站获得大鼠的COL1A1 cDNA序列, 根据Whitehead研究所的siRNA设计软件设计3条理论上最佳的siRNA序列, 相应的双链DNA被插入pGPU6/GFP/Neo质粒中, 即pGPU6/GFP/Neo-shRNA-A、pGPU6/GFP/Neo-shRNA-B和pGPU6/GFP/Neo-shRNA-C. 为得到高效沉默COL1A1-siRNA, 以脂质体LipofectAMINE2000, 将1、2、3、4 μg DNA质粒转染至HSC-T6细胞中, 并观察转染效果. 将最佳沉默siRNA导入HSC-T6细胞, RT-PCR分析各组的COL1A1 mRNA表达水平.
结果: 靶向COL1A1 mRNA的3个shRNA重组质粒载体pGPU6/GFP/Neo-shRNA-A、pGPU6/GFP/Neo-shRNA-B和pGPU6/GFP/Neo-shRNA-C经测序分析, shRNA编码序列与设计的片段完全一致, 经酶切凝胶电泳证实载体构建成功. 1、2、3、4 μg组转染效率分别为16.7%、20.3%、23.5%和22.3%, 以2 μg siRNA为最佳剂量, pGPU6/GFP/Neo-shRNA-A、pGPU6/GFP/Neo-shRNA-B和pGPU6/GFP/Neo-shRNA-C对COL1A1 mRNA的抑制率分别为16.6%, 63.3%和80.3%.
结论: 筛选出的pGPU6/GFP/Neo-shRNA-C表达质粒能高效地抑制转染细胞COL1A1 mRNA的表达, 从而为肝纤维治疗提供新的方法和材料.
引文著录: 芦军, 赵金满, 孟艳, 余永红. COL1A1-shRNA表达质粒构建及抑制COL1A1表达的有效序列的筛选. 世界华人消化杂志 2008; 16(15): 1622-1627
Revised: March 19, 2008
Accepted: May 20, 2008
Published online: May 28, 2008
AIM: To construct and select procollagen type 1 alpha 1 (COL1A1) short hairpin RNA (shRNA) expression plasmid that can inhibit COL1A1 mRNA expression in rat hepatic stellate cell (HSC).
METHODS: Rat COL1A1 cDNA sequence was obtained from NCBI website. Three small interfering RNA sequences were selected through online design of the Whitehead Institute. The corresponding double-stranded DNA was used to construct pGPU6/GFP/Neo plasmids, namely pGPU6/GFP/Neo-shRNA-A, pGPU6/GFP/Neo-shRNA-B and pGPU6/GFP/Neo-shRNA-C. HSC-T6 cells were transfected with a green fluorescent protein (GFP)-labeled siRNA to assess the transfection efficiency. To get most effective and optimal dosage siRNA, the three plasmids (1, 2, 3, 4 μg) were transfected into HSC-T6 cells with Lipofectamine 2000 respectively, and the untreated HSC-T6 cells were used as controls. The expression of COL1A1 mRNA was detected by reverse transcription-polymerase chain reaction (RT-PCR) after the most effective and optimal dosage was used.
RESULTS: The expression plasmids targeting on COL1A1 mRNA were successfully constructed, and confirmed by agarose electrophoresis and sequence analysis. The transfection efficiencies at a dose of 1, 2, 3, and 4 μg were approximately 16.7%, 20.3%, 23.5%, and 22.3%, and 2 μg was considered as the most optimal dosage in each group. The inhibitory rates of COL1A1 mRNA levels in the HSC-T6 cells transfected with pGPU6/GFP/Neo-shRNA-A, pGPU6/GFP/Neo-shRNA-B, and pGPU6/GFP/Neo-shRNA-C were 16.6%, 63.3%, and 80.3%, respectively, when 2 μg siRNA plasmid was used.
CONCLUSION: The constructed expression plasmid pGPU6/GFP/Neo-shRNA-C can effectively inhibit the expression of COL1A1 mRNA, providing a new method and material for the treatment of liver fibrosis.
- Citation: Lu J, Zhao JM, Meng Y, Yu YH. Construction of COL1A1-shRNA expression plasmid and screening of effective sequences to inhibit COL1A1 expression. Shijie Huaren Xiaohua Zazhi 2008; 16(15): 1622-1627
- URL: https://www.wjgnet.com/1009-3079/full/v16/i15/1622.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i15.1622
肝纤维化是各种病因引起的慢性肝病, 使肝脏中胶原蛋白的增生和降解失衡, 进而导致肝脏内纤维结缔组织异常沉积的病理过程, 最终可导致肝硬化. 在肝纤维化中细胞外基质(extracellular matrix, ECM)主要成分是胶原, 其中Ⅰ型胶原为主要成分, 现已证明肝星状细胞(hepatic stellate cell, HSC)激活是产生Ⅰ型胶原导致纤维化发生发展的中心环节, 通过减少HSC中前Ⅰ型胶原的产生, 逆转或减慢肝纤维化的发展[1-2]. RNAi技术是一种新型发展起来的技术手段, 现今被广泛应用到疾病治疗研究中. RNAi是一种进化上保守的基因组水平的免疫监控机制, 通过RNaseⅢ内切核酸酶Dicer的作用产生小的(21-23 nt)短小扰性RNA(short interfering RNA, siRNA), 介导其互补同源mRNA序列的特异性降解, 能高效、特异地阻抑细胞内源或外源性靶基因的表达[3-5]. 本研究构建以U6启动子控制下的能产生针对COL1A1的短发夹环状小干扰RNA(short hairpin RNA, shRNA)表达质粒, shRNA能在RNA多聚酶Ⅲ(Pol H1)启动子的控制下从DNA模板合成, RNA Pol HI能介导3端以4-5个T结尾的小的、非编码的转录的合成. 拟用构建质粒转染大鼠肝星转细胞, 筛选出能高效沉默Ⅰ型胶原序列.
主要试剂真核表达载体pGPU6/GFP/Neo购自上海吉玛公司; E.coli DH5α购自Invitrogen公司; 质粒抽提试剂盒购自Qiagen公司; 逆转录试剂盒购自Promega公司; TRIzol(r)试剂购自Invitrogen公司; 限制性内切酶BamHⅠ、BbsⅠ、T4连接酶、Taq酶, 脂质体转染试剂盒LipofectAMINE2000和DMEM购自宝生生物工程有限公司; DNA2000Marker, G418(Gibco公司); 小牛血清购自杭州四季青生物公司; 大鼠肝星状T6细胞由中国肿瘤医院生物检测中心惠赠.
1.2.1 目的基因RNAi靶点设计和合成shRNA: 在GenBank中找到大鼠HSC的α1(Ⅰ)mRNA基因序列, 序列号为NM_053304(GeneID: 29393), 应用Whitehead研究所的siRNA设计软件, 通过计算机网络(网站为http://jura.wi.mit.edu/bioc/siRNA)辅助设计siRNA, 结合RNAi效率预测公式进行评估, 包括分析自由能特性, 与mRNA的空间可接触性, 排除偏靶(off-target)效应等, 最终筛选出3条理论上最佳的siRNA序列Col1a1-A: 5'-GAGTATGGAAGCGAAGGTTCC-3', 3'-CTCATACCTTCGCTTCCAAGG-5'. Col1a1-B: 5'-ACAAGGTGACAGAGGCATAAA-3', 3'-TGTTCCACTGTCTCCGTATTT-5'; Col1a1-C:5'-TGATGGTTCTCCTGGCAAAGA-3', 3'-ACTACCAAGAGGACCGTTTCT-5'; 同时设阴性对照. 作为阴性对照的siRNA应和选中的siRNA序列有相同的组成, 但和mRNA没有明显的同源性. 将选中的siRNA序列打乱设计阴性对照序列5'-GTTCTCCGAACGTGTCACGT-3', 5'-ACGTGACACGTTCGGAGAA-3', 同样检查结果以保证和靶细胞中其他基因没有同源性. shRNA模板中的loop结构选用了TTCAAGAGA以避免形成终止信号, shRNA的转录终止序列采用T6结构. 正义链模板的5'端添加了CACC, 与BbsⅠ酶切后形成的黏端互补; 反义链模板的5'端添加了GATC, 与BamHⅠ酶切后形成的黏端互补; 如果siRNA的第一个碱基不是G, 则在CACC后补加一个G(以上正义链和反义链由上海吉玛生物公司合成). 将DNA oligo分别用TE(pH8.0)溶解, 浓度为100 μmol/L. 取相应的正义链和反义链oligo溶液, 按照如下配比配置退火反应体系, 在PCR仪上按照如下程序进行退火处理: 95℃ 5 min; 85℃ 5 min; 75℃ 5 min; 70℃ 5 min; 4℃保存. 退火处理后得到的shRNA模板, 用于连接反应.
1.2.2 酶切和连接反应: 载体pGPU6/GFP/Neo经BamHⅠ, BbsⅠ双酶切, 37℃酶切1 h, 琼脂糖电泳, 使用Agarose Gel DNA Purification Kit Ver2.0 回收, 电泳检测估算浓度, 稀释浓度至50 mg/L. 将退火片段与经酶切后的载体混和进行连接反应, 置22℃水浴反应过夜过夜.
1.2.3 重组质粒的转化、筛选和鉴定: 各取连接产物5 μL接种到大肠杆菌DH5a感受态细胞中进行转化, 分别接种于卡那霉素抗性的LB平板上, 37℃恒温箱培养过夜. 分别挑取单克隆菌落接种于LB培养液中(含卡那霉素终浓度为30 mg/L), 37℃恒温摇床培养过夜, 用小提试剂盒小量提取质粒, 并分别用BamHⅠ, PstⅠ分别酶切鉴定. 阳性重组载体应该可以被BamHⅠ切开, 而不能被PstⅠ切开. 收集酶切鉴定正确的重组质粒, 送上海英骏生物技术有限公司进行测序鉴定. 将构建成功的载体分别命名为pGPU6/GFP/Neo-shRNA-A、pGPU6/GFP/Neo-shRNA-B、pGPU6/GFP/Neo-shRNA-C、阴性对照载体pGPU6/GFP/Neo-shRNA-N. 将鉴定证实的细菌扩增培养, 摇至对数生长期, 每0.8 mL过夜培养细菌加0.2 mL无菌甘油, 混匀后-70℃冰箱保存.
1.2.4 HSC-T6细胞培养及转染: 将大鼠肝星状细胞株(HSC-T6)细胞接种至225 cm2培养瓶中, 每瓶加入15 mL DMEM混合培养液(含100 mL/L FBS及100 kU/L青霉素、100 g/L链霉素、4 mmol/L谷氨酰胺及l mol/L HEPES), 置37℃, 50 mL/L恒温CO2培养箱中, 细胞为贴壁生长, 每3天以2.5 g/L胰蛋白酶消化传代. 转染前一天24孔板培养细胞, 设定1、2、3、4 μg, 不同量的DNA质粒, 每个四个复孔, 按照脂质体转染试剂盒LipofectAMINE2000操作说明转染细胞, 质粒和脂质体以1:4配比, 转染后48 h, 消化细胞, 流式细胞仪测定转染效率. 选取最佳剂量进行转染, 转染后24 h传代, 加入G418进行筛选, G418浓度为600 mg/L, 2 wk后挑取单克隆株扩大培养. 获得稳定表达细胞系, 分别建立表达pGPU6/GFP/Neo-shRNA-A、pGPU6/GFP/Neo-shRNA-B、pGPU6/GFP/Neo-shRNA-C和阴性对照的单克隆大鼠肝星状细胞株.
1.2.5 半定量RT-PCR测不同pGPU6/GFP/Neo-shRNA表达质粒对COL1A1的影响: 细胞总RNA提取按TRIzol操作说明书进行, COL1A1 mRNA和β-actin的引物用引物设计软件Primer Premier5.0设计, 其中β-actin为内参照. 引物由上海闪晶生物技术有限公司合成, 分别为Col1a1 F: CATAAAGGGTCATCGTGGC Col1a1 R:TCAGGCTCTTGAGGGTAGTGT; β-Actin-F: 5-TGACCCAGATCATGTTTGAGACC-3 β-Actin-R: 5-CAGTGGTACGACCAGAGGCA-3. 根据一步法RT-PCR试剂盒说明检测COL1A1. 并摄影后应用计算机图像分析系统行灰度扫描. 以与β-actin的PCR产物DNA条带灰度值之比(COL1A1/β-actin)作为反映COL1A1 mRNA水平的相对指标, 计算抑制率. 抑制率的计算为: 以条带灰度代表DNA的含量. 干扰载体对COL1A1 mRNA表达的抑制率采用下式计算: 抑制率(%) = (1-A1×A2c/A1c×A2)×100. A1: 转染干扰载体细胞的COL1A1 mRNA含量; A2: 转染干扰载体细胞的β-actin mRNA的含量; A1c: 转染shRNA-N细胞的COL1A1 mRNA含量. A2c: 转染shRNA-N载体细胞β-actin mRNA含量.
统计学处理 实验数据以mean±SD表示, 采用SPSS.13软件分析数据, 组间比较采用多组单因素方差分析(ANOVA), P<0.05为差异有统计学意义.
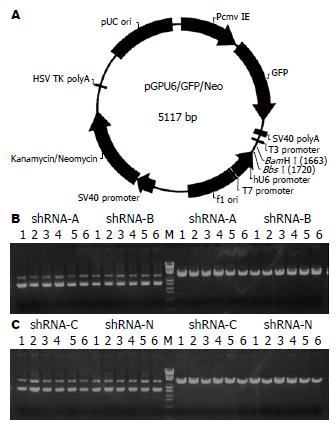
限制性内切酶PstⅠ酶和BamHⅠ双酶切质粒pGPU6/GFP/Neo-shRNA(图1A), 然后各取5 μL酶切产物在10 g/L的琼脂糖凝胶上进行电泳(图1B-C). 图1B中M为l amda/E.co130Ⅰ; 左侧为pGPU6/GFP/Neo-shRNA-A (1-6)和pGPU6/GFP/Neo-shRNA-B(1-6)PstⅠ酶切结果; 其右侧为pGPU6/GFP/Neo-shRNA-A和pGPU6/GFP/Neo-shRNA-B(1-6)BamHⅠ酶切结果. 图1C中M为lamda/E.co130Ⅰ; 左侧为pGPU6/GFP/Neo-shRNA-C(1-6)和pGPU6/GFP/Neo-shRNA-N(1-6)PstⅠ酶切结果; 其右侧为pGPU6/GFP/Neo-shRNA-C(1-6)和pGPU6/GFP/Neo-shRNA-N(1-6)BamHⅠ酶切结果.
重组质粒经自动基因测序仪测序(上海英骏生物技术有限公司). 结果与设计序列完全相符, 所含目的基因序列准确无误, 重组质粒构建成功. 真核表达载体pGPU6/GFP/Neo-shRNA带有一突变的绿色荧光蛋白的报告基因, 能发出高亮度的绿色荧光, 应用流式细胞仪分析转染效率显示, 1、2、3、4 μg不同量的DNA质粒转染效率分别为16.7%±1%、20.3%±2%、23.5%±2%、22.3%±3%, 以2 μg为最佳剂量.
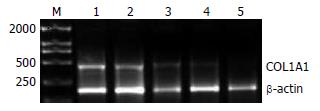
半定量RT-PCR检测HSC内COL1A1 mRNA水平, 结果显示空白组和pGPU6/GFP/Neo-shRNA-N无显著差异(P>0.05), pGPU6/GFP/Neo-shRNA-A、B、C、N各组之间均有显著性差异(P<0.001, 表1, 图2).
| 分组 | 灰度比 | 抑制率(%) |
| 空白组 | 0.91±0.03 | 0.10±0.10 |
| pGPU6/GFP/Neo-shRNA-N | 0.90±0.03 | 0.00±0.03 |
| pGPU6/GFP/Neo-shRNA-A | 0.75±0.05 | 16.6±0.40 |
| pGPU6/GFP/Neo-shRNA-B | 0.33±0.04 | 63.3±0.30 |
| pGPU6/GFP/Neo-shRNA-C | 0.18±0.06 | 80.3±0.60 |
肝纤维化是慢性肝病共有的病理改变, 其本质是以胶原为主的ECM合成增多, 而降解相对减少, 两者失去动态平衡, 致使过多ECM沉积于肝内[6]. 研究表明ECM中主要成分是胶原, 正常肝脏胶原蛋白占总蛋白5%-10%, 其中Ⅰ型胶原蛋白占总胶原蛋白的40%-50%, Ⅲ型胶原蛋白占总胶原蛋白的40%-50%, 肝纤维化时胶原蛋白可占总蛋白的50%甚至更多, 其中Ⅰ型胶原蛋白占总胶原蛋白升至60%-70%, Ⅲ型胶原蛋白降至20%-30%. 可见肝纤维化时主要是肝脏中Ⅰ型胶原过度增加的结果. 进一步研究发现HSC合成分泌Ⅰ型胶原为主, 且整个肝脏胶原合成的数量以HSC合成的胶原为主, 现已证明HSC激活是产生Ⅰ型胶原导致纤维化发生发展的中心环节, 与肝脏内细胞, 基质, 介质相互作用构成了复杂的网络系统, 造成肝纤维化的发生发展[7-11]. 理论上,如果设法能阻断HSC中α1(Ⅰ)链的合成, HSC中缺少α1(Ⅰ)链, 而不能和α2(Ⅰ)链结合产生前Ⅰ型胶原, 从而达到减少Ⅰ型胶原合成的目的, 有效阻止肝纤维化进展.
RNAi作为一种新型的基因研究手段和治疗手段, 逐渐被广泛应用到生命科学当中. 在肝纤维化基因研究和治疗中, Kim et al[12]利用以质粒为载体的TGF-β1 siRNA注入到CCl4肝纤维化大鼠体内, 结果发现Ⅰ型胶原蛋白和α-平滑肌动蛋白(α-SMA)表达水平下降, 肝脏损害减轻. 我们可以看出RNA干扰对于肝纤维化治疗是一个很好的策略. 由于RNAi具有高度特异性、高效性、高成功率和模板选择性、效果可预测并且迅速、经济, 具有良好的应用前景. 有效的基因沉默, 需要设计和筛选特异、高效的靶基因RNAi序列, 以及通过有效方法把有效序列导入目的细胞是RNA干扰试验成功的关键[13-15]. 我们在设计RNA干扰有效序列是通过计算机网络进行第一步设计, 然后根据Tuschl新算法的设计原则既(N4)A(N6)T(N2)H(N5)W(N2), 其中N为任意核苷酸, A为腺嘌呤, T为胸腺嘧啶, H为不是鸟嘌呤, W为是A而不是G或C(胞嘧啶), 选取和上述原则相近的序列, 排除偏靶(off-target)效应, 最终筛选出3条理论上最佳的序列, 结果和原则最为相近的序列沉默效果最好, 而其他较差, 考虑和序列作用靶点有关[16-21]. 通过实验我们可以看到结合多种筛选方式选择有效序列, 能大大提高筛选成功率, 节约实验成本, 节省实验时间. 将siRNAs转入细胞是RNA干扰关键技术之一, 常规方法有: 脂质体转染、电穿孔法、微注射法[22-24]. 电穿孔法和微注射法操作复杂, 并且细胞死亡率较高, 而且需要较好的设备, 虽然脂质体(本课题使用LipfectamineTM 2000)转染试剂介导的转染, 操作简单, 而且不需要复杂的设备, 可以直接转染小的siRNA, 又可以转染质粒DNA, 且适合多种细胞类型使用, 但也存在对于不同细胞转染效率不一, 本课题转染细胞株效率不高, 进行G418稳定筛选, 工作量加大, 实验成本增加. 因此进一步研究高效导入HSC工具, 是十分必要的, 近年来相关实验, 应用腺相关病毒和慢病毒作为载体越来越多, 但腺病毒的免疫效应需进一步改善, 而以慢病毒为载体, 目的片段被整合到基因组当中, 这是否造成肿瘤的发生需要进一步研究[25-27].
总之, 本实验的siRNA设计是成功的, 除pGPU6/GFP/Neo-shRNA-A抑制无明显效果外, pGPU6/GFP/Neo-shRNA-B和pGPU6/GFP/Neo-shRNA-C均产生了很好的抑制效果, 在COL1A1 mRNA水平均能较显著抑制COL1A1的表达, 而且设计的shRNA-C靶点抑制效率达到80.3%, 表明我们筛选出了具有高效沉默COL1A1的小干扰RNA表达质粒, 为深入研究肝纤维化的RNAi靶向基因治疗体内实验奠定了基础.
肝纤维化发生机制是肝损害持续存在, 机体发生的修复反应, 造成细胞外基质合成, 降解与沉积不平衡的病理过程, 而胶原是细胞外基质得主要成分, 其中尤以Ⅰ型胶原, 如果能减少其合成, 就能延缓肝纤维化向肝硬化的转变. RNAi技术是一种新型发展起来的技术手段, 能高效、特异地阻抑细胞内源或外源性靶基因的表达.
冯志杰, 主任医师, 河北医科大学第二医院消化内科.
有报道经门静脉注射肝纤维化大鼠CTGF siRNA发现, 治疗组大鼠肝组织CTGF mRNA及蛋白表达显著下调, 肝组织炎症、坏死及纤维化显著减轻. Kim et al将TGFβ1 siRNA用于CCl4肝损伤大鼠模型同样发现, 肝脏表达TGFβ1、β-SMA、Ⅰ型胶原含量均明显下降.
本研究通过RNA干扰的方法抑制大鼠肝星状细胞COL1A1 mRNA的表达, 从而减少Ⅰ型胶原的合成.
利用RNA干扰抑制COL1A1 mRNA的表达, 为进一步治疗肝脏纤维化提供了方法和途径.
本文选材新颖, 技术先进, 论据充分, 国内外相关报道较少, 有一定的应用价值.
编辑: 程剑侠 电编:郭海丽
| 2. | Prosser CC, Yen RD, Wu J. Molecular therapy for hepatic injury and fibrosis: where are we? World J Gastroenterol. 2006;12:509-515. [PubMed] |
| 3. | Paul CP, Good PD, Winer I, Engelke DR. Effective expression of small interfering RNA in human cells. Nat Biotechnol. 2002;20:505-508. [PubMed] [DOI] |
| 4. | Brummelkamp TR, Bernards R, Agami R. A system for stable expression of short interfering RNAs in mammalian cells. Science. 2002;296:550-553. [PubMed] [DOI] |
| 5. | Sarem M, Znaidak R, Macias M, Rey R. Hepatic stellate cells: it's role in normal and pathological conditions. Gastroenterol Hepatol. 2006;29:93-101. [PubMed] |
| 6. | Rockey DC. Antifibrotic therapy in chronic liver disease. Clin Gastroenterol Hepatol. 2005;3:95-107. [PubMed] [DOI] |
| 7. | Safadi R, Friedman SL. Hepatic fibrosis--role of hepatic stellate cell activation. MedGenMed. 2002;4:27. [PubMed] |
| 8. | Shen H, Fan J, Burczynski F, Minuk GY, Cattini P, Gong Y. Increased Smad1 expression and transcriptional activity enhances trans-differentiation of hepatic stellate cells. J Cell Physiol. 2007;212:764-770. [PubMed] [DOI] |
| 9. | Parsons CJ, Takashima M, Rippe RA. Molecular mechanisms of hepatic fibrogenesis. J Gastroenterol Hepatol. 2007;22Suppl 1:S79-S84. [PubMed] [DOI] |
| 10. | Brandao DF, Ramalho LN, Ramalho FS, Zucoloto S, Martinelli Ade L, Silva Ode C. Liver cirrhosis and hepatic stellate cells. Acta Cir Bras. 2006;21 Suppl 1:54-57. [PubMed] [DOI] |
| 11. | Blum HE. Molecular therapy and prevention of liver diseases. Adv Med Sci. 2007;52:29-36. [PubMed] |
| 12. | Kim KH, Kim HC, Hwang MY, Oh HK, Lee TS, Chang YC, Song HJ, Won NH, Park KK. The antifibrotic effect of TGF-beta1 siRNAs in murine model of liver cirrhosis. Biochem Biophys Res Commun. 2006;343:1072-1078. [PubMed] [DOI] |
| 13. | Harborth J, Elbashir SM, Vandenburgh K, Manninga H, Scaringe SA, Weber K, Tuschl T. Sequence, chemical, and structural variation of small interfering RNAs and short hairpin RNAs and the effect on mammalian gene silencing. Antisense. Nucleic Acid Drug Dev. 2003;13:83-105. [PubMed] [DOI] |
| 14. | Hamada M, Ohtsuka T, Kawaida R, Koizumi M, Morita K, Furukawa H, Imanishi T, Miyagishi M, Taira K. Effects on RNA interference in gene expression (RNAi) in cultured mammalian cells of mismatches and the introduction of chemical modifications at the 3'-ends of siRNAs. Antisense Nucleic Acid Drug Dev. 2002;12:301-309. [PubMed] [DOI] |
| 15. | Ohnishi Y, Tokunaga K, Hohjoh H. Influence of assembly of siRNA elements into RNA-induced silencing complex by fork-siRNA duplex carrying nucleotide mismatches at the 3'- or 5'-end of the sense-stranded siRNA element. Biochem Biophys Res Commun. 2005;329:516-521. [PubMed] [DOI] |
| 16. | Hossbach M, Gruber J, Osborn M, Weber K, Tuschl T. Gene silencing with siRNA duplexes composed of target-mRNA-complementary and partially palindromic or partially complementary single-stranded siRNAs. RNA Biol. 2006;3:82-89. [PubMed] |
| 17. | Vanitharani R, Chellappan P, Fauquet CM. Short interfering RNA-mediated interference of gene expression and viral DNA accumulation in cultured plant cells. Proc Natl Acad Sci U S A. 2003;100:9632-9636. [PubMed] [DOI] |
| 18. | Miyagishi M, Taira K. Development and application of siRNA expression vector. Nucleic Acids Res Suppl. 2002;113-114. [PubMed] |
| 19. | Miyagishi M, Taira K. U6 promoter-driven siRNAs with four uridine 3' overhangs efficiently suppress targeted gene expression in mammalian cells. Nat Biotechnol. 2002;20:497-500. [PubMed] [DOI] |
| 20. | Fire A, Xu S, Montgomery MK, Kostas SA, Driver SE, Mello CC. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 1998;391:806-811. [PubMed] [DOI] |
| 21. | Elbashir SM, Harborth J, Lendeckel W, Yalcin A, Weber K, Tuschl T. Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells. Nature. 2001;411:494-498. [PubMed] [DOI] |
| 22. | Elbashir SM, Lendeckel W, Tuschl T. RNA interference is mediated by 21- and 22-nucleotide RNAs. Genes Dev. 2001;15:188-200. [PubMed] [DOI] |
| 23. | Elbashir SM, Martinez J, Patkaniowska A, Lendeckel W, Tuschl T. Functional anatomy of siRNAs for mediating efficient RNAi in Drosophila melanogaster embryo lysate. EMBO J. 2001;20:6877-6888. [PubMed] [DOI] |
| 24. | Elbashir SM, Harborth J, Weber K, Tuschl T. Analysis of gene function in somatic mammalian cells using small interfering RNAs. Methods. 2002;26:199-213. [PubMed] [DOI] |
| 25. | Reynolds A, Leake D, Boese Q, Scaringe S, Marshall WS, Khvorova A. Rational siRNA design for RNA interference. Nat Biotechnol. 2004;22:326-330. [PubMed] [DOI] |
| 26. | Qu Z, Lou D, Pan Y. The role of IkappaBalpha in TNF-alpha-induced apoptosis in hepatic stellate cell line HSC-T6. J Huazhong Univ Sci Technolog Med Sci. 2007;27:407-410. [PubMed] [DOI] |
| 27. | Mao HX, Yi ZW, Wu XC, Dang XQ, He XJ, Cao Y, Mo SH. Effects of siRNAs silencing transforming growth factor-beta1 on fibronectin in SD rat masangial cells. Zhonghua Erke Zazhi. 2007;45:494-497. [PubMed] |