修回日期: 2007-09-18
接受日期: 2007-09-28
在线出版日期: 2007-10-18
目的: 评估镇江地区H pylori临床株的cagA基因阳性率、了解菌株CagA蛋白的可能分型、 其羧端可变区EPIYA的数目以及与临床结果之间有无关联.
方法: 培养分离来自临床胃十二指肠疾病患者的H. pylori, 提取基因组DNA, 通过PCR方法检测H. pylori cagA基因的状况. 随机选择不同病种的cagA基因阳性(cagA+)的PCR产物, 通过转化质粒, 进行cagA+ PCR产物测序, 通过ExPASY-Tranlation软件获得cagA基因相应的CagA蛋白的氨基酸序列. 国际标准株NCTC11637的cagA基因以及CagA蛋白序列通过搜索NCBI(www.ncbi.nlm.nih.gov)数据库获得.
结果: 60株H. pylori临床株中, 56例为cagA+, 阳性率达93.3%. 20例测序的结果表明, CagA蛋白结构可分为2种类型, 19株东亚型和1株西方型. 所有19株东亚型均为Yamaoka分型的A型, 所有20例菌株的EPIYA的数目均为3个, 与临床结果无关联.
结论: cagA基因阳性率在不同病种之间无差异. 镇江地区H. pylori临床株的CagA蛋白的一级结构, 与日本、韩国菌株一样, 主要为东亚型, 其CagA蛋白羧端可变区EPIYA数目均为3个, 与临床结果无关联.
引文著录: 吴莺, 张尤历, 范钰, 魏金文, 何亚龙, 沈琰, 陈劲频, 印亦萍, 王文兵. 镇江地区H. pylori的cagA基因及其3'可变区结构的变化. 世界华人消化杂志 2007; 15(29): 3147-3151
Revised: September 18, 2007
Accepted: September 28, 2007
Published online: October 18, 2007
AIM: To evaluate the expression of cagA gene of Helicobacter pylori strains isolated from Chinese patients living in Zhenjiang, Jiangsu Province, their CagA protein type, and the relationship between the number of EPIYA located in CagA protein C terminus and the clinical outcome.
METHODS: After H. pylori isolated from patients with gastrointestinal diseases were cultured, the genomic DNA was extracted and PCR was performed to examine the expression of the variable region of the cagA gene. The DNA fragments from 20 isolated strains with cagA+ were sequenced.
RESULTS: CagA gene expression was found in 56 of 60 patients. According to the amino acid sequences of CagA protein variable region, the isolates fell into two groups: the East-Asia type that contained 19 isolated strains belonging to Yamaoka's A type, and one strain that belonged to the Western type. All 20 isolated strains possessed three EPIYA motifs, and had no relationship between the amount of EPIYA and clinical outcome.
CONCLUSION: It is no distinguish that the expression of CagA gene between different diseases. CagA proteins of H. pylori isolated from patients living in Zhenjiang all belong to the East-Asia type, the same as strains isolated from Korea and Japan. There are three EPIYA motifs in the CagA C-terminal variable region of all H. pylori strains, and there is no relationship between the amount and clinical outcome.
- Citation: Wu Y, Zhang YL, Fan Y, Wei JW, He YL, Shen Y, Chen JP, Yin YP, Wang WB. Study of the 3' terminal variable region of the cagA gene in Helicobacter pylori isolated from patients living in Zhenjiang. Shijie Huaren Xiaohua Zazhi 2007; 15(29): 3147-3151
- URL: https://www.wjgnet.com/1009-3079/full/v15/i29/3147.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i29.3147
H. pylori的cagA基因编码的CagA蛋白是目前研究最多的细菌毒力因子之一, 研究认为CagA蛋白与H. pylori感染的临床结果相关[1-2]. 有研究发现, H. pylori cagA基因3'可变区的结构存在差异, 从而导致了CagA蛋白大小、以及生物功能的不同. 而造成cagA基因3'可变区结构差异的主要原因是重复序列的数目, 这个差异可能影响H. pylori感染的临床后果[2-3]. Stein et al[4]的研究发现, 易位后的CagA蛋白在宿主细胞内经过酪氨酸磷酸化, 引起宿主细胞支架的重排、细胞延伸效应和细胞活力的增加, 这种变化与胃癌的发生相关. 而CagA蛋白的酪氨酸磷酸化位点, 位于羧端的Glu-Pro-Ile-Tyr-Ala(EPIYA)基序上[5]. Naito et al[6]的研究发现, CagA蛋白羧端的EPIYA基序的数目与CagA蛋白的磷酸化能力和CagA蛋白的毒力呈正相关. 因此, CagA蛋白的EPIYA序列重复的多态性可很大程度影响CagA活性的强度和持续时间, 决定CagA毒力因子的强弱, 而这个毒力可能直接导致胃癌的发生. 本研究通过对镇江地区H. pylori临床株的cagA基因的检测, 以及对部分cagA+菌株PCR产物的序列测定分析, 了解镇江地区cagA基因的阳性率、cagA基因3'可变区的差异以及相应CagA蛋白氨基酸序列的特征、EPIYA基序的数目与临床结果关联.
60株H. pylori菌株, 从2004-06/2006-02江苏大学附属医院消化科胃镜室进行胃镜检察的患者中分离(男54例,女6例。平均年龄47.3+2.1岁), 每人取胃窦黏膜3块, 一块用于H. pylori的分离、培养, 一块用于快速尿素酶试验, 1块用于组织学检查. 慢性胃炎13例, 消化性溃疡38例(胃溃疡11例, 十二指肠球部溃疡27例), 胃窦腺癌9例. 国际标准株NCTC11637(cagA+)由英国胃肠病学会主席、英国诺丁汉大学王后医学中心教授Atherton馈赠. TaqDNA聚合酶及配套缓冲液、100 bp DNA ladder均购自Fermentas公司, dNTP购自南京大治生物有限公司, 空肠弯曲菌培养基、微需氧产气袋、选择性抗生素购自德国Merk公司, PCR引物由上海生工生物工程技术服务有限公司合成, PCR仪为HBPX-220型(英国产).
1.2.1 Hpylori培养及DNA抽提: 患者胃窦黏膜标本接种于含100 mL/L羊血和混合抗生素的哥伦比亚琼脂培养基上, 37℃微需氧环境(50 mL/L O2、100 mL/L CO2、850 mL/L N2)中培养3-5 d. 转种后, 培养3 d, 经菌落形态、菌落涂片、生化反应(快速尿素酶试验、氧化酶试验、和触酶试验)证实, 收集菌体并重悬于10 g/L SDS的STE中, 用酚/氯仿/异丙醇法抽提DNA , 700 mL/L的酒精洗涤2次, 室温晾干后加入适量TE缓冲液, 4℃冰箱过夜溶解, -20℃保存备用.
1.2.2 PCR检测cagA基因: 引物设计见参考文献[7], 上游引物Cag2序列为5'-GGAACCCTAGTCGGTAATG, 下游引物CagA 26R序列为5'-GCTTTAGCTTCTGATACC. 25 µL反应体系含TaqDNA聚合酶, 2×10-4 mol/L dNTPs, 0.2 mmol/L每条引物, 1.5 µL DNA模板, 10 mmol/L Tris-HCl(pH8.3), 50 mmol/L KCl, 25 mmol/L MgCl2. PCR扩增条件为95℃预变性3 min, 95℃变性30 s, 54 ℃退火30 s, 72 ℃延伸3 min, 循环35次, 72℃终延伸3 min. PCR反应完毕后, 取5 µL反应产物在15 g/L琼脂糖凝胶中电泳,凝胶成像系统紫外光下观察结果并照相.
1.2.3 cagA DNA质粒转换和测序: 在56例cagA+的菌株中, 分别选择胃癌菌株4例、消化性溃疡9例、慢性胃炎7例, 对其PCR产物用纯化试剂盒回收, 再与pMD18-T载体连接, 转化感受态细胞进行蓝白斑筛选. 挑出白色单菌落扩增, 用SDS-碱裂解法抽提质粒, 分别用BamHⅠ、EcoRⅠ酶切, 并电泳鉴定. 对阳性克隆进行测序, 测序由上海生工生物工程技术服务有限公司完成. 标准株11637 cagA的序列, 以"Helicobacter pylori"、"cagA"主题词检索Pubmed的Nucleotide数据库获得, 其核酸序列GenBank(http://www.ncbi.nlm.nih.gov)接受号: AB015416; 蛋白序列GenBank接受号: BAB20926.1
统计学处理 数据分析采用χ2检验, P<0.05为有显著性差异.
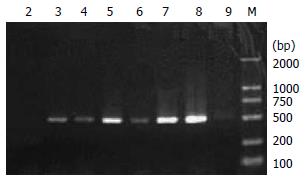
60例H. pylori菌株, 56例cagA基因阳性, 阳性率93.3%; 对照株11 637 cagA及56例cagA+的镇江H. pylori株均可扩增出单一的518 bp片段, 未见非特异性扩增产物(图1). 消化性溃疡, 胃窦腺癌H. pylori分离株的cagA基因阳性率与慢性胃炎比较, 差异无显著性(P>0.05, 表1).
| 分组 | n | cagA基因 | P |
| 慢性胃炎 | 13 | 12(92.3) | |
| 十二指肠球部溃疡 | 27 | 26(96.3) | >0.05 |
| 胃溃疡 | 11 | 10(90.0) | >0.05 |
| 胃窦腺癌 | 9 | 8(88.9) | >0.05 |
| 总计 | 60 | 56(93.3) |
选取慢性胃炎7例, 胃溃疡4例, 十二指肠球部溃疡5例, 胃窦腺癌4例cagA+的PCR产物进行测序, 部分菌株测序结果见图2. 本组菌株的测序特点有6点: (1)大多数镇江菌株的cagA基因3'可变区结构相似, 但存在碱基的插入、缺失、替代; (2)镇江地区菌株均具有3个EPIYA重复区; (3)19个镇江菌株的第3个EPIYA后有1个DFD序列, 与西方菌株11637的DDL序列明显不同; (4)西方菌株11637的FPLKRHDKVDDLSKV氨基酸序列在19株镇江菌株中被KIASAGKGVGGFSGA氨基酸序列取代, 与报道的日本菌株特征一致[3], 具有东亚地区菌株的特征; (5)一株镇江地区的菌株W248, 来自于十二指肠球部溃疡的患者, 表现出与西方菌株11 637相似的特征: 主要序列为两个西方特异性FPLKRHDKVDDLSKV序列而非东方特异性KIASAGKGVGGFSGA序列, 第3个EPIYA后为DDL序列而非东亚的DFD序; (6)19株表现为东亚型的镇江菌株均表现为Yamaoka所描述的A型(R1-R2-R1-R3-R1), 而没有发现其所描述的B型、C型、D型. 一株表现为西方型的菌株, 则不能按此标准分型. 未发现Yamaoka所描述的与严重胃病相关的C型, 也未发现cagA基因3'可变区的结构变化与临床相关.
50%以上的世界人口感染H. pylori, 大多数表现为无症状, 仅15%-20%的感染者可能发展为慢性胃炎、消化性溃疡、胃窦腺癌, 这可能与宿主的个体遗传差异、环境因素以及细菌的毒力有关. CagA蛋白是目前H. pylori研究最多的毒力因子之一. CagA蛋白由H. pylori cagA基因编码, 分泌后, 经IV分泌系统进入细菌黏附的胃上皮细胞, 在其羧基端EPIYA序列上进行酪氨酸磷酸化, 磷酸化的CagA引起细胞骨架的重排、诱导细胞形态学的变化(蜂鸟表型)和细胞动力的增强, 造成细胞异常的增殖和运动, 最终导致癌变[4,8]. 因此cagA阳性(cagA+)的H. pylori菌株较cagA-的H. pylori菌株毒力强. 而不同来源的H. pylori菌株的cagA基因羧基端可变区存在重复序列的变化, 并因此导致cagA基因结构的多态性以及细菌毒力(致病性)不同. 因此, cagA 3'端可变区及其CagA蛋白EPIYA基序与临床相关性关系的研究, 成为目前H. pylori的研究热点之一.
本研究显示, 镇江地区cagA基因菌株的cagA基因阳性率为93.3%(56/60), 与国内以及日本的报道相符[9-11]. 相对于西方, 中国和日本同属胃癌高发的东亚地区, cagA基因在东西方之间阳性率的不同, 可能是造成东亚地区胃癌高发的原因. 但在作者分离的菌株中, 并没有发现不同病种之间, cagA基因的阳性率存在显著性差异(P>0.05, 表1), 说明cagA+基因在镇江地区不能作为细菌毒力强弱的标志. 因此, 有必要进一步研究cagA基因的多态性以及由此造成其所编码CagA蛋白的多态性, 以及CagA蛋白致病性的差异.
对来自不同病种的20株菌株cagA+ PCR产物测序, 可以推断其相应CagA蛋白的一级氨基酸序列结构, 从而分析CagA蛋白的多态性. 本研究显示, 镇江地区菌株的CagA蛋白, 根据其一级结构分为2个亚型: 东亚型和西方型. 东亚型和西方型CagA蛋白羧端可变区序列的主要区别表现为: (1)东亚型的主要序列是KIASAGKGVGGFSGA, FPLKRHDKVDDLSKV; (2)第3个EPIYA后, 东亚型是DFD, 而西亚型是DDL序列. 在测序的20株菌株中, 19株(95.0%, 19/20)表现为东亚型, 1株(5.0%, 1/20)表现为西方型, 并且东亚型菌株与日本菌株相似[11], 这和同属胃癌高发的东亚地区一致. 我们的19株东亚型, 参照Yamaoka的划分, 均为A型, 未见与胃癌发病有关联的C型. 虽然我们有4株菌株来自于胃腺癌, 也未发现Yamaoka的B型和D型, 显示中国镇江地区菌株以Yamaoka的A型占主流. 在20株菌株中, 有一株主要序列与西方菌株的标准株11 637主要序列相似, 故为西方型, 来自于与胃癌无关的十二指肠球部溃疡的患者. 至今尚未发现有东亚菌株表现为西方菌株的报道, 其机制有待进一步明确, 而且表现为西方菌株的W248菌株不能根据Yamaoka法进行分型[11].
Stein的研究证实, CagA蛋白的酪氨酸磷酸化位点位于其羧基端的EPIYA基序上. CagA蛋白EPIYA基序上的酪氨酸残基, 在宿主细胞内进行酪氨酸磷酸化后, 成为SHP-2的SH2区的停泊位点[4], 因此EPIYA基序有两个功能: 与细胞膜的连接和进行酪氨酸磷酸化. 这两个功能均涉及CagA蛋白的活化以激活细胞内信号转导途径, 显示EPIYA基序是cagA基因+H. pylori感染的关键性治疗目标[5]. 而CagA蛋白与SHP-2的连接能力的强弱决定其细胞形态学转化能力, 即生物学活性的强弱. 研究显示, 东亚菌株的CagA蛋白较西方菌株相比, 与SHP-2的连接能力更强, 因而毒力更强[8,12,13], 这与东亚地区为胃癌高发区以及胃癌高死亡率一致. 多数H. pylori菌力株具有3个串联的EPIYA基序, 也有2和4个的报道[7,9]. 本实验所测序的20株镇江地区H. pylori菌株CagA蛋白的EPIYA数目菌为3个, 并没有发现EPIYA的数目与疾病之间存在相关性.
因此, cagA基因阳性在镇江地区的H. pylori中不能作为细菌毒力强弱的指标, 镇江地区的CagA蛋白可以分为东亚型和西方型, 镇江菌株95.%(19/20)表现为东亚型. CagA蛋白羧基端可变区的EPIYA序列的数目与临床结果无关.
感谢江苏大学生命科学研究院王文兵博士给予的技术帮助和胃镜室沈琰医师在标本采集中提供的帮助.
50%以上的世界人口感染H. pylori, 大多数表现为无症状, 仅15%-20%的感染者发展为胃肠疾病和胃癌. 因此, H. pylori菌株的遗传多样性, 尤其毒力因子的遗传多样性(致病性差异)成为目前国内外H. pylori研究的焦点.
CagA(cytotoxin-associated per-tain A)蛋白, 是H. pylori最重要的标志性毒素之一, 在胃上皮细胞内发生酪氨酸磷酸化, 从而引起细胞信号转导通路的改变, 而这个磷酸化反应, 被认为是H. pylori毒力增强的标志, 与致病力密切相关, 而位于CagA蛋白羧端的EPIYA序列的重复数目和类型, 可能与CagA蛋白的磷酸化程度有关.
西方学者认为cagA基因阳性(cagA+)H. pylori菌株较cagA基因阴性(cagA-)菌株毒力更强, 但日本的学者认为日本与西方H. pylori菌株的cagA基因及其相应的CagA蛋白存在结构差异, 并且这个差异可能与胃癌的发生相关.
本文研究了60株中国H. pylori菌株和一株西方标准H. pylori的cagA基因状态及其相应的CagA蛋白的分型、CagA蛋白端EPIYA序列的重复数目和类型, 以及与临床结果的关联.
本文研究发现东西方菌株存在结构差异, 并且中国菌株存在西方型的变异. 该菌株来自一名DU患者, 应该进一步研究东西方菌株毒力差异的原因.
EPIYA序列: 是指CagA蛋白羧端的Glu-Pro-Ile-Tyr-Ala(EPIYA)基序, 位于其上的酪氨酸残基是CagA蛋白的磷酸化位点, 与CagA蛋白的致病性有关.
本文立题新颖, 陈述清晰, 分析有据, 有一定的学术价值和参考价值.
编辑: 程剑侠 电编:郭海丽
| 1. | Covacci A, Censini S, Bugnoli M, Petracca R, Burroni D, Macchia G, Massone A, Papini E, Xiang Z, Figura N. Molecular characterization of the 128-kDa immunodominant antigen of Helicobacter pylori associated with cytotoxicity and duodenal ulcer. Proc Natl Acad Sci U S A. 1993;90:5791-5795. [PubMed] |
| 2. | Azuma T, Yamakawa A, Yamazaki S, Fukuta K, Ohtani M, Ito Y, Dojo M, Yamazaki Y, Kuriyama M. Correlation between variation of the 3' region of the cagA gene in Helicobacter pylori and disease outcome in Japan. J Infect Dis. 2002;186:1621-1630. [PubMed] |
| 3. | Higashi H, Tsutsumi R, Fujita A, Yamazaki S, Asaka M, Azuma T, Hatakeyama M. Biological activity of the Helicobacter pylori virulence factor CagA is determined by variation in the tyrosine phosphorylation sites. Proc Natl Acad Sci U S A. 2002;99:14428-14433. [PubMed] |
| 4. | Stein M, Bagnoli F, Halenbeck R, Rappuoli R, Fantl WJ, Covacci A. c-Src/Lyn kinases activate Helicobacter pylori CagA through tyrosine phosphorylation of the EPIYA motifs. Mol Microbiol. 2002;43:971-980. [PubMed] |
| 5. | Higashi H, Yokoyama K, Fujii Y, Ren S, Yuasa H, Saadat I, Murata-Kamiya N, Azuma T, Hatakeyama M. EPIYA motif is a membrane-targeting signal of Helicobacter pylori virulence factor CagA in mammalian cells. J Biol Chem. 2005;280:23130-23137. [PubMed] |
| 6. | Naito M, Yamazaki T, Tsutsumi R, Higashi H, Onoe K, Yamazaki S, Azuma T, Hatakeyama M. Influence of EPIYA-repeat polymorphism on the phosphorylation-dependent biological activity of Helicobacter pylori CagA. Gastroenterology. 2006;130:1181-1190. [PubMed] |
| 7. | Zhang Y, Argent RH, Letley DP, Thomas RJ, Atherton JC. Tyrosine phosphorylation of CagA from Chinese Helicobacter pylori isolates in AGS gastric epithelial cells. J Clin Microbiol. 2005;43:786-790. [PubMed] |
| 8. | Hatakeyama M, Higashi H. Helicobacter pylori CagA: a new paradigm for bacterial carcinogenesis. Cancer Sci. 2005;96:835-843. [PubMed] |
| 11. | Yamaoka Y, Kodama T, Kashima K, Graham DY, Sepulveda AR. Variants of the 3' region of the cagA gene in Helicobacter pylori isolates from patients with different H. pylori-associated diseases. J Clin Microbiol. 1998;36:2258-2263. [PubMed] |
| 12. | Ando T, Goto Y, Maeda O, Watanabe O, Ishiguro K, Goto H. Causal role of Helicobacter pylori infection in gastric cancer. World J Gastroenterol. 2006;12:181-186. [PubMed] |
| 13. | Azuma T. Helicobacter pylori CagA protein variation associated with gastric cancer in Asia. J Gastroenterol. 2004;39:97-103. [PubMed] |