修回日期: 2007-04-01
接受日期: 2007-04-21
在线出版日期: 2007-06-18
目的: 研究痢疾杆菌福氏2a入侵前和入侵后3 h的人上皮细胞差异表达的新基因.
方法: 采用增毒的痢疾杆菌福氏2a 2457T侵袭HeLa细胞, 检查HeLa细胞中细菌的数量并用RT-PCR方法从HeLa细胞中提取总RNA, 制备探针. 采用含有约3000个代表新基因的芯片进行芯片杂交, 分析杂交结果. 将所获得的新的156条EST序列为种子序列用cDNA微阵列实验, 以人类EST数据库为基础库, 应用SiClone软件进行EST拼接、校对并尽可能延长获得大片段或全长cDNA. 选取基因组未注释的编码区序列作为新基因进行RT-PCR实验验证.
结果: 共鉴定到≥3倍或≤0.33的差异表达的代表新基因的EST序列45条, 其中25个延伸序列鉴定为与重要的肠道功能相关的已知基因, 并有3个在痢疾杆菌侵袭期间高表达的存在显著差异的新EST序列, 他们分别名为: NPCCKH12、ADBCSB02和HTBAMG05, 这3个基因是新的人基因或假基因.
结论: 鉴定出了在HeLa细胞感染痢疾杆菌前后差异表达的新EST序列, 为进一步研究痢疾杆菌与寄主相互作用奠定了基础.
引文著录: 黄留玉, 史兆兴, 袁静, 胡福泉. HeLa细胞感染痢疾杆菌前后差异表达的新EST序列的电子延伸及验证. 世界华人消化杂志 2007; 15(17): 1905-1913
Revised: April 1, 2007
Accepted: April 21, 2007
Published online: June 18, 2007
AIM: To investigate and test differential mRNA expression of new ESTs within HeLa epithelial cells following infection with Shigella flexneri 2457T.
METHODS: HeLa cells were incubated with S. flexneri 2a 2457T. A methylene blue assay was performed to examine the ratio of bacterial infection. Total RNA was extracted from HeLa cells and mRNA was isolated for use as probes. A cDNA microarray was assembled with about 3000 cDNA clones representing the same number of independent cDNA clusters, which were unknown-gene ESTs. Using 156 EST sequences obtained from cDNA microarray analysis as seed sequences, the Siclone software was applied for splicing, proofing, and extending EST sequences as long as possible. To validate the correctness of sequences after extension and to confirm the accuracy of the differential expression of genes from the microarray analysis, three new genes were selected and their transcription levels in HeLa cells were analyzed before and after Shigella infection using semi-quantitative reverse transcription-polymerase chain reaction (RT-PCR).
RESULTS: Forty-five ESTs were determined as being differentially expressed, with ≥ 3-fold or ≤ 0.33-fold changes, and 25 of these were identified as known genes involved in several important intestinal functions. Interestingly, three strikingly different cDNA fragments from unknown ESTs, name NPCCKH12, ADBCSB0 and HTBAMG05, were cloned by RT-PCR, sequenced and their expression levels were confirmed by semi-quantitative RT-PCR. We confirmed that they were new human or pseudo genes.
CONCLUSION: Three new ESTs from HeLa cells, differentially expressed after S. flexneri 2a infection, were identified. This investigative strategy is useful for obtaining information to be applied as a basis for further study of the interactions between Shigella and epithelial cells.
- Citation: Huang LY, Shi ZX, Yuan J, Hu FQ. Electronic extension and identification of new HeLa cell ESTs differentially expressed after Shigella flexneri 2a infection. Shijie Huaren Xiaohua Zazhi 2007; 15(17): 1905-1913
- URL: https://www.wjgnet.com/1009-3079/full/v15/i17/1905.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i17.1905
病原菌致病是在与真核宿主细胞相互作用过程中实现的. 痢疾杆菌在结肠上皮细胞中的繁殖和扩散是导致痢疾的重要环节之一[1]. 志贺氏菌感染引起痢疾发病机制为所有病原性痢疾杆菌都具有一个可以编码侵袭质粒抗原IpaACDB毒力大质粒[2], 通过Mxi-Spa-Ⅲ型分泌系统(TTSS)将细菌蛋白注射到肠道上皮细胞内[3], 通过胃肠屏障到达结肠, 侵入结肠上皮细胞开始增殖并向邻近细胞扩散, 最终引起结肠炎症、上皮细胞死亡、溃疡及液体的增多产生脓血及黏液等[4-5]. 肠上皮细胞作为痢疾杆菌的宿主细胞, 在受到痢疾杆菌的入侵之后为适应新的环境会诱发新的生存或抵御机制. 在该过程中, 基因表达将会发生明显的变化. 以前的研究主要集中于通过免疫染色推断寄主的形态学的变化[6-7], 但目前对痢疾杆菌侵袭上皮细胞后, 宿主细胞基因发生的变化及宿主的免疫保护机制还不十分清楚. 基因表达谱(转录组)是研究细胞表型和功能的一个重要工具. 与基因组不同, 表达谱是高度动态的, 细胞所处环境改变或受到病原微生物入侵时, 其形态和生理状况都会发生改变, 甚至当细胞处于复制、分裂等正常的生理活动时, 基因的转录情况也会变化很大, 而这些都是由于基因表达的不同引起的. 因此, 对这种基因表达差异的检测不仅会深入认识痢疾发生发展的机制, 也会为预防和治疗痢疾带来新的思路. 转录组学可能提供一些未知表达序列标签(EST)的信息, 但如果这些重要的序列太短, 那就很难把他们评判为差异基因. 尽管利用传统的方法可能可以研究这些EST序列, 通过从cDNA文库中筛选获得较长的序列, 然而, 这个方法是很复杂的而且仍处于试验阶段, 尤其是大量的信息获得应该通过采用高通量的技术. 在病原菌与寄主细胞相互作用的研究中经常会出现未知的EST序列, 同时大量的生物信息资源为未知EST序列的分析提供了便利的方法. GenBank 或 EMBL等公开的数据库中存在大量的EST序列, 他们许多序列相互重叠并可能代表了相同的cDNA序列, 因此短的EST序列可以利用生物软件被延伸. 根据这个原理, 通过序列图谱和基因克隆[6]可以设计电子PCR[8]. 我们采用新的策略快速鉴定未知EST序列进行定量转录组学分析, 用于研究上皮细胞和痢疾杆菌相互作用, 利用含有约3000个新基因的表达谱芯片检测了痢疾杆菌入侵诱导3 h的人上皮细胞新EST序列基因表达的差异, 对差异表达的全新EST序列进行了电子延伸克隆和相应的验证, 这些发现也为进一步进行寄主-病原菌相互作用的生化分析奠定了基础和框架.
大肠杆菌DH5α和野生型痢疾杆菌福氏2a毒株2457T均为本室保存. 实验前, 痢疾杆菌福氏2a 2457T用豚鼠角膜实验鉴定其毒性并进一步增毒. 人子宫上皮细胞株HeLa细胞为本室保存.
1.2.1 HeLa细胞侵袭实验[9]: 人子宫上皮细胞株HeLa细胞培养在含有100 mL/L FBS无支原体感染的RPMI 1640培养基中. 进行感染实验时, 将2457T福氏2a痢疾杆菌培养至指数生长期, 取约108CFU的菌用PBS洗涤3次后, 悬浮于不含血清的RPMI 1640培养液中, 用来侵袭约106个的HeLa细胞, 37℃作用3 h, PBS冲洗3次感染后的细胞, 加入新鲜培养基(含20 mg/L的庆大霉素)以杀死细胞外的细菌, 再培养3或24 h. 收集细胞并用RNA稳定试剂RNAlater(Qiagen公司产品)保存样品. 另外, 感染实验时进行染色, 用甲醇:冰醋酸(3:1)固定15 min, 美蓝染色10 min, 检测HeLa细胞中细菌的数量.
1.2.2 cDNA微阵列的构建: 含有约3000个代表新基因和对照基因的芯片克隆由上海中科开瑞公司提供. 利用通用引物PCR扩增所需片段, 并用琼脂糖凝胶电泳检测PCR扩增产物的质量. 将PCR产物异丙醇沉淀后溶于变性溶液(1.5 mol/L NaCl, 0.5 mol/L NaOH), 利用芯片制作仪(BiorRobotics, Cambridge, UK)将100 μL含PCR产物的溶液点到8 cm×12 cm的Hybond N尼龙膜上(Amersham Pharmacia, Buckinghamshire, UK), 每个点的直径约为0.4 mm, 每个cDNA片段在同一张膜的不同位置点2次. 以λDNA和pUC18 DNA作为阴性对照. 每张8 cm×12 cm的尼龙膜上包含8个看家基因(分别编码核糖体蛋白S9、β肌丝蛋白、3磷酸甘油脱氢酶、次黄嘌呤磷酸核糖转移酶、RPL13A蛋白、泛素C、磷脂酶A2和UCHL1蛋白)作为膜内对照基因, 每个基因均匀地点在每张膜的12个不同的位置. 如果杂交结果中某个基因的12个点的最大差异倍数大于1.5倍, 则认为杂交结果是无效的.
1.2.3 RNA的提取和检测: 采用标准的TRIzol RNA提取方法抽提对照组和实验组(包括感染后3和24 h)的HeLa细胞总RNA, 利用poly(dT)resin(Qiagen, Hilden, Germany)分离纯化mRNA, 并检测mRNA的质量和纯度, 使其纯度符合要求(A260/280>1.9). 总RNA质量检测(Lab on chip检测)操作步骤参考人oligo基因表达谱芯片使用手册, Lab-on-chip参照Agilent 2100分析仪操作手册, 制备凝胶, 按照软件提示进行RNA电泳操作, 评价RNA质量.
1.2.4 探针的制备: 取大约1-2 μg mRNA用于反转录探针制备和纯化, 以200 μCi的α-33P为标记物, 用MMLV反转录酶进行反转录, 取反转录反应产物的1/10进行PCR反应(94℃ 30 s, 55℃ 1 min, 72℃ 1.5 min, 35个循环).
1.2.5 杂交及洗涤: 芯片用20 mL预杂交液(6×SSC, 5 g/L SDS, 5×Denhardt和100 mg/L变性鲑精DNA)在68℃预杂交3 h, 随后用洗液(0.1×SSC, 5 g/L SDS)在65℃下严格洗涤1 h, 与33P标记的cDNA在6 mL杂交液(6×SSC, 5 g/L SDS和100 mg/L鲑精DNA)中68℃杂交过夜.
1.2.6 检测及分析: 将杂交好的芯片置于Phosphor Screen上过夜, 扫描用FLA-3000A Plate/Fluorescent Image Analyzer (Fuji Photo Film, Tokyo, Japan), 图像分析采用Array Gauge软件(Fuji Photo Film, Tokyo, Japan). 根据没有cDNA位置的背景灰度, 对芯片上cDNA点的灰度进行背景消减. 只有灰度值大于10的cDNA点才认为是有意义的信号, 足以保证与背景灰度有显著性差异(>99.9%).
1.2.7 电子延伸: 首先对人的EST数据库进行预处理, 去除载体等混杂序列, 得到干净的人类EST序列数据库. 然后以cDNA微阵列实验所获得的156条新的EST序列为种子序列, 以人类EST数据库为基础库, 应用Siclone软件进行EST拼接、校对并尽可能延长获得大片段或全长cDNA(称为Contig). 用Sim4软件将延长并校对的Contig序列与人基因组序列对准, 检验拼接的正确程度, 并可与基因组注释结果进行比较. 用Estscan软件, 预测没有注释的新的Contig序列的真正阅读框, 重构编码区序列, 并进行启动子等调控序列的预测.
1.2.8 RT-PCR分析: 选取基因组未注释的编码区序列作为新基因进行RT-PCR实验验证. 利用RNeasy Mini Kit(Qiagen, Hilden, Germany)提取细胞总RNA, 并用Oligo(dT)和MMLV反转录酶将约1 μg的总RNA反转录为单链cDNA. 在50 μL PCR扩增体系中加入1 μL cDNA作为模板, 以不同基因的序列设计引物进行PCR(94℃ 30 s, 52-60℃ 1 min, 72℃ 2-3 min, 30个循环). 得到的PCR产物纯化后连接到pMD18-T载体上测序, BLAST分析.
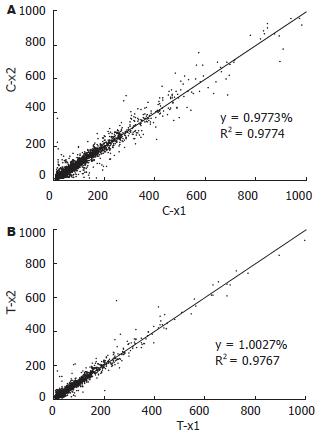
从人类cDNA文库中随机挑取的克隆经过末端测序和与Unigene数据库比对, 鉴定该DNA序列为代表未知基因的新EST序列. 代表约3000个新基因的cDNA克隆构成了cDNA微阵列体系. 为了评估cDNA微阵列体系的重复性好坏, 独立地进行了重复性实验. 制作不同批次的cDNA芯片, 用相同的mRNA样品制作的cDNA探针进行杂交, 并通过数据分析计算相关系数R2(用来表示基因表达模式的相似性), 经检测R2值约为0.97(图1), 表明在3000个基因中只有0.2%的基因两次杂交的结果差异大于2倍. 由此表明, 本实验构建的cDNA微阵列系统具有很高的可重复性.
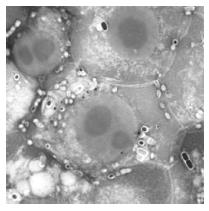
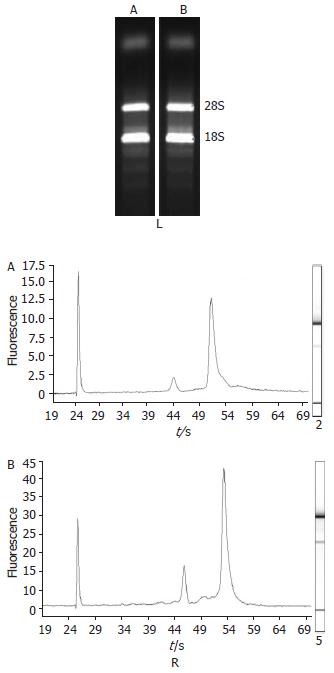
采用痢疾杆菌入侵HeLa细胞3 h后, 进行美蓝染色, 检测HeLa细胞中细菌的数量, 结果见图2, 镜检每个细胞内含有细菌数约10个, 表明痢疾杆菌已完全侵袭上皮细胞内. 我们对痢疾杆菌感染后3 h和未感染2个样品mRNA进行总RNA的质量控制, 见图3. 采用琼脂糖电泳和较高灵敏度的Lab-on-chip进一步对总RNA进行质量检测, 通过对图谱的分析发现没有明显的RNA降解(图3), 因此可以判定10个样品的总RNA完整性良好, 可以进行探针标记.
严格按照方法中描述的质量控制标准筛选cDNA阵列中差异表达基因, 部分扫描图的结果见图4. 对数据进行分析比较后得到有3倍以上差异的EST序列进行了测序(结果未显示). 痢疾杆菌感染3 h后与对照相比, 有11个代表新基因的EST序列表达上调, 最高倍数达到14.60, 只有2个EST序列表达下调, 差异最大为0.146.
为了能够获得差异表达新基因, 对代表新基因的EST序列进行了电子延伸, 其中痢疾杆菌感染3 h后的差异EST序列包含了3倍以上差异表达的代表新基因的EST序列45条, 对这些EST序列进行电子延伸, 延伸后的Contig序列长度由436-2993 bp. 对这些Contig序列与人基因组序列对准, 检验拼接的正确程度, 并可与基因组注释结果进行比较, 这些序列分布在多个染色体上, 在6号和17号染色体上聚集有较多的序列, 并且多数序列是在同一染色体上邻近的多个序列(2-9个)匹配, 提示这些序列可能是经过mRNA剪接得到的.
通过GenBank BLAST分析, 有25条序列与已知的基因或cDNA序列高度匹配, 20个序列是未注释的, 可能是这些基因的表达序列标签, 也可能是这些基因的不同的剪接体. 因为未注释序列可能是新基因, 我们对EST序列进行了结构域编码序列库搜索. 25个序列具有正确的开放阅读框, 分别是人DEAD(Asp-Glu-Ala-Asp)多肽3, SNRPN, CLCN5, 黏附因子B2, FOXJ3, RPS2, actinβ, cofilin, talin, gelsolin, cDNA FLJ46584 fis, cDNA DKFZp686N23124, TPD52, TPP2, C10orf9, ABCA8, COMMD3, COL4A5, Vof16, CLECSF2, Nedd4 结合蛋白Ⅰ, calpain5, 钙转运ATPase Ⅰ, MOBKL2B, unc-84同源体A. 鉴定到的差异表达的EST延伸序列主要的功能分类是与几个重要的肠道功能, 包括痢疾杆菌诱导的凋亡、细胞骨架、离子转运、离子通道功能、黏膜屏障防御、异型生物质代谢以及上皮细胞的保护机制. 与离子转运、离子通道和细胞凋亡相关基因大多表达量上调, 这可能是痢疾杆菌引起腹泻的原因.
鉴定到的差异基因在分类中大多属于细胞骨架相关蛋白, 如actin, talin, cofilin和gelsolin, 其次是离子转运蛋白如calpain-5, Nedd4结合蛋白Ⅰ和钙转运蛋白ATPaseⅠ. 在痢疾杆菌2457T侵袭反应中寄主上皮细胞最显著变化的是TPP Ⅱ, 表达量上调了7.01倍, 其参与了由痢疾杆菌2457T触发的细胞凋亡途径. 此外, 另一个重要的功能基因包括CLECSF2和Vof16也显示了重要的基因水平变化, 反应了上皮细胞的保护性机制.
用Estscan软件, 预测没有注释的新的Contig序列的真正阅读框, 重构编码区序列, 预测到存在正确阅读框的序列有3条序列, 分别命名为NPCCKH12、ADBCSB02和HTBAMG05. 然后对这3条序列进行RT-PCR的克隆验证分析, 经测序后得到的序列与电子延伸得到的序列相比稍有差异. 克隆NPCCKH12和克隆ADBCSB02没有已知的基因与之相匹配, 为新的基因序列; 克隆HTBAMG05的延伸序列与褐鼠局部缺血相关基因Vof16有90%的同源性.
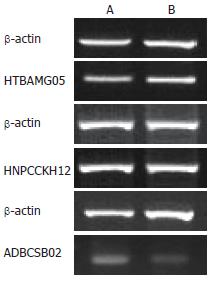
为了验证cDNA微阵列分析中3个延伸后的序列差异表达的准确性, 本研究进行了半定量RT-PCR的差异表达分析, 结果见图5. 克隆ADBCSB02(感染后下调6.25倍)和克隆HTBAMG05(感染后上调2.17倍)代表的EST序列在RT-PCR实验中反映出了在cDNA微阵列分析中的表达差异; 克隆NPCCKH12(感染后下调2.38倍)代表的EST序列差异表达在RT-PCR实验中不能明显地反映出来. 由此可知, cDNA微阵列分析的大部分数据是可信的.
在肠道内, 痢疾杆菌会引发急性感染而破坏组织, 细菌进一步侵袭胞质或形成膜结合复合物, 导致寄主免疫反应下降. 目前对痢疾病原学的研究焦点是痢疾杆菌与上皮细胞的相互作用[5]. 最近Pedron et al[9]利用affymetrix微阵列技术对S.flexneri 5a(M90T)侵袭上皮细胞的转录学反应进行了详细的研究, 证明了上皮细胞的前感染过程. 这些结论都是检测了已知基因的表达变化, 但对于未知基因的作用尚不清楚. 然而, 痢疾杆菌侵袭上皮细胞新的保护或反应机制可能和一些未知功能基因密切相关.
为了了解未知基因表达的改变, Eckmann et al[10]对沙门氏菌侵袭肠上皮细胞的约4300个EST序列进行微阵列分析, 他们发现几个细胞因子、激酶、转录因子和HLA Ⅰ表达上调, 认为微阵列技术是研究病原菌-寄主相互作用的有效方法[10]. 因此, 我们也应用了cDNA微阵列技术检测短的痢疾杆菌侵袭上皮细胞HeLa后新EST序列的差异表达, 并使用Siclone软件进行了电子延伸. 结果显示: (1)大约50% EST 延伸序列是和已知基因直接相关; (2)强烈暗示了上皮细胞对痢疾杆菌的侵袭具有新的反应机制. 通过cDNA阵列分析和EST序列的电子延伸, 具有差异表达的ESTs中约一半为已知基因, 参与了富含肌动蛋白actin支架、离子运输、细胞凋亡和其他重要途径.
最显著差异EST序列之一是编码tripeptidyl peptidaseⅡ(TPPⅡ)的基因, 其表达量上调了7.01倍. TPPⅡ是一个高分子量丝氨酸蛋白酶, 以同型寡聚体存在于细胞质或细胞膜中[9], 起蛋白质降解的作用. 例如, 在淋巴瘤细胞中, 采用蛋白酶体抑制剂, TPPⅡ替代了一些蛋白酶体的功能[11], TPPⅡ类蛋白水解系统发挥作用以补偿蛋白酶体的失活. 除了参与通常蛋白质降解的功能外, TPPⅡ在凋亡途径中也扮演了特殊的作用. 在由Shigella flexneri触发巨噬细胞凋亡的过程中, TPPⅡ参与到Caspase的上游, 促进了pro-caspase-1的成熟[12]. 同时, 在我们的实验中, TPPⅡ在痢疾杆菌侵袭细胞后表达量上调, 是否他也起到蛋白质降解或凋亡的作用还需要进一步研究. 如果TPPⅡ参与细胞凋亡, 将与凋亡调控蛋白密切相连, 如Caspase-类凋亡调控蛋白CLARP 和两个生长因子, EGF受体的配体amphiregulin 和TGF-α, 他们在痢疾感染上皮细胞Caco-2中被强烈诱导表达[9]. TPPⅡ如何参与痢疾杆菌诱导的凋亡目前尚不清楚. 已知痢疾杆菌侵袭素IpaB可以诱导巨噬细胞凋亡, 并直接结合和激活Caspase-1, 因此, TPPⅡ可能的作用是: (1)与IpaB和Caspase-1形成复合物, 直接促进Caspase的活性; (2)降解IpaB-caspase-1复合物引发细胞死亡的抑制子; (3)断裂作为细胞凋亡途径中一部分的Caspase-1底物. TPPⅡ抑制子是Ala-Ala-Phe-chloromethylketone(AAF-cmk)和clasto-lactacystin b-lactone(lactacystin), 也是在侵袭时保护细胞的蛋白酶体的抑制子. 在本实验中, 我们鉴定到一些蛋白酶体如calpain-5明显上调, 因此, 我们认为TPPⅡ可能通过降解AAF-cmk抑制子, 参与了痢疾杆菌诱导的凋亡途径, 对于确定TPPⅡ在凋亡中的作用以及其凋亡前底物的鉴定还需要更详细的研究.
痢疾杆菌利用寄主细胞骨架蛋白进入到细胞内. 在痢疾杆菌侵袭上皮细胞后, 本研究发现了几个与肌动蛋白鞭毛翻转相关基因, 他们的表达是被特殊的方式调控的, 这是以前未报道过的. Actin、talin、cofilin和gelsolin在感染后的表达水平提高了, 而支架黏附因子B2的表达水平下降. 在痢疾杆菌黏附到中国鼠卵细胞时, 细胞骨架蛋白talin连接了肌动蛋白微纤丝与细胞质膜, 结合integrins形成肌动蛋白的核心聚集到F-actin, 并与细胞运动中负调控蛋白vinculins结合[13]. 在本研究中, 我们鉴定到HeLa细胞的talin表达上调.
细胞骨架重排的连续, 对于HeLa细胞的趋向性和趋化性很重要[14]. Cofilin和gelsolin属于痢疾杆菌切断肌动蛋白的组分, 在痢疾杆菌感染细胞中过表达. 已知Cofilin在吞噬菌作用中[15], 与痢疾杆菌相互作用, 并提高Na+, K+-ATPase的活性, 促进离子转运作用[16], 可以通过控制肌动蛋白聚合因子/cofilin过程, 快速连接和分裂肌动蛋白微纤丝. 肌动蛋白微纤丝的分裂可以释放肌动蛋白单体和cofilin, 并且在cofilin去磷酸化后2种组分都可以结合到新的微纤丝中[17]. 在细胞骨架中肌动蛋白解聚物丰度的提高表明在寄主细胞和痢疾杆菌的能使具有解聚能力的蛋白失活的TTSS-效应子蛋白之间进行着复杂的分子竞争, 这与Philip et al[18]采用蛋白质组学方法分析人细胞对EPEC反应的结论是一致的.
鉴定到的另一个重要的EST序列是膜相关基因: ABCA8、UNC84A、MOBKL2B和SAFB2. ABCA8是一个新的异质生物质ATP-结合(ABC)转运子超家族的成员[19], ABC转运蛋白可以将各种分子转运到细胞外和细胞内膜[20], ABC基因被分为7个不同的亚家族, 包括: ABC1, MDR/TAP, MRP, ALD, OABP, GCN20, 和White. ABCA8属于ABC1亚家族, 广泛存在于多细胞真核生物中, 其功能目前仍未确定, 但推测ABC转运子可能与细胞对痢疾杆菌存在的环境条件的适应性有关[20]. 另一个膜相关基因-编码一个新的100 kD转膜蛋白UNC84A, 位于内部核膜上, 其基因序列与Caenorhabditis elegans Unc-84A高度相似[21], 他可能与核层相互作用[22]. MOBKL2B和SAFB2是2个细胞骨架相关蛋白, MOBKL2B蛋白与酵母菌Mob1蛋白具有相似性, Mob1是在极化纺锤体的复制和有丝分裂检测调控起关键作用的蛋白激酶[23]. 另一个基因SAFB2, 推测编码953-氨基酸的蛋白, 与支架黏附因子B1(SAFB1)具有75%的同源性. SAFB1和SAFB2包括一个高度保守的SAF框和RRM结构域, 他们都有一个核定位信号. 这些蛋白在许多细胞加工中发挥作用, 包括染色质形成、转录调控、RNA剪拼以及应激反应[24]. SAFB2通过与vinexin相互作用参与了细胞骨架的信号转导[25]. 我们认为, 在本实验中SAFB2的高表达与痢疾杆菌进入上皮细胞期间细胞骨架信号密切相关.
此外, 本研究还鉴定到calpain-5, Nedd4结合蛋白 I (Na+通道), 氯化物通道5的表达上调, 控制肿瘤蛋白D52和钙-转运(Calcium-transporting)ATPase Ⅰ的转录水平下调. 到目前为止, Shigella诱发腹泻的分子机制尚不清楚. 我们鉴定到寄主蛋白酶calpain-5显著上调, 可能破坏了细胞紧密结合的完整性, 并降低细菌穿过上皮的抗性. 当具有高Ca2+储存能力的细胞器如内质网(endoplosmic reticulum, ER)和线粒体, 由于应激或受到损伤时, 造成Ca2+外泄, 使细胞质中Ca2+浓度提高. 一些研究提出Shigella诱发的腹泻依赖于HCO3-分泌, 也就暗示了对钙流量的依赖[18]. HeLa细胞受痢疾杆菌侵袭后, 胞内Ca2+的浓度提高表明了Ca2+从线粒体中的泄露. 我们检测到钙激活的对粘液素调控[26]的氯化物通道5的表达量提高.
Nedd4结合蛋白I在被痢疾杆菌侵袭后表达上调. Nedd4是一个泛素连接酶, 可以靶向到上皮细胞的钠通道, 调控盐和液体的动态平衡, 并且通过Nedd4的WW结构域与离子通道相互作用[27]. 因此, 由蛋白酶体介导的特异离子转运子的降解很可能在痢疾杆菌引发的腹泻中起关键作用. 在ER上肿瘤蛋白D52P(TPD52)通过一个不同的基序结合Ca2+, ER中Ca2+的积累引起TPD52表达水平的降低. 我们发现质膜钙转运 ATPaseⅠ和肿瘤蛋白D52P下调, 可能也是引起发炎和腹泻的一个因素.
EST序列MDSDAH01鉴定为CLECSF2基因. 这个基因编码的蛋白是C-型血凝素/C-型血凝素类结构域(CTL/CTLD)超家族成员, 位于染色体12p13天然致死基因复合物区域[28]. 该家族成员具有一个共同的蛋白折叠和多种功能, 如细胞黏附, 细胞-细胞信号、糖蛋白翻转等, 普遍认为是参与对外源因子的防御机制, 识别健康和病原感染细胞, 对潜在的病原产生天然免疫反应[29]. 当淋巴细胞具有活性时, CLECSF2或AICL基因的表达水平是上调的. 编码2型转膜蛋白可能的功能是作为一个细胞被诱导激活的抗原[30-31], 该诱导激活抗原在上皮细胞内将引起新的免疫反应. 所以, CLECSF2基因在感染早期的上调预示他在上皮细胞的保护性机制中可能起重要的作用. 这种保护机制的现象在Caco-2对Shigella的转录组学研究中也观察到了[9]. 此外, 在细胞解毒作用中的基因, 如CYP异构体和乙醛脱氢酶(aldehyde dehydrogenase), 我们鉴定到他们的表达量上调. 我们的数据说明S. flexneri 2457T可能调节了一些重要的天然免疫蛋白的表达.
有趣的是, 对NPCCKH12、ADBCSB02和HTBAMG05 3个片段经测序后得到的序列与电子延伸得到的序列相比稍有差异. 克隆NPCCKH12和克隆ADBCSB02没有已知的基因与之相匹配, 为新的基因序列; 克隆HTBAMG05的延伸序列与褐鼠局部缺血相关基因Vof16有90%的同源性. 我们得到的这3个基因是新的人基因或假基因, 其相关信息已提交GenBank, 登录号为nos.UAY776160U, UAY776161U和UAY776162U. 我们认为他们在细胞对痢疾杆菌2457T侵袭反应中起重要的作用.
本研究为了大规模获得新的功能基因, 利用cDNA微阵列技术和电子延伸技术进行了尝试性研究. 从取得的结果可以看到, 我们不仅鉴定到了很多新基因, 也鉴定到了与已知基因高度同源的基因. 这一结果说明我们的尝试性研究思路获得了初步的成功, 为全面深入开展痢疾杆菌和上皮细胞之间相互作用的研究奠定了基础. 利用cDNA微阵列技术研究微生物和宿主之间的相互作用已经成为近期的研究热点之一[6-8]. 通过这样的研究, 可以得到大量的基因表达谱差异的信息. 这些信息既有已知基因, 也有未知基因. 对于已知基因的表达差异, 可以通过生物信息学手段(包括聚类分析、信号通路分析等)寻找总结规律. 但要对未知的EST序列进行深入的分析, 就需要先获得较长的序列. 通过筛选全长基因文库获得全长cDNA序列是早期的经典方法, 该方法的缺点是操作烦琐, 时间周期较长, 成功的几率也较低. 目前拥有大量EST序列的数据库已经建立起来, 从生物信息学的原理出发, 利用数据库中的EST序列进行电子延伸, 可以使很多基因片段得以延伸, 这样可以迅速地对未知基因的功能进行研究. 电子延伸技术已经在基因组研究方面发挥了重要作用. 这些高度同源基因可能是已知基因的不同剪接体, 也可能是新的基因, 这需要进一步的实验进行研究.
痢疾是全世界广泛流行并高发的一种急性肠道传染病, 其在结肠上皮细胞中的繁殖和扩散是导致痢疾的重要环节之一. 结肠上皮细胞作为痢疾杆菌的宿主细胞, 在受到痢疾杆菌的入侵之后为适应新的环境而会诱发产生新的生存或抵御机制. 但目前对痢疾杆菌侵袭上皮细胞后, 宿主细胞基因发生的变化及宿主的免疫保护机制还不十分清楚.
痢疾杆菌侵入肠上皮细胞引起细胞大量死亡是造成痢疾发生的关键环节. 因此, 要了解痢疾杆菌的致病机制, 必须在基因水平研究宿主细胞在与痢疾杆菌相互作用过程中的应答, 从而提高对痢疾杆菌致病机制的认识.
2003年, Pedronetal利用affymetrix微阵列技术对S. flexneri5a(M90T)侵袭上皮细胞的转录学反应进行了详细的研究[9], 证明了上皮细胞的前感染过程. 这些结论都是检测了已知基因的表达变化, 但对于未知基因的作用尚不清楚. 然而, 痢疾杆菌侵袭上皮细胞新的保护或反应机制可能和一些未知功能基因密切相关.
在本研究采用新的策略快速鉴定未知EST序列进行定量转录组学分析, 用于研究上皮细胞和痢疾杆菌相互作用, 利用含有约3000个新基因的表达谱芯片检测了痢疾杆菌入侵诱导3h的人上皮细胞新EST序列基因表达的差异, 对其中3个差异表达的全新EST序列进行了电子延伸克隆和相应的验证. 这些发现也为进一步进行寄主-病原菌相互作用的生化分析奠定了基础和框架.
研究结果为寻找上皮细胞新的生存或抵御通路提供了思路, 也为进一步进行寄主-病原菌相互作用的生化分析奠定了基础和框架.
本文实验设计严谨、方法先进, 实验证据较充实, 统计学数据可靠, 结果客观, 有一定的学术和实际应用价值.
编辑: 张海宁 电编:郭海丽
| 1. | Andrews GP, Hromockyj AE, Coker C, Maurelli AT. Two novel virulence loci, mxiA and mxiB, in Shigella flexneri 2a facilitate excretion of invasion plasmid antigens. Infect Immun. 1991;59:1997-2005. [PubMed] |
| 2. | Grassl GA, Finlay BB. Shigella rewrites host transcriptional responses. Nat Immunol. 2007;8:15-16. [PubMed] |
| 3. | Bach S, Makristathis A, Rotter M, Hirschl AM. Gene expression profiling in AGS cells stimulated with Helicobacter pylori isogenic strains (cagA positive or cagA negative). Infect Immun. 2002;70:988-992. [PubMed] |
| 4. | Ren Q, Robertson SJ, Howe D, Barrows LF, Heinzen RA. Comparative DNA microarray analysis of host cell transcriptional responses to infection by Coxiella burnetii or Chlamydia trachomatis. Ann N Y Acad Sci. 2003;990:701-713. [PubMed] |
| 5. | Hess S, Peters J, Bartling G, Rheinheimer C, Hegde P, Magid-Slav M, Tal-Singer R, Klos A. More than just innate immunity: comparative analysis of Chlamydophila pneumoniae and Chlamydia trachomatis effects on host-cell gene regulation. Cell Microbiol. 2003;5:785-795. [PubMed] |
| 6. | Tran Van Nhieu G, Bourdet-Sicard R, Dumenil G, Blocker A, Sansonetti PJ. Bacterial signals and cell responses during Shigella entry into epithelial cells. Cell Microbiol. 2000;2:187-193. [PubMed] |
| 7. | Zhang Y, Lai M, Gu X, Luo M, Shao L. Reg IV, a differentially expressed gene in colorectal adenoma. Chin Med J (Engl). 2003;116:918-922. [PubMed] |
| 8. | Schuler GD. Sequence mapping by electronic PCR. Genome Res. 1997;7:541-550. [PubMed] |
| 9. | Pedron T, Thibault C, Sansonetti PJ. The invasive phenotype of Shigella flexneri directs a distinct gene expression pattern in the human intestinal epithelial cell line Caco-2. J Biol Chem. 2003;278:33878-33886. [PubMed] |
| 10. | Eckmann L, Smith JR, Housley MP, Dwinell MB, Kagnoff MF. Analysis by high density cDNA arrays of altered gene expression in human intestinal epithelial cells in response to infection with the invasive enteric bacteria Salmonella. J Biol Chem. 2000;275:14084-14094. [PubMed] |
| 11. | Hilbi H, Jozsa E, Tomkinson B. Identification of the catalytic triad in tripeptidyl-peptidase II through site-directed mutagenesis. Biochim Biophys Acta. 2002;1601:149-154. [PubMed] |
| 12. | Geier E, Pfeifer G, Wilm M, Lucchiari-Hartz M, Baumeister W, Eichmann K, Niedermann G. A giant protease with potential to substitute for some functions of the proteasome. Science. 1999;283:978-981. [PubMed] |
| 13. | Hilbi H, Puro RJ, Zychlinsky A. Tripeptidyl peptidase II promotes maturation of caspase-1 in Shigella flexneri-induced macrophage apoptosis. Infect Immun. 2000;68:5502-5508. [PubMed] |
| 14. | Watarai M, Kamata Y, Kozaki S, Sasakawa C. rho, a small GTP-binding protein, is essential for Shigella invasion of epithelial cells. J Exp Med. 1997;185:281-292. [PubMed] |
| 15. | Melki R, Batelier G, Soulie S, Williams RC Jr. Cytoplasmic chaperonin containing TCP-1: structural and functional characterization. Biochemistry. 1997;36:5817-5826. [PubMed] |
| 16. | Bierne H, Gouin E, Roux P, Caroni P, Yin HL, Cossart P. A role for cofilin and LIM kinase in Listeria-induced phagocytosis. J Cell Biol. 2001;155:101-112. [PubMed] |
| 17. | Lee K, Jung J, Kim M, Guidotti G. Interaction of the alpha subunit of Na,K-ATPase with cofilin. Biochem J. 2001;353:377-385. [PubMed] |
| 18. | Chen H, Bernstein BW, Bamburg JR. Regulating actin-filament dynamics in vivo. Trends Biochem Sci. 2000;25:19-23. [PubMed] |
| 19. | Hardwidge PR, Rodriguez-Escudero I, Goode D, Donohoe S, Eng J, Goodlett DR, Aebersold R, Finlay BB. Proteomic analysis of the intestinal epithelial cell response to enteropathogenic Escherichia coli. J Biol Chem. 2004;279:20127-20136. [PubMed] |
| 20. | Tsuruoka S, Ishibashi K, Yamamoto H, Wakaumi M, Suzuki M, Schwartz GJ, Imai M, Fujimura A. Functional analysis of ABCA8, a new drug transporter. Biochem Biophys Res Commun. 2002;298:41-45. [PubMed] |
| 21. | Antal M, Bordeau V, Douchin V, Felden B. A small bacterial RNA regulates a putative ABC transporter. J Biol Chem. 2005;280:7901-7908. [PubMed] |
| 22. | Bray JD, Chennathukuzhi VM, Hecht NB. Identification and characterization of cDNAs encoding four novel proteins that interact with translin associated factor-X. Genomics. 2002;79:799-808. [PubMed] |
| 23. | Dreger M, Bengtsson L, Schoneberg T, Otto H, Hucho F. Nuclear envelope proteomics: novel integral membrane proteins of the inner nuclear membrane. Proc Natl Acad Sci U S A. 2001;98:11943-11948. [PubMed] |
| 24. | Ota T, Suzuki Y, Nishikawa T, Otsuki T, Sugiyama T, Irie R, Wakamatsu A, Hayashi K, Sato H, Nagai K. Complete sequencing and characterization of 21,243 full-length human cDNAs. Nat Genet. 2004;36:40-45. [PubMed] |
| 25. | Oesterreich S. Scaffold attachment factors SAFB1 and SAFB2: Innocent bystanders or critical players in breast tumorigenesis? J Cell Biochem. 2003;90:653-661. [PubMed] |
| 26. | Townson SM, Dobrzycka KM, Lee AV, Air M, Deng W, Kang K, Jiang S, Kioka N, Michaelis K, Oesterreich S. SAFB2, a new scaffold attachment factor homolog and estrogen receptor corepressor. J Biol Chem. 2003;278:20059-20068. [PubMed] |
| 27. | Bertrand CA, Danahay H, Poll CT, Laboisse C, Hopfer U, Bridges RJ. Niflumic acid inhibits ATP-stimulated exocytosis in a mucin-secreting epithelial cell line. Am J Physiol Cell Physiol. 2004;286:C247-255. [PubMed] |
| 28. | Henry PC, Kanelis V, O'Brien MC, Kim B, Gautschi I, Forman-Kay J, Schild L, Rotin D. Affinity and specificity of interactions between Nedd4 isoforms and the epithelial Na+ channel. J Biol Chem. 2003;278:20019-20028. [PubMed] |
| 29. | Renedo M, Arce I, Montgomery K, Roda-Navarro P, Lee E, Kucherlapati R, Fernandez-Ruiz E. A sequence-ready physical map of the region containing the human natural killer gene complex on chromosome 12p12.3-p13.2. Genomics. 2000;65:129-136. [PubMed] |
| 30. | Ebner S, Sharon N, Ben-Tal N. Evolutionary analysis reveals collective properties and specificity in the C-type lectin and lectin-like domain superfamily. Proteins. 2003;53:44-55. [PubMed] |
| 31. | Eichler W, Ruschpler P, Wobus M, Drossler K. Differentially induced expression of C-type lectins in activated lymphocytes. J Cell Biochem. 2001;81:201-208. [PubMed] |