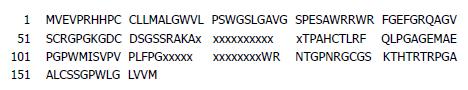修回日期: 2006-01-09
接受日期: 2006-01-14
在线出版日期: 2006-02-28
目的: 克隆HCV p7蛋白反式调节未知功能新基因p7TP2, 构建其真核表达载体, 并应用生物信息学初步探讨其结构及功能.
方法: 应用PCR技术从HepG2细胞提取的cDNA扩增p7TP2基因, 选用pGEM-T载体进行TA克隆, 通过PCR, 限制性酶切分析及测序进行鉴定, 再将其亚克隆到真核表达载体pcDNATM3.1/myc-His A, 通过PCR, 限制酶切分析进行鉴定, 并应用生物信息学初步分析其物理化学性质、蛋白质结构域、功能及染色体定位.
结果: 成功从HepG2细胞提取的cDNA中扩增出p7TP2基因, 编码区为495核苷酸(nt), 编码产物为164氨基酸残基(aa), 经核苷酸序列数据库(GenBank)同源序列的搜寻, 与已知基因序列和蛋白序列之间没有显著同源性, 属于未知功能新基因, 并成功进行TA克隆, 酶切、测序均正确, 并进一步亚克隆至pcDNATM3.1/myc-His A真核表达载体, 生物信息学分析此基因位于8q24.3,Mr17 146.5, 理论pI: 9.26, 半衰期为30 h(体外哺乳类网状细胞), 属于不稳定蛋白, 疏水指数较高, 含有潜在的三个螺旋区域, 四个β折叠, 四个蛋白激酶C磷酸化位点, 一个酪蛋白激酶Ⅱ磷酸化位点, 5个N-肉豆蔻酰位点, 预测可能为具有不紧密的球蛋白结构, 具有信号肽及两个跨膜结构域.
结论: 发现了HCV p7反式调节新的靶基因, 构建了pcDNATM3.1/myc-His A真核表达载体.
引文著录: 袁菊, 郭江, 成军, 陶明亮, 蓝贤勇, 洪源, 靳亚平, 毛羽. HCV p7蛋白反式调节基因p7TP2的克隆化及生物信息学分析. 世界华人消化杂志 2006; 14(6): 581-587
Revised: January 9, 2006
Accepted: January 14, 2006
Published online: February 28, 2006
AIM: To clone and identify a new gene p7TP2 transregulated by hepatitis C virus p7 protein, and construct its recombinant eukaryotic expression vector to explore its function and structure.
METHODS: p7TP2 gene was amplified by reverse chain polymerase chain reaction (RT-PCR) using HepG2 cDNA as template and inserted into the vector pGEM-T by TA cloning. Recombinant eukaryotic expression vector (pcDNATM3.1/myc-His A-p7TP2) was then constructed by subcloning technique which were all confirmed by restriction enzyme digestion analysis and sequencing. Bioinformatic methods were used to analyze its possible physical and chemical characters, structure, function and chromosome location.
RESULTS: p7TP2 gene was successfully amplified by RT-PCR, consisting of 495 nucleotides and coding a protein of 164 amino acid residues. After searching in GenBank, we found the new gene had no significant homology with the known genes. The DNA fragment was inserted into the vector pGEM-T by TA cloning, and the recombinant eukaryotic expression vector (pcDNATM3.1/myc-His A-p7TP2) was then constructed and confirmed correct after restriction enzyme digestion analysis and sequencing. Bioinformatic analysis showed that the new gene was located at chromosome 8q24.3 with Mr17 146.5 and theoretical pI 9.26. The estimated half-life period was 30 h (mammalian reticulocytes, in vitro). The protein was unstable with a high aliphatic index, consisting of 3 helix regions, 4 strand regions, 4 protein kinase C phosphorylation sites, 1 casein kinase Ⅱ phosphorylation site and 5 N-myristoylation sites. It was predicted that the new protein may be globular, but not as compact as a domain, containing signal peptide and two transmembrane regions.
CONCLUSION: A new gene is recognized as the new target transregulated by HCV p7 protein, and its recombinant eukaryotic expression vector (pcDNATM3.1/myc-His A-p7TP2) is successfully constructed.
- Citation: Yuan J, Guo J, Cheng J, Tao ML, Lan XY, Hong Y, Jin YP, Mao Y. Cloning and bioinformatics analysis of p7TP2 gene transregulated by hepatitis C virus p7 protein. Shijie Huaren Xiaohua Zazhi 2006; 14(6): 581-587
- URL: https://www.wjgnet.com/1009-3079/full/v14/i6/581.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i6.581
丙型病毒性肝炎是由丙型肝炎病毒(HCV)感染引起的世界性传染病[1-6]. 全世界约有1.7亿HCV感染者, 是HIV感染者的5倍. HCV感染后约70%转化为慢性并与肝硬化肝衰竭和肝癌的发生密切相关. HCV的基因组含有一个长的开放阅读框架(ORF), 编码一个约3 010个氨基酸残基(aa)组成的多蛋白, 该多蛋白被细胞和病毒蛋白酶切割产生至少10个病毒基因产物[7]. p7是由HCV基因组2 580-2 768核苷酸(nt)之间的基因编码的由63个aa组成的一个小蛋白, 关于其功能研究较少. 为了研究p7蛋白对于宿主细胞基因的上调、下调作用, 郭江 et al应用基因表达谱芯片技术研究p7蛋白对于宿主细胞基因的上调、下调作用, 同时进行了系统的筛选研究, 发现并鉴定了一系列p7蛋白反式调节的靶基因, 其中包括一些未知功能的新基因. 为探讨其生物学功能及其作用, 我们构建其真核表达载体, 并对其生物学功能进行预测分析, 为今后更加广泛深入地研究新基因的生物学功能打下基础, 并为研究HCV p7的反式调节作用、丙型肝炎发病机制创造了条件.
肝母细胞瘤细胞系HepG2及感受态大肠杆菌DH5α为本室保存, Taq酶(鼎国生物), T4 DNA连接酶、pGEM-T, IPTG及X-α-gal购于Promega公司, 玻璃奶回收试剂盒购自博大泰克公司, 引物合成及DNA序列测定均由Invitrogen公司完成, EcoRⅠ,BamHⅠ购自TaKaRa生物公司, pcDNATM3.1/myc-His A由北大人民医院魏来教授惠赠.
郭江 et al利用基因芯片技术构建HCV p7蛋白反式调节基因, 挑选克隆测序, 与GenBank数据库进行初步比较, 发现一个新基因, 根据基因起始密码子的Kozak规则和终止密码子下游保守的多聚腺苷酸信号序列, 电子拼接推定该基因的开放读码框架获得相应的全长编码基因, 将该未知功能基因命名为p7TP2, 发现其开放读码框长495 bp, 编码164个氨基酸残基.
根据p7TP2的全长编码基因, 设计2条引物, 有义链引物5'-GAA TTC CA ATG GTG GAG GTG CCC AGG-3', 引入EcoRⅠ酶切位点, 反义链引物5'-GGA TCC CAT GAC GAC CAG GCC CAG-3', 引入BamHⅠ酶切位点, 提取HepG2细胞的总RNA, 进行反转录, 以反转录产物cDNA为模板进行PCR, PCR参数如下: 95 ℃ 5 min预变性, 94 ℃ 30 s变性, 72 ℃ 1 min退火及延伸, 共35个循环, 72 ℃延伸10 min. 将PCR产物在10 g/L琼脂糖凝胶中电泳, 切取目的片段, 玻璃奶法回收PCR产物, 与pGEM-T载体连接, 转化DH5α感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养12-16 h. 挑取阳性菌落, 增菌. 提取质粒进行限制性酶切分析鉴定, 选择经鉴定的克隆送测序. 挑取上述白色菌落扩大培养, 碱裂解法提取质粒, 选择酶切鉴定阳性克隆, 并回收纯化目的p7TP2片段, 同时将pcDNATM3.1/myc-His A真核表达载体进行EcoRⅠ, BamHⅠ双酶切反应并回收纯化, 以T4连接酶构建pcDNATM3.1/myc-His A-p7TP2重组载体, 按上述方法常规转化感受态细菌, 提取质粒, 作EcoRⅠ, BamHⅠ双酶切鉴定.
将该基因与NCBI进行BLASTN比对分析, 确定其染色体定位, 应用ProtParam工具(http://www.expasy.ch/tools/protparam.html)对p7TP2蛋白质一级结构的生物信息学预测, 包括蛋白质的相对分子量、理论pI值、消光系数、半衰期、不稳定系数以及总平均亲水性等, 应用nnpredict(http://www.cmpharm.ucsf.edu/-nomi/nnpredict.html)预测氨基酸序列中各个氨基酸对应的二级结构的程序. 应用PredictProtein(http://www.embl-heidelberg.de/predictprotein/predictprotein.html)对蛋白质结构进行分析, 应用ProtScale(http://www.expasy.org/tools/protscale.html)做蛋白质的疏水性分析, 应用SignalP-信号肽预测工具(http://www.cbs.dtu.dk/services/SignalP/)对其进行蛋白质结构信号肽分析, 有助于蛋白质功能域的区分及蛋白质细胞定位. 应用Tmpred-跨膜结构分析(http://www.ch.embnet.org/software/TMPRED_form.html), 对于正确认识蛋白质结构、甚至功能具有重要的意义, COILS(http://www.ch.embnet.org/software/COILS_form.html)预测蛋白质中卷曲螺旋结构.
新基因开放读码框长495 bp, 编码164个氨基酸残基(图1), 该序列被美国核苷酸序列数据库GenBank收录, 收录号码为AY819648.
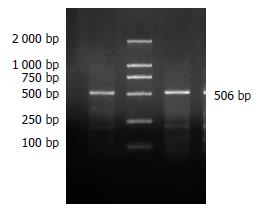
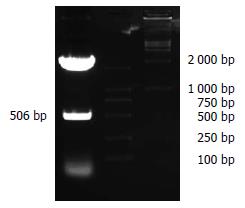
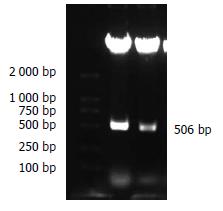
以HepG2细胞cDNA文库为模板, PCR反应后经10 g/L琼脂糖凝胶电泳, 可见长度为506 bp左右的电泳条带(图2). 利用pGEM-T vector SystemⅠ将纯化PCR产物进行TA克隆, 经EcoRⅠ, BamHⅠ双酶切得到两条带(图3), 然后送测序验证是正确的克隆, 命名为pGEM-T-p7TP2, 进一步将pcDNATM3.1/myc-His A和pGEM-T-p7TP2作EcoRⅠ, BamHⅠ双酶切, 回收目的片段, 构建重组真核表达载体pcDNATM3.1/myc-His A-p7TP2, 经酶切鉴定重组载体在506 bp左右见1条带(图4), 证明pcDNATM3.1/myc-His A-p7TP2构建成功.
将基因测序结果与NCBI进行BLASTN比对分析, p7TP2基因位于人染色体8q24.3, ProtParam分析Mr17 146.5, 理论pI: 9.26, 消光系数38 235 M-1 cm-1(276 nm); 39 581 M-1 cm-1(278 nm); 39 980 M-1 cm-1 (279 nm); 40 190 M-1 cm-1(280 nm); 39 560 M-1 cm-1(282 nm)是在6.0 mol/L盐酸胍, 0.02 mol/L磷酸盐缓冲液, pH 6.5以半胱氨酸来计算, 半衰期为30 h(体外哺乳类网状细胞), 大于20 h(酵母体内), 大于10 h(埃希菌属大肠杆菌体内), 不稳定系数是54.82, 一般认为该蛋白是不稳定的, 疏水性指数是60.12, 总平均亲水性-0.243. nnpredict预测的二级结构为----------HHHH---E---------------HHHHH---------------------------HHHH----------------------------------EE---------------------------------------------E-------EEE--
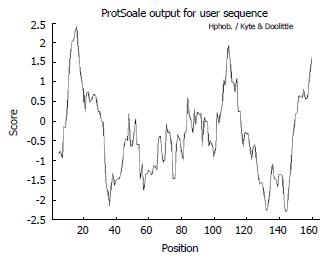
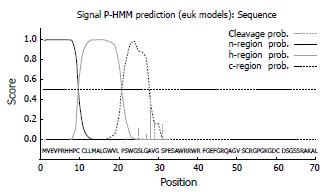
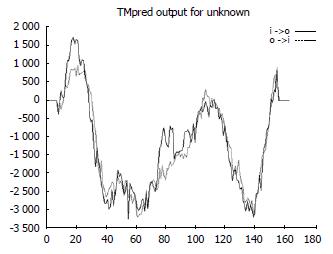
(H表示螺旋, E表示β折叠, 用"-"表示转角结构), PredictProtein预测包含潜在的蛋白激酶C磷酸化位点(51/SCR, 64/SSR, 87/TLR, 128/TWR),酪蛋白激酶Ⅱ磷酸化位点(116/SAAE), N-肉豆蔻痫位点(24/GSLGAV, 30/GSPESA, 63/GSSRAK, 76/GQPQGL, 149/GAALCS), 含2个低复杂性区域结构(图5), 对序列进行PROF预测, 预测包含提交的蛋白质的二级结构含量同疾病分类、蛋白质亲水表面统计、残基组成、预测的二级结构等. OBS-sec表示二级结构的观测值; PROF-sec表示预测的二级结构; Rel-sec为预测二级结构的可信度, 数值越大表示越可信; SUB-secPROF表示预测的二级结构的集合. 所有区域平均精确度大于82%, H为螺旋; E为折叠; L为随机卷曲; O-3-acc表示相对亲水表面的观测值, 共有3种状态, b = 0%-9%, i = 9%-36%, e = 36%-100%. P-3-acc表示预测的相对亲水表面. Rel-acc表示预测亲水表面的可信度. SUB-acc表示预测的亲水表面的集合(图6), 并预测可能为球蛋白, 但结构并不是特别紧密, 蛋白质疏水性预测最大值是2.422, 最小值是-2.289, 在8-32, 100-118, 150-160区域的氨基酸具有很强的疏水性, 其次是84-95的区域具有一定的疏水性(图7). 用SignalP-信号肽预测工具预测该蛋白信号肽概率0.991, 锚定蛋白概率0.005, 最大分隔位点概率是0.471, 位于27与28氨基酸之间(图8). 对其进行跨膜结构的分析包括两个强跨膜螺旋(从11-27, 跨膜方向由内向外; 148-167, 跨膜方向由外向内)(图9), 无明显的卷曲螺旋结构.
HCV p7是一个小的疏水性多蛋白, 编码基因位于结构蛋白和非结构蛋白之间, 可在黑脂质膜中形成六聚体阳离子通道, 推测其功能与病毒离子孔道蛋白(viroporin)类似, 可使细胞内膜结构不稳定化, 以利于成熟的病毒颗粒释放[8-9], 这一离子通道可被金刚烷胺和长烷基链的亚氨基糖衍生物抑制[10-12], p7对病毒复制并不是关键的, 但对感染性病毒的产生是必不可少的. p7仍有许多功能没有研究清楚, 对其反式调节作用的研究更少[13]. 为进一步研究p7的功能, 明确p7在丙型肝炎发病机制中的作用, 郭江 et al[14]曾利用抑制性消减杂交技术(suppression subtractive hybridization, SSH)和基因芯片技术筛选HCV p7反式激活的靶基因, 推测其在体内可能存在的功能线索. 基因表达谱芯片技术是近年来新发展起来的一项研究差异基因表达的分子生物学前沿技术, 通过将大量的基因特异的探针火气cDNA片段固定在一块固相载体上, 与待测样品杂交后用激光共聚焦荧光检测系统进行结果扫描、监测, 对来源不同的个体、组织、发育阶段、分化阶段、病变及刺激条件下的细胞内的mRNA或cDNA进行分析, 获得大量有价值的生命信息[15-16], 并可以发现未知基因.
确定一个新基因的编码序列, 研究该基因的表达与调控, 编码蛋白的结构与功能, 确定新基因的生物学和医学意义, 是医学分子生物学研究领域中一项具有挑战性的工作[17]. 我们发现了p7TP2基因, 这是一种未知功能的新基因, 对于这一新基因结构与功能的研究目前还是空白, p7TP2基因开放读码框长495 bp, 编码164个氨基酸残基, 该序列被美国核苷酸序列数据库GenBank收录, 收录号码为AY819648, 将基因测序结果与NCBI进行BLASTN比对分析, p7TP2基因位于人染色体8q24.3, ProtParam分析Mr 17 146.5, 理论pI: 9.26, 半衰期为30 h(体外哺乳类网状细胞), 大于20 h(酵母体内), 大于10 h(埃希菌属大肠杆菌体内), 不稳定系数是54.82, 属于不稳定蛋白, 疏水指数较高60.12, 总平均亲水性-0.243, 在8-32, 100-118, 150-160区域的氨基酸具有很强的疏水性, 含有三个螺旋区域, 四个β折叠, 四个潜在的蛋白激酶C磷酸化位点(51/SCR, 64/SSR, 87/TLR, 128/TWR), 一个酪蛋白激酶Ⅱ磷酸化位点(116/SAAE), 5个N-肉豆蔻酰位点(24/GSLGAV, 30/GSPESA, 63/GSSRAK, 76/GQPQGL, 149/GAALCS), 其中蛋白激酶C磷酸化位点与介导胞外分泌有关[18], 酪蛋白激酶Ⅱ磷酸化位点与调节细胞凋亡和细胞周期有关[19], N-豆蔻酰化位点可能定位于内质网或线粒体外膜[20]. 用SignalP-信号肽预测工具预测该蛋白信号肽概率0.991, 锚定蛋白概率0.005, 对其进行跨膜结构的分析包括两个强跨膜螺旋(从11-27, 跨膜方向由内向外; 148-167, 跨膜方向由外向内). 本实验将该基因构建到真核表达载体pcDNATM3.1/myc-His A, 为新基因的更一步研究奠定基础, 同时也为研究HCV p7蛋白的生物学功能, 阐明HCV的致病机制提供了新的方向[21].
生物信息学技术伴随计算机网络的发展而产生, 其强大的功能使其已经成为生命科学研究的重要手段[22-26], 一方面基因和蛋白质序列数据库的不断累积和计算机技术的进展催生了生物信息学这一崭新学科的诞生, 同时生物信息学技术对于经典的分子生物学技术具有重要的推动作用. 本项研究, 首先应用基因芯片技术, 筛选克隆了人的p7TP2编码基因, 随后利用核苷酸数据库的生物信息学分析技术, 确定了基因序列和蛋白质一级结构序列. 不仅如此, 利用生物信息学技术, 还确定了该基因的二级结构和功能域及染色体定位, 为进一步研究带该基因的结构和功能、表达与调控, 生物学意义和医学意义奠定了坚实的基础.
丙型病毒性肝炎是由丙型肝炎病毒(HCV)感染引起的世界性传染病. 全世界约有1.7亿HCV感染者是HIV-1感染者的5倍, 但目前还没有特效的治疗技术和方法, HCV p7是一个小的疏水性多蛋白, 编码基因位于结构蛋白和非结构蛋白之间, 可在黑脂质膜中形成六聚体阳离子通道, 推测其功能与病毒离子孔道蛋白(viroporin)类似, 可使细胞内膜结构不稳定化, 以利于成熟的病毒颗粒释放, p7对病毒复制并不是关键的, 但对感染性病毒的产生是必不可少的.
对p7蛋白的生物学功能有所研究, 但其反式调节机制尚不是很明确. p7TP2是利用基因芯片技术筛选得到的p7蛋白反式调节的靶基因, 其结构和功能的研究均未有报道. 对其进一步的研究期望能在HCV的发病机制方面有所突破.
1 Ramesh Prabhu, Nutan Khalap, Roberto Burioni, Massimo Clementi, Robert F. Garry, and Srikanta Dash. Inhibition of Hepatitis C Virus Nonstructural Protein, Helicase Activity, and Viral Replication by a Recombinant Human Antibody Clone. Am J Pathol 2004 165: 1163-1173
2 Jae-Hwan Nam, Kristina Faulk, Ronald E. Engle, Sugantha Govindarajan, Marisa St. Claire, and Jens Bukh. In Vivo Analysis of the 3' Untranslated Region of GB Virus B after In Vitro Mutagenesis of an Infectious cDNA Clone: Persistent Infection in a Transfected Tamarin. J. Virol 2004 78: 9389-9399
本文利用PCR技术克隆了p7TP2的全基因序列, 并利用生物信息学技术分析p7TP2基因组序列、编码蛋白的结构、蛋白质功能结构单位等.
新基因p7TP2的克隆和功能研究, 为今后更加广泛深入地研究新基因的生物学功能打下基础, 并为研究HCV p7的反式调节作用、丙型肝炎发病机制创造了条件.
反式调节: 基因表达的调节方式分为顺式调节和反式调节, 分子结构内部的基因表达调节元件的作用形式称为顺式调节, 而分子间的基因表达调节元件的作用形式称为反式调节.
此课题是国家重点基础研究项目, 是HCV肝炎病毒基因, 克隆、生物信息学功能的基础性研究为阐明HCV的致病机制提供了新的方向. 生物信息学技术是生命科学研究的重要手段, 利用生物信息学技术为进一步研究该基因的结构和功能、表达与调控、生物学意义和医学意义结合奠定了坚实的基础.
电编: 张敏 编辑:潘伯荣
| 1. | 邵 清, 成 军, 白 雪帆, 王 琳, 张 健, 梁 耀东, 刘 敏, 李 强. 酵母双杂交技术筛选白细胞中HCV NS3蛋白结合蛋白基因. 世界华人消化杂志. 2003;11:1897-1900. [DOI] |
| 3. | 郝 春秋, 冯 志华, 周 永兴, 聂 青和, 李 谨革, 贾 战生, 梁 雪松, 谢 玉梅, 曹 义战, 康 文臻. 复制缺陷型HCV C基因腺病毒表达载体的构建包装及鉴定. 世界华人消化杂志. 2003;11:144-147. [DOI] |
| 4. | 梁 雪松, 连 建奇, 周 永兴, 聂 青和, 郝 春秋. IRES特异性IRNA对HCV IRES启动蛋白翻译细胞内抑制作用. 世界华人消化杂志. 2003;11:157-160. [DOI] |
| 5. | 孙 利, 周 永兴, 郝 春秋, 冯 志华, 赵 君, 胡 沛臻, 付 勇, 马 福成, 常 吉庆, 王 九平. DNA疫苗对小鼠HCV-C皮下移植瘤的防治. 世界华人消化杂志. 2003;11:165-168. [DOI] |
| 6. | 王 建军, 成 军, 刘 妍, 杨 倩, 纪 冬, 党 晓燕, 王 春花. 丙型肝炎病毒核心蛋白反式激活基因TAHCCP2的克隆. 世界华人消化杂志. 2003;11:1893-1896. [DOI] |
| 7. | Lo SY, Selby M, Tong M, Ou JH. Comparative studies of the core gene products of two different hepatitis C virus isolates: two alternative forms determined by a single amino acid substitution. Virology. 1994;199:124-131. [PubMed] |
| 8. | Griffin SD, Beales LP, Clarke DS, Worsfold O, Evans SD, Jaeger J, Harris MP, Rowlands DJ. The p7 protein of hepatitis C virus forms an ion channel that is blocked by the antiviral drug, Amantadine. FEBS Lett. 2003;535:34-38. [PubMed] |
| 9. | Pavlovic D, Neville DC, Argaud O, Blumberg B, Dwek RA, Fischer WB, Zitzmann N. The hepatitis C virus p7 protein forms an ion channel that is inhibited by long-alkyl-chain iminosugar deriva-tives. Proc Natl Acad Sci USA. 2003;100:6104-6108. [PubMed] |
| 10. | Friebe P, Bartenschlager R. Genetic analysis of sequences in the 3' nontranslated region of hepatitis C virus that are important for RNA replication. J Virol. 2002;76:5326-5338. [PubMed] |
| 11. | Griffin SD, Harvey R, Clarke DS, Barclay WS, Harris M, Rowlands DJ. A conserved basic loop in hepatitis C virus p7 protein is required for aman-tadine-sensitive ion channel activity in mamma-lian cells but is dispensable for localization to mitoch-ondria. J Gen Virol. 2004;85:451-461. [PubMed] |
| 12. | Branza-Nichita N, Durantel D, Carrouee-Durantel S, Dwek RA, Zitzmann N. Antiviral effect of N-butyldeoxynojirimycin against bovine viral diarrhea virus correlates with misfolding of E2 envelope proteins and impairment of their association into E1-E2 heterodimers. J Virol. 2001;75:3527-3536. [PubMed] |
| 13. | Sakai A, Claire MS, Faulk K, Govindarajan S, Emer-son SU, Purcell RH, Bukh J. The p7 poly-peptide of hepatitis C virus is critical for infectivity and contains functionally important genotype-specific sequences. Proc Natl Acad Sci USA. 2003;100:11646-11651. [PubMed] |
| 14. | 郭 江, 成 军, 纪 冬, 赵 龙凤, 王 建军, 刘 妍, 黄 燕萍. 应用抑制性消减杂交技术克隆和筛选HCVp7蛋白的反式调节基因. 世界华人消化杂志. 2004;12:2590-2593. [DOI] |
| 15. | Yang GP, Ross DT, Kuang WW, Brown PO, Weigel RJ. Combining SSH and cDNA microarrays for rapid identification of differentially expressed genes. Nucleic Acids Res. 1999;27:1517-1523. [PubMed] |
| 16. | Golub TR, Slonim DK, Tamayo P, Huard C, Gaasen-beek M, Mesirov JP, Coller H, Loh ML, Downing JR, Caligiuri MA. Molecular classification of cancer: class discovery and class prediction by gene expression monitoring. Science. 1999;286:531-537. [PubMed] |
| 18. | Morgan A, Burgoyne RD, Barclay JW, Craig TJ, Prescott GR, Ciufo LF, Evans GJ, Graham ME. Reg-ulation of exocytosis by protein kinase C. Biochem Soc Trans. 2005;33:1341-1344. [PubMed] |
| 19. | Yamane K, Kinsella TJ. Casein kinase 2 regulates both apoptosis and the cell cycle following DNA damage induced by 6-thioguanine. Clin Cancer Res. 2005;11:2355-2363. [PubMed] |
| 20. | Colombo S, Longhi R, Alcaro S, Ortuso F, Sprocati T, Flora A, Borgese N. N-myristoylation determines dual targeting of mammalian NADH-cytochrome b5 reductase to ER and mitochondrial outer mem-branes by a mechanism of kinetic partitioning. J Cell Biol. 2005;168:735-745. [PubMed] |
| 22. | Li X, Zhang Y. Bioinformatics data distribution and integration via Web Services and XML. Genomics Proteomics Bioinformatics. 2003;1:299-303. [PubMed] |
| 23. | Friedhoff P. Mapping protein-protein interactions by bioinformatics and cross-linking. Anal Bioanal Chem. 2005;381:78-80. [PubMed] |
| 24. | Pazos F, Guijas D, Valencia A, De Lorenzo V. Meta-Router: bioinformatics for bioremediation. Nucleic Acids Res. 2005;33:D588-D592. [PubMed] |
| 25. | Cattley S. A review of bioinformatics degrees in Australia. Brief Bioinform. 2004;5:350-354. [PubMed] |
| 26. | Cooper MS, Sommers-Herivel G, Poage CT, McCar-thy MB, Crawford BD, Phillips C. The Zebrafish DVD Exchange Project: a bioinformatics initiative. Methods Cell Biol. 2004;77:439-457. [PubMed] |