修回日期: 2005-11-30
接受日期: 2005-12-10
在线出版日期: 2006-02-18
目的: 观察靶向siRNA能否抑幽门螺杆菌(H. pylori )vacA基因表达.
方法: 合成靶向 H. pylori vacA基因5对特异siRNA(实验组: vacA-s1, vacA-s2, vacA-s3, vacA-s4, vacA-s5)和1对非特异siRNA(对照组). 通过电穿孔法使siRNA转化至H. pylori内, 观察转化效率和转化1, 6, 12, 24, 48 h mRNA和蛋白表达的抑制率, 并将PCR产物克隆和测序 .
结果: 电穿孔转化效率平均为89%. vacA-s2、vacA-s4组在转化1 h vacA mRNA表达抑制率达最大值, 1, 6, 12 h抑制率分别为65%和77%, 43%和50%, 17%和9%, 24和48 h时vacA mRNA表达无抑制效应, 与vacA-s1、vacA-s3、vacA-s5、vacA-s6组相比有显著差异(P<0.05), 因为vacA-s1、vacA-s3、vacA-s5、vacA-s6组转化后各时间点mRNA表达无变化. vacA-s2和vacA-s4组在转化1, 6, 12, 24 h时vacA蛋白表达抑制率分别为26%和17%, 47%和40%, 70%和75%, 33%和30%, 与vacA-s1、vacA-s3、vacA-s5、vacA-s6组相比有显著差异(P<0.05); 转化48 h则无抑制效应. PCR产物克隆和测序与相应报道序列比较, 同源性为99%.
结论: 靶向siRNA可以通过电穿孔法转化并特异地抑制H. pylori vacA基因表达.
引文著录: 赵刚, 詹文华, 严燕国, 马晋平, 彭俊生, 董文广, 蔡世荣, 何裕隆. 靶向siRNA抑制幽门螺杆菌vacA表达. 世界华人消化杂志 2006; 14(5): 470-475
Revised: November 30, 2005
Accepted: December 10, 2005
Published online: February 18, 2006
AIM: To investigate whether RNA interference (RNAi) induced by small interfering RNA (siRNA) can suppress the expression of vacA in Helicobacter pylori.
METHODS: Five pairs of siRNA targeted vacA, named vacA-s1, vacA-s2, vacA-s3, vacA-s4, and vacA-s5, and one pair non-specific siRNA were synthesized and then transferred into H. pylori using electroporation method. Before and 1, 6, 12, 24, and 48 h after electroporation, the expression of vacA gene and protein were detected by reverse transcription polymerase chain reaction (RT-PCR) and Western blot, respectively. PCR products were also cloned and sequenced.
RESULTS: siRNA was transformed into H. pylori with an average efficiency of 89% by electroporation. The inhibitory rates of vacA mRNA expression in vacA-s2 and vacA-s4 transferred H. pylori were 65% and 77% at 1 h, 43% and 50% at 6 h, and 17% and 9% at 12 h, respectively, which were significantly higher than those in vacA-s1, vacA-s3, vacA-s5, and vacA-s6 transferred ones (P < 0.05). No inhibition of vacA mRNA was observed in vacA-s2 and vacA-s4 transferred H. pylori at 24 and 48 h. The expression of vacA mRNA had no changes at all time points in vacA-s1, vacA-s3, vacA-s5, and vacA-s6 transferred bacteria. The inhibitory rates of vacA protein in vacA-s2 and vacA-s4 transferred H. pylori were 26% and 17% at 1 h, 47% and 40% at 6 h, 70% and 75% at 12 h, and 33% and 30% at 24 h, respectively, which were markedly higher than those in vacA-s1, vacA-s3, vacA-s5, and vacA-s6 transferred ones (P< 0.05). There was no inhibition of vacA protein 48 h after electroporation. After the PCR products were sequenced, we found them 99% homologous to the reported sequence.
CONCLUSION: It is feasible to suppress the expression of vacA in H. pylori by targeting siRNA using electroporation.
- Citation: Zhao G, Zhan WH, Yan YG, Ma JP, Peng JS, Dong WG, Cai SR, He YL. Small interfering RNA targeted inhibition of vacA expression in Helicobacter pylori. Shijie Huaren Xiaohua Zazhi 2006; 14(5): 470-475
- URL: https://www.wjgnet.com/1009-3079/full/v14/i5/470.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i5.470
目前已发现H. pylori的毒力因子有多种, 如Cag致病岛[1,2]、尿素酶(ureB)[3,4]、空泡毒素A(vacA)[5,6]等, 其中能使胃上皮细胞空泡变性的vacA是该菌重要的致病因子之一. RNA干扰(RNA interference, RNAi)技术是利用双链RNA (double stranded RNA)诱导的序列特异的转录后基因沉默(posttranscriptional gene silencing, PTGS)[7], 他是由19-21个核苷酸大小的小分子干扰RNA(small interfering RNA, siRNA)引起的. RNAi现象广泛存在于多种生物体内[8-10], RNAi在基因功能研究、基因调控研究和基因治疗等领域展示了巨大的应用前景[11-15]. 本研究依据RNAi原理, 以H. pylori的vacA基因为靶点, 用vacA高表达的国际标准H. pylori NCTC11637菌株为研究对象, 设计、筛选针对vacA基因的 siRNA片段, 观察靶向siRNA抑制H. pylori vacA基因表达.
国际标准H. pylori NCTC11637(cagA+/ vacA+)菌株购自中国疾病预防控制中心, 空肠弯曲菌培养基溶于水后加新鲜脱纤维羊血(5%), 混匀铺在无菌培养皿上, 冷却凝固后接种H. pylorimL/L O 置于37 ℃ 1002mL/L CO , 502mL/L , 850 N2mL/L CO mL/L胎牛血清的DMEM高糖培养液中, 37 ℃ 50 d. 胃黏膜上皮细胞株(GES-1)购自北京肿瘤研究所, GES-1培养于100 微厌氧培养箱内培养32恒温培养. 根据siRNA基本设计原则, 利用siRNA序列搜索软件, 由上海吉玛公司设计并化学合成针对H. pylori NCTC11637菌株的vacA基因(AJ239564)的5对特异siRNA和1对非特异siRNA. 5对特异siRNA序列为: vacA-s1 5'-GCUAGGCAUUAUUGGGUCATT-3', 5'-UGACCCAAUAAUGCCUAGCTT-3', vacA-s2: 5'-GCAAUGGAACAAGCUUGAATT-3', 5'-UUCAAGCUUGUUCCAUUGCTT-3', vacA-s3: 5'-GCUUGAAGUGGAUAUGAAATT-3', 5'-UUUCAUAUCCACUUCAAGCTT-3', vacA-s4: 5'-GGAUAUGAAAGACGCUGUATT-3', 5'-UACAGCGUCUUUCAUAUCCTT-3', vacA-s5: 5'-GGGACUUAUAAACUUUCAGTT-3', 5'-CUGAAAGUUUAUAAGUCCCTT-3'. 一对非特异siRNA序列vacA-s6为: 5'-AGUGGAUUUCAACGCUAAATT -3', 5'-UUUAGCGUUGAAAUCCACUCT -3'并将vacA-s1和vacA-s6mmol/L MgCl mmol/L NaCl, 5 mmol/L Hepes, 30 的5'-FAM荧光标记. 应用Blast(http://www.ncbi.nlm.nih.gov/blast)在EST数据库查询上述序列, 证实5对特异siRNA序列除vacA基因外, 未发现与另外任何基因同源. One step RT-PCR Kit购自Takara公司, Total RNA Isolation Kit(GIBCO). vacA一抗为羊单抗, 二抗为鼠抗羊, 购自美国Santa Cruz公司. ECL增强化学发光试剂盒购自美国Amersham公司. Electroporater 2510型为Eppendorf公司. Milipore公司超滤离心管. 细菌裂解液HNM: 10 mmol/L Hepes, 30 mmol/L NaCl, 5 mmol/L MgCl2, pH 7.0, 10 mg/L DNA酶, 2 g/ L溶菌酶.
1.2.1 电穿孔法转化及转化效率: 共分为6组, 实验组: vacA-s1, vacA-s2, vacA-s3, vacA-s4, vacA-s5, 对照组: vacA-s6d的 . 将培养3 d的H. pylori菌落用100 mL/L无菌冰甘油溶液冲下, 反复漂洗, 4 ℃ 4 000 r/min离心15 min, 弃上清, 100 mL/L无菌冰甘油重悬, 吸管吹匀, 以吸光度值来调整H. pylori浓度至109 个/L. siRNA 11 μg溶解于5 mL DEPC水并加入95 μL菌液中, 置于电穿孔杯内(4 ℃预冷), 2 500 V, 5 ms电穿孔法转化. 再将转化后的H. pylori置于37 ℃ 100 mL/L, O2, 50 mL/L CO2, 850 mL/L N2 微厌氧培养箱内培养48 h. 将5'-FAM荧光标记的vacA-s1和vacA-s6 经同样方法转化, 2 mL无菌冰甘油溶液洗涤后荧光显微镜下观察(×400倍), 每组10个视野计数发绿色荧光的H. pylori占总H. pylori数的百分比为平均转化效率(%), 转化效率(%) = 荧光H. pylori/总H. pylori.
1.2.2 提取RNA及RT-PCR: 每组分别于转化前、转化后1, 6, 12, 24, 48 h取H. pylori样本提取RNA, RT-PCR扩增的引物序列: 上游5'-TTCAGACTTTATCAATAAG-3', 下游5'-TCCTGCAAAAGATTGTTTG-3', 扩增片段长(259 bp). RT-PCR反应体系: 10×One Step RNA Buffer 2.5 μL, MgCl2(25 mmol/L) 5 μL, dNTP Mixture(10 mmol/L)2.5 μL, Rnase Inhibitor(40 MU/L)0.5 μL, AMV Rtase XL (5 MU/L)0.5 μL, AMV-Optimized Taq(5 MU/L) 0.5 mL. 上游特异性引物(10 μmol/L)0.5 μL, 下游特异性引物(10 μmol/L)0.5 μL, 模板RNA 2 μL, RNase Free dH2O 10.5 μL, Total体积25 μL/Sample. 反应条件: 50 ℃ 30 min; 94 ℃ 2 min; (94 ℃ 1 min; 52 ℃ 1 min; 72 ℃ 1 min)×30; 72 ℃ 7 min. RT-PCR扩增产物经15 g/L琼脂糖电泳溴化乙啶染色确定. 内参选用gyrB基因(DNA旋转酶B亚单位基因), 根据Genbank查到的H. pylori的gyrB基因序列, 经同源性分析, 设计引物为: 上游5'-CGCTAAAGAAAGTGGCACGAC-3', 下游: 5'-TGCGCGTTTCTTCATCCAT-3'. 扩增片段长267 bp, 反应条件及体系同上. 产物取5 μL在15 g/L琼脂糖凝胶(含0.5 mg/L溴化乙锭)电泳, 根据内对照gyrB基因与vacA的RT-PCR产物的电泳条带相对强度, 用Glykobandcan4.5软件确定mRNA的抑制率. 扩增259 bp长度的m区上下游引物为: 5'-TCAATATCAACAAGCTC-3', 5'-CCGCATGCTTTAATGTC-3'. 采用上海博彩生物科技有限公司(BBST)T-A克隆试剂盒将扩增的目的片断克隆至pUCm-T载体中. 重组质粒转化与E.coli DH5a株并扩增, 碱变性法提取质粒. 委托BBST公司测定插入片断的核苷酸序列, 并与报道的核苷酸序列进行比较.
1.2.3 Western blot: 电穿孔转化后1, 6, 12, 24, 48 h各组分别取1×106个H. pylori, 3 000 r/min离心5 min, 弃上清, 加入10倍于沉淀的细菌裂解液反复冻融3次, 15 000 r/min离心20 min, 取上清液经超滤浓缩40倍, 电泳后置12% SDS-PAGE行蛋白分离后, 电转移至硝酸纤维素膜, 50 mL/L脱脂奶粉的TTBS封闭1 h, 以一抗稀释vacA为1:1 000, 内对照vacA为1:5 000, 4 ℃孵育过夜. 二抗采用辣根过氧化物酶(HRP)标记的鼠抗羊抗体为1:1 000, 室温孵育3 h, 用化学发光法(ELC)显色. 待目的蛋白检测完毕后, 1×TBST重新洗膜, 以检测内对照Actin蛋白表达. 用Glykobandcan4.5软件分析Actin与目标蛋白条带灰度比值, 确定其抑制率.
1.2.4 GES-1与H. pylori超滤液共培养: 11 μg特异vacA-s2和非特异vacA-s6电穿孔法转化109个/L H. pylori后, 分别用超滤离心管过滤除菌并浓缩成0.1 mL, GES-1以每孔1×104细胞数分别接种于96孔板上, 每次更换DMEM高糖培养液时均加入上述超滤液10 μL, 共培养1 wk后透射电镜观察GES-1超微结构变化. 流式细胞仪检测细胞周期相分布: 将与H. pylori超滤液共培养后的GES-1收获, PBS洗涤, 700 mL/L冰乙醇固定, 碘化丙啶染色, 流式细胞仪检测.
统计学处理 实验数据用mean±SD表示, 多个样本均数比较用单因素方差分析(one-way ANOVA), 多个实验组与一个对照组比较用最小显著差法(the least significant difference, LSD)分析. 全部资料上机经SPSS 10.0软件包处理, P<0.05为统计学上差异有显著性.
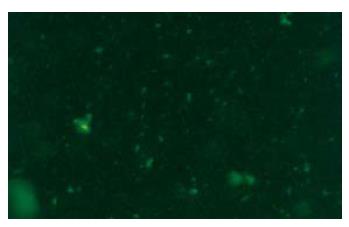
电穿孔法转化后, 在荧光显微镜下(×400, 图1)观察发现H. pylori内出现绿色荧光物质, 冰甘油溶液洗涤后绿色荧光亦无变化, vacA-s1和vacA-s6电穿孔转化效率86%-95%, 平均89%, vacA-s1和vacA-s6两组电穿孔转化效率比较无统计学差别(P>0.05).
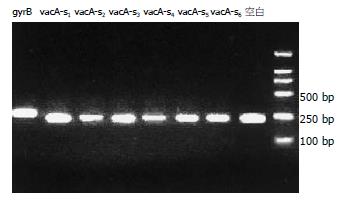
vacA引物的基因片断经电泳和EB染色后, 结果显示扩增片断大小与所设计的大小完全一致, 为229 bp. VacA-s1和vacA-s6各组siRNA对H. pylori vacA的mRNA抑制效应: vacA-s1, vacA-s3, vacA-s5, vacA-s6转化后各时间点mRNA表达无变化; vacA-s2、vacA-s4组在转化1 h vacA mRNA表达明显抑制并达最大值, 1, 6, 12 h抑制率分别为65%和77%, 43%和50%, 17%和9%(与vacA-s1, vacA-s3, vacA-s5, vacA-s6组比较, P<0.05), 转化24, 48 h vacA mRNA表达则为无抑制效应(图2).
GGGGAATGCCGCTAGGCATTATTGGGTCAAAGGCGGGCAATGGAACAAGCTTGAAGTGGATATGAAAGACGCTGTAGGGACTTATAAACTTTCAGGCCTTATCAACTTTACTGGTGGGGATTTAGATGTCAATATGCAAAAAGCCACTTTGCGCTTGGGCCAATTCAATGGCAATTCTTTCACAAGCTATAAGGATAGTGCTGATCGCACCACGAGAGTGGATTTCAAC, 产物与报道相应序列比较, 无碱基插入或缺失现象, 同源性为99%.
vacA-s1, vacA-s3, vacA-s5, vacA-s6转化后各时间点vacA蛋白表达无变化; vacA-s2, vacA-s4组在转化1, 6, 12, 24 h vacA蛋白表达抑制率分别为26%和17%, 47%和40%, 70%和75%, 33%和30%(与vacA-s1, vacA-s3, vacA-s5, vacA-s6组比较, P<0.05), 转化48 h则无抑制效应(图3).
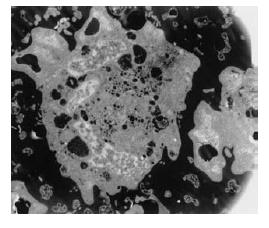
GES-1空泡样变, 空泡几乎充满细胞质, 细胞核固缩, 细胞出现变性、坏死, 细胞质内出现大量溶酶体和胞内体(图4). 而vacA-s2组则无明显上述变化. 细胞周期相分布: vacA-s2组GES-1经流式细胞仪检测均显示以S期细胞为主, G2/M 期细胞、G1/G0期细胞变化不明显, 而GES-1和vacA-s6组转化H. pylori超滤液共培养1 wk后S期细胞明显减少(由61.2%减少到31.0%), G1/G0期细胞显著增加(由36.0%增加到64.7%), G2/M期细胞亦有少量增加(由2.8%增加到4.3%).
迄今, H. pylori确切致病机制未完全明了, 总的说来, 主要由H. pylori产生的毒素、毒性酶及其代谢产物(尿素酶、过氧化氢酶、黏液酶、蛋白酶和脂多糖、生物胺等)的直接破坏作用和介导炎症反应, 诱发宿主的局部和全身免疫反应导致胃黏膜损伤. 目前vacA在众多毒力因子中是被研究最多与进展最快的一种, vacA作用晚期胞内体上的空泡型ATP酶,一旦与目标结合反应, 晚期胞内体便互相融合, 失去细胞器的功能. 另外空泡型ATP酶被毒素刺激后, 在空泡内产生酸性环境, 而环境中的一些渗透性弱碱物质, 例如氨, 在酸性环境中聚集, 不能再渗透过膜, 而在细胞器中通过渗透作用吸收水份使空泡肿胀、变大能够引起晚期溶酶体与晚期胞内体的融合与积累, 并最终形成空泡, 使细胞发生空泡变性[11]. 我们发现GES-1与vacA-s6组在转化H. pylori超滤液共培养后S期细胞明显减少, G1/G0期细胞显著增加, G2/M期细胞亦有少量增加. vacA抑制细胞增殖机制是其能够破坏上皮细胞的正常细胞骨架, 而上皮细胞的骨架对维持细胞的增殖与迁移是极重要的. Pai et al[16]发现纯化的vacA能抑制正常兔胃黏膜细胞肌动蛋白弹性纤维的形成和打乱微管的结构, 显著减少黏着斑激酶的磷酸化与表达. 这些发现证实了vacA破坏了细胞骨架和干扰了细胞信号转导通路. 他们又用vacA处理胃黏膜单层上皮细胞的伤口模型, 发现vacA抑制上皮的重新形成与细胞的增殖, 同时减少Rac21蛋白的表达与分布和c3介导的Rho因子APD的核糖化. 从而进一步证实了vacA破坏了细胞功能与信号转导所依赖的细胞骨架[17].
RNAi技术近年发展迅速, 已成为分子生物学研究的主要技术手段之一, 在人类基因功能研究、信号转导研究及基因治疗方面已显示出巨大的前景. 目前, 利用RNAi进行白血病[18,19]、艾滋病[20-23]、乙型肝炎[24-26]、丙型肝炎[27,28]的治疗研究已取得一定进展, 即成功地下调了相关基因的表达. 我们通过RNAi下调vacA基因及蛋白的表达以治疗H. pylori. 本研究中使用电穿孔法转化, 特异和非特异的siRNA均可以成功地转化H. pylori内, 转化率可达89%, 并可通过5'-FAM荧光标记siRNA转化H. pylori加以证实. 在各组不同序列siRNA中, vacA-s2、vacA-s4h时vacA蛋白表达抑制率最大, 分别为70%和75%, 而其他四组则无干扰效应. 尽管RNAi在其他诸多实验中有着出色的表现, 但在本实验中转化效率及抑制率尚低, 且RNAi抑制mRNA和蛋白的表达时间作用短暂, 且并非设计的每一对特异siRNA皆有干扰效应. 其原因可能是由于设计siRNA针对 h时vacA mRNA表达明显抑制并达最大值, 抑制率分别为65%和77%, 两组在转化12 组在转化1H. pylori vacA基因的靶位点不正确所致, 且由于电穿孔法转化方式所致siRNA在H. pylori内不能长久稳定的表达. 因此, 采用其他方式进行转化, 如脂质体载体法转化、逆转录病毒载体介导转化法等, 开发具有高转化效率、高基因容量和较低毒性的新型载体是RNAi技术应用于基因治疗关键[29,30]
RNAi技术近年发展迅速, 已成为分子生物学研究的主要技术手段之一, 在人类基因功能研究、信号转导研究及基因治疗方面已显示出巨大的前景. 目前, 利用RNAi进行白血病、艾滋病、乙型肝炎、丙型肝炎的治疗研究已取得一定进展, 即成功地下调了相关基因的表达.本研究依据RNAi原理, 观察靶向siRNA抑制H. pylori vacA基因表达.
RNA干扰技术是利用双链RNA诱导的序列特异的转录后基因沉默(PTGS), 他是由19-21个核苷酸大小的小分子干扰RNA(siRNA)引起的.
本研究依据RNAi原理, 通过电穿孔法将针对vacA基因的siRNA转化入H. pylori, 发现siRNA可特异地抑制vacA基因表达.
RNA干扰(RNA interference, RNAi)技术是利用双链RNA (double stranded RNA)诱导的序列特异的转录后基因沉默(posttran-scriptional gene silencing, PTGS), 他是由19-21个核苷酸大小的小分子干扰RNA(small interfering RNA, siRNA)引起的, RNAi现象广泛存在于多种生物体内.
RNAi技术已成为分子生物学研究的主要技术手段之一, 在人类基因功能研究、信号转导研究及基因治疗方面已显示出巨大的前景, 故本研究有重要意义.
电编: 张敏 编辑:潘伯荣 审读:张海宁
| 1. | Couturier MR, Tasca E, Montecucco C, Stein M. Interaction with CagF is required for translocation of CagA into the host via the Helicobacter pylori type IV secretion system. Infect Immun. 2006;74:273-281. [PubMed] |
| 2. | Perrone M, Munoz L, Camorlinga M, Correnti M, Cavazza ME, Lecuna V, Torres J. Importance of IgG anti-CagA antibodies of Helicobacter pylori in Venezuelan patients with gastric diseases. Invest Clin. 2005;46:357-367. [PubMed] |
| 3. | Eaton KA, Gilbert JV, Joyce EA, Wanken AE, Theve-not T, Baker P, Plaut A, Wright A. In vivo comple-mentation of ureB restores the ability of Helicobacter pylori to colonize. Infect Immun. 2002;70:771-778. [PubMed] |
| 4. | Datta S, Chattopadhyay S, Chowdhury A, Santra A, Saha DR, Ramamurthy T, Bhattacharya SK, Berg DE, Nair GB, Mukhopadhyay AK. Diagnosis and genotyping of Helicobacter pylori by polymerase chain reaction of bacterial DNA from gastric juice. J Gastroenterol Hepatol. 2005;20:1253-1259. [PubMed] |
| 5. | Skibinski DA, Genisset C, Barone S, Telford JL. The cell-specific phenotype of the polymorphic vacA midregion is independent of the appearance of the cell surface receptor protein tyrosine phosphatase beta. Infect Immun. 2006;74:49-55. [PubMed] |
| 6. | Ricci V, Chiozzi V, Necchi V, Oldani A, Romano M, Solcia E, Ventura U. Free-soluble and outer membr-ane vesicle-associated VacA from Helicobacter pylori: Two forms of release, a different activity. Biochem Biophys Res Commun. 2005;337:173-178. [PubMed] |
| 8. | Cogoni C, Macino G. Homology-dependent gene silencing in plants and fungi: a number of varia-tions on the same theme. Curr Opin Microbiol. 1999;2:657-662. [PubMed] |
| 9. | Yang D, Lu H, Erickson JW. Evidence that process-ed small dsRNAs may mediate sequence-specific mRNA degradation during RNAi in Drosophila embryos. Curr Biol. 2000;10:1191-1200. [PubMed] |
| 10. | Wianny F, Zernicka-Goetz M. Specific interference with gene function by double-stranded RNA in early mouse development. Nat Cell Biol. 2000;2:70-75. [PubMed] |
| 11. | Molinari M, Galli C, Norais N, Telford JL, Rappuoli R, Luzio JP, Montecucco C. Vacuoles induced by Helicobacter pylori toxin contain both late endosomal and lysosomal markers. J Biol Chem. 1997;272:25339-25344. [PubMed] |
| 12. | Sabariegos R, Gimenez-Barcons M, Tapia N, Clotet B, Martinez MA. Sequence homology required by human immunodeficiency virus type 1 to escape from short interfering RNAs. J Virol. 2006;80:571-577. [PubMed] |
| 13. | Grunweller A, Hartmann RK. RNA interference as a gene-specific approach for molecular medicine. Curr Med Chem. 2005;12:3143-3161. [PubMed] |
| 14. | Larsen SR, Rasko JE. Lymphoproliferative disor-ders: prospects for gene therapy. Pathology. 2005;37:523-533. [PubMed] |
| 15. | Schutz S, Sarnow P. Interaction of viruses with the mammalian RNA interference pathway. Virology. 2006;344:151-157. [PubMed] |
| 16. | Pai R, Cover TL, Tarnawski AS. Helicobacter pylori vacuolating cytotoxin (VacA) disorganizes the cytoskeletal architecture of gastric epithelial cells. Biochem Biophys Res Commun. 1999;262:245-250. [PubMed] |
| 17. | Pai R, Sasaki E, Tarnawski AS. Helicobacter pylori vacuolating cytotoxin (VacA) alters cytoskeleton-associated proteins and interferes with re-epithelia-lization of wounded gastric epithelial monolayers. Cell Biol Int. 2000;24:291-301. [PubMed] |
| 18. | Ma XX, Wang C, Wei J, Qin YW, Yan SK, Gao YR, Cai Q. Inhibitory effect of RNA interference on chronic myeloid leukemia bcr/abl oncogene expre-ssion. Zhonghua Xueyexue Zazhi. 2005;26:359-362. [PubMed] |
| 19. | Withey JM, Marley SB, Kaeda J, Harvey AJ, Cromp-ton MR, Gordon MY. Targeting primary human leukaemia cells with RNA interference: Bcr-Abl targeting inhibits myeloid progenitor self-renewal in chronic myeloid leukaemia cells. Br J Haematol. 2005;129:377-380. [PubMed] |
| 20. | Wilda M, Fuchs U, Wossmann W, Borkhardt A. Killing of leukemic cells with a BCR/ABL fusion gene by RNA interference (RNAi). Oncogene. 2002;21:5716-5724. [PubMed] |
| 21. | Barnor JS, Miyano-Kurosaki N, Yamaguchi K, Abumi Y, Ishikawa K, Yamamoto N. Lentiviral-mediated delivery of combined HIV-1 decoy TAR and Vif siRNA as a single RNA molecule that cleaves to inhibit HIV-1 in transduced cells. Nucleo-sides Nucleotides Nucleic Acids. 2005;24:431-434. [PubMed] |
| 22. | Anderson J, Akkina R. HIV-1 resistance conferred by siRNA cosuppression of CXCR4 and CCR5 coreceptors by a bispecific lentiviral vector. AIDS Res Ther. 2005;2:1. [PubMed] |
| 23. | Amini S, Mameli G, Del Valle L, Skowronska A, Reiss K, Gelman BB, White MK, Khalili K, Sawaya BE. p73 Interacts with human immunodeficiency virus type 1 Tat in astrocytic cells and prevents its acetylation on lysine 28. Mol Cell Biol. 2005;25:8126-8138. [PubMed] |
| 24. | Jacque JM, Triques K, Stevenson M. Modulation of HIV-1 replication by RNA interference. Nature. 2002;418:435-438. [PubMed] |
| 25. | Radhakrishnan SK, Layden TJ, Gartel AL. RNA interference as a new strategy against viral hepati-tis. Virology. 2004;323:173-181. [PubMed] |
| 26. | Giladi H, Ketzinel-Gilad M, Rivkin L, Felig Y, Nuss-baum O, Galun E. Small interfering RNA inhibits hepatitis B virus replication in mice. Mol Ther. 2003;8:769-776. [PubMed] |
| 27. | Shlomai A, Shaul Y. Inhibition of hepatitis B virus expression and replication by RNA interference. Hepatology. 2003;37:764-770. [PubMed] |
| 28. | Prabhu R, Vittal P, Yin Q, Flemington E, Garry R, Robichaux WH, Dash S. Small interfering RNA effectively inhibits protein expression and negative strand RNA synthesis from a full-length hepatitis C virus clone. J Med Virol. 2005;76:511-519. [PubMed] |
| 29. | An DS, Xie Y, Mao SH, Morizono K, Kung SK, Chen IS. Efficient lentiviral vectors for short hairpin RNA delivery into human cells. Hum Gene Ther. 2003;14:1207-1212. [PubMed] |
| 30. | Stevenson M. Therapeutic potential of RNA interf-erence. N Engl J Med. 2004;351:1772-1777. [PubMed] |