修回日期: 2005-11-24
接受日期: 2005-11-25
在线出版日期: 2006-02-08
目的: 应用甲基化敏感性代表性差异分析法(methylation-sensitive representational difference analysis, MS-RDA)筛选胃癌和正常胃组织间甲基化差异DNA片段.
方法: 通过MS-RDA筛选胃癌和正常胃组织间DNA甲基化差异片段, 经克隆、测序后进行生物信息学分析.
结果: 获得3个甲基化差异片段, 分别为CRS1308, CRS1309, CRS1310序列, 其中CRS1309和CRS1310已被GenBank收录, 登陆号分别为AY887106和AY887107, CRS1309序列与LOC440683基因第11外显子、LOC440887基因的3'端, DRD5基因启动子和外显子区域均有很高的相似性(分别为98%, 99%, 94%), CRS1310序列与1999年Minoru Toyota在人类结肠癌中分离出的核糖体RNA上的甲基化差异性CpG岛有很高的相似性(98%).
结论: 胃癌和正常胃组织间DNA甲基化存在差异, MS-RDA可有效分析这两种不同组织间甲基化的差异, 筛选出有意义的甲基化差异片段.
引文著录: 廖爱军, 苏琦, 王珣, 曾斌, 石巍. 应用MS-RDA筛选胃癌甲基化差异片段. 世界华人消化杂志 2006; 14(4): 365-369
Revised: November 24, 2005
Accepted: November 25, 2005
Published online: February 8, 2006
AIM: To isolate the differentially methylated DNA sequences between gastric cancer and normal gastric mucosa.
METHODS: The differentially methylated DNA sequences between gasrtic cancer and normal gastric mucosa were isolated by methylation-sensitive representational difference analysis (MS-RDA). Similarities between the separated fragments and the human genomic DNA were analyzed with Building Loads Analysis and System Thermodynamics (BLAST) program.
RESULTS: Three differentially methylated DNA sequences, CRS1308, CRS1309, and CRS1310, were obtained. CRS1309 and CRS1310 had been accepted by GenBank. CRS1309 was highly similar to the eleventh exon of LOC440683 (98%), 3' end of LOC440887 (99%), and promoter and exon region of DRD5 (94%). CRS1310 was consistent (98%) with a CpG island, in ribosomal RNA, isolated from colorectal cancer by Minoru Toyota in 1999.
CONCLUSION: The methylation degree is different between gastric cancer and normal gastric mucosa. The differentially methylated DNA sequences can be isolated effectively by MS-RDA.
- Citation: Liao AJ, Su Q, Wang X, Zeng B, Shi W. Isolation of differentially methylated DNA sequences of gastric cancer by methylation-sensitive representational difference analysis. Shijie Huaren Xiaohua Zazhi 2006; 14(4): 365-369
- URL: https://www.wjgnet.com/1009-3079/full/v14/i4/365.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i4.365
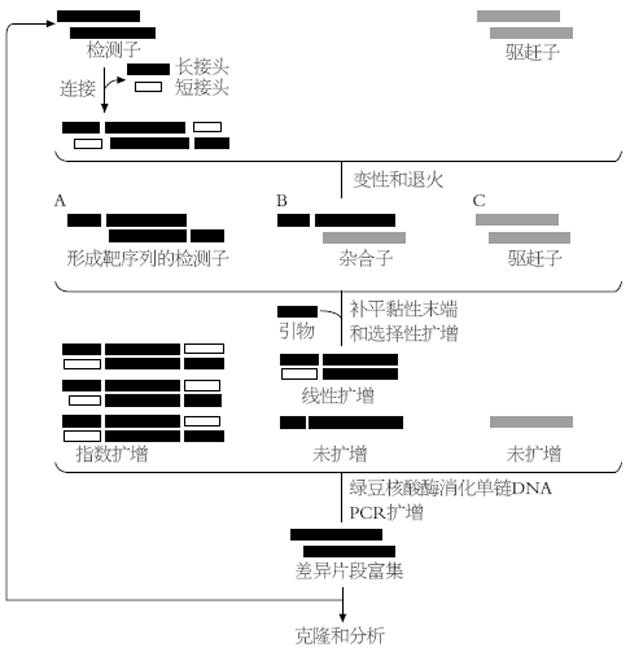
1993年Lisitsyn et al[1]在消减杂交的基础上发展了一种代表性差异分析(RDA)的方法, 该方法通过限制性内切酶消化基因组DNA, 降低基因组的复杂性, 经多轮消减杂交结合PCR后使差异性的靶基因片段得到大量的富集, 后来, Ushijima et al[2]采用甲基化敏感性限制性内切酶消化基因组DNA, 而在RDA的基础上形成了MS-RDA(甲基化敏感性代表性差异分析), 该方法被用来筛选肿瘤中发生异常甲基化的基因. 我们运用MS-RDA方法筛选胃癌与正常胃组织间DNA甲基化差异片段, 为进一步研究胃癌新的相关抑癌基因提供理论依据.
胃腺癌患者胃窦部肿瘤边缘黏膜及其癌旁(>5 cm)正常胃黏膜各1-3 g, 用锡箔、纱布包好置于液氮中保存, 组织标本术后经病理检查结果证实.
取100 mg组织匀浆, 加入1.5 mL DNA提取液及胰RNA酶(Fermentas公司)(10 g/L)20 μL, 混匀, 37℃孵育1 h, 加入蛋白酶K(Fermentas公司)(20 g/L)40 μL, 上下颠倒混匀, 37℃消化3 h后溶液变得清亮, 使用经典的酚氯仿抽提法提取基因组DNA. 在10 μg基因组DNA中加入10 μL甲基化敏感性限制性内切酶HpaⅡ(Fermentas公司)(10MU/L), 混匀稍离心后, 37℃酶切20 h. 未甲基化的-CCGG-位点被切割成黏性末端, 已甲基化的位点未被切割. 在酶切产物中加入RHpaⅡ24和RHpaⅡ11接头(表1), 50℃孵育1 min, 然后以1 min降1℃的速度逐步冷却到10℃, 使其自身退火形成RHpaⅡ24/11接头, 在T4DNA连接酶(Takara公司)作用下, 16℃连接14-16 h[1]. 以上述连接接头后产物接头中RHpaⅡ24为引物在LA Taq酶(Takara公司)作用下进行PCR扩增, 95℃变性1 min, 72℃退火3 min, 26个循环后72℃延伸10 min, 使带有R接头的DNA片段即扩增子得到有效富集[2], 10 g/L琼脂糖凝胶电泳观察扩增子.
| 接头名称 | 序列 |
| RHpa Ⅱ24 | 5'-AGC ACT CTC CAG CCT CTC ACC GAC-3' |
| RHpa Ⅱ11 | 5'-CGG TCG GTG AG-3' |
| JHpa Ⅱ24 | 5'-ACC GAC GTC GAC TAT CCA TGA AAC-3' |
| JHpa Ⅱ11 | 5'-CGG TTT CAT GG-3' |
| NHpa Ⅱ24 | 5'-AGG CAA CTG TGC TAT CCG AGG GAC-3' |
| NHpa Ⅱ11 | 5'-CGG TCC CTC GG-3' |
| SHpa Ⅱ24 | 5'-ACT TCT ACG GCT GAA TTC CGC CAC-3' |
| SHpa Ⅱ11 | 5'-CGG TGT CGG AAT-3' |
1.2.1 消减杂交: 胃癌扩增子和胃正常扩增子在同裂酶MspⅠ酶(Fermentas公司)酶切作用下切除R接头, 酶切产物用DNA片段纯化试剂盒(Takara公司)纯化以去除混于其中的R接头. 将胃癌扩增子去R接头纯化后的产物连上JHpaⅡ24/11接头(表1)作为检测子, 胃正常扩增子去R接头产物作为驱赶子进行的杂交称为正交, 反之称为反交, 检测子和驱赶子按1∶40的比例混合沉淀后溶于4 μL杂交缓冲液, 然后96℃变性10 min, 67℃杂交24 h, 杂交反应完成后, 取1/10体积杂交产物为模板, 以JHpaⅡ24为引物进行PCR反应, 13个循环后加入Mung Bean Nuclease(Takara公司)消化单链核苷酸, 酚/氯仿抽提后继续PCR反应20-30个循环[2]. 将上述第1轮正交和反交的产物用MspⅠ消化, 经DNA片段纯化试剂盒纯化后, 依次换以N/S接头, J接头分别进行随后的第2轮, 第3轮杂交和扩增. 检测子和驱赶子的比例由第1轮的1∶40分别改为1∶400, 1∶4 000[3], 获得胃癌和正常胃组织间差异甲基化DNA片段(图1).
1.2.2 克隆测序及相似性分析: 将最终的消减杂交产物进行15 g/L琼脂糖凝胶电泳, 紫外投射仪下将目的条带切胶, 经琼脂糖凝胶回收试剂盒(Takara公司)回收后与pGEM-T载体(Promega公司)在4℃下连接12 h, 将连接产物加入高效感受态制备试剂盒(Fermentas公司)制备的JM109感受态细菌进行转化复苏后, 将菌液涂布于含有氨苄青霉素的LB固体培养基上, 37℃培养箱中培养14 h, 挑取阳性克隆, 在LB液体培养基中37℃摇菌8 h, 用碱法提取质粒DNA, 经PCR法鉴定目的片段已转化到细菌质粒中, 将菌液送Takara公司测序, 测序结果经BLAST系统进行相似性分析.
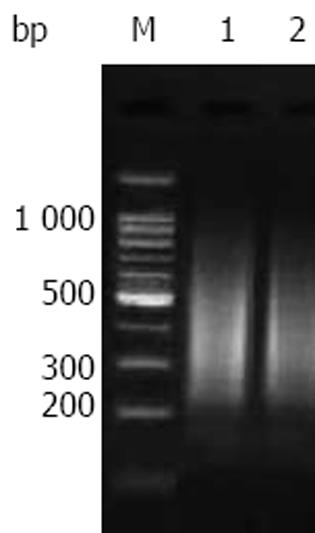
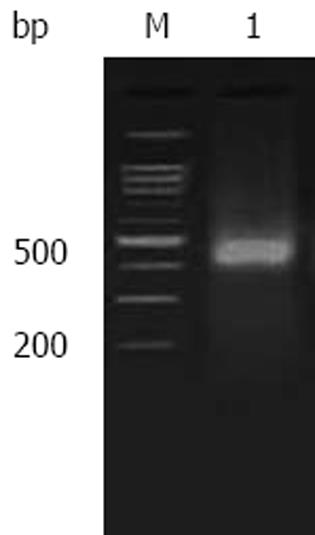
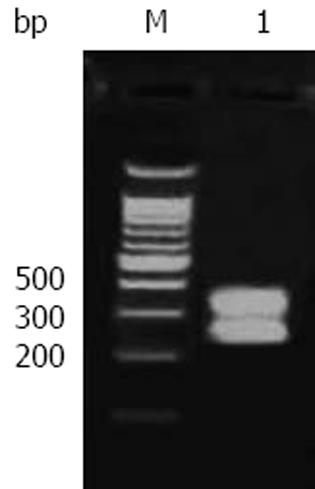
胃癌组织和正常胃组织基因组DNA的HpaⅡ酶切产物加RHpaⅡ24/11接头, 经PCR扩增有效富集的扩增子(图2). 扩增子在200-1 000 bp之间为明亮的弥散的条带. 将胃癌组织作为检测子, 胃正常组织作为驱赶子经过3轮的正交反应后便可在400 bp左右见1条清晰的差异性条带(图3). 以胃正常组织作为检测子, 胃癌组织作为驱赶子进行2轮反交后可在400 bp左右及200-300 bp间见2条明亮清晰的差异条带(图4).
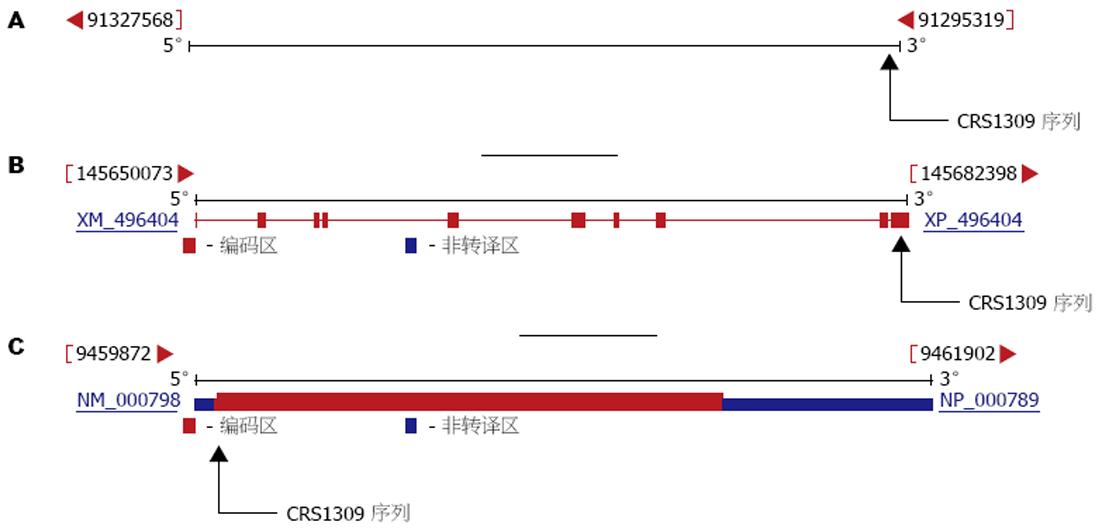
正交和反交后的3条甲基化差异DNA片段经pGEM-T克隆后, 分别挑取阳性克隆进行序列测通分析. 其中正交的1条差异片段CRS1308的测序结果不理想, 存在较多的误读和失读, 考虑为GC含量丰富, 模板存在复杂的二级结构导致测序时峰值叠加, 测序仪无法准确识别, 表2中的相关数据仅供参考. 反交的2条差异片段CRS1309和CRS1310已被GenBank收录, 登陆号分别为AY887106和AY887107, 经BLAST分析后发现, 其中AY887106序列与1q21.1的LOC440683基因第11外显子有98%的相似性, 与2p11.1的LOC440887基因的3'端有99%的相似性, 与4p16.1位置的DRD5基因的启动子和外显子区域有94%的相似性, AY887106序列与LOC440887基因、LOC440683基因和DRD5基因的位置关系如图5. AY887107序列位于核糖体RNA上与1999年Toyota et al[4]在人类结肠癌中分离出的甲基化差异性CpG岛有98%的相似性. BLAST分析结果归纳为表2.
| 片段 | 筛选 (次数) | 长度 (bp) | GC (%) | CG/GC | 相似性 | |||
| 染色体定位 | S | E | 相似率 (%) | |||||
| CRS1308 | 2 | 431 | 46.6 | 0.653 8 | 18q21.1-2 | 272 | 1e-69 | 86 |
| CRS1309 (AY887106) | 2 | 264 | 46.6 | 0.700 0 | 2p11.1 | 521 | e-145 | 99 |
| 1q21.1 | 498 | e-138 | 98 | |||||
| 4p16.1 | 435 | e-119 | 94 | |||||
| CRS1310 (AY887107) | 2 | 395 | 50.6 | 0.928 6 | 8q13.3 | 745 | 0 | 98 |
DNA甲基化通过影响基因转录、促进基因突变、增加染色体结构的不稳定性等多方面机制促进胃癌的发生、发展, 是表遗传学修饰的一种重要方式, 研究胃癌组织与正常胃组织基因的甲基化分布的差异, 不仅可探讨甲基化在胃癌发生中的作用, 同时也可筛选出与胃癌发生有关的新的相关基因, 为胃癌研究提供新的指标. 早期使用Southern杂交和限制性标志的基因扫描(restriction landmark genomic scanning)也能确定异常甲基化基因, 但是前者仅局限于已知基因, 后者则存在操作复杂以及分离得到的基因难以克隆的缺点[5]. Ushijima et al[2]创建MS-RDA的同时也成功的筛选出了小鼠肝肿瘤中差异性甲基化片段. MS-RDA采用甲基化敏感性四碱基限制性内切酶消化基因组DNA, 可按甲基化分布的差异最大限度降低基因组DNA的复杂性后[3]酶切产物连接RHpaⅡ24/11接头进行PCR, 使整个基因组较短的酶切片段有效富集. 由于长片段难以经PCR扩增, 而短片段扩增效率高, 这就为随后的消减杂交的效率打下了基础, 同时将"检测子"和"驱赶子"两者之间的差异转化为限制性内切酶位点之间的差异[1], 我们得到的扩增子是位于200-1 000 bp之间的弥散条带. 将"检测子"和"驱赶子"进行多轮消减杂交结合PCR扩增, 其中的靶序列即差异甲基化片段为同聚体, 两端均有接头成指数扩增, 而"驱赶子"和"检测子"相同序列形成的异源二聚体因只有单链接头只能成线性扩增, 这样二者间的相同序列被彻底抵消, 从而使"驱赶子"中缺乏而存在于"检测子"中的甲基化差异片段呈指数扩增而得到有效的富集, 因此MS-RDA中"驱赶子"和"检测子"间的差异比较不是酶切片段长度的比较而是两者间甲基化差异信息的比较, 因而可有效的筛选出甲基化差异片段. MS-RDA的实验方法较为繁复, 费用较高, 我们无法进行大样本量的筛选, 仅采用一例标本进行研究, 目的是希望通过该实验为胃癌发病机制研究提供一个新的研究靶点, 为后续研究提供一个方向和平台. 我们采用的标本为同一个体的胃癌组织及其癌旁正常组织, 可排除由于个体差异及其他干扰因素而导致的假阳性结果, 因此我们筛选出的片段可以反映该个体在胃癌发展过程中相关基因甲基化的差异. 我们从GenBank提供的信息显示, 与AY887106序列高度相关的LOC440683基因和LOC440887基因都是目前研究甚少的新基因, LOC440683基因的最新信息更新认为其可能为多巴胺受体D5基因的假基因, 是物种进化过程中的产物, LOC440887基因被认为可能与髓细胞/淋巴细胞或多系混合性白血病有关, 编码ALR(急性淋巴母细胞性白血病[ALL]-1相关)样蛋白, 并与位于染色体7q36上的MLL3基因具有密切的相关性. Tan et al[6]发现在髓性白血病中发现MLL3基因所在的染色体区域频繁缺失, 认为MLL3基因的缺失在恶性白血病的发生发展中起着重要的作用. 2002年, Ruault et al[7]发现1, 2, 13和21号染色体的着丝粒部位存在MLL3基因的部分拷贝, 而LOC440887基因正是位于2号染色体的着丝粒部位, 很可能为其部分拷贝. 我们已在NCBI的网站上对MLL3基因进行了Digital Northern分析, 结果认为MLL3基因在人胃组织中有表达, 并在胃癌及正常胃组织间存在一定的表达差异, 我们目前正在进行相关的实验室研究. AY887107序列位于核糖体RNA上, 与1999年Toyota et al[4]在人类结肠癌中分离出的甲基化差异性CpG岛有98%的相似性, 进一步表明该位点在胃肠道肿瘤中较易发生甲基化.
MS-RDA由于周期较短、富集效率高且假阳性低的特点而广泛的应用于基因筛选和克隆方面. 2001年, Takai et al[8]应用MS-RDA对两种肺癌细胞株EBC-1和LK-2中异常甲基化基因筛选发现HTR1B基因沉默以及EDN1基因的低表达. 2003年, Miyamoto et al[9]应用MS-RDA发现人类结肠癌、乳腺癌、肺癌、胰腺癌中3-OST-2基因的沉默均与甲基化相关. 2004年, Hagihara et al[10]应用MS-RDA在人类胰腺癌中筛选出111个甲基化差异片段, 其中35个位于已知基因的5'区域. 但是MS-RDA同时也存在步骤繁多, 作为"检测子"和"驱赶子"样本要求具有高度同源性, 最后富集的片段小于1 Kb的缺点, 有可能使部分信息丢失, 但从本研究结果表明, MS-RDA能有效分离两种组织的差异甲基化DNA片段, 是研究胃癌中DNA甲基化作用, 筛选新的抑癌基因的一种较好的方法.
研究胃癌组织与正常胃组织基因的甲基化分布的差异, 不仅可探讨甲基化在胃癌发生中的作用, 同时也可筛选出与胃癌发生有关的新的相关基因, 为胃癌研究提供新的指标. MS-RDA通过采用甲基化敏感性四碱基限制性内切酶消化基因组DNA, 按甲基化分布的差异最大限度降低了基因组DNA的复杂性, 使整个基因组较短的酶切片段得到了有效富集.
从本研究结果表明, MS-RDA能有效分离两种组织的差异甲基化DNA片段, 是研究胃癌中DNA甲基化作用, 筛选新的抑癌基因的一种较好的方法.
本研究利用MSRDA方法筛选胃癌甲基化差异片段, 方法先进, 研究设计合理, 充实了胃癌发生过程中基因变异的材料, 为胃癌的研究提供了新的指标.
电编: 张敏 编辑: 潘伯荣
| 1. | Lisitsyn N, Lisitsyn N, Wigler M. Cloning the differences between two complex genomes. Science. 1993;259:946-951. [PubMed] [DOI] |
| 2. | Ushijima T, Morimura K, Hosoya Y, Okonogi H, Tatematsu M, Sugimura T, Nagao M. Establishment of methylation-sensitive-representational difference analysis and isolation of hypo- and hypermethy-lated genomic fragments in mouse liver tumors. Proc Natl Acad Sci USA. 1997;94:2284-2289. [PubMed] [DOI] |
| 3. | Hubank M, Schatz DG. Identifying differences in mRNA expression by representational difference analysis of cDNA. Nucleic Acids Res. 1994;22:5640-5648. [PubMed] [DOI] |
| 4. | Toyota M, Ho C, Ahuja N, Jair KW, Li Q, Ohe-Toyota M, Baylin SB, Issa JP. Identification of differ-entially methylated sequences in colorectal cancer by methylated CpG island amplification. Cancer Res. 1999;59:2307-2312. [PubMed] |
| 5. | Parkes V, Modha N, Ulrich JM, Jones T, Francis GE. DNA-protein interaction sites in differentiating cells. II. A subset of alphoid repetitive sequences with retinoic acid induced protein attachment and an unusual purine-pyrimidine 'signature'. Exp Hematol. 1996;24:568-579. [PubMed] |
| 6. | Tan YC, Chow VT. Novel human HALR (MLL3) gene encodes a protein homologous to ALR and to ALL-1 involved in leukemia, and maps to chro-mosome 7q36 associated with leukemia and develo-pmental defects. Cancer Detect Prev. 2001;25:454-469. [PubMed] |
| 7. | Ruault M, Brun ME, Ventura M, Roizes G, De Sario A. MLL3, a new human member of the TRX/MLL gene family, maps to 7q36, a chromosome region frequently deleted in myeloid leukaemia. Gene. 2002;284:73-81. [PubMed] [DOI] |
| 8. | Takai D, Yagi Y, Wakazono K, Ohishi N, Morita Y, Sugimura T, Ushijima T. Silencing of HTR1B and reduced expression of EDN1 in human lung cancers, revealed by methylation-sensitive repre-sentational difference analysis. Oncogene. 2001;20:7505-7513. [PubMed] [DOI] |