修回日期: 2005-12-01
接受日期: 2005-12-10
在线出版日期: 2006-01-28
目的: 观察Runx3基因在人胃癌组织及相应正常胃组织中的表达分布特点, 并对其表达下调的机制进行初步研究.
方法: 对25例胃癌标本进行DNA、RNA和蛋白的提取, 利用单链构象多态性(SSCP)和微卫星法, 检测Runx3基因突变和缺失情况. 用MSP(methylation specific PCR)检测25例胃癌标本及细胞株(MGC803, SGC7901)的甲基化状态. 蛋白印迹法(Western blot)和逆转录聚合酶链反应(RT-PCR), 分别检测Runx3蛋白质和mRNA水平.
结果: 在25例胃癌标本中发现一例杂合性缺失, 未发现Runx3第三外显子突变. MSP法检测胃癌标本中55%(14/25)发生启动子区域的甲基化, 同时在胃癌细胞株MGC803中Runx3基因发生了部分甲基化. Western blot和RT-PCR发现Runx3在胃癌组织中表达明显低于相应正常胃组织(蛋白: 50 178±14 404 vs 95 020±43 136, P<0.01; mRNA: 0.66±0.31 vs 1.06±0.34, P<0.01).
结论: Runx3基因在胃癌组织中表达较正常组织明显下调, 提示该基因与胃癌的发生和发展有关. Runx3基因表达下调的机制主要是因为启动子区域甲基化.
引文著录: 曾超, 贺修胜, 罗桥, 赵帅, 邓敏. Runx3 基因在胃癌中的表达及其表达下调机制. 世界华人消化杂志 2006; 14(3): 250-255
Revised: December 1, 2005
Accepted: December 10, 2005
Published online: January 28, 2006
AIM: To investigate level of Runx3 expression in gastric carcinoma, and to explore the main mechanism of Runx3 down-regulation.
METHODS: DNA, RNA and protein were extracted from 25 gastric carcinoma specimens. Single strand conformation polymorphism (SSCP) and microsatellites were used to detect the mutation and loss of heterozygosity (LOH) of Runx3 gene. The methylation status of Runx3 gene was examined by methylation-specific polymerase chain reaction (MS-PCR). The Runx3 mRNA and protein were detected by reverse transcription polymerase chain reaction (RT-PCR) and Western blot.
RESULTS: LOH was detected in only 1 of 25 specimens, and no mutation in exon3 of Runx3 was found. Methylation of Runx3 promoter region was confirmed in 55% (14/25) specimens and part of gastric cancer cell line MGC803. RT-PCR and Western blot indicate expression of Runx3 in gastric carcinoma were lower than that in corresponding normal stomach tissue.
CONCLUSION: The expression of Runx3 gene is down-regulated in gastric carcinoma, suggesting a possible involvement of Runx3 gene in carcinogenesis and progression of gastric cancer. The main mechanism of this down-regulation in Runx3 expression was related to the methylation of its promoter region.
- Citation: Zeng C, He XS, Luo Q, Zhao S, Deng M. Runx3 gene expression in gastric carcinoma and its mechanism of down-regulation. Shijie Huaren Xiaohua Zazhi 2006; 14(3): 250-255
- URL: https://www.wjgnet.com/1009-3079/full/v14/i3/250.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i3.250
Runx家族成员包括Runx1、Runx2和Runx3. Runx1基因是血形成所必需的[1], 其功能障碍与30%急性白血病有关. Runx2基因是骨形成所必需的[2,3], 其功能异常导致常染色体显现的骨疾病, 如锁骨头发育异常. Runx3基因定位于人染色体1p36.1区域[4], 是Runx家族重要成员之一, 其编码基因是近年来新发现的一个抑癌基因, 并且很可能是引起胃癌的关键性基因. 研究发现, 早期胃癌中Runx3基因表达下降约40%, 而在进展期胃癌细胞中, Runx3基因表达下降超过90%[5]. 我们同时检测胃癌中Runx3基因mRNA和蛋白的表达及其与临床病理的联系, 以探讨Runx3在胃癌发生发展中的作用.
南华大学附属第一医院2004-05/2005-03手术切除的25例胃癌及其相应正常组织. 25例标本中, 男性15例, 女性10例, 年龄45-68岁, 平均66岁. 其中细胞分化为未分化及低分化的18例, 中分化及高分化的7例, 全部标本均经病理组织学切片证实. 胃癌细胞株MGC-803、SGC-7901均取自本所.
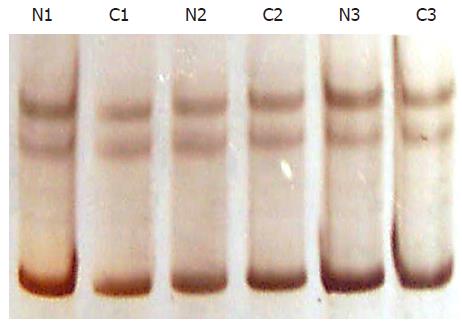
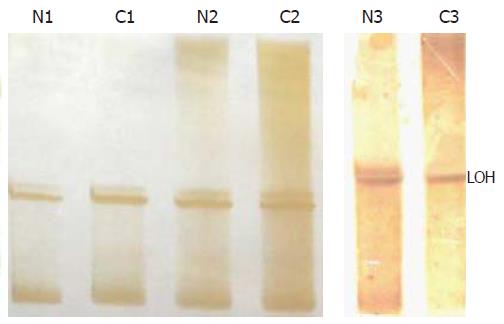
1.2.1 PCR-SSCP和LOH: 对Runx3基因第三外显子进行扩增, 引物序列参照文献[6]. 第三外显子PCR反应条件为94℃ 40 s, 58℃ 30 s, 72℃ 1 min, 反应35个循环. 扩增产物经99℃ 10 min 变性后行聚丙烯酰胺凝胶电泳, 硝酸银染色, 通过观察泳动条带是否移位以判断突变情况(图1). PCR扩增微卫星位点D1S1460, PCR反应条件为94℃ 40 s, 56.6℃ 30 s, 72℃ 1 min, 反应35个循环, 扩增产物经99℃ 10 min变性后行聚丙烯酰胺凝胶电泳, 硝酸银染色, 观察条带有无杂合性缺失.
1.2.2 DNA提取: 用酚-氯仿法提取胃癌组织及胃癌细胞株的DNA.
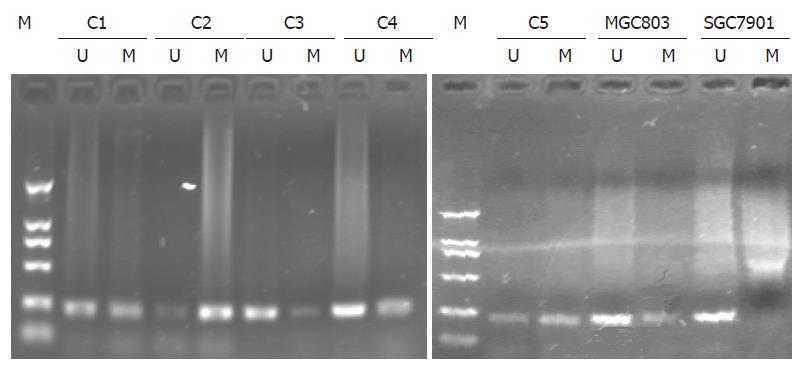
1.2.3 MSP分析Runx3基因甲基化状态: 采用MSP法对胃癌Runx3基因甲基化状态进行了检测, 用氢氧化钠和亚硫酸氢钠对提取的DNA进行修饰, DNA样本处理用CpGenomeTM DNA Modification Kit(CHEMICON, CAD). 最后采用Runx3基因特异的甲基化引物和未甲基化引物对修饰后的DNA进行扩增, 扩增引物序列参照文献[6].
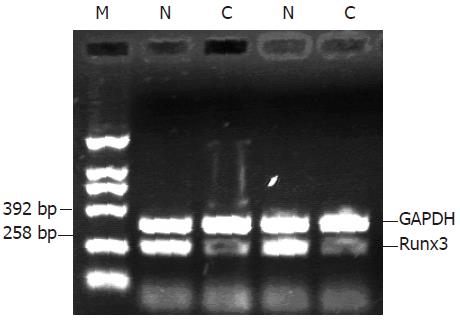
1.2.4 RT-PCR: 采用Trizol总RNA提取液, 取100 mg组织提取总RNA, 10 g/L琼脂糖凝胶电泳分析其完整性, 用紫外分光光度仪测其吸光度(A)值和浓度. 鸟类成髓细胞瘤病毒(AMV)逆转录酶(Promege公司产品)进行逆转录. 取5 mL逆转录产物用于PCR反应. PCR反应的Runx3基因和作为内参照的GAPDH均由大连宝生物公司合成. Runx3产物大小258 bp, 上游引物序列TGGCAGGCAATGACGA, 下游引物序列为CAGGGAACGGCTTGGT; GAPDH产物大小393 bp, 上游引物序列为GGATTTGGT CGTATTGGGCG, 下游引物序列为TACTTCTCATGGTTCACAC. Runx3与GAPDH反应同管进行, 反应终体积为50 mL, 反应条件为94℃ 1 min, 56.5℃ 45 s, 72℃ 1 min, 共34个循环. PCR产物在含0.5 mg/L溴化乙锭的10 g/L琼脂糖凝胶上电泳, 应用AlphalmagerTM2200凝胶成像系统对条带进行扫描分析. Runx3表达量以Runx3的A值比同管反应GAPDH的A值衡量.
1.2.5 Western blot: 取200 mg的组织按组织和细胞裂解液1:5加入1 mL裂解液(0.1 mol/L NaCl, 0.01 mol/L Tris-HCL pH 7.6; 0.001 mol/L EDTA, pH 8.0, 1 mg/L Leuptin, 100 mg/L PMSF), 匀浆器匀浆提取总蛋白, BCA法蛋白定量. 取100 mg 总蛋白变性10 min后, 以聚丙烯酰胺凝胶电泳, 蛋白电泳分离后经电转移槽转移至PVDF膜. 5%脱脂奶粉室温封闭1 h, PVDF膜置于1:250稀释的一抗(羊抗人, Runx3单克隆抗体, Santa Cruz公司产品)稀释液中, 4℃过夜, TBST洗膜30 min, 再置于1:4 500稀释的二抗(抗羊, IgG-HRP, 北京中杉金桥生物公司产品)稀释液中, 37℃孵育1 h, TBST洗膜45 min后加入ECL发光剂显影曝光.
统计学处理 采用SPSS 11.5统计软件, 计量资料的组间比较采用t检验.
对25例胃癌进行了PCR-SSCP, 均未出现异常泳动条带(图1), LOH发现1例杂合性缺失(图2).
对25例胃癌标本及胃癌细胞株MGC803、SGC7901进行检测, 结果发现55%(14/25)的胃癌标本有不同程度的甲基化, 并且在胃癌细胞株MGC803中检测到启动子区域部分甲基化(图3).
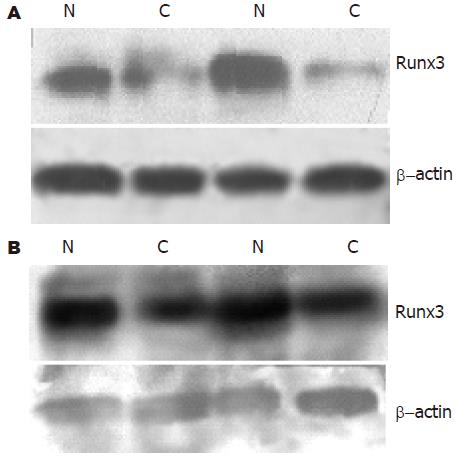
距癌原发灶10 cm以上, Runx3基因mRNA和蛋白的表达 在全部的胃癌组织中均可以检测到mRNA和蛋白的表达, 胃癌组织及相应正常组织中Runx3 mRNA的表达为0.66±0.31和1.06±0.34; Runx3蛋白的表达量分别为50 178±14 404和95 020±43 136. Runx3基因mRNA在胃癌组织中的表达均低于相应正常组织, 差异具有显著性(P<0.01, 图4). 低分化胃癌中, Runx3蛋白在正常胃黏膜中表达明显高于相应的胃癌组织. 高分化胃癌中, Runx3蛋白在正常胃黏膜中表达高于相应的胃癌组织, 但差异并不明显(图5).
直线相关分析显示胃癌组织中Runx3基因mRNA和蛋白的表达具有相关性(rs = 0.820, P<0.05).
将胃癌按不同的临床病理特征分组比较Runx3 mRNA和蛋白的表达, 结果显示, Runx3 mRNA 和蛋白的表达与胃癌的进展程度及分化程度有关(P<0.05), 而与性别无关(表1).
| 病理特征 | n | Runx3 mRNA (mean±SD) | P值 | Runx3蛋白 (mean±SD) | P值 |
| 性别 | |||||
| 男性 | 15 | 0.66±0.1 | 47950±7000 | ||
| 女性 | 10 | 0.63±0.2 | >0.05 | 47857±7204 | >0.05 |
| 进展程度 | |||||
| 早期 | 9 | 0.96±0.1 | 74 499±17 372 | ||
| 进展期 | 16 | 0.52±0.1 | <0.05 | 40 345±8 472 | <0.05 |
| 分化程度 | |||||
| 未分化及低分化 | 18 | 0.48±0.3 | 48 462±10 762 | ||
| 中分化及高分化 | 7 | 0.86±0.6 | <0.05 | 68 049±19 176 | <0.05 |
尽管世界上一些地区胃癌发病率在降低, 但他仍是导致癌症患者死亡的主要疾病之一[7]. 胃癌的发生是一个多因素、多步骤、多基因参与的过程. 他涉及到基因的不稳定性, 端粒的活化, 癌基因的激活[8-10], 抑癌基因的失活[11-13]. 对胃癌的研究表明, 胃癌的发病是由饮食、感染、环境和遗传等各种因素所引起的[14-16].
Li et al[6]发现大约有45%-60%人胃癌细胞中有Runx3的表达下降或缺失. 与之相比, 目前虽然已经发现约10种与胃癌相关的抑癌基因如"p53", "c-met"等[17-18], 但他们与胃癌的相关性偏小, 而Runx3很可能是与胃癌发生相关的一个关键性基因. Levanon et al[5]发现当Runx3+/-小鼠配对后, 大约20%-25%新生小鼠是Runx3-/-, 他的重量大小与其父母无差别. 然而, 75% Runx3-/-新生小鼠由于血糖过低, 没有存活超过10天, 并发现Runx3-/-小鼠胃壁的厚度是野生型的2倍. Runx3蛋白是TGF-β信号通路下游的一个转录因子, TGF-β是许多细胞的有效抑制因子[19](如: 上皮细胞、内皮细胞、淋巴细胞等). TGF-β信号紊乱将导致许多肿瘤的发生. 有实验表明, Smad复合物须在Runx3蛋白的指导下, 才能从胞质内转入特定的靶位点[20], 与Runx蛋白共同转录激活靶基因, 从而对细胞的分化、细胞周期调控、凋亡和恶性转化起作用.
我们检测了胃癌中Runx3基因mRNA和蛋白表达. 结果表明, 胃癌中Runx3 mRNA和蛋白的表达呈正相关, 这提示胃癌中Runx3 mRNA和蛋白的表达具有一致性. 同时我们发现Runx3基因mRNA和蛋白的表达在胃癌中明显低于相应正常组织, 这提示Runx3基因可能不仅与胃癌的发生有关. 在不同进展和分化程度的胃癌中, Runx3表达也明显下调, 表明Runx3基因与胃癌的演进也密切相关.
我们通过PCR-SSCP对Runx3第三外显子突变进行了检测, 未出现异常泳动条带, 表明Runx3基因表达下调不是由于突变造成的. 由于不同长度的微卫星序列标记了等位基因, 因此应用微卫星多态标记非常适用于检测杂合性缺失[11,21,22]. 我们检测了25例胃癌组织杂合性缺失状况, 仅发现一例胃癌标本的杂合性缺失. 抑癌基因启动子区域的高甲基化可导致基因转录失活及其表达下调[23], CpG岛甲基化在多种肿瘤中均有发生[24-26]. Li et al[6]研究发现Runx3基因失活机制是杂合性缺失和启动子区域甲基化. 我们对25例胃癌标本进行检测, 发现55%的胃癌标本发生了不同程度的甲基化, 表明在本实验组中Runx3基因表达下调主要机制是启动子区域甲基化. Oshimo et al[27]研究80例胃癌标本及7例胃癌细胞株, 45例相应的正常黏膜Runx3基因的CpG岛甲基化情况. 同时用定量RT-PCR检测50例胃癌中Runx3基因表达水平, 结果证实Runx3启动子区域高甲基化在71/80例胃癌中出现, 启动子区域的高甲基化在肠型胃癌中最常见. Kim et al[28]发现在慢性胃炎中Runx3甲基化为8.1%, 肠化时为28.1%, 胃腺瘤中为27.3%. Runx3基因甲基化在非肿瘤的胃黏膜中仅发生于77岁以上的老人[29]. 因此, Runx3的甲基化可能是胃癌特异性的标记物, 可作为一种很有前途的分子诊断的标记. 用去甲基化药物处理激活Runx3基因的表达, 也许可能成为临床上治疗胃癌的一种有效的策略.
Lietal研究表明, 45%-60%的胃癌中Runx3表达均明显下调, 而与之相比, 其他胃癌相关抑癌基因与胃癌发生的相关性偏小, 因此推测Runx3可能是引起胃癌的关键性基因.
关于Runx3基因的研究国内报道较少, 本研究检测到胃癌标本中Runx3基因表达明显下调, 并发现下调机制主要是启动子区域高甲基化.
Runx3基因可作为一个有效的分子靶标, 从而为临床治疗胃癌提供坚实的理论依据.
文章具有一定新颖性和可读性, 达到国内先进水平.
编辑: 菅鑫妍 审读: 张海宁 电编: 张敏
| 1. | Okuda T, van Deursen J, Hiebert SW, Grosveld G, Downing JR. AML1, the target of multiple chromosomal translocations in human leukemia, is essential for normal fetal liver hematopoiesis. Cell. 1996;84:321-330. [PubMed] |
| 2. | Komori T, Yagi H, Nomura S, Yamaguchi A, Sasaki K, Deguchi K, Shimizu Y, Bronson RT, Gao YH, Inada M. Targeted disruption of Cbfa1 results in a complete lack of bone formation owing to maturational arrest of osteoblasts. Cell. 1997;89:755-764. [PubMed] |
| 3. | Otto F, Thornell AP, Crompton T, Denzel A, Gilmour KC, Rosewell IR, Stamp GW, Beddington RS, Mundlos S, Olsen BR. Cbfa1, a candidate gene for cleidocranial dysplasia syndrome, is essential for osteoblast differentiation and bone development. Cell. 1997;89:765-771. [PubMed] |
| 4. | Levanon D, Negreanu V, Bernstein Y, Bar-Am I, Avivi L, Groner Y. AML1, AML2, and AML3, the human members of the runt domain gene-family: cDNA structure, expression, and chromosomal localization. Genomics. 1994;23:425-432. [PubMed] |
| 5. | Levanon D, Brenner O, Otto F, Gorner Y, Runx3 knock out and stomach cancer. EMBO reports. 2003;4:560-564. |
| 6. | Li QL, Ito K, Sakakura C, Fukamachi H, Inoue K, Chi XZ, Lee KY, Nomura S, Lee CW, Han SB. Casual relationship between the loss ofRunx3 expression and gastric cancer. Cell. 2002;109:113-124. [PubMed] |
| 7. | Correa P. Human gastric carcinogenesis: a multistep and multifactorial process-First American Cancer Society Award Lecture on Cancer Epidemiology and Prevention. Cancer Res. 1992;52:6735-6740. [PubMed] |
| 8. | Yasui W, Yokozaki H, Fujimoto J, Naka K, Kuniyasu H, Tahara E. Genetic and epigenetic alter-ations in multistep carcinogenesis of the stomach. J Gastroenterol. 2000;35:111-115. [PubMed] |
| 9. | Hara T, Ooi A, Kobayashi M, Mai M, Yanagihara K, Nakanishi I. Amplification of c-myc, K-sam, and c-met in gastric cancers: detection by fluores-cence in situ hybridization. Lab Invest. 1998;78:1143-1153. [PubMed] |
| 10. | Yu J, Miehlke S, Ebert MP, Hoffmann J, Breidert M, Alpen B, Starzynska T, Stolte Prof M, Malfertheiner P, Bayerdorffer E. Frequency of TPR-MET rearran-gement in patients with gastric carcinoma and in first-degree relatives. Cancer. 2000;88:1801-1806. [PubMed] |
| 11. | Chan AW, Chan MW, Lee TL, Ng EK, Leung WK, Lau JY, Tong JH, Chan FK, To KF. Promoter hyper-methylation of Death-associated protein-kinase gene associated with advance stage gastric cancer. Oncol Rep. 2005;13:937-941. [PubMed] |
| 12. | Lee SW, Tomasetto C, Sager R. Positive selection of candidate tumor-suppressor genes by subtractive hybridization. Proc Natl Acad Sci USA. 1991;88:2825-2829. [PubMed] |
| 13. | Li JY, Zheng HC, Yang L, Xu L, Yang XF, Gao H, Zhang YC, Xin Y. Altered expression of PTEN gene and LOH of its epigenetic microsatellite in gastric carcinoma. Zhonghua Zhongliu Zazhi. 2004;26:389-392. [PubMed] |
| 14. | Sugimura T. Nutrition and dietary carcinogens. Carcinogenesis. 2000;21:387-395. [PubMed] |
| 15. | Saito D. Present state of Japanese intervention trial of H. pylori. Nippon Rinsho. 2003;61:50-55. [PubMed] |
| 16. | Berman SH, Wandersman A. Fear of cancer and knowledge of cancer: a review and proposed relevance to hazardous waste sites. Soc Sci Med. 1990;31:81-90. [PubMed] |
| 17. | Kim JH, Takahashi T, Chiba I, Park JG, Birrer MJ, Roh JK, De Lee H, Kim JP, Minna JD, Gazdar AF. Occurrence of p53 gene abnormalities in gastric carcinoma tumors and cell lines. J Natl Cancer Inst. 1991;83:938-943. [PubMed] |
| 18. | Rege-Cambrin G, Scaravaglio P, Carozzi F, Giordano S, Ponzetto C, Comoglio PM, Saglio G. Karyo-typic analysis of gastric carcinoma cell lines carrying an amplified c-met oncogene. Cancer Genet Cytogenet. 1992;64:170-173. [PubMed] |
| 19. | Miyazono K, Suzuki H, Imamura T. Regulation of TGF-beta signaling and its roles in progression of tumors. Cancer Sci. 2003;94:230-234. [PubMed] |
| 20. | Zaidi SK, Sullivan AJ, van Wijnen AJ, Stein JL, Stein GS, Lian JB. Integration of Runx and Smad regulatory signals at transcriptionally active subnuclear sites. Proc Natl Acad Sci USA. 2002;99:8048-8053. [PubMed] |
| 21. | An C, Choi IS, Yao JC, Worah S, Xie K, Mansfield PF, Ajani JA, Rashid A, Hamilton SR, Wu TT. Prognostic significance of CpG island methylator phenotype and microsatellite instability in gastric carcinoma. Clin Cancer Res. 2005;11:656-663. [PubMed] |
| 22. | Grundei T, Vogelsang H, Ott K, Mueller J, Scholz M, Becker K, Fink U, Siewert JR, Hofler H, Keller G. Loss of heterozygosity and microsatellite instability as predictive markers for neoadjuvant treatment in gastric carcinoma. Clin Cancer Res. 2000;6:4782-4788. [PubMed] |
| 23. | Fang DC, Wang RQ, Yang SM, Yang JM, Liu HF, Peng GY, Xiao TL, Luo YH. Mutation and methylation of hMLH1 in gastric carcinomas with microsatellite instability. World J Gastroenterol. 2003;9:655-659. [PubMed] |
| 25. | Leung SY, Yuen ST, Chung LP, Chu KM, Chan AS, Ho JC. hMLH1 promoter methylation and lack of hMLH1 expression in sporadic gastric carcinomas with high-frequency microsatellite instability. Cancer Res. 1999;59:159-164. [PubMed] |
| 26. | Leung WK, Yu J, Ng EK, To KF, Ma PK, Lee TL, Go MY, Chung SC, Sung JJ. Concurrent hypermethylation of multiple tumor-related genes in gastric carcinoma and adjacent normal tissues. Cancer. 2001;91:2294-2301. [PubMed] |
| 27. | Oshimo Y, Oue N, Mitani Y, Nakayama H, Kitadai Y, Yoshida K, Ito Y, Chayama K, Yasui W. Frequent loss of RUNX3 expression by promoter hypermethylation in gastric carcinoma. Pathobiology. 2004;71:137-143. [PubMed] |
| 28. | Kim TY, Lee HJ, Hwang KS, Lee M, Kim JW, Bang YJ, Kang GH. Methylation of RUNX3 in various types of human cancers and premalignant stages of gastric carcinoma. Lab Invest. 2004;84:479-484. [PubMed] |
| 29. | Waki T, Tamura G, Sato M, Terashima M, Nishizuka S, Motoyama T. Promoter methylation status of DAP-kinase and RUNX3 genes in neoplastic and non-neoplastic gastric epithelia. Cancer Sci. 2003;94:360-364. [PubMed] |