修回日期: 2006-04-10
接受日期: 2006-05-09
在线出版日期: 2006-06-18
目的: 观察缺氧诱导因子-1α(hypoxia inducible factor-1α, HIF-1α)短发夹状RNA(short hairpin RNA, shRNA)对人食管鳞癌细胞系Eca-109中HIF-1α基因的沉默效应, 筛选有效干扰靶点.
方法: 设计合成干扰缺氧诱导因子-1α RNA寡核苷酸片段, 经退火、连接等步骤克隆至线性化的PGCsi真核表达载体上, 测序鉴定. 应用LipofectamineTM 2000将该质粒分别转染Eca-109细胞和293T细胞, 其中Eca-109抗性细胞又经2-4 wk的G418筛选. 荧光显微镜评估转染效率, 荧光定量RT-PCR检测HIF-1α mRNA表达情况, Western blot检测HIF-1α蛋白表达情况.
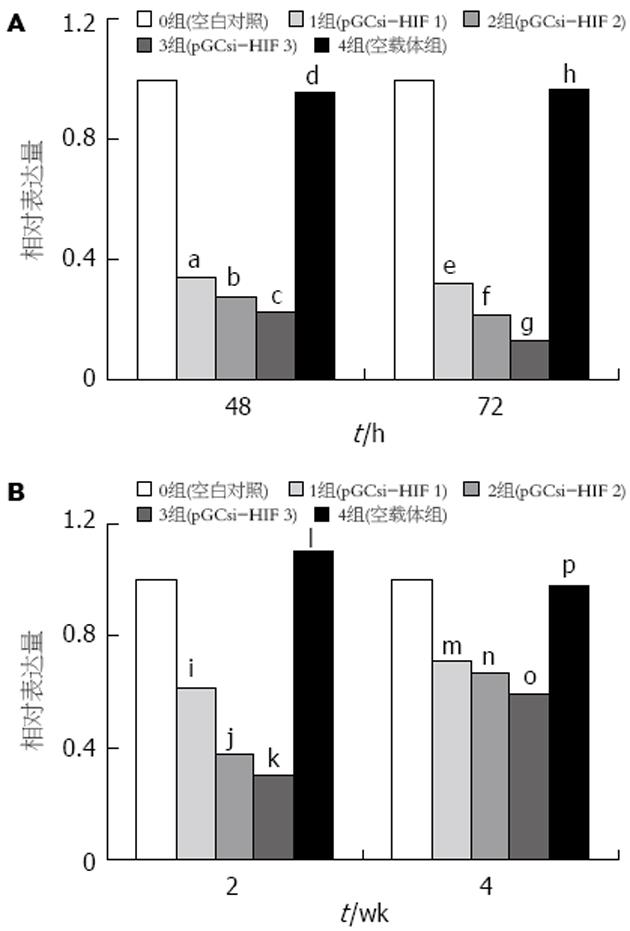
结果: 成功构建了3个靶点的重组质粒pGCsi-H1-shHIF, 293T细胞平均转染效率为约85.4%, Eca-109细胞的平均转染效率约73.2%. 荧光定量RT-PCR和Western blot显示瞬时转染这3种重组质粒的293T细胞和筛选2 wk的Eca-109细胞HIF-1α mRNA和蛋白表达均较对照组有不同程度的下降, 其中2号和3号靶点的干扰效应尤为明显. 在瞬时转染72 h后的293T细胞中, 其抑制率分别达到了78.5%、86.9% (P = 0.000, P = 0.000 vs 72 h空白对照组), 筛选2 wk的Eca-109细胞中, 3号靶点对HIF-1α mRNA的抑制率也达到了69.7% (P = 0.000 vs 2 wk空白对照组).
结论: 重组质粒pGCsi-shHIF能有效抑制食管鳞癌细胞Eca-109中HIF-1α基因的表达, 经不同细胞中的瞬时和稳定转染筛选出了有效干扰靶位.
引文著录: 肖斌, 施瑞华, 杜琰萍, 朱宏, 凌亭生, 张国新, 林艳, 郝波. RNA干扰体外抑制人食管鳞癌细胞Eca-109缺氧诱导因子-1α的表达. 世界华人消化杂志 2006; 14(17): 1654-1661
Revised: April 10, 2006
Accepted: May 9, 2006
Published online: June 18, 2006
AIM: To investigate inhibitory effect of hypoxia inducible factor-1α (HIF-1α) short hairpin RNA (shRNA) on the expression of HIF-1α in human esophageal squamous cancer cell line Eca-109, and to screen suitable interference targets in HIF-1α gene.
METHODS: Three pairs of oligonucleotides targeting HIF-1α were cloned into linearized PGCsi eukaryotic expression vector after annealing. The recombinant plasmids were identified by sequencing. Human esophageal squamous cancer cells Eca-109 and embryonic kidney cells 293T were transfected with the recombinant plasmids, respectively. Fluorescence microscopy was used to observe the effect of RNA interference and evaluate the efficacy of transfection. The transcription of HIF-1α gene and the expression of HIF-1α protein were determined by real-time quantitative reverse transcription-polymerase chain reaction (RT-PCR) and Western blot, respectively.
RESULTS: DNA sequencing showed that three recombinant plasmids (pGCsi-HIF 1, 2, 3) were successfully constructed. The mean efficacy of transfection was 85.4% in 293T cells, and it was 73.2% in Eca-109 cells. The transcription of HIF-1α gene and the expression of HIF-1α protein were down-regulated by RNA interference. The pGCsi-HIF 2 and pGCsi-HIF 3 showed higher interfering efficiency than pGCsi-HIF 1. The inhibition rates of pGCsi-HIF 2 and pGCsi-HIF 3 were 78.5% and 86.9% (P = 0.000, P = 0.000 vs control group) in 293T cells 72 h after transient transfection, respectively. The inhibition rate of pGCsi-HIF 3 was also 69.7% (P = 0.000 vs control group) in Eca-109 cells after screening for 2 wk.
CONCLUSION: The recombinant plasmid pGCsi-shHIF can effectively suppress the expression of HIF-1α in esophageal squamous cancer cells Eca-109, and the efficient interfering targets in HIF-1α gene are screened out.
- Citation: Xiao B, Shi RH, Du YP, Zhu H, Ling TS, Zhang GX, Lin Y, Hao B. RNA interference inhibits expression of hypoxia inducible factor-1α in human esophageal squamous cancer cell line Eca-109 in vitro. Shijie Huaren Xiaohua Zazhi 2006; 14(17): 1654-1661
- URL: https://www.wjgnet.com/1009-3079/full/v14/i17/1654.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i17.1654
缺氧诱导因子-1(hypoxia inducible factor-1, HIF-1)是在哺乳动物体内广泛分布的核蛋白, 是目前公认的在细胞缺氧自适应调节过程中重要的转录因子之一. HIF-1属于bHLH-PAS(basic helix-loop-helix-Per/ARNT/Sim)转录因子家族, 是由α亚基和β亚基组成的异二聚体, 其中HIF-1α的表达与缺氧有关, 是调控下游转录的活性亚基, 其表达随细胞内氧浓度的下降而增加. 对缺氧的耐受是肿瘤细胞能够在实体瘤组织中存活和快速增殖的基础, 在多种肿瘤组织中检出了HIF-1α的过表达[1], 进一步研究发现, HIF-1α还与肿瘤血管生成、能量代谢、离子和儿茶酚胺代谢、肿瘤细胞凋亡、侵袭转移和耐药性的产生有关[2-3]. HIF-1α在核内与HIF-1β结合形成异二聚体, 通过与下游靶基因上含共有序列5'-RCGTG-3'的缺氧反应元件(hypoxia response element, HRE)结合, 促进靶基因如血管内皮生长因子(vascular endothelial growth factor, VEGF)、血红素加氧酶(hemeoxygenase 1, HO-1)、诱导型NO合酶(iducible nitricoxide synthase, iNOS)、转铁蛋白及其受体、胰岛素样生长因子Ⅱ(insulin-like growthing factor Ⅱ, IGF-Ⅱ)及多种糖酵解相关酶等基因的转录, 从而启动一系列的代偿反应, 使肿瘤细胞在缺血缺氧的恶劣环境下存活并保持旺盛的增殖活力. 目前HIF-1α的研究已应用到肿瘤的诊断、预后评估和基因靶向治疗等方面. 我们通过构建针对HIF-1α mRNA的shRNA真核表达载体并转染人食管鳞癌细胞Eca-109和胚肾细胞293T, 以RNA干扰手段抑制HIF-1α的表达, 经实时荧光定量RT-PCR和Western blot分别在核酸和蛋白水平上验证干扰效率, 从而筛选出针对HIF-1α mRNA的有效靶位, 为进一步研究HIF-1α在食管鳞癌发生、发展过程中的作用以及在肿瘤基因治疗中的应用打下基础.
pGCsi-H1-GFP环形质粒(上海吉凯公司)为携带cGFP报道基因并不干扰任何内源基因的短发夹状RNA载体, 同时携有H1启动子及氨苄、新霉素抗性. 人食管鳞癌细胞Eca-109及胚肾细胞293T购自中科院上海细胞所, 细胞培养液为含100 mL/L胎牛血清的DMEM培养液. 胎牛血清及DMEM均为Hyclone公司产品, 感受态大肠杆菌DH5α由本实验室保种. 限制性内切酶BamH Ⅰ、Hind Ⅲ和T4 DNA连接酶为Fermantas公司产品, 脂质体细胞转染试剂LipofectamineTM 2000购自Invitrogen公司, 质粒抽提纯化试剂盒(3SHL Spin Column Plasmid Maxi-Prep Kit V4)购自上海申能博彩生物科技有限公司, 小鼠HIF-1α mAb为Chemicon公司产品(MAB5382), HRP标记羊抗鼠二抗为Rockland公司产品, 鼠抗人的Tubulin-α mAb为Sigma公司产品, PVDF膜购自Roche公司, BCA蛋白定量试剂盒、化学发光试剂盒均购自Pierce公司, 总RNA抽提试剂Tri-Blue购自上海申能博彩生物科技有限公司, RNA逆转录试剂盒(#K1622)、Taq酶(#EP 0404)、10 mmol/L dNTP Mix (#R0191)均为Fermentas公司产品, SYBR Green Ⅰ购自上海捷瑞生物工程有限公司(RS0975). 相差倒置荧光显微镜为Carl Zeiss公司产品, 荧光实时定量PCR仪(RG-3000)为Corbett Research公司产品, 蛋白电泳及转膜系统为Bio-Rad公司产品.
1.2.1 HIF-1α shRNA表达载体的构建、扩增与纯化: 根据人HIF-1α基因序列(GenBank No. NM001530)设计合成3对HIF-1α-siRNA寡核苷酸片段, 序列分别为: 1号靶正义链: 5'-GATCCCGAGGAAGAACTATGAACATAA TTCAAGAGA TTATGTTCATAGTTCTTCCTC TTTTTGGAT-3', 反义链: 5'-AGCTATCCAAAAAGAGGAAGAACTATGAACATAA TCTCTTGAA TTATGTTCATAGTTCTTCCTCGG-3'; 2号靶正义链: 5'-GATCCC GCACAGGCCACATTCACGTATA TTCAAGAGA TATACGTGAATGTGGCCTGTGCTTTTTGGAT-3', 反义链: 5'-AGCTATCCAAAAAGCACAGGCCACATTCACGTATA TCTCTTGAA TATACGTGAATGTGGCCTGTGCGG-3'; 3号靶正义链: 5'-GATCCC GACTGATGACCAGCAACTTGA TTCAAGAGA TCAAGTTGCTGGTCATCAGTC TTTTTGGAT-3', 反义链: 5'-AGCTATCCAAAAAGACTGATGACCAGCAACTTGA TCTCTTGAA TCAAGTTGCTGGTCATCAGTCGG-3'(斜体为LOOP). 每对寡核苷酸两端分别带有BamH Ⅰ和Hind Ⅲ酶切位点. 将各1 μL的正向和反向siRNA寡核苷酸片段与48 μL的退火缓冲液混合; 混合物在90℃温育4 min, 70℃温育10 min. 缓慢冷却退火的寡核苷酸片段至10℃. 同时用Hind Ⅲ和BamH Ⅰ限制酶将1 μL的pGCsi载体线性化. 将2 μL退火后的siRNA寡核苷酸片段加到T4 DNA连接酶缓冲液中, 接着再加1 μL pGCsi载体、5 μL DEPC水、1 μL T4 DNA连接酶, 室温下温育过夜. 构建成的质粒分别称为: pGCsi-HIF 1, pGCsi-HIF 2和pGCsi-HIF 3. 常规方法将重组质粒及pGCsi空载体转化入感受态宿主细胞DH5α, 细菌在含氨苄青霉素的LB平板37℃培养过夜后挑单克隆, 接种至LB培养基(10 g/L胰蛋白酶胨, 5 g/L酵母提取物, 10 g/L氯化钠), 37℃振摇培养过夜, 次日按说明书操作以试剂盒进行质粒纯化抽提, 测浓度后保存于-20℃备用. 部分质粒送英骏公司测序.
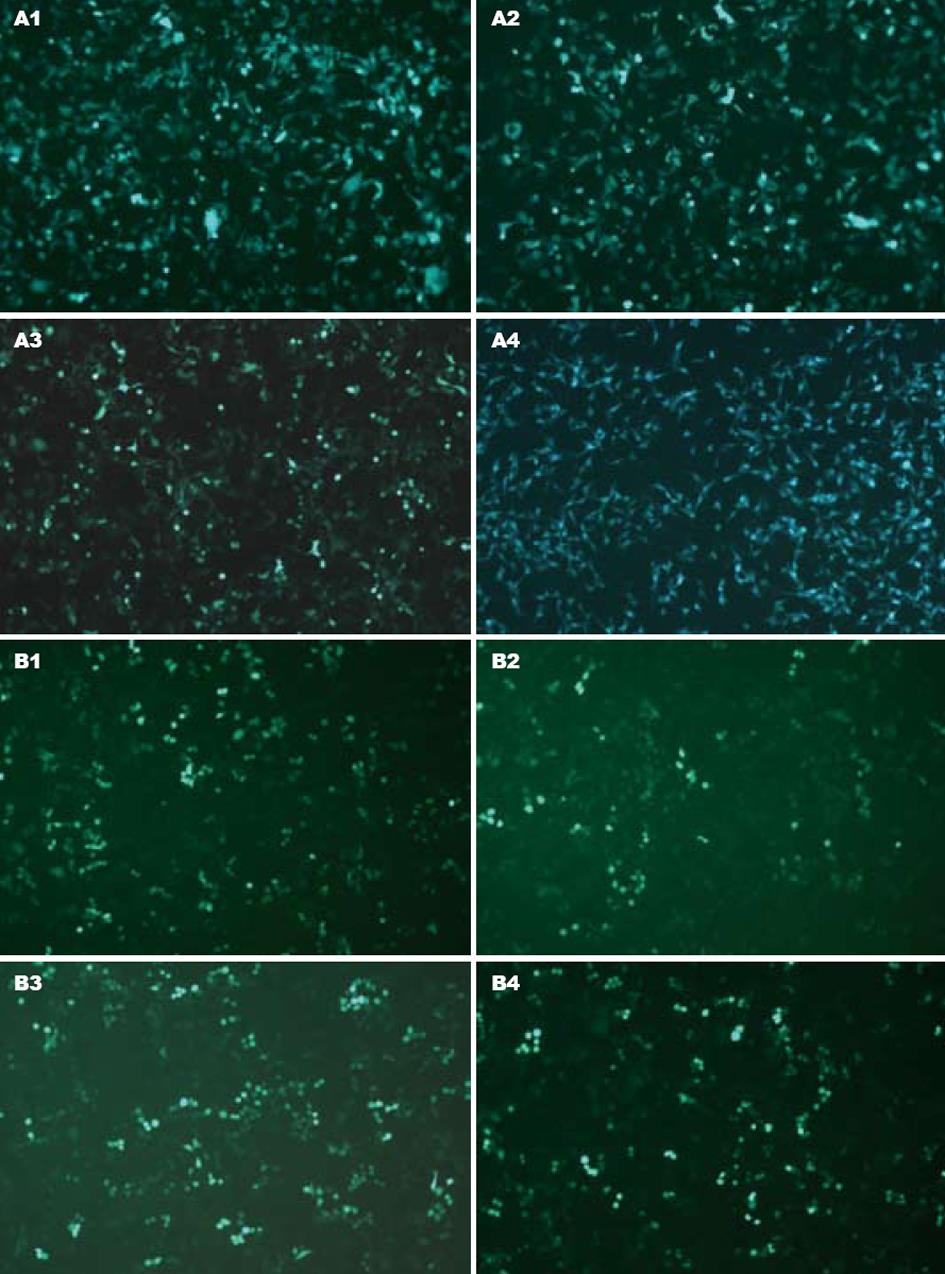
1.2.2 细胞转染、培养和观察: 两种细胞各分5组, 分别为0组空白对照组、1组转染pGCsi-HIF 1质粒组、2组转染pGCsi-HIF 2质粒组、3组转染pGCsi-HIF 3质粒组、4组转染空载体组. 转染前一天取对数生长期的Eca-109细胞及293T细胞2.5×105个接种于35 mm培养皿, 培养于不含抗生素的DMEM培养液(含100 mL/L胎牛血清), 次日观察细胞长至约90%聚合开始转染. 取质粒4 μg, 脂质体LipofectamineTM 2000 10 μL稀释于不含抗生素的DMEM, 室温孵育20 min后加入培养皿, 培养于37℃培养箱, CO2浓度为50 mL/L. 6 h后更换新鲜的含100 mL/L胎牛血清, 100 kU/L青霉素, 100 mg/L链霉素的DMEM培养液. 其后每2 d换液. 293T细胞做瞬时转染, 分别于48, 72 h收集细胞, Eca-109细胞转染后24 h按1∶9的比例传代, 48 h后开始用含800 mg/L G418的选择性培养液进行筛选, 2 wk后挑取克隆在400 mg/L G418培养液中继续传代培养2-4 wk, 分别收集该时段细胞进行检测. 细胞转染后48 h在荧光显微镜下观察绿色荧光显色情况并照相, 镜下分别计算10个40倍视野下发绿色荧光的细胞占总细胞数的百分率作为细胞转染效率的近似值.
1.2.3 实时定量PCR: 收集各组细胞, 采用一步法提取细胞总RNA(Tri-blue), 随后取RNA 2.5 μg按试剂盒说明书以小鼠M-MuLV逆转录酶进行逆转录, 转录后的cDNA中取1个样品进行梯度稀释作为绘制标准曲线的样本, 与其余样品同时进行QPCR, 在25 μL体系中加入HIF-1α的上、下游引物各0.12 μmol/L, Taq酶0.625 U, dNTP Mix 0.2 mmol/L, SYBR Green Ⅰ终浓度为0.3×, 以β-actin作为内参照同时扩增, 每样品均做复孔, 另有2管为不加模板的空白对照. 94℃ 5 min预变性, 94℃ 20 s, 58℃ 20 s, 72℃ 30 s, 80℃ 10 s读板, 扩增35个循环, 最后72℃延伸10 min. 应用Rotor-Gene Real-Time Analysis Software 6.0软件分析数据得到CT值, 绘制标准曲线并根据公式amount of target = 2-△△CT计算得出相对样品模板量[4]. 所用引物以Primer-5软件根据引物设计要求自行设计, 交由上海申能博彩生物科技有限公司合成, HIF-1α上游引物: 5'-GCA AGA CTT TCC TCA GTC GAC ACA-3', 下游引物: 5'-GCA TCC TGT ACT GTC CTG TGG TGA-3'; β-actin上游引物: 5'-CCA GCC ATG TAC GTT GCT ATC-3', 下游引物: 5'-CAG GTC CAG ACGCAGGATGGC-3'. 扩增结束后取PCR产物10 μL上样, 10 g/L琼脂糖凝胶电泳, EB染色, Tanon凝胶成像系统摄片并分析结果.
1.2.4 Western blot: 收细胞, 冰PBS洗2次, 加入细胞裂解液[20 mmol/L Tris (pH 7.5), 4 mmol/L EDTA, 20 g/L SDS] 150 μL裂解细胞, 超声破碎后12 000 r/min, 离心5 min, 取上清, 用BCA蛋白定量试剂盒检测总蛋白浓度后, 以蛋白上样量60 μg, 行稳流100 g/L SDS-PAGE电泳, 稳压冰浴电转至PVDF膜, 50 g/L脱脂奶粉室温封闭1 h后TBST洗膜, 加入鼠抗HIF-1α mAb(工作浓度1∶400) 4℃孵育过夜. 次日TBST洗膜后再加HRP标记的羊抗鼠二抗(工作浓度为1∶10 000)室温孵育1 h, TBST洗膜. ECL化学发光法显影, 暗室曝光. 以α-Tubulin(单抗工作浓度为1∶4000)为内参照.
统计学处理 所得数据均以mean±SD表示, 采用SPSS 11.0软件进行统计分析. 单因素方差分析, LSD法检验各组与对照组间差异, P<0.05认为两组数据存在显著性差异. 感光胶片条带以Tanon Gis软件进行灰度值分析, 以目的条带与内参照α-Tubulin条带的比值代表目的基因蛋白的表达水平.
质粒抽提后送上海英骏生物技术有限公司测序, 测序结果证实与设计相符, 插入shRNA序列100%正确(测序结果略).
转染后48 h于倒置荧光显微镜下观察绿色荧光表达情况(图1), 按发绿色荧光的细胞占总细胞数的百分率计算, 293T细胞转染空载体及3个重组质粒的平均转染效率为约85.4%, Eca-109细胞的平均转染效率约73.2%.
转染3种重组质粒的细胞HIF-1α mRNA相对表达量均小于1, 认为3种shRNA在2细胞中均发挥了不同程度的干扰作用, 其中pGCsi-HIF 2和pGCsi-HIF 3的干扰效应尤为明显, 在瞬时转染72 h后的293T细胞中, 抑制率分别达到了78.5%, 86.9%(P均<0.01, fP = 0.000, gP = 0.000 vs 72 h空白对照组, 图2A), 在转染后经G418筛选2 wk的Eca-109细胞中, 质粒pGCsi-HIF 3对HIF-1α mRNA的抑制率也达到了69.7%(P<0.01, kP = 0.000 vs 2 wk空白对照组, 图2B), 接近70%. 而在筛选4 wk的Eca-109细胞中, HIF-1α mRNA的表达似乎有不同程度的上升, 相对于同时间段的空白对照细胞而言, 3种质粒的抑制率仅为29.3%, 33.0%, 40.4%(P均<0.05, mP = 0.016, nP = 0.008, oP = 0.002 vs 4 wk对照组, 图2B). 此外, 转染空载体的各组细胞HIF-1α mRNA表达量均接近1, 与空白对照组无显著性差异(dP = 0.525, hP = 0.412, iP = 0.372, pP = 0.848), 说明转染空载体细胞中未发生针对HIF-1α mRNA的干扰效应.
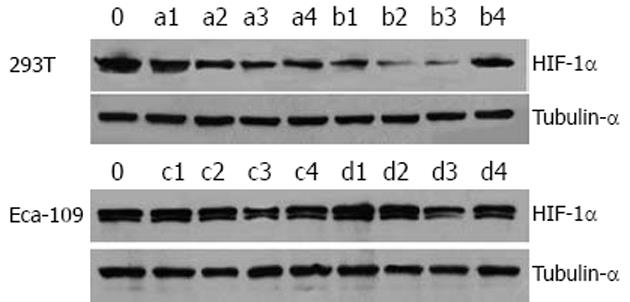
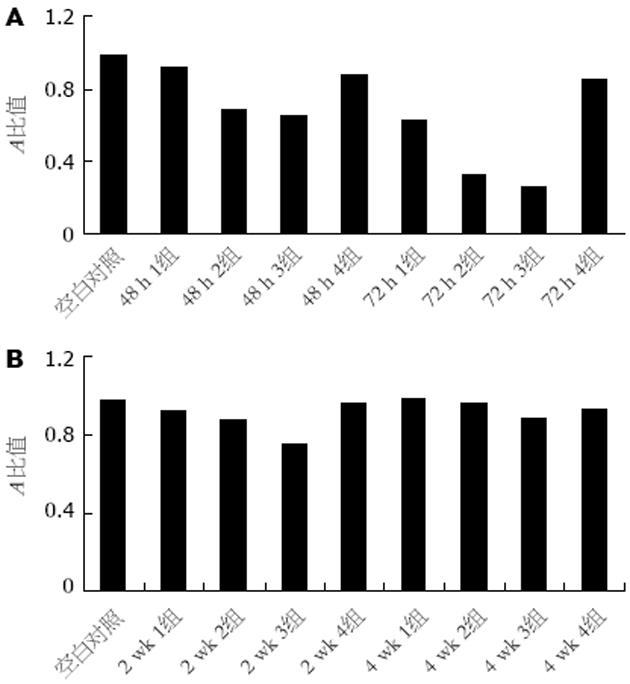
在瞬时转染的293T细胞中转染pGCsi-HIF 2, pGCsi-HIF 3两种质粒的实验组蛋白表达量较空白对照均有不同程度的低减, 其中转染后72 h的HIF-1α蛋白表达水平尤其低下, 此外, 在经G418筛选的Eca-109细胞中, 转染pGCsi-HIF 3质粒的细胞HIF-1α蛋白表达较空白对照组也有一定程度的降低, 但这种抑制效应不如瞬时转染的293T细胞中明显, 同时检测的仅转染空质粒的对照组(4组)和空白对照组(0组)相比HIF-1α蛋白表达未见明显差异(P>0.05, 图3). 灰度扫描结果与上述结果相符合(图4). 这说明重组载体pGCsi-HIF 2、pGCsi-HIF 3通过RNA干扰有效抑制了HIF-1α蛋白的表达, 其中pGCsi-HIF 3的干扰效应尤其强烈. 结果与荧光实时定量PCR一致.
我国是食管癌的高发区, 死亡率居世界第一, 病理类型中鳞癌占绝大多数, 约90%左右. 食管癌确诊时往往已届晚期, 大多已无手术机会, 放化疗对鳞癌的疗效甚差, 故而通常治疗困难, 预后不佳. 因此, 研究发病机制、提高早期诊断率、寻找新的治疗途径和预后判断标准就成为食管癌防治研究的关键. 作为生长迅速的实体肿瘤, 食管癌组织内常常存在缺氧微环境和代谢紊乱状态, 在缺氧数小时内, 肿瘤细胞即可分泌大量促血管生成因子如血管内皮生长因子(VEGF)等, 从而使血管内皮细胞大量增殖, 肿瘤组织中新生血管生成, 同时细胞内无氧代谢增加以使其在恶劣环境下得以生存并保持增殖活性, 在此过程中, 缺氧诱导因子-1α(HIF-1α)发挥了重要作用[5-6], 目前认为HIF-1α是调节VEGF转录最重要的核转录因子之一[7]. 研究表明, HIF-1α在人类大多数肿瘤的缺氧部位表达明显上调[8]. 不仅如此, HIF-1α在细胞代谢、转录调控、凋亡抑制、侵袭性和耐药性等肿瘤相关生物学行为中都扮演着重要角色. 多种抑癌基因失活或原癌基因激活也会诱导HIF-1α的高表达[9]. 在结构上HIF-1α具有氧浓度依赖性功能域(ODD), 常氧下该功能域与E3泛醌连接酶结合通过泛素-蛋白酶体途径将HIF-1α迅速降解, 半衰期不到5 min[10], 而缺氧时, 细胞内脯氨酸羟化酶(prolyl hydroxylase domain, PHDs)受抑制, HIF-1α的泛素降解途径受阻, HIF-1α在核内积聚, 同时缺氧也可通过PI3K和MAPK途径将HIF-1α磷酸化, 增加其稳定性, 从而使HIF-1α蛋白表达增加, 并与b亚单位形成有活性的异二聚体HIF-1, 再与下游靶基因上缺氧反应元件(HRE)结合, 调控下游60余种靶基因转录, 最终启动各种生理病理过程[11-13]. 在不同肿瘤组织中进行的研究表明, HIF-1α不仅在肿瘤的局部和转移灶中高表达, 而且与肿瘤的良恶性、发生进展演进过程、侵袭、转移能力及预后状况直接有关[1-2,14], 此外, 在基因治疗方面, Koukourakis et al[15]在早期食管癌中进行的研究发现HIF-1α在早期癌组织中亦有明显升高, 高表达HIF-1α者完全缓解(CR)率低, 同时这种升高与癌肿对光动力治疗的敏感性呈正相关, Sun et al[16]通过反义HIF-1α的作用完全而持久地消除了直径0.1 cm的肿瘤, 并诱导了NK细胞依赖的杀瘤效应. 业已证明, 针对肿瘤多种信号转导通路的抑癌剂大多间接抑制了HIF-1的作用[17]. HIF-1α在肿瘤的病理过程中起着"中心枢纽"和"最后通路"的作用, 进一步研究HIF-1α在肿瘤细胞中的表达及其功能将为其在肿瘤治疗、早期诊断和预后判断的应用奠定基础[18].
RNA干扰是生物进化过程中基因组水平上的免疫监控机制[19], 他作为一种特异性基因沉默技术, 正越来越多地在反向遗传学研究、细胞信号转导通路研究和某些疾病, 如病毒感染、肿瘤及多种遗传相关性疾病的发病机制和治疗学研究中发挥着重要作用. 相对于传统上反义寡核苷酸和核酶等基因沉默手段来说, 由于siRNA具有RNA双链结构和3'端双核苷酸悬垂, 较难被核酶降解, 所以RNA干扰具有更好的稳定性和更优越的抑制效果[20], 是高通量基因功能研究的得力工具. 不仅如此, RNA干扰的研究为疾病的基因治疗开拓了一个全新的途径, 通过抑制过表达的致瘤基因、干扰细胞周期调节蛋白E及其相关基因阻遏细胞分裂以及通过抑制抗凋亡基因促进细胞凋亡可能对肿瘤的治疗带来重大突破, 以RNA干扰抑制耐药基因及多药耐药基因以改善肿瘤对化疗的反应性也有利于肿瘤的治疗[21]. 此外, 也有利用RNA干扰手段抑制肿瘤细胞的侵袭和转移的研究[22-24]. 体内RNA干扰的难点在于如何将siRNA安全输入并富集于有效部位, 在这方面有学者做了一些有意义的尝试, Zhang et al[25]使用带有转铁蛋白受体和胰岛素受体抗体的系统, 将针对表皮生长因子受体(EGFR)的shRNA载体通过血脑屏障和肿瘤细胞膜转入小鼠脑胶质细胞瘤中, 结果使肿瘤中EGFR的表达明显下调, 干扰后进展期颅内肿瘤小鼠的生存期较对照组延长了88%. 至今已有多项针对肿瘤的干扰实验取得明显效果, 如对胃癌、肝癌、白血病、淋巴瘤、口腔癌、脑癌的基因治疗和逆转化疗敏感性的研究[26-30], 但目前在食管鳞癌细胞中的干扰报道尚少. 我们设计了3对HIF-1α-siRNA寡核苷酸片段, 并通过分子克隆技术成功构建了质粒pGCsi-HIF 1、pGCsi-HIF 2和pGCsi-HIF 3. 经脂质体LipofectamineTM 2000转染入食管鳞癌细胞系Eca-109挑克隆筛选稳定转染细胞, 同时选择转染效率较高的胚肾细胞293T进行瞬时转染靶点筛选. 研究结果显示, 3种shRNA在两细胞中均发挥了不同程度的干扰作用, 其中pGCsi-HIF 2和pGCsi-HIF 3对HIF-1α发生了明显抑制, HIF-1α的表达量较空白对照组和转染空载体组的差异有统计学意义, pGCsi-HIF 3差异尤为明显, 在筛选2 wk的Eca-109细胞中也出现了这种差异. 这表明2号、3号靶位可能是有效的, 而3号靶位的干扰效率是这3个靶点当中最高的. 在筛选4 wk的Eca-109细胞中对HIF-1α基因沉默效应似乎出现了减弱的现象, 我们分析可能与挑克隆的方法有关, 大平皿挑细胞团的方法得到的并非纯合克隆, 其中部分虽带有抗生素抗性基因而未敲除HIF-1α基因的细胞经较长时间的培养呈现出生长优势, 影响了HIF-1α沉默细胞的增殖, 导致总体水平上核酸和蛋白表达水平的增加. 目前我们已另采用有限稀释法进行单克隆化操作筛选稳定转染细胞株, 进一步对HIF-1α基因在食管鳞癌发生、发展和基因治疗中的作用进行研究. 我们构建的HIF1-α短发夹状RNA表达载体在食管鳞癌细胞系Eca-109中特异性地抑制了HIF1-α基因的表达, 这为今后深入研究HIF1-α在食管鳞癌及其他肿瘤发病、进展机制和基因治疗的探索打下了基础.
HIF-1α与肿瘤的增殖、血管生成、代谢、侵袭转移等多种生物学行为有关, 多种肿瘤组织中存在HIF-1α的高表达,现已成为肿瘤诊断、基因治疗研究的热点之一. 但通过分子生物学手段在食管鳞癌中研究HIF-1α功能的研究尚不多见.
RNA干扰是近年来兴起的高效、高特异性基因沉默技术, 该技术广泛应用于基因功能研究. 目前食管鳞癌中的干扰报道不多.
HIF-1α基因研究微观上已进入亚分子水平(参考文献[11-13]), 宏观上在提高早期肿瘤检出率、提高对放、化疗、光动力治疗敏感性及肿瘤基因治疗等方面均有积极的尝试(参考文献[15-17]). 而应用RNA干扰手段进行基因治疗的研究更是层出不穷( 参考文献[19-30]).
利用RNA干扰手段研究食管鳞癌中HIF-1α基因功能, 抑制效应高,特异性好, 该手段将为食管癌相关基因研究开辟广阔前景.
本研究构建了3种针对HIF-1α基因的质粒载体并验证了其在食管鳞癌细胞中对该基因的抑制效应, 这为进一步研究食管鳞癌中HIF-1α基因功能和以该基因为靶点的基因治疗提供了技术平台.
该文采用RNA干扰技术, 研究其对食管鳞癌细胞Eca-109, HIF-1a表达的抑制作用,为研究食管癌中该基因的功能奠定了基础, 选题新颖, 方法学先进,结果可信, 有较强的科学性.
电编: 张敏 编辑:潘伯荣
| 1. | Zhong H, De Marzo AM, Laughner E, Lim M, Hilton DA, Zagzag D, Buechler P, Isaacs WB, Semenza GL, Simons JW. Overexpression of hypoxia-inducible factor 1alpha in common human cancers and their metastases. Cancer Res. 1999;59:5830-5835. [PubMed] |
| 2. | Semenza GL. Expression of hypoxia-inducible factor 1: mechanisms and consequences. Biochem Pharmacol. 2000;59:47-53. [PubMed] [DOI] |
| 3. | Vaupel P. The role of hypoxia-induced factors in tumor progression. Oncologist. 2004;9 Suppl 5:10-17. [PubMed] [DOI] |
| 4. | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 2001;25:402-408. [PubMed] [DOI] |
| 5. | Mizukami Y, Li J, Zhang X, Zimmer MA, Iliopoulos O, Chung DC. Hypoxia-inducible factor-1-indepen-dent regulation of vascular endothelial growth factor by hypoxia in colon cancer. Cancer Res. 2004;64:1765-1772. [PubMed] [DOI] |
| 6. | Harris AL. Hypoxia-a key regulatory factor in tumour growth. Nat Rev Cancer. 2002;2:38-47. [DOI] |
| 7. | Sanchez-Elsner T, Botella LM, Velasco B, Corbi A, Attisano L, Bernabeu C. Synergistic cooperation between hypoxia and transforming growth factor-beta pathways on human vascular endothelial growth factor gene expression. J Biol Chem. 2001;276:38527-38535. [PubMed] [DOI] |
| 8. | Pugh CW, Ratcliffe PJ. Regulation of angiogenesis by hypoxia: role of the HIF system. Nat Med. 2003;9:677-684. [PubMed] [DOI] |
| 9. | Zhong H, Chiles K, Feldser D, Laughner E, Hanrahan C, Georgescu MM, Simons JW, Semenza GL. Modulation of hypoxia-inducible factor 1alpha expression by the epidermal growth factor/phosphatidylinositol 3-kinase/PTEN/AKT/FRAP pathway in human prostate cancer cells: implications for tumor angiogenesis and therapeutics. Cancer Res. 2000;60:1541-1545. [PubMed] |
| 10. | Huang LE, Arany Z, Livingston DM, Bunn HF. Activation of hypoxia-inducible transcription factor depends primarily upon redox-sensitive stabiliza-tion of its alpha subunit. J Biol Chem. 1996;271:32253-32259. [PubMed] [DOI] |
| 11. | Mazure NM, Brahimi-Horn MC, Berta MA, Benizri E, Bilton RL, Dayan F, Ginouves A, Berra E, Pouyssegur J. HIF-1: master and commander of the hypoxic world. A pharmacological approach to its regulation by siRNAs. Biochem Pharmacol. 2004;68:971-980. [PubMed] [DOI] |
| 12. | Lee JW, Bae SH, Jeong JW, Kim SH, Kim KW. Hypoxia-inducible factor (HIF-1)alpha: its protein stability and biological functions. Exp Mol Med. 2004;36:1-12. [PubMed] [DOI] |
| 13. | Maynard MA, Qi H, Chung J, Lee EH, Kondo Y, Hara S, Conaway RC, Conaway JW, Ohh M. Multiple splice variants of the human HIF-3 alpha locus are targets of the von Hippel-Lindau E3 ubiquitin ligase complex. J Biol Chem. 2003;278:11032-11040. [PubMed] [DOI] |
| 14. | Swinson DE, Jones JL, Cox G, Richardson D, Harris AL, O'Byrne KJ. Hypoxia-inducible factor-1 alpha in non small cell lung cancer: relation to growth factor, protease and apoptosis pathways. Int J Cancer. 2004;111:43-50. [PubMed] [DOI] |
| 15. | Koukourakis MI, Giatromanolaki A, Skarlatos J, Corti L, Blandamura S, Piazza M, Gatter KC, Harris AL. Hypoxia inducible factor (HIF-1a and HIF-2a) expression in early esophageal cancer and response to photodynamic therapy and radiotherapy. Cancer Res. 2001;61:1830-1832. [PubMed] |
| 16. | Sun X, Kanwar JR, Leung E, Lehnert K, Wang D, Krissansen GW. Gene transfer of antisense hypoxia inducible factor-1 alpha enhances the therapeutic efficacy of cancer immunotherapy. Gene Ther. 2001;8:638-645. [PubMed] [DOI] |
| 17. | Semenza GL. Development of novel therapeutic strategies that target HIF-1. Expert Opin Ther Targets. 2006;10:267-280. [PubMed] [DOI] |
| 18. | Semenza GL. HIF-1 and tumor progression: patho-physiology and therapeutics. Trends Mol Med. 2002;8:S62-67. [PubMed] [DOI] |
| 19. | Hutvagner G, Zamore PD. RNAi: nature abhors a double-strand. Curr Opin Genet Dev. 2002;12:225-232. [PubMed] [DOI] |
| 21. | Izquierdo M. Short interfering RNAs as a tool for cancer gene therapy. Cancer Gene Ther. 2005;12:217-227. [PubMed] [DOI] |
| 22. | Patel KN, Maghami E, Wreesmann VB, Shaha AR, Shah JP, Ghossein R, Singh B. MUC1 plays a role in tumor maintenance in aggressive thyroid carcinomas. Surgery. 2005;138:994-1001; discussion 1001-1002. [PubMed] [DOI] |
| 23. | Wyatt CA, Geoghegan JC, Brinckerhoff CE. Short hairpin RNA-mediated inhibition of matrix metalloproteinase-1 in MDA-231 cells: effects on matrix destruction and tumor growth. Cancer Res. 2005;65:11101-11108. [PubMed] [DOI] |
| 24. | Celetti A, Testa D, Staibano S, Merolla F, Guarino V, Castellone MD, Iovine R, Mansueto G, Somma P, De Rosa G. Overexpression of the cytokine osteopontin identifi-es aggressive laryngeal squamous cell carcinomas and enhances carcinoma cell proliferation and invasiveness. Clin Cancer Res. 2005;11:8019-8027. [PubMed] [DOI] |
| 25. | Zhang Y, Zhang YF, Bryant J, Charles A, Boado RJ, Pardridge WM. Intravenous RNA interference gene therapy targeting the human epidermal growth factor receptor prolongs survival in intracranial brain cancer. Clin Cancer Res. 2004;10:3667-3677. [PubMed] [DOI] |
| 26. | Chen XH, Lan B, Qu Y, Zhang XQ, Cai Q, Liu BY, Zhu ZG. Inhibitory effect of Polo-like kinase 1 depletion on mitosis and apoptosis of gastric cancer cells. World J Gastroenterol. 2006;12:29-35. [PubMed] [DOI] |
| 27. | Huang SL, Wu Y, Yu H, Zhang P, Zhang XQ, Ying L, Zhao HF. Inhibition of Bcl-2 expression by a novel tumor-specific RNA interference system increases chemosensitivity to 5-fluorouracil in Hela cells. Acta Pharmacol Sin. 2006;27:242-248. [PubMed] [DOI] |
| 28. | Damm-Welk C, Fuchs U, Wossmann W, Borkhardt A. Targeting oncogenic fusion genes in leukemias and lymphomas by RNA interference. Semin Cancer Biol. 2003;13:283-292. [PubMed] [DOI] |
| 29. | Kudo Y, Kitajima S, Ogawa I, Kitagawa M, Miyauchi M, Takata T. Small interfering RNA targeting of S phase kinase-interacting protein 2 inhibits cell growth of oral cancer cells by inhibiting p27 degradation. Mol Cancer Ther. 2005;4:471-476. [PubMed] |