修回日期: 2006-01-19
接受日期: 2006-01-25
在线出版日期: 2006-05-28
目的: 克隆HBeAg结合蛋白未知功能新基因HBeBP4的剪切体基因HBeBP4A, 构建其真核表达载体, 并应用生物信息学初步探讨其结构及功能.
方法: 应用PCR技术从HepG2细胞提取的cDNA扩增HBeBP4基因的剪切体基因HBeBP4A, 选用pGEM-Teasy载体进行TA克隆, 通过PCR, 限制性酶切分析及测序进行鉴定, 再将其亚克隆到真核表达载体pcDNATM3.1/myc-HisA, 通过PCR, 限制酶切分析进行鉴定, 并应用生物信息学初步分析其物理化学性质, 蛋白质结构和功能.
结果: 成功扩增出HBeBP4剪切体基因, 命名为HBeBP4A. 利用生物信息学分析推定其ORF为1104个核苷酸(nt), 编码产物为367个氨基酸残基(aa).
结论: 发现了HBeAg结合蛋白4(HBeBP4)基因的剪切体HBeBP4A, 构建了pcDNATM3.1/myc-HisA真核表达载体.
引文著录: 张健康, 郭江, 成军, 王丹琼, 伦永志, 赵龙凤, 蓝贤勇, 洪源, 毛羽. HBeAg结合蛋白4基因剪切体HBeBP4A的克隆化. 世界华人消化杂志 2006; 14(15): 1462-1465
Revised: January 19, 2006
Accepted: January 25, 2006
Published online: May 28, 2006
AIM: To clone a new gene, binding protein 4 spliced variant HBeBP4A of hepatitis B virus e antigen (HBeAg), and to explore its function and structure by bioinformatical analysis.
METHODS: HBeBP4A was amplified by reverse transcription-polymerase chain reaction (RT-PCR) using HepG2 cDNA as template and inserted into pGEM-T easy vector by TA cloning. Recombinant eukaryotic expression vector (pcDNATM3.1/myc-HisA-HBeBP4A) was constructed by subcloning followed by restriction enzyme digestion analysis and sequencing. Bioinformatical methods were used to analyze its possible physical and chemical characteristics, structure, and function.
RESULTS: Spliced form of HBeBP4, named HBeBP4A, was amplified successfully by RT-PCR from HepG2 cDNA. Bioinformatical analysis showed that its new open reading frame (ORF) is 1104 bp in length and translated a protein containing 367 amino acid residues.
CONCLUSION: A new gene, spliced variant HBeBP4A of HBeAg binding protein 4, is recognized, and its recombinant eukaryotic expression vector is constructed.
- Citation: Zhang JK, Guo J, Cheng J, Wang DQ, Lun YZ, Zhao LF, Lan XY, Hong Y, Mao Y. Cloning of spliced variant HBeBP4A of hepatitis B virus e antigen binding protein 4. Shijie Huaren Xiaohua Zazhi 2006; 14(15): 1462-1465
- URL: https://www.wjgnet.com/1009-3079/full/v14/i15/1462.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i15.1462
HBeAg是核壳蛋白的分泌型, 研究发现HBeAg对HBV的感染和复制无重要作用, 有学者提出他可能具有免疫调节的作用而导致HBV感染的持续[1-3]. HBeAg可阻断细胞毒性T淋巴细胞优先清除对HBcAg特异的Th1细胞及Th1细胞介导的抗-HBc抗体反应, 使免疫应答转换为Th2细胞亚型, 使HBV逃避免疫清除, 致感染慢性化[4-7]. 但HBeAg与肝细胞之间的相互作用机制尚不清楚, 此方面的研究较少. 陆荫英 et al[8]利用酵母双杂交系统3, 发现了HBeAg结合蛋白4-HBeBP4, 为探讨其生物学功能及其作用, 我们应用PCR技术对其进行扩增与克隆, 构建其真核表达载体, 从寻找HBeAg与肝细胞间的相互作用蛋白入手, 对其生物学功能进行预测分析, 为今后更加广泛深入地研究新基因的生物学功能打下基础, 并希望在研究HBeAg的确切生物学功能方面有所发现[9-11].
肝母细胞瘤细胞系HepG2及大肠杆菌DH5α为本室保存; Taq酶购于鼎国生物公司; T4 DNA连接酶, pGEM-Teasy, IPTG及X-gal购于Promega公司; 玻璃奶回收试剂盒购自博大泰克公司; EcoRⅤ, HindⅢ, BamHⅠ, SacⅠ, NheⅠ, DNA Marker DL15000和DL2000购自TaKaRa生物公司; pcDNATM3.1/myc-HisA由北京大学人民医院肝病研究所魏来教授惠赠; 引物合成及DNA序列测定由Invitrogen公司完成. 陆荫英 et al[8]利用酵母双杂交系统3, 构建了pGBKT7-HBeAg诱饵表达质粒, 在预转化的人肝cDNA文库中"钓"出与HBeAg有相互作用的蛋白基因39种, 5种为未知基因, 在GenBank中未发现与之同源的表达基因序列, 根据基因起始密码子的Kozak规则和终止密码子下游保守的多聚腺苷酸信号序列, 电子拼接推定未知基因的开放读码框架获得相应的全长编码基因, 将其中之一命名为HBeAg结合蛋白4(HBeBP4), 开放读码框长1374 bp, 编码457个氨基酸残基, 并获得GenBank注册号AY134474, 目前该基因在GenBank中已升级为GLYCTK基因, GenBank注册号NM_145262.
根据HBeBP4的全长编码基因, 设计序列特异性的含有限制性核酸内切酶EcoRⅤ/HindⅢ(下划线序列)位点的引物P1、P2. P1: 5'-CGG ATA TCA TGG CTG CAG CCC TGC AG-3', P2: 5'-CGA AGC TTC CGA GGC CGC AGG AAC AAG-3', 提取HepG2细胞的总RNA进行反转录, 以反转录产物cDNA为模板进行PCR, PCR参数如下: 95 ℃ 6 min预变性, 94 ℃ 50 s变性, 66 ℃退火1 min, 72 ℃延伸2 min, 共35个循环, 72 ℃延伸10 min, 最后4 ℃保存. 将PCR产物在10 g/L琼脂糖凝胶中电泳, 切取目的片段, 玻璃奶法回收PCR产物, 与pGEM-Teasy载体16 ℃连接过夜, 转化进DH5α感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养12-16 h. 挑取阳性菌落, 增菌. 碱裂解法提取质粒进行EcoRⅤ/HindⅢ与BamHⅠ/SacⅠ两次双酶切鉴定, 证明目的基因片段已正确插入pGEM-Teasy载体中后送测序. 选择经酶切与测序鉴定的pGEM-Teasy-HBeBP4A重组质粒和pcDNATM3.1/myc-His A真核表达空质粒分别进行EcoRⅤ/HindⅢ双酶切反应, 10 g/L琼脂糖凝胶电泳纯化回收并在16 ℃以T4连接酶连接两个目的基因片段, 按上述方法常规转化感受态细菌, 提取pcDNATM3.1/myc-HisA-HBeBP4A重组质粒作EcoRⅤ/HindⅢ与BamHⅠ/SacⅠ两次双酶切鉴定. 利用在线http://www.ebi.ac.uk/clustalw/软件将Teasy vector所测基因序列与HBeBP4及其升级基因GLYCTK进行序列比对, 确定该序列为HBeBP4基因的剪切体; 然后利用NCBI数据库中BLASTn比对分析, 确定剪切体的新开放读码框(ORF), 命名为HBeBP4A基因, 并在NCBI中登陆获取GenBank注册号.
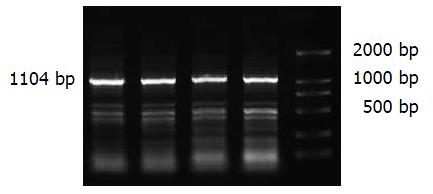
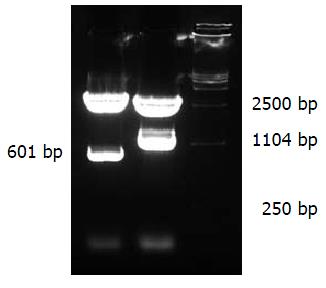
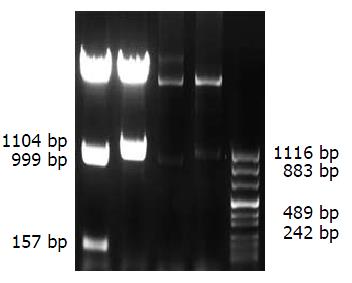
新基因HBeBP4A与HBeBP4及其升级基因GLYCTK序列比对结果发现, 在441 bp处有1个G-A的点突变, 在1077-1549 bp之间缺失473 bp, 确定该序列为HBeBP4基因的剪切体, 其开放读码框长1104 bp, 编码367个氨基酸残基, 该序列已在美国核苷酸序列数据库GenBank注册, 注册号DQ352863. 以HepG2细胞cDNA文库为模板, PCR反应后经10 g/L琼脂糖凝胶电泳, 可见长度为1104 bp左右的电泳条带(图1). 利用pGEM-Teasy vector SystemⅠ将纯化PCR产物进行TA克隆, 经EcoRⅤ/HindⅢ酶切, 得到约3027 bp, 1104 bp两条带, BamHⅠ/SacⅠ鉴定酶切, 得到约3531 bp, 601 bp两条带(图2), 命名为pGEM-Teasy-HBeBP4A, 进一步构建的pcDNATM3.1/myc-HisA-HBeBP4A重组真核表达载体, 经EcoRⅤ/HindⅢ酶切, 得到约5473 bp, 1104 bp两条带,HindⅢ/NheⅠ鉴定酶切, 得到约5422 bp, 999 bp, 157 bp三条带(图3), 证明pcDNATM3.1/myc-HisA-HBeBP4A构建成功.
近年来由于乙肝疫苗的广泛应用对HBV感染起到了一定的阻断作用, 但全世界仍有超过3.5亿人处于HBV慢性感染状态, 成为重要的传染源, 其中有很大部分要转化为肝硬化、肝癌[12-15]. 在HBV感染的漫长过程中大多数感染者由于HBeAg的阴转及抗HBe的出现而伴随病毒复制明显减少, 但少数HBeAg阴性的感染者体内病毒仍有持续或间断的高水平复制, 并伴有肝细胞坏死性炎症及进行性纤维化. 既往的研究结果普遍认为HBeAg是一种免疫调节因子, 可调节宿主的免疫应答, 抑制宿主T细胞的细胞毒活性, 形成对HBV感染的免疫耐受性[16-18]. 另一方面由于HBeAg含有病毒高度保守的体液和细胞抗原靶位, 与HBcAg有部分共同的序列, 有相同的抗原靶位, 当HBeAg发生突变时, 失去血清中HBeAg的调节, 表达HBcAg的肝细胞经受增强的T细胞的细胞毒作用, 可导致病情加重[19-23]. 血中抗HBe长期阳性的患者发生肝硬化肝癌的机率较高, 原因不清, 这些发现使得HBeAg的生物学功能变得更复杂. 寻找肝细胞中HBeAg的相互作用蛋白并进一步探明其机制对于明确上述问题有着重要意义.
陆荫英 et al[24-30]利用酵母双杂交系统3, 构建了pGBKT7-HBeAg诱饵表达质粒在预转化的人肝cDNA文库中钓出与HBeAg有相互作用的蛋白基因39种, 其中一种未知基因在GenBank中未发现与之同源的表达基因序列. 根据基因起始密码子的Kozak规则和终止密码子下游保守的多聚腺苷酸信号序列, 电子拼接推定该基因的开放读码框架获得相应的全长编码基因, 将该未知功能基因命名为HBeBP4, 发现其开放读码框长1374 bp, 编码457个氨基酸残基, 并获得GenBank注册号(AY134474). 目前该基因在GenBank中已升级为GLYCTK基因, GenBank注册号NM_145262. 本研究以HBeBP4基因序列为参考, 应用PCR及生物信息学技术设计引物对其进行克隆研究过程中, 我们意外地发现了该基因的剪切体, 其开放读码框长1104 bp, 编码367个氨基酸残基, 命名为HBeBP4A[31-32]. 新基因HBeBP4A与HBeBP4及其升级基因GLYCTK序列比对结果发现, 在441 bp处有1个G-A的点突变, 在1077-1549 bp之间缺失473 bp, 该序列已在美国核苷酸序列数据库GenBank注册, 注册号DQ352863. 确定一个新基因的编码序列, 研究该基因的表达与调控, 编码蛋白的结构与功能, 确定新基因的生物学和医学意义, 是医学分子生物学研究领域中一项具有挑战性的工作[33]. 我们发现了HBeAg的肝细胞结合蛋白新基因HBeBP4A, 这是一种未知功能的新基因, 对于这一新基因结构与功能的研究目前还是空白. 提示我们对HBeAg生物学功能极其在HBV致病机制的认识存在很大的局限性, 目前的研究着眼点可能遗漏了很多重要的方面, 或许有更关键的因素没有被发现而使研究工作误入歧途, 新基因的发现同时也为今后的科研工作指明了新的方向.
乙型病毒性肝炎是由HBV感染引起的世界性传染病, 目前还没有特效的治疗方法. HBV属嗜肝DNA 病毒, 其基因组功能单位十分密集, 高度压缩, 重复利用, 目前己确定有4个主要的开放读码框, 编码至少7个蛋白, 包括3个表面抗原: 包膜蛋白前-S1、前-S2、S; 核心抗原(HBcAg); e抗原(HBeAg); 病毒多聚酶(P); X蛋白(HBxAg). HBeAg 是核壳蛋白的分泌型, 存在于HBV Dane氏颗粒的核心, 既往研究认为HBeAg对HBV的感染和复制无重要作用, 但HBeAg 与肝细胞之间的具体作用机制尚不清楚, 研究HBeAg与肝细胞间的相互作用蛋白, 可能是解决HBV感染的有效途径之一.
酵母双杂交(yeast two hybrid)技术是利用酵母遗传学方法分析蛋白质之间的相互作用. HBeBP4是利用酵母双杂交技术筛选得到的HBeAg肝细胞结合蛋白, 其结构和功能的研究均未有报道. 对其进一步的研究期望能在HBV的发病机制方面有所突破.
本文利用PCR技术在对HBeBP4克隆时意外地发现了该基因的剪切体HBeBP4A, 并利用生物信息学技术分析对比了二者的基因组序列、编码蛋白的结构功能, 确定这是一个HBeAg与肝细胞结合新的靶基因, 已获GenBank注册DQ352863.
新基因HBeBP4A的克隆和功能研究, 为今后更加广泛深入地研究新基因的生物学功能打下基础, 并为研究HBeAg与肝细胞间的相互作用、乙型肝炎发病机制创造了条件.
剪切体: 由于mRNA编辑产生了不同的多肽, 进而形成不同蛋白质, 这些蛋白质就互称为剪切体(splice variants).
本文报道的HBeAg结合蛋白4剪切体HBeBP4A基因的克隆, 内容新颖, 有一定参考借鉴价值.
电编: 张敏 编辑:潘伯荣
| 1. | Milich DR, Chen MK, Hughes JL, Jones JE. The secreted hepatitis B precore antigen can modulate the immune response to the nucleocapsid: a mechanism for persistence. J Immunol. 1998;160:2013-2021. [PubMed] |
| 2. | Chu CM, Liaw YF. Natural history of chronic hepatitis B virus infection: an immunopathological study. J Gastroenterol Hepatol. 1997;12:S218-222. [PubMed] [DOI] |
| 3. | Ou JH. Molecular biology of hepatitis B virus e anti-gen. J Gastroenterol Hepatol. 1997;12:S178-187. [PubMed] [DOI] |
| 5. | Riedl P, Stober D, Oehninger C, Melber K, Reimann J, Schirmbeck R. Priming Th1 immunity to viral core particles is facilitated by trace amounts of RNA bound to its arginine-rich domain. J Immunol. 2002;168:4951-4959. [PubMed] [DOI] |
| 6. | Chen M, Sallberg M, Thung SN, Hughes J, Jones J, Milich DR. Modeling the T-helper cell response in acute and chronic hepatitis B virus infection using T-cell receptor transgenic mice. Antiviral Res. 2001;52:99-111. [PubMed] [DOI] |
| 7. | Barth H, Klein R, Berg PA, Wiedenmann B, Hopf U, Berg T. Induction of T helper cell type 1 response and elimination of HBeAg during treatment with IL-12 in a patient with therapy-refractory chronic hepatitis B. Hepatogastroenterology. 2001;48:553-555. [PubMed] |
| 9. | Wang XZ, Jiang XR, Chen XC, Chen ZX, Li D, Lin JY, Tao QM. Seek protein which can interact with hepatitis B virus X protein from human liver cDNA library by yeast two-hybrid system. World J Gastroenterol. 2002;8:95-98. [PubMed] [DOI] |
| 10. | Nagpal S, Ghosn CR, Chandraratna RA. Identifica-tion of nuclear receptor interacting proteins using yeast two-hybrid technology. Applications to drug discovery. Methods Mol Biol. 2001;176:359-376. [PubMed] |
| 11. | Serebriiskii IG, Toby GG, Finley RL Jr, Golemis EA. Genomic analysis utilizing the yeast two-hybrid system. Methods Mol Biol. 2001;175:415-454. [PubMed] [DOI] |
| 12. | Ohkubo K, Kato Y, Ichikawa T, Kajiya Y, Takeda Y, Higashi S, Hamasaki K, Nakao K, Nakata K, Eguchi K. Viral load is a significant prognostic factor for hepatitis B virus-associated hepatocellular carcinoma. Cancer. 2002;94:2663-2668. [PubMed] [DOI] |
| 13. | Miranda J, Cabezas C. Hepatitis B among health workers. Rev Gastroenterol Peru. 2001;21:128-135. [PubMed] |
| 14. | Torbenson M, Thomas DL. Occult hepatitis B. Lancet Infect Dis. 2002;2:479-486. [PubMed] [DOI] |
| 15. | Marx G, Martin SR, Chicoine JF, Alvarez F. Long-term follow-up of chronic hepatitis B virus infection in children of different ethnic origins. J Infect Dis. 2002;186:295-301. [PubMed] [DOI] |
| 16. | Kakimi K, Isogawa M, Chung J, Sette A, Chisari FV. Immunogenicity and tolerogenicity of hepatitis B virus structural and nonstructural proteins: implications for immunotherapy of persistent viral infections. J Virol. 2002;76:8609-8620. [PubMed] [DOI] |
| 17. | Lau GK, Nanji A, Hou J, Fong DY, Au WS, Yuen ST, Lin M, Kung HF, Lam SK. Thymosin-alpha1 and famciclovir combination therapy activates T-cell response in patients with chronic hepatitis B virus infection in immune-tolerant phase. J Viral Hepat. 2002;9:280-287. [PubMed] [DOI] |
| 18. | Merkle H, Deutschle T, Gastrock-Balitsch I, Nusser P, Knehr S, Reifenberg K. H-2(d) mice born to and reared by HBeAg-transgenic mothers do not develop T cell tolerance toward the hepatitis B virus core gene products. Virology. 2000;273:149-159. [PubMed] [DOI] |
| 19. | Chen M, Sallberg M, Thung SN, Hughes J, Jones J, Milich DR. Nondeletional T-cell receptor transgenic mice: model for the CD4(+) T-cell repertoire in chronic hepatitis B virus infection. J Virol. 2000;74:7587-7599. [PubMed] [DOI] |
| 21. | Milich DR. Do T cells "see" the hepatitis B core and e antigens differently? Gastroenterology. 1999;116:765-768. [PubMed] [DOI] |
| 22. | Diepolder HM, Ries G, Jung MC, Schlicht HJ, Gerlach JT, Gr ner N, Caselmann WH, Pape GR. Differential antigen-processing pathways of the hepatitis B virus e and core proteins. Gastroenterology. 1999;116:650-657. [PubMed] [DOI] |
| 23. | Papatheodoridis GV, Hadziyannis SJ. Diagnosis and management of pre-core mutant chronic hepatitis B. J Viral Hepat. 2001;8:311-321. [PubMed] [DOI] |
| 26. | 陆 荫英, 李 克, 成 军, 王 琳, 刘 妍, 张 玲霞. 乙型肝炎病毒前-S2基因酵母表达载体的构建及表达. 胃肠病学和肝病学杂志. 2002;11:222-224. |
| 27. | 王 琳, 李 克, 成 军, 陆 荫英, 王 刚, 刘 妍, 钟 彦伟, 段 惠娟, 洪 源. 筛选与克隆肝再生增强因子结合的蛋白基因. 世界华人消化杂志. 2002;10:161-164. [DOI] |
| 28. | 李 克, 王 琳, 成 军, 张 玲霞, 段 惠娟, 陆 荫英, 杨 继珍, 刘 妍, 洪 源, 夏 小兵. 酵母双杂交技术筛选克隆HCV核心蛋白结合蛋白基因1. 世界华人消化杂志. 2001;9:1379-1383. [DOI] |
| 29. | 李 克, 王 琳, 成 军, 张 玲霞, 陆 荫英, 李 莉, 刘 妍, 段 惠娟. 丙型肝炎病毒NS2基因酵母双杂交"饵"载体构建及表达. 世界华人消化杂志. 2002;10:129-132. [DOI] |
| 30. | 李 克, 王 琳, 成 军, 陆 荫英, 张 玲霞, 牟 劲松, 洪 源, 刘 妍, 段 惠娟, 王 刚. 丙型肝炎病毒核心蛋白与染色体转位蛋白的相互作用. 中华医学杂志. 2002;82:673-677. |