修回日期: 2005-01-15
接受日期: 2005-01-26
在线出版日期: 2005-03-15
目的: 体外研究干扰素-α2b(IFN-α2b)作用下乙肝病毒(HBV)突变机制.
方法: 以不同浓度IFN-α2b反复作用于HBV分泌型HepG2.2.15细胞, Western blot方法检测培养细胞上清中AFP, 提取培养细胞上清中HBV基因组DNA, 以聚合酶联反应扩增HBV C基因, 克隆入pGEM-T easy载体, 鉴定后行测序分析.
结果: Western blot方法检测各组培养细胞上清中AFP, 实验组AFP未见明显减少; 序列分析表明, 对照组前C/C基因克隆有两种类型, 同源性93%, 有10个氨基酸的差别, 其中6个氨基酸相比, 氨基酸的性质相同; 实验组同样具有这两种克隆, 其中优势株所占比例分别为12/15、14/14和9/15, 高剂量组与对照组相比优势株所占比例差异有统计学意义(P<0.05); 实验组高剂量IFN组出现了四个插入/缺失突变克隆, 造成前C/C基因移框突变, 其中C31-2插入突变发生在前C区, 另外3个突变均发生在C区, 与优势株相比平均同源性97%以上.
结论: 在rhIFN-α高剂量条件选择下, 能检测到HepG2.2.15 HBV前C/C基因插入或缺失突变株, 突变株与优势株相比基因序列同源性平均在97%以上.
引文著录: 王卫峰, 周晓东. 干扰素作用下乙型肝炎病毒的变异机制. 世界华人消化杂志 2005; 13(6): 724-728
Revised: January 15, 2005
Accepted: January 26, 2005
Published online: March 15, 2005
AIM: To investigate the mutation of hepatitis B virus (HBV) with the treated with interferon (IFN)-α2b.
METHODS: Different doses of IFN-α2b were added to the HepG2.2.15 cell culture. The α-fetoprotein (AFP) in the supernatant was assayed by Western blot. HBV DNA was isolated from the culture medium. HBV C gene amplified by PCR was cloned by T-A cloning into pGEM-T easy vector. The recombinatant was sequenced with T7/SP6 primer, and analyzed by DNA software.
RESULTS: AFP in the culture medium was detected, which was not reduced after IFN-α treatment. There were two subtypes of preC/C gene in the control group. The homogeneity was 93%, with 10 amino acids different, 6 of which were of identical characteristics. The same subtypes were found in the experiment group. The proportion of the dominant strain gene was altered in the high dose IFN group. There were 4 insertion/ deletion mutations in the high dose IFN experiment group, which resulted in reading frame shift. The mean homogeneity was more than 97%.
CONCLUSION: HBV pre-C/C gene insertion/deletion mutants exist in HepG2.2.15 cells treated with high dose IFN-α. Compared with the dominant strain, the mean homogeneity of the mutant strain was more than 97%.
- Citation: Wang WF, Zhou XD. Mutation of hepatitis B virus with treatment of interferon. Shijie Huaren Xiaohua Zazhi 2005; 13(6): 724-728
- URL: https://www.wjgnet.com/1009-3079/full/v13/i6/724.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v13.i6.724
乙型肝炎病毒(HBV)变异是导致HBV持续性感染的一个重要原因. 在IFN-α临床治疗过程中, HBV突变株被检测到的概率增加[1-10], 但突变株产生的机制目前存在着不同意见[11]. 通过体外研究IFN作用下HBV突变现象将有助于揭示IFNα直接分子作用在HBV突变株产生过程中的权重, 为探讨IFN致HBV C基因变异的多重机制提供科学的实验依据.
HepG2.2.15细胞由广州中医药大学病毒室惠赠; HepG2细胞系、JM109菌种为林百欣医学研究中心保存; DMEM培养基、胎牛血清、G418、谷氨酰胺、蛋白质预染Marker购自美国Invitrogen公司; IFNα2b, 兔抗人AFP, Pfu聚合酶购自晶美实验有限公司; DL2000(DNA ladder), 1kb DNA ladder购自广州威佳公司; SDS-PAGE试剂、蛋白酶K、TaqDNA聚合酶购自上海生物工程有限公司; QIAquick gel extraction kit购自基因公司; pGEM-T easy vector购自美国Promega公司; E. Z. N. A plasmid minipep kit购自美国Omega公司; NotⅠ内切酶购自MBI公司.
将HepG2.2.15细胞悬液调整为4×108/L, 分组接种于六孔板, 每孔2 mL, 培养3 d, 细胞生长状态良好, 该培养细胞株用于实验研究. IFN作用浓度为100, 500, 1 000 kU/L共3个剂量组, 对照组不加IFN, HepG2细胞作为空白对照, 每组3个复孔. 实验组每日换液, 培养液含不同浓度IFN, 连续培养20 d, 最后1次换液(用无血清DMEM培养液)后细胞继续培养48 h, 留取的上清立即于-20℃保存待测. 采用台盼蓝排斥试验检测细胞活力. 培养上清中AFP的检测采用Western blot方法, 对实验结果进行图像分析. 细胞上清中HBV DNA的提取通过蛋白酶K消化, 酚、氯仿纯化, 从培养细胞收获的上清中提取病毒基因组DNA, 溶于TE溶液中, -20℃保存待测. 根据Genebank中HBV ayw亚型全基因序列, 选择保守区域设计出特异性引物: P1 5' -GGGAGGAGATTAG GTTAA -3'; P2 5'-GTACAGTAGAAGAATAAAGC-3'. 取上清HBV DNA提取物5 μL, Pfu聚合酶2.5U及适量引物、dNTP、三蒸水、PCR缓冲液组成50 μL反应体系, 在如下条件下反应: 94℃ 5 min, 然后94℃ 1 min, 55℃ 1 min, 72℃ 1 min, 共25个循环, 最后72℃延伸7 min. 取纯化后的PCR产物7 μL、10×缓冲液(含Mg2+)1 μL、dATP 1 μL、Taq酶1 μL、70℃反应30 min. 取适量反应产物与pGEM-T easy载体连接, 然后氯化钙法转入受体菌JM109中. 经蓝白斑筛选获得可能插入目的DNA的白色菌落, 提取质粒并进行酶切(NotⅠ内切酶)和PCR鉴定(条件同上). 将鉴定阳性者送上海申有公司测序. 测序结果用DNA软件分析.
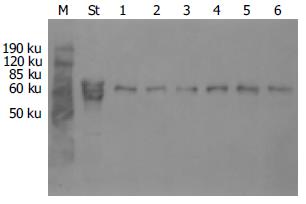
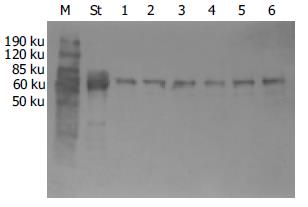
用Western blot方法检测AFP, 在70 ku位置上的蛋白能特异性地被AFP抗体识别(图1, 2); 对免疫杂交后条带作图像分析, 对照组AFP%作为100%, 各实验组AFP%分别为: 低剂量组114%, 中剂量组94%, 高剂量组118%. 方差分析P = 0.870, 即各组之间差异不显著.
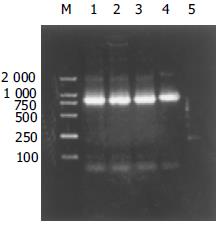
DNA 通过上述方法从培养细胞收获的上清中提取病毒基因组DNA, 溶于TE溶液中, 经UV-2201型紫外分光光度计测定其A260 = 0.022, A280 = 0.012, 浓度为1.1 g/L, A260/A280 = 1.83, 表明DNA纯度较高. 以提取物为模板, 按上述方法扩增HBV C基因, PCR产物用12 g/L琼脂糖凝胶电泳检测, 可见大小约770 bp的片断, 多次实验此结果均稳定出现(图3).
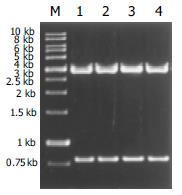
将HBV C基因与pGEM-T easy载体连接后转化至受体菌JM109; 经蓝白斑筛选, 挑取白色菌落, 用OBI质粒提取kit提取质粒; 由于插入片断两端含有NotⅠ酶切位点, 作NotⅠ单酶切可以释放出插入基因(图4), PCR鉴定可以扩增出770 bp大小片断, 均与预期相符, 可以初步确定pHBV是pGEM-T easy与HBV C基因连接的重组体.
C区克隆基因大小为770 bp, 前C区编码29个氨基酸, C区编码183个氨基酸, 包含有完整的编码序列. 对照组前C/C基因克隆有两种类型, 优势株所占比例14/15, 同源性93%, 两种类型基因有10个氨基酸的差别, 其中6个氨基酸对比氨基酸的性质相同(表1); 实验组同样具有这两种克隆, 其中优势株所占比例分别为12/15、14/14和9/15; 用统计学方法比较实验组和对照组中优势株所占比例, Fisher精确概率检验法(Fisher's exact test), 低剂量组P = 0.33; 中剂量组P = 1.00; 高剂量组P = 0.04.高剂量组与对照组相比优势株所占比例差异有统计学意义; 实验组高剂量IFN组出现了4个插入/缺失突变克隆, 造成前C/C基因移框突变, 其中C31-2插入突变发生在前C区, 另外3个突变均发生在C区, 与优势株相比平均同源性97%以上(表2).
| 克隆号 | 位置核苷酸(nt) | 类型突变 | 突变后果 |
| C31-2 | nt 13 | 插入 | 密码子25ATC→TAA, e抗原不能表达 |
| C32-4 | nt 108 | 缺失 | 密码子60CTA→TAG, c蛋白提前终止 |
| C32-5 | nt 283 | 缺失 | 密码子95ATG→TGA, c蛋白提前终止 |
| C31-4 | nt 598 | 插入 | 密码子199后的氨基酸序列改变 |
C基因测序结果显示, 在对照组和实验组均存在着不同的HBV毒株, 这表明HepG2.2.15细胞内HBV具有异质性. 目前研究结果表明, 在慢性HBV感染者体内可发现3种类型的基因异质性[12]. 第一类, 不同的HBV基因型(A-F)在人群中稳定传播; 第二类, HBV感染者体内的DNA序列发生自发性突变, 主要是在HBV DNA复制过程中, 由于HBV DNA逆转录酶的不精确性而造成的, 如多种类型的变异体形成; 第三类, 是由于在某些外来干预因素的作用下进行选择, 使得某些类型的突变体存活下来, 改变了病毒的基因表型, 最常见的变化是增加了病毒复制的活性, 或对于抗病毒药物产生了耐药性. 后两类HBV异质性孕育出了准种的概念, 准种是用于表现遗传物质异质性的概念, 其定义是物种的基因组DNA或RNA的碱基序列在统计学上高度一致, 但个体之间又存有差异的一组群体. 产生的原因是由于RNA多聚酶或逆转录酶不具有校对功能, 从而产生一群存在不同位点变异的基因组DNA或RNA. 在宿主免疫压力的作用下, 或在药物的干预下, 准种群经过筛选, 遗留下优势种群耐受宿主内环境, 并继续变异.1993年有学者[13-14]指出HBV在患者体内变异程度达到了准种的定义标准, 近年来一些研究支持HBV准种假说. 更有学者[15-29]研究了经拉米夫啶治疗前后的准种漂变现象.
了解HBV基因异质性与准种特点, 对于研究HBV基因变异是非常重要的, 首先, 他使我们认识到机体内HBV变异是受两方面共同作用的结果, 其内因是HBV逆转录酶存在校读偏差, 外因是存在免疫压力和药物的干扰, 在内因和外因的共同作用下HBV不断的发生变异, 因此HBV变异是绝对的, 而突变的程度和速度是相对的. 其次, 了解HBV基因异质性与准种特点, 有助于全面理解HBV基因突变的特点和规律. 如果从准种这一角度来看HBV的基因变异, 因为很难控制所得到的HBV基因片段总是来源于同一病毒株, 因此关于HBV基因突变的生物学意义及临床意义应持谨慎态度. 准种最大的特点是从患者体内整个病毒群体水平上去认识病毒的生物学特征及临床意义, 不再注重某种单个的所谓野生型或变异株的意义. 尽管目前已在不同病变和不同感染状态的感染者发现了许多变异株, 但对其生物学意义大多还是推测性的, 可以肯定因果关系的证据极为有限. 因此, 认为评估HBV准种群体的异质性程度可能比鉴定单个特殊的病毒变异株更重要, 这种异质性赋予了HBV群体更强的生存适应能力.
在高剂量IFN组发现有4个克隆发生了插入或缺失变异(4/15), 插入或缺失的碱基数不是3的倍数, 引起移框突变, 造成HBcAg和/或HBeAg的不完整表达或不表达. 而在对照组及其他实验组没有见到这种类型的突变株, 显示这种突变和高剂量IFN高度相关. 另外, 根据准种理论, 研究HBV变异不仅要分析单个突变株的意义, 更要从病毒群体水平去认识病毒的生物学特征及临床意义. 因此我们分析了高剂量组内优势株的比例在IFN作用下的变化, 结果表明, 与对照组相比, 优势株的比例发生了变化, 这种变化具有统计学意义, 即可以认为, 受高剂量IFN干扰的影响, 优势株在群体中的比例发生了变化.
Radecke et al[1]在研究高剂量IFN治疗对前C/C基因影响时, 发现HBcAg有稳定的氨基酸替代. 表明高剂量IFN对HBcAg免疫显性表位有选择作用. 但这种选择作用是靠IFN的免疫调节机制还是其直接抗病毒机制, 作者没有加以阐述. 在本实验中, 高剂量IFN组发现核心基因有4个克隆出现插入或缺失突变, 与Radecke et al的研究结果一致. 虽然这些突变位置较为分散, 突变部位没有规律, 但他们共同的结果是造成HBcAg部分表达或不表达. 由于HBcAg是HBV的重要结构蛋白, 具有多种功能, 他的不完整表达必然会影响病毒颗粒的包装过程, 如果出现在体内, 还可以逃避宿主免疫系统的攻击, 降低机体的清除作用. 由于体外实验中缺乏机体内的免疫环境, IFN对HBV的作用只表现为直接分子作用机制, 因此推测这种突变的机制是IFN的直接抗病毒效应.
在这种效应下, 这些突变株的产生我们分析有两种可能性. 第一, 这种变异株在IFN作用前已经出现, 是原有准种的一种, 由于野生株对IFN比较敏感, 在IFN作用下逐渐减少, 使得这种对IFN不敏感的变异株比例相对增加, 能够被检测出来. 在其他病毒研究中发现, 一些病毒如麻疹病毒、单纯疱疹病毒和淋巴细胞性脉络丛脑膜炎病毒在治疗前就存在一些耐受IFN的毒株, 这些毒株对IFN的耐受作用使得病毒感染持续存在[30-32], 这种现象支持了上述观点. 第二, 变异株是在IFN作用开始后出现的, 即由原来的野生株突变而来, 是一种新出现的突变株, 这种观点抛弃准种理论, 认为该突变株的出现是一种真正意义上的突变现象, 而不是选择的结果. 突变的发生和病毒的复制率以及聚合酶的错配率有关, 有研究表明, 杂交实验检测阳性者, 病毒复制水平1011个颗粒/日, HBV聚合酶的错配率在10-3-10-5之间, 即病毒复制一个循环出现单个碱基错配的可能性为10-3-10-5, 如果每日产生1011个HBV颗粒, 那么1 d中就可以产生106-109个单碱基突变株.
Sells et al人在对2.2.15细胞研究中发现, 细胞培养上清中可以检测到HBsAg、HBeAg以及完整的Dane颗粒, 胞质中可以检测到HBV各种复制中间体, 特别重要的是, 在胞核中发现了共价闭合环状HBV DNA游离体, 即cccDNA. 由于自然感染状态下cccDNA是细胞核内HBV复制的模板, 因此, 在2.2.15细胞核中检测到cccDNA表明这种人工构建细胞系中存在着HBV自身复制的过程, 这为体外研究HBV在各种状态下生物学特性及其变化提供了可能.
2.2.15细胞生长状态对HBV复制很重要, 生长期细胞内游离型HBV颗粒较少, 静止期细胞内HBV复制活跃, 是生长期细胞内病毒量的10-100倍. HBV复制中间体在细胞融合后一段时间快速积累增加几百倍, 这一时期是HBV核心颗粒积累的过程, 对IFN作用较为敏感.
本实验中以不同剂量IFN反复作用于2.2.15细胞, 达到一定时间后, 收获细胞上清, 通过免疫印迹的方法检测上清中AFP的含量, 并将实验组与对照组中的AFP比较, 结果显示各实验组中AFP与对照组相比未见减少, 表明IFN在实验组剂量下主要发挥抗病毒作用, 对2.2.15细胞增生及蛋白合成没有抑制作用.
蛋白酶K消化-饱和酚: 氯仿(1: 1)抽提法提取细胞上清中的HBV DNA, PCR法扩增靶片段, 采用TA克隆的方法将单个实验组内可以获得的克隆尽可能多地存储在载体中. 利用 pGEM-Teasy载体固有的NotI酶切分析, 发现插入片段与PCR扩增的靶片段长度相当; 随机选择菌落测序, 用clustalx软件对测序结果进行分析. Pfu酶的错配率为2.6×10-6, 而我们扩增的靶片段长度仅770 bp, 因此Pfu酶引起的碱基错配不会影响到实验的真实性.
编辑: 张海宁 电编: 潘伯荣
| 1. | Radecke K, Protzer U, Trippler M, Meyer Zum Buschenfelde KH, Gerken G. Selection of hepatitis B virus variants with aminoacid substitutions inside the core antigen during interferon-alpha therapy. J Med Virol. 2000;62:479-486. [PubMed] |
| 2. | Zampino R, Marrone A, Cirillo G, del Giudice EM, Utili R, Karayiannis P, Liang TJ, Ruggiero G. Sequential analysis of hepatitis B virus core promoter and precore regions in cancer survivor patients with chronic hepatitis B before, during and after interferon treatment. J Viral Hepat. 2002;9:183-188. [PubMed] |
| 3. | Cabrerizo M, Bartolome J, Otero M, Ruiz-Moreno M, Carreno V. Sequence variation of hepatitis B virus precore-core open reading frame isolated from serum and liver of children with chronic hepatitis B before and after interferon treatment. J Med Virol. 1999;58:208-214. [PubMed] |
| 4. | Karasawa T, Aizawa Y, Zeniya M, Kuramoto A, Shirasawa T, Toda G. Genetic heterogeneity in the precore region of hepatitis B virus in hepatitis B e antigen-negative chronic hepatitis B patients: spontaneous seroconversion and interferon-induced seroconversion. J Med Virol. 1995;45:373-380. [PubMed] |
| 5. | Laskus T, Rakela J, Persing DH. Nucleotide sequence analysis of precore and proximal core regions in patients with chronic hepatitis B treated with interferon. Dig Dis Sci. 1995;40:1-7. [PubMed] |
| 6. | Lampertico P, Manzin A, Rumi MG, Paolucci S, Del Ninno E, Clementi M, Colombo M. Hepatitis B virus precore mutants in HBeAg carriers with chronic hepatitis treated with interferon. J Viral Hepat. 1995;2:251-256. [PubMed] |
| 7. | Seo Y, Yoon S, Hamano K, Nakaji M, Yano Y, Katayama M, Ninomiya T, Hayashi Y, Kasuga M. Response to interferon-alpha in chronic hepatitis B with and without precore mutant strain detected by mutation site-specific assay. J Clin Gastroenterol. 2004;38:460-464. [PubMed] |
| 8. | Chen RY, Bowden S, Desmond PV, Dean J, Locarnini SA. Effects of interferon alpha therapy on the catalytic domains of the polymerase gene and basal core promoter, precore and core regions of hepatitis B virus. J Gastroenterol Hepatol. 2003;18:630-637. [PubMed] |
| 9. | Gunther S, Paulij W, Meisel H, Will H. Analysis of hepatitis B virus populations in an interferon-alpha-treated patient reveals predominant mutations in the C-gene and changing e-antigenicity. Virology. 1998;244:146-160. [PubMed] |
| 10. | Fattovich G, McIntyre G, Thursz M, Colman K, Giuliano G, Alberti A, Thomas HC, Carman WF. Hepatitis B virus precore/core variation and interferon therapy. Hepatology. 1995;22:1355-1362. [PubMed] |
| 11. | Rehermann B, Pasquinelli C, Mosier SM, Chisari FV. Hepatitis B virus(HBV)sequence variation of cytotoxic T lymphocyte epitopes is Not common in patients with chronic HBV infection. J Clin Invest. 1995;96:1527-1534. [PubMed] |
| 12. | 成 军. 病毒性肝炎研究的新进展-第三届国际肝炎及肝病学术研讨会系列报道. 国外医学·流行病学传染病学分册. 2001;28:273-278. |
| 13. | Blum HE. Hepatitis B virus: significance of naturally occurring mutants. Intervirology. 1993;35:40-50. [PubMed] |
| 14. | Carman W, Thomas H, Domingo E. Viral genetic variation: hepatitis B virus as a clinical example. Lancet. 1993;341:349-353. [PubMed] |
| 15. | Mutimer D, Pillay D, Dragon E, Tang H, Ahmed M, O'Donnell K, Shaw J, Burroughs N, Rand D, Cane P. High pre-treatment serum hepatitis B virus titre predicts failure of lamivudine prophylaxis and graft re-infection after liver transplantation. J Hepatol. 1999;30:715-721. [PubMed] |
| 16. | Li ZG, Chen LY, Huang J, Qiao P, Qiu JM, Wang SQ. Quantification of the relative levels of wild-type and lamivudine-resistant mutant virus in serum of HBV-infected patients using microarray. J Viral Hepat. 2005;12:168-175. [PubMed] |
| 17. | Natsuizaka M, Hige S, Ono Y, Ogawa K, Nakanishi M, Chuma M, Yoshida S, Asaka M. Long-term follow-up of chronic hepatitis B after the emergence of mutations in the hepatitis B virus polymerase region. J Viral Hepat. 2005;12:154-159. [PubMed] |
| 18. | Pas SD, Noppornpanth S, van der Eijk AA, de Man RA, Niesters HG. Quantification of the newly detected lamivudine resistant YSDD variants of Hepatitis B virus using molecular beacons. J Clin Virol. 2005;32:166-172. [PubMed] |
| 19. | Lin CL, Tsai SL, Lee TH, Chien RN, Liao SK, Liaw YF. High frequency of functional anti-YMDD and -mutant cytotoxic T lymphocytes after in vitro expansion correlates with successful response to lamivudine therapy for chronic hepatitis B. Gut. 2005;54:152-161. [PubMed] |
| 20. | Thabut D, Thibault V, Bernard-Chabert B, Mouquet C, Di Martino V, Le Calvez S, Opolon P, Benhamou Y, Bitker MO, Poynard T. Long-term therapy with lamivudine in renal transplant recipients with chronic hepatitis B. Eur J Gastroenterol Hepatol. 2004;16:1367-1373. [PubMed] |
| 21. | Nogales MC, Serrano MC, Suarez E, Corpas R, Perez L, Claro R, Jarana R, Romero-Gomez M, Martin-Mazuelos E. Determination of hepatitis B virus genotype and detection of lamivudine-resistance mutations. Gastroenterol Hepatol. 2004;27:515-520. [PubMed] |
| 22. | 李 兵, 周 伯平, 彭 劲甫, 陈 立炎, 李 丽雄, 王 召钦, 王 敏, 刘 赛云, 蒋 小玲. 基因芯片高通量检测乙型肝炎病毒拉米夫定耐药株及对其突变热点的分析. 中华肝脏病杂志. 2004;12:673-676. [PubMed] |
| 23. | Torre F, Mattioli F, Campo N, Delfino A, Basso M, Pelli N, Martelli A, Picciotto A. Serum lamivudine levels in the presence of a lamivudine-resistant HBV mutant. Dig Liver Dis. 2004;36:677-681. [PubMed] |
| 24. | Lindstrom A, Odeberg J, Albert J. Pyrosequencing for detection of lamivudine-resistant hepatitis B virus. J Clin Microbiol. 2004;42:4788-4795. [PubMed] |
| 25. | Yuen MF, Yuan HJ, Sablon E, Wong DK, Chan AO, Wong BC, Lai CL. Long-term follow-up study of Chinese patients with YMDD mutations: significance of hepatitis B virus genotypes and characteristics of biochemical flares. J Clin Microbiol. 2004;42:3932-3936. [PubMed] |
| 26. | Santos EA, Sucupira MV, Arabe J, Gomes SA. Hepatitis B virus variants in an HIV-HBV co-infected patient at different periods of antiretroviral treatment with and without lamivudine. BMC Infect Dis. 2004;4:29. [PubMed] |
| 27. | Tanaka Y, Yeo AE, Orito E, Ito K, Hirashima N, Ide T, Sata M, Mizokami M. Prognostic indicators of breakthrough hepatitis during lamivudine monotherapy for chronic hepatitis B virus infection. J Gastroenterol. 2004;39:769-775. [PubMed] |
| 28. | Punia P, Cane P, Teo CG, Saunders N. Quantitation of hepatitis B lamivudine resistant mutants by real-time amplification refractory mutation system PCR. J Hepatol. 2004;40:986-992. [PubMed] |
| 29. | Liaw YF, Chien RN, Yeh CT. No benefit to continue lamivudine therapy after emergence of YMDD mutations. Antivir Ther. 2004;9:257-262. [PubMed] |
| 30. | Carrigan DR, Knox KK. Identification of interferon-resistant subpopulations in several strains of measles virus: positive selection by growth of the virus in brain tissue. J Virol. 1990;64:1606-1615. [PubMed] |