修回日期: 2004-04-11
接受日期: 2004-04-20
在线出版日期: 2004-08-15
目的: 初步筛选和鉴定大肠癌转移相关基因, 阐述大肠癌转移的分子机制.
方法: 利用抑制消减杂交技术(suppression subtractive hybridization, SSH), 在一对来自同一亲本、转移能力不同的人大肠癌细胞株SW620和SW480间, 已成功构建了人大肠癌转移促进基因和转移抑制基因的两个cDNA消减文库. 从以上两个文库中随机挑取约200个菌液PCR片段, 利用Differential screening(DS)方法筛选真正差异表达的cDNA片段, 之后通过Northern blot对部分差异cDNA片段的DS筛选结果进行验证. 最后对筛选出的差异cDNA片段进行测序和同源检索, 分析其性质并初步探讨其在大肠癌中转移中的可能机制.
结果: 约200个菌液PCR片段经过DS筛选后, 共获得29个真正差异表达cDNA片段, 利用Northern blot对其中4种cDNA片段的表达情况进行验证, 结果与DS筛选一致. 然后对29个差异cDNA片段进行测序和同源性分析, 得到25个差异表达基因, 10个为已知基因, 其中5个基因在高转移细胞株SW620中差异表达, 可能有促进转移的作用, 包括热休克蛋白10(heat shock protein10, HSP10)、细胞色素氧化酶II (cytochrome C oxidaseII, COXII)、有丝分裂调控蛋白dis3人同源物(mitotic control protein dis3 homolog, DIS3)、骨骼肌α1-肌动蛋白(skeletal muscle actin alpha1, ACTA1)及血浆淀粉样蛋白A(serum amyloid A, SAA); 另外5个基因的表达仅见于在SW480低转移细胞株, 具有潜在的转移抑制作用, 包括egl nine homolog 2 (EGLN2)、真核翻译起始因子4A(eukaryotic translation initiation factor 4A, EIF4A)、人细胞色素氧化酶III的类似物(similar to cytochrome c oxidase III, COXIII)、谷胱甘肽S转移酶3(glutathione S-transferase mu 3, GSTM3) 及线粒体DNA(mitochondrion DNA, mtDNA). 10个已知基因基本上是一些与细胞生长分化、代谢合成、转录、凋亡及信号转导等相关的基因. 除DIS3和SAA外, 其余基因与大肠癌转移的关系在本研究中首次被报道. 此外, 还筛选到15个未知基因片段, 通过与人基因组序列进行比对, 发现其中6个基因定位于5号染色体上, 包括4个可能促进大肠癌转移的基因和2个抑制基因. 目前这15个未知基因片段已经被GeneBank dbEST库收录, GeneBank登陆号为CD485499-CD485513.
结论: DS技术是一种筛查表达文库的有效途径; 细胞的生长分化、转录、凋亡、信号转导等变化可能在大肠癌转移过程中发挥重要作用; 5号染色体上可能存在多个与大肠癌转移相关的新基因位点.
引文著录: 梁莉, 丁彦青, 李欣, 杨玉芳, 肖军, 张进华, 赵培冉. 人大肠癌转移相关基因片段的筛选和鉴定. 世界华人消化杂志 2004; 12(8): 1800-1805
Revised: April 11, 2004
Accepted: April 20, 2004
Published online: August 15, 2004
AIM: To primarily screen and identify metastasis-associated genes of colorectal carcinoma and to illustrate the molecular mechanisms of metastasis.
METHODS: Suppression subtractive hybridization was performed between a pair of high metastatic cell line SW620 and low metastatic cell line SW480 of human colorectal carcinoma, which originated from the same parent. Two subtracted cDNA libraries for metastasis accelerating genes or metastasis suppressor genes of human colorectal carcinoma had been then constructed. About 200 bacterial PCR fragments were picked out randomly to obtain the differentially expressed cDNA fragments by differential screening (DS) method. After that, expression of partial differentially expressed cDNA fragments was validated by Northern blot. Then the differentially expressed cDNA fragments were sequenced. Finally BLAST searching and literature review were done to analyze their characters and illustrate the possible mechanisms in the process of metastasis of colorectal carcinoma.
RESULTS: After Differential screening of about 200 bacterial PCR fragments, totally 29 differentially expressed cDNA fragments were obtained. Northern blot was then performed to validate the expression of 4 cDNA fragments. The results were consistent with DS findings. 29 differentially expressed cDNA fragments were then sequenced and Blast analysis showed that 25 differentially expressed gene fragments were obtained. There were 10 known genes, among which 5 genes were differentially expressed in high metastatic cell lines SW620 and thus may accelerate metastasis, including heat shock protein10, cytochrome C oxidaseII, mitotic control protein dis3 homolog, skeletal muscle actin alpha1 and serum amyloid A. In addition, another 5 genes were differentially expressed in SW480 cell lines and may have the potential to suppress metastasis, which included egl nine homolog 2, eukaryotic translation initiation factor 4A, similar to cytochrome C oxidase Ⅲ, glutathione S transferase mu 3 and mitochondrion DNA. These 10 known genes are mainly related with cell growth and differentiation, metabolism and synthesis, transcription, apoptosis, signal transduction and so on. Except DIS3 and SAA, other genes were first reported on their relationship with metastasis of colorectal carcinoma in the studies. In addition, 15 unknown genes were screened in this study. After BLAST analysis with human genome sequences, 6 unknown genes were found to be located on chromosome 5, including 4 candidate metastasis accerating genes and 2 metastasis suppressor genes of colorectal carcinoma. Those 15 unknown genes have been collected by the GeneBank dbEST database with the accession number of CD485499-CD485513 respectively.
CONCLUSION: Differential screening method is the efficient way to screen the expressive cDNA library. Changes in cell growth and differentiation, transcription, apoptosis and signal transduction may play an important role in metastasis of colorectal carcinoma. Chromosome 5 might exist many new genes locations related with metastasis of colorectal carcinoma.
- Citation: Liang L, Ding YQ, Li X, Yang YF, Xiao J, Zhang JH, Zhao PR. Screen and identification of human colorectal carcinoma metastasis-associated genes. Shijie Huaren Xiaohua Zazhi 2004; 12(8): 1800-1805
- URL: https://www.wjgnet.com/1009-3079/full/v12/i8/1800.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i8.1800
转移是大肠癌患者死亡的主要原因, 其分子机制尚未完全明了. 抑制消减杂交(suppression subtractive hybridization, SSH)是目前应用最为有效的一种差异表达基因克隆技术. 我们已利用该技术, 在一对来自同一亲本、转移能力不同的人大肠癌细胞株SW480和SW620间构建了大肠癌转移相关消减cDNA文库, 为筛选差异表达基因及发现新基因奠定了基础[1]. 本研究利用Differential screening方法, 并结合生物信息学, 对大肠癌转移相关基因进行筛选和鉴定, 为阐述大肠癌转移的分子机制提供了新的线索.
人大肠癌高转移细胞株SW620和低转移细胞株SW480购自美国ATCC细胞库, ATCC号分别为CCL-227和CCL-228, 两种细胞株来自同1例结肠腺癌患者, 遗传背景相同. 用含DMEM和100 mL/L胎牛血清的培养基于37 ℃, 50 mL/L CO2条件下常规培养细胞.
两种细胞株mRNA的分离提取参照QuickPrep micro mRNA Purification Kit(购自Pharmacia公司)说明书进行操作. 抑制消减杂交参照PCR-Select TM cDNA Subtraction Kit (购自Clontech公司)说明书进行操作. 正向杂交组(A组)以大肠癌高转移细胞株SW6200为检测子(tester), 低转移细胞株SW480为驱动子(driver); 反向杂交组(R组)则相反. 消减cDNA文库的构建及鉴定按pGEM-T Vector System I(购自Promega)说明书进行操作. 将PCR扩增产物与pGEM-T Easy Vector连接, 转化JM109感受态细菌, 在含X-gal/IPTG的氨苄青霉素琼脂培养板上, 37 ℃过夜培养. 挑取白色菌落, 扩菌. 以Nested Primer 1及2R作引物进行菌液PCR扩增, 电泳观察PCR产物是否为单一片段. 各取纯化的正向杂交产物A、反向杂交产物R、正向未消减产物(1-C)、反向未杂交产物(2-C)1 μL进行PCR Dig 探针合成, 反应条件同第2次PCR, 其中5×PCR DIG Mix包括1 mmoL/L dATP, dCTP和dGTP, 0.65 mmoL/L dTTP, 0.35 mmoL/L Dig-dUTP. 随机挑取PCR阳性样品热变性后, 分别加样于四张硝酸纤维素膜上相应的位置, 80 ℃干烤2 h. 按照0.2 mL/cm2膜面积的比例加入预杂交液(6×SSC, 5×Denhardt试剂, 5 g/L SDS、100 mg/L变性鲑精DNA, 500 g/L去离子甲酰胺), 42 ℃预杂交8 h. 热变性Dig标记的4种PCR探针, 按0.2 mL/cm2的比例加至预杂交液中, 42 ℃杂交16 h. 最后按Dig DNA Labeling and Detection Kit(购自Roche公司)说明书进行洗膜和显色. 将阳性克隆送交上海博亚公司进行商业测序, 将测序结果用Blast软件与GeneBank公布的基因进行同源性比较. 用Qiaex II Agorose Gel Extration Kit(购自Gene公司)回收4种待验证的cDNA扩增片段和内参照GAPDH片段, 对其进行随机引物法Dig探针标记(Dig DNA Labeling and Detection Kit, Roche公司), 再按NorthernMaxTM(购自Ambion公司)说明书进行Northern blot分析, 取Tester和Driver总RNA各约30 μg, 尼龙膜为Amersham公司产品.
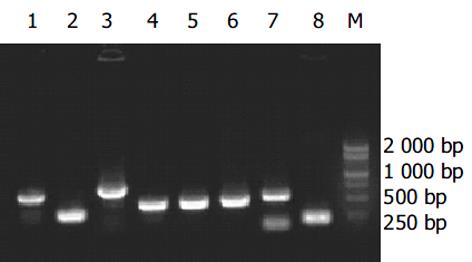
在一对人大肠癌高、低转移细胞株SW620和SW480间, 分别进行正向(A组)和反向消减杂交(R组). 经过蓝白斑筛选, A组获得235个白色菌落, R组有232个白色菌落. 利用菌液PCR法扩增挑取的全部白色克隆, 两组分别有98%或91%的白色克隆含有插入片段, 大小约为200-700 bp(图1).
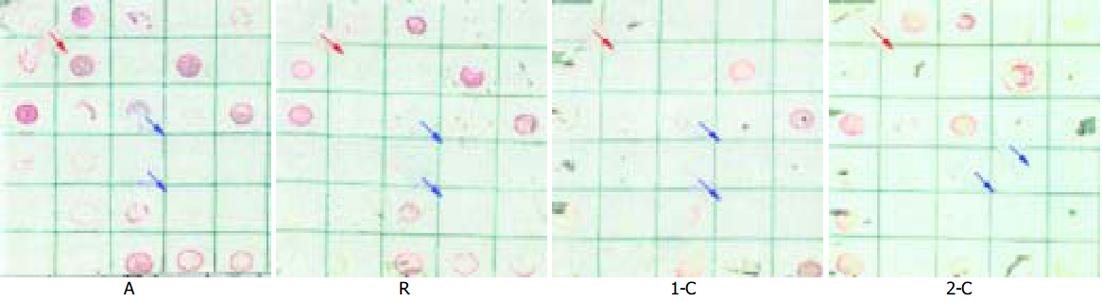
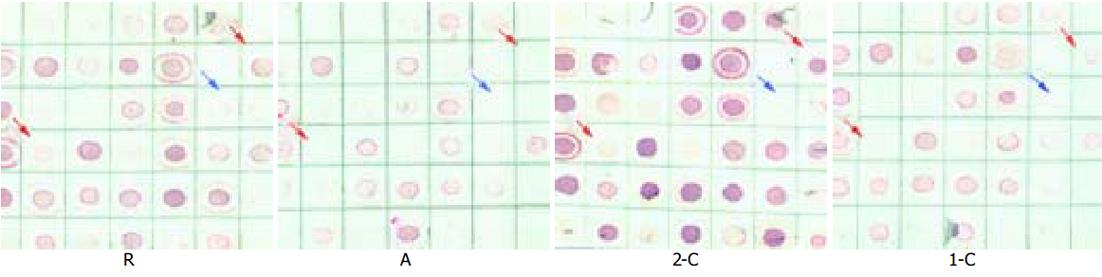
DS技术同时采用1-C, 2-C, A, R与同一个目的基因进行杂交, 这4个探针的杂交信号之间必然存在强弱、有无的差异. 表1为DS方法杂交结果判读标准. 在正向消减A组中, 这4种探针中上调差异表达基因的杂交信号强弱将呈现以下差异: R<2-C≤1-C<A(图2). 而在反向消减R组, 杂交信号强弱的规律则是: A<1-C≤2-C<R(图3). 若差异基因仅与正向消减A组的R探针或仅与反向消减R组的A探针杂交, 则强烈提示该基因呈低丰度表达.
| 探针类型 | 杂交结果分析 | |||
| A | R | 1-C | 2-C | |
| + | - | + | - | 阳性差异表达结果 |
| + | - | - | - | 阳性差异表达基因(强烈提示低丰度表达差异基因) |
| + | - | + | + | 多为假阳性结果,但是也可能模板与不同长度地探针杂交造成结果难以判断,多为多基因家族成分 |
| ++ | + | + | ± | 绝少情况为差异表达基因 |
| + | + | + | + | 非阳性差异表达基因 |
| - | - | - | - | 非阳性差异表达基因 |
按照以上标准, 从A, R组随机挑选100个白色克隆的菌液PCR产物进行鉴定, 2组共筛选出29个差异表达cDNA片段, 其中A组15个, R组14个, 其中低丰度表达的片段4个(A140, R57, R159, R163).
将DS筛选出的29个差异片段测序, 经分析A87与A186, R119与R154、R142与R163分属同一个克隆. 利用GeneBank中与核苷酸和人基因组的BLAST比对功能, 发现R32与R145属于同一个基因的不同片段, 因此共获得25个差异基因, 其中已知基因10个(表2).
| Clone(bp) | Known gene description | Acc1ession | Identities | Expression |
| A80(400) | Heat Shock 10kD | NM_002157 | 200/209 (95%) in sw620 | Protein1 (HSP10) |
| A98(138) | Cytochrome C | NM_173705 | 75/75(100%) in sw620 | OxidaseII (COXII) |
| A108(181) | Mitotic Control Protein | NM_014953 | 130/131(99%) in sw620 | dis3 homolog(DIS3) |
| A164(300) | skeletal muscle actin | NM_001100 | 167/168(99%) in sw620 | alpha 1 (ACTA1) |
| A184(301) | Serum amyloid A(SAA) | NM_030754 | 276/276 (100%) in sw620 | |
| R159(91) | egl nine homolog 2 | NM_080732 | 90/91(98%) in sw480 | (EGLN2) |
| R57(450) | Eukaryotic Translation | NM_001416 | 448/450(99%) in sw480 | Initiation Factor 4A(ETIF4A) |
| R178(298) | Glutathione S-transferase | AF043105 | 260/264(98%) in sw480 | mu 3 (GSTM3) |
| R211(155) | Similar to Cytochrome c | BC013930 | 153/155(98%) in sw480 | Oxidase III(COXIII) |
| R32(214) | Haplotype As9y | AY195792 | 213/213(100%) in sw480 | mitochondrion |
此外还筛选出了15个未知基因片段, 将其与人基因组序列进行同源性比对, 发现与5号染色体序列同源的基因有6个, 分别有2个基因与3, 18号染色体序列同源, 另外, 还有基因与4, 6, 10, 11, 16号染色体序列同源. 目前这15个未知片段已经被GeneBank dbEST库收录, GeneBank登陆号为CD485499-CD485513.
5种探针分别与转至膜上的两种细胞RNA杂交, 结果如图4. SW620和SW480两种细胞株中, 内参照GAPDH均呈阳性表达; 低丰度表达的A140仅见于SW620; A98在两种细胞中均有表达, 但在SW620的表达水平较高; R57, R23的表达仅见于SW480. 以上结果与DS筛选结果基本符合.
转移是大肠癌患者死亡的主要原因, 其分子机制至今尚未完全阐明. 研究报道已有百余种基因参与调控大肠癌的转移过程, 这些基因为阐述转移的分子机制提供了重要的线索, 但他们仍远远不能解释转移过程的复杂性和多样性. 此外, 其检测手段多限于一种或几种基因的表达分析, 不能反映基因间的相互关系及发现新基因. 对大肠癌转移相关基因进行克隆, 不仅有利于阐明大肠癌转移的分子机制, 而且还能为临床上预测转移、基因治疗和预后判断寻找有效的靶点.
近年来基因克隆技术发展迅速, 数种基于消减杂交策略的基因克隆技术相继问世, 使克隆、分离大肠癌转移相关基因成为可能. 1996年Diatchenko et al建立的基于抑制PCR基础上的SSH技术, 凭借其高水平富集、低背景值和对高、低丰度的目的基因均可有效富集的特性, 是目前应用最为有效的一种差异表达基因克隆技术, 已广泛应用在肿瘤发生发展[2-4]、信号转导[5]、凋亡[6]及治疗[7]等多种研究领域. 在肿瘤转移方面, SSH已被用于肺癌、骨肉瘤、乳腺癌等转移相关基因的克隆[8-11]. Zhang et al[12]利用SSH技术在人经典型NCI-H69和变异型、侵袭力更强的NCI-N417 小细胞肺癌细胞株间, 进行反向和正向两轮杂交, 筛查出42个具有不同侵袭能力的基因.
在以往的研究中, 我们以一对来自同一亲本, 高、低转移能力不同的人大肠癌细胞株SW620和SW480为实验对象, 该对细胞株国内外通用、遗传背景一致, 仅转移表型存在差异, 是进行大肠癌晚期差异表达研究的一种重要实验模型[13]. 通过组间"双向"差减, 成功构建了人大肠癌转移促进基因或抑制基因的两个cDNA消减文库, 分别含有235个和232个白色克隆. 两个文库中, 90%以上的白色克隆均含有插入片段[1].
消减文库构建后, 如何高效、便捷、高通量地差异筛选该文库, 是研究者面临的一个关键问题. 以往研究多用Northern blot和反向Northern印迹对随机挑取的少数片段进行检测, 其筛查量有限. cDNA微阵列用于文库筛查具有明显的优越性[14], 但成本较高、对小插入片段的检出率还有待提高. Wang et al于1991年提出了Differential Screening技术, 该技术思路和反向Northern blot相似, 除采用tester和driver mRNA反转录成的cDNA为探针, DS还使用了tester和driver未消减产物1-C、2-C的PCR扩增产物作为探针, 四种探针同时与目的片段进行杂交. 相对于其他杂交手段, DS对低丰度差异表达基因具有很高的灵敏性. 这是由于未消减产物分别来源于tester和driver cDNA酶切、连接后的产物, 不仅包含了与tester和driver mRNA相同的遗传信息, 而且其中的低丰度差异基因因得到了消减"富集"和PCR特异性放大, 指数级增强了低丰度基因的杂交信号, 这对于完整捕捉基因差异表达信息具有重要意义.
本研究从两个消减文库中随机挑取了近200个白色克隆, 进行DS筛选, 共筛选出29个差异表达cDNA片段, Northern blot验证了部分筛选结果的可靠性, 证实了DS技术是一种筛查表达文库的有效途径. 以上片段经测序及BLAST比对分析, 获得了25个差异基因, 10个为已知基因, 其中5个基因在高转移细胞株SW620中差异表达, 可能有促进转移的作用, 包括HSP10、COXII、DIS3、ACTA1及SAA; 另外5个基因的表达仅见于在SW480低转移细胞株, 具有潜在的转移抑制作用, 包括EGLN2、EIF4A、COXIII、GSTM3及mtDNA.
HSP是细胞受各种应激原刺激后诱导产生的一组应激蛋白, 具有维持蛋白稳定、促进细胞生存等功能, 近年来发现HSP与肿瘤的发生发展密切相关, HSP27和HSP70不仅参与胃癌[15]、肝癌[16]等癌变过程, 其过表达还能促进肿瘤细胞的侵袭和转移潜能, 与患者预后密切相关[17]. 但HSP10的作用机制尚不清楚. COX是线粒体呼吸链上的终止酶, 催化将还原型细胞色素C上的电子转运至氧分子这一过程. COX3是一种确认的预示家族性息肉病和家族性结肠癌的危险因子, 体内研究表明相对于正常黏膜, 结肠腺瘤和腺癌中COX3的平均表达水平进行性降低, 其抑制作用的机制可能与限制COX功能的发挥, 从而导致一些酶活性的降低有关[18]. COX2可能通过降低癌细胞对凋亡刺激的敏感性以促进肿瘤细胞的转移[19], 该蛋白可能是联系P53与线粒体功能失调的重要蛋白之一[20]. DIS3是参与有丝分裂调控的重要因子, 有研究发现与DIS3高度同源的基因在大肠癌原发灶及转移灶的表达远远高于周围正常黏膜, 提示其表达增强可能与一些大肠癌的恶性表型相关[21]. ACTA1属细胞骨架蛋白, 已有文献报道肌动蛋白及其调控蛋白的表达异常与肿瘤细胞的迁移、浸润及转移相关[22]. SAA是一种急性期反应蛋白, 他能影响细胞的黏附、迁移、增生及聚集等, 提示他在很多生理和病理过程中均有重要作用[23]. 目前其在转移中的潜在作用尚有争议[24], 本研究支持了该基因在大肠癌转移中的作用.
EGLN2编码prolyl羟化酶异构体, 他能介导氧依赖性的转录因子-α-低氧诱导因子靶定蛋白酶体的过程[25-26], 其在转移过程中的作用尚有待确认. EIF4A控制着翻译起始, 是细胞增生和癌变的一个关键点, 研究发现该基因在人黑色素瘤细胞中持续高表达, 将其反义重组子转染至黑色素细胞中, 细胞的增生能力明显降低[27]. 本研究结果提示EIF4A可能具有抑制转移的作用, 与上述报道不一致, 尚有待进一步的确认. GSTM3在对外源性物质、环境因素及癌化合物的解毒过程中发挥重要作用. 已有报道突变的GSTM3能增加对膀胱癌[28]、乳腺癌[29]等的易感性, 含有野生型纯合子GSTM3的个体能明显抵抗癌的发生. 近年来不少学者提出mtDNA参与癌变过程[30-31]. 该突变还与非小细胞肺癌的阶段性演进和预后相关[32], mtDNA的改变可能导致癌细胞的代谢异常及凋亡能力等改变, 从而参与转移过程[33].
总之, 10个筛选出来的已知基因, 基本上是一些与细胞生长分化、代谢合成、转录、凋亡及信号转导等相关的基因, 其功能的多样性也证实了肿瘤转移机制的复杂性. 除DIS3和SAA外, 其余基因与大肠癌转移的关系在本研究中首次被报道, 他们在大肠癌转移过程中的作用机制还有待进一步研究.
此外, 还筛选出了15个未知基因. 通过与人基因组序列进行比对, 发现其中6个基因定位于5号染色体上, 包括4个可能促进大肠癌转移的基因和2个抑制基因. 在大肠癌发生发展过程中, 常发生特定染色体区域片段的丢失, 其中以含有抑制基因MCC和APC的5q和18q的等位丢失最为常见[34]. 张放et al[35]发现5号染色体上存在着高频的杂和缺失, 可能有未知的抑癌基因存在; 其中在5q13.3-31.1区域中, 有与大肠癌发生密切相关的APC, MCC等抑制基因[36]. 本研究提示5号染色体上可能存在多个与大肠癌转移相关的新基因位点, 以上基因的性质和功能还有待进一步研究.
编辑: N/A
| 1. | Liang L, Ding YQ, Li X, Yang GZ, Xiao J, Lu LC, Zhang JH. Construction of a metastasis-associated gene subtracted cDNA library of human colorectal carcinoma by suppression subtraction hybridization. World J Gastroenterol. 2004;10:1301-1305. [PubMed] |
| 2. | Luo MJ, Lai MD. Identification of differentially expressed genes in normal mucosa, adenoma and adenocarcinoma of colon by SSH. World J Gastroenterol. 2001;7:726-731. [PubMed] |
| 3. | Fox SA, Loh S, Thean AL, Garlepp MJ. Identification of differentially expressed genes in murine mesothelioma cell lines of differing tumorigenicity using suppression subtractive hybridization. Biochim Biophys Acta. 2004;1688:237-244. [PubMed] [DOI] |
| 4. | Wentzensen N, Wilz B, Findeisen P, Wagner R, Dippold W, von Knebel Doeberitz M, Gebert J. Identification of differentially expressed genes in colorectal adenoma compared to normal tissue by suppression subtractive hybridization. Int J Oncol. 2004;24:987-994. [PubMed] [DOI] |
| 5. | Asai T, Tomita Y, Nakatsuka S, Hoshida Y, Myoui A, Yoshikawa H, Aozasa K. VCP (p97) regulates NFkappaB signaling pathway, which is important for metastasis of osteosarcoma cell line. Jpn J Cancer Res. 2002;93:296-304. [PubMed] [DOI] |
| 6. | Kostic C, Shaw PH. Isolation and characterization of sixteen novel p53 response genes. Oncogene. 2000;19:3978-3987. [PubMed] [DOI] |
| 7. | Sakamoto H, Mashima T, Kizaki A, Dan S, Hashimoto Y, Naito M, Tsuruo T. Glyoxalase I is involved in resistance of human leukemia cells to antitumor agent-induced apoptosis. Blood. 2000;95:3214-3218. [PubMed] |
| 8. | Koo TH, Lee JJ, Kim EM, Kim KW, Kim HD, Lee JH. Syntenin is overexpressed and promotes cell migration in metastatic human breast and gastric cancer cell lines. Oncogene. 2002;21:4080-4088. [PubMed] [DOI] |
| 9. | Wang T, Lu Y, Liu S. [Suppression subtractive hybridization in the cloning of gene fragments in relation to lung cancer metastasis]. Zhonghua Zhong Liu Za Zhi. 2001;23:296-300. [PubMed] |
| 10. | Yin J, Fan Q, Hao X, Fan D. [Identification of genes associated with human osteosarcoma metastasis suppression using suppression subtractive hybridization]. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 2002;19:213-217. [PubMed] |
| 11. | Devoogdt N, Hassanzadeh Ghassabeh G, Zhang J, Brys L, De Baetselier P, Revets H. Secretory leukocyte protease inhibitor promotes the tumorigenic and metastatic potential of cancer cells. Proc Natl Acad Sci U S A. 2003;100:5778-5782. [PubMed] [DOI] |
| 12. | Zhang L, Cilley RE, Chinoy MR. Suppression subtractive hybridization to identify gene expressions in variant and classic small cell lung cancer cell lines. J Surg Res. 2000;93:108-119. [PubMed] [DOI] |
| 13. | Hewitt RE, McMarlin A, Kleiner D, Wersto R, Martin P, Tsokos M, Stamp GW, Stetler-Stevenson WG. Validation of a model of colon cancer progression. J Pathol. 2000;192:446-454. [PubMed] [DOI] |
| 14. | Yang GP, Ross DT, Kuang WW, Brown PO, Weigel RJ. Combining SSH and cDNA microarrays for rapid identification of differentially expressed genes. Nucleic Acids Res. 1999;27:1517-1523. [PubMed] [DOI] |
| 15. | Kapranos N, Kominea A, Konstantinopoulos PA, Savva S, Artelaris S, Vandoros G, Sotiropoulou-Bonikou G, Papavassiliou AG. Expression of the 27-kDa heat shock protein (HSP27) in gastric carcinomas and adjacent normal, metaplastic, and dysplastic gastric mucosa, and its prognostic significance. J Cancer Res Clin Oncol. 2002;128:426-432. [PubMed] [DOI] |
| 16. | Zhong X, Ran YL, Lou JN, Hu D, Yu L, Zhang YS, Zhou Z, Yang ZH. Construction of human liver cancer vascular endothelium cDNA expression library and screening of the endothelium-associated antigen genes. World J Gastroenterol. 2004;10:1402-1408. [PubMed] [DOI] |
| 17. | Farkas B, Hantschel M, Magyarlaki M, Becker B, Scherer K, Landthaler M, Pfister K, Gehrmann M, Gross C, Mackensen A. Heat shock protein 70 membrane expression and melanoma-associated marker phenotype in primary and metastatic melanoma. Melanoma Res. 2003;13:147-152. [PubMed] [DOI] |
| 18. | Heerdt BG, Augenlicht LH. Effects of fatty acids on expression of genes encoding subunits of cytochrome c oxidase and cytochrome c oxidase activity in HT29 human colonic adenocarcinoma cells. J Biol Chem. 1991;266:19120-19126. [PubMed] |
| 19. | Wu H, Rao GN, Dai B, Singh P. Autocrine gastrins in colon cancer cells Up-regulate cytochrome c oxidase Vb and down-regulate efflux of cytochrome c and activation of caspase-3. J Biol Chem. 2000;275:32491-32498. [PubMed] [DOI] |
| 20. | Zhou S, Kachhap S, Singh KK. Mitochondrial impairment in p53-deficient human cancer cells. Mutagenesis. 2003;18:287-292. [PubMed] [DOI] |
| 21. | Lim J, Kuroki T, Ozaki K, Kohsaki H, Yamori T, Tsuruo T, Nakamori S, Imaoka S, Endo M, Nakamura Y. Isolation of murine and human homologues of the fission-yeast dis3+ gene encoding a mitotic-control protein and its overexpression in cancer cells with progressive phenotype. Cancer Res. 1997;57:921-925. [PubMed] |
| 22. | Jawhari AU, Buda A, Jenkins M, Shehzad K, Sarraf C, Noda M, Farthing MJ, Pignatelli M, Adams JC. Fascin, an actin-bundling protein, modulates colonic epithelial cell invasiveness and differentiation in vitro. Am J Pathol. 2003;162:69-80. [PubMed] [DOI] |
| 23. | Urieli-Shoval S, Linke RP, Matzner Y. Expression and function of serum amyloid A, a major acute-phase protein, in normal and disease states. Curr Opin Hematol. 2000;7:64-69. [PubMed] [DOI] |
| 24. | Parle-McDermott A, McWilliam P, Tighe O, Dunican D, Croke DT. Serial analysis of gene expression identifies putative metastasis-associated transcripts in colon tumour cell lines. Br J Cancer. 2000;83:725-728. [PubMed] [DOI] |
| 25. | Lieb ME, Menzies K, Moschella MC, Ni R, Taubman MB. Mammalian EGLN genes have distinct patterns of mRNA expression and regulation. Biochem Cell Biol. 2002;80:421-426. [PubMed] [DOI] |
| 26. | Taylor MS. Characterization and comparative analysis of the EGLN gene family. Gene. 2001;275:125-132. [PubMed] [DOI] |
| 27. | Eberle J, Fecker LF, Bittner JU, Orfanos CE, Geilen CC. Decreased proliferation of human melanoma cell lines caused by antisense RNA against translation factor eIF-4A1. Br J Cancer. 2002;86:1957-1962. [PubMed] [DOI] |
| 28. | Schnakenberg E, Breuer R, Werdin R, Dreikorn K, Schloot W. Susceptibility genes: GSTM1 and GSTM3 as genetic risk factors in bladder cancer. Cytogenet Cell Genet. 2000;91:234-238. [PubMed] [DOI] |
| 29. | Mitrunen K, Jourenkova N, Kataja V, Eskelinen M, Kosma VM, Benhamou S, Vainio H, Uusitupa M, Hirvonen A. Glutathione S-transferase M1, M3, P1, and T1 genetic polymorphisms and susceptibility to breast cancer. Cancer Epidemiol Biomarkers Prev. 2001;10:229-236. [PubMed] |
| 30. | Shao JY, Gao HY, Li YH, Zhang Y, Lu YY, Zeng YX. Quantitative detection of common deletion of mitochondrial DNA in hepatocellular carcinoma and hepatocellular nodular hyperplasia. World J Gastroenterol. 2004;10:1560-1564. [PubMed] [DOI] |
| 31. | Kumimoto H, Yamane Y, Nishimoto Y, Fukami H, Shinoda M, Hatooka S, Ishizaki K. Frequent somatic mutations of mitochondrial DNA in esophageal squamous cell carcinoma. Int J Cancer. 2004;108:228-231. [PubMed] [DOI] |
| 32. | Matsuyama W, Nakagawa M, Wakimoto J, Hirotsu Y, Kawabata M, Osame M. Mitochondrial DNA mutation correlates with stage progression and prognosis in non-small cell lung cancer. Hum Mutat. 2003;21:441-443. [PubMed] [DOI] |
| 33. | Amuthan G, Biswas G, Ananadatheerthavarada HK, Vijayasarathy C, Shephard HM, Avadhani NG. Mitochondrial stress-induced calcium signaling, phenotypic changes and invasive behavior in human lung carcinoma A549 cells. Oncogene. 2002;21:7839-7849. [PubMed] [DOI] |
| 34. | Sugai T, Habano W, Nakamura S, Sato H, Uesugi N, Orii S, Itoh C, Katoh R. Allelic losses of 17p, 5q, and 18q loci in diploid and aneuploid populations of multiploid colorectal carcinomas. Hum Pathol. 2000;31:925-930. [PubMed] [DOI] |
| 35. | Zhang F, Zhou C, Ling Y, Qiu G, Bai S, Liu W, He L, Peng Z. [Allelic analysis on chromosome 5 in sporadic colorectal cancer patients]. Zhonghua Zhong Liu Za Zhi. 2002;24:458-460. [PubMed] |
| 36. | Ko JM, Cheung MH, Kwan MW, Wong CM, Lau KW, Tang CM, Lung ML. Genomic instability and alterations in Apc, Mcc and Dcc in Hong Kong patients with colorectal carcinoma. Int J Cancer. 1999;84:404-409. [PubMed] [DOI] |