修回日期: 2004-09-13
接受日期: 2004-09-30
在线出版日期: 2004-12-15
目的: 筛选与克隆HBcAg激活基因, 了解其在体内的调节功能线索及机制.
方法: 以分子生物学技术构建HBcAg的真核表达载体pcDNA3.1(-)-HBcAg, 以表达质粒pcDNA3.1(-)-HBcAg转染HepG2细胞, 以空载体pcDNA3.1(-)为平行对照, 制备转染后的细胞裂解液, 提取mRNA. 应用基因表达谱芯片技术对差异表达mRNA进行检测和分析.
结果: HBcAg表达质粒pcDNA3.1(-)-HBcAg经酶切鉴定和DNA测序鉴定正确. 经基因表达谱芯片分析, 29种基因的表达水平上调, 17种基因的表达水平下调.
结论: 筛选到一些与细胞内信号传导、免疫调节、细胞凋亡、蛋白质翻译合成、肿瘤发生相关的HBcAg反式调节的靶基因.
引文著录: 徐志强, 成军, 张鸿飞, 王建军, 刘妍, 纪冬. 应用表达谱芯片技术筛选HBcAg反式调节基因. 世界华人消化杂志 2004; 12(12): 2886-2890
Revised: September 13, 2004
Accepted: September 30, 2004
Published online: December 15, 2004
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2004; 12(12): 2886-2890
- URL: https://www.wjgnet.com/1009-3079/full/v12/i12/2886.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i12.2886
乙型肝炎病毒(HBV)属于嗜肝DNA病毒, 是一种严重危害人类健康的致病因子. 不仅引起急、慢性病毒性肝炎, 而且与肝纤维化(LF)、肝细胞癌(HCC)的发生发展密切相关[1]. HBcAg是由HBV DNA C基因区编码的一种结构性蛋白, 有保守的三维结构, 可因血清型不同而有183-185个氨基酸残基(aa), 在乙型肝炎病毒(HBV)的生活周期中, 病毒核心抗原(HBcAg)、病毒mRNA和DNA聚合酶共同构成核心颗粒, 在核心颗粒中完成病毒DNA的合成[2-3]. 基因表达谱芯片技术(cDNA microarray)是由大量目的基因片段有序、密集地固定于玻片或尼龙膜上制成芯片, 将两组组织或细胞的mRNA逆转录成cDNA, 掺入荧光标记, 同时与芯片杂交, 通过扫描分析每一位置的荧光信息可以快速有效地检测到二者间差异表达的基因[4-7]. 为从不同角度对HBcAg的反式调节基因进行验证及了解HBcAg对于肝细胞基因表达谱的影响, 我们应用基因表达谱芯片技术检测肝癌细胞系在HBcAg基因转染后差异表达的基因谱的变化.
HepG2细胞及感受态E.coli JM109(本室保存), pcDNA3.1(-)真核表达载体(Invitrogen); FuGENE6转染试剂, 总RNA提取试剂Trizol购自Gibco公司, mRNA Purification试剂盒购自Amersham Pharmacia Biotech, 表达谱芯片由上海联合基因有限公司提供. pcDNA3.1(-)-HBcAg表达载体由本室构建.
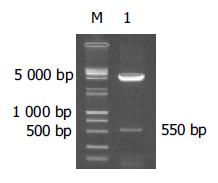
以质粒pCP10中所含的HBV ayw亚型的DNA序列为模板, 用PCR方法扩增HBcAg基因片段. 上游引物为: 5'-GCT AGC CAT GGA CAT CGA CCC TTA TA-3', 下游引物为: 5'-GGT ACC CTA ACA TTG AGG TTC CCG AG-3'. PCR产物经10 g/L琼脂糖凝胶电泳, 切胶, 以Geneclean Ⅱ kit(Bio 101公司)回收纯化550 bp DNA条带.
在35 mm培养皿中常规培养HepG2细胞, 细胞生长至对数期时, 以脂质体转染试剂FuGENE分别将2 μg pcDNA3.1(-)-HBcAg和空载体pcDNA3.1(-)转染HepG2细胞, 48 h后收获细胞, 每5×106个细胞加入1 mL Trizol试剂, 立即于液氮中保存.
使用Trizol试剂一步法提取PFA及0.9% NaCl处理的Jurkat细胞总RNA(分别标记为实验组和对照组), 样品经分光光度计检测吸光度A值, 并行热稳定实验, 于-20 ℃和70 ℃保温1 h后, 经琼脂糖凝胶电泳检测28 s、18 s条带变化. 以Qiagen公司Oligotex mRNA Midi Kit纯化得mRNA. 操作按说明书进行, 并行电泳检测.
参照Schena et al[1]方法逆转录标记cDNA探针并纯化. Cy3-dUTP标记对照组细胞mRNA(5 μg), Cy5-dUTP标记实验组细胞mRNA(5 μg). 乙醇沉淀后溶解在20 μL 5×SSC+2g/L SDS杂交液中.
芯片包含的1 152个cDNA由上海联合基因有限公司提供, 包括原癌基因和抑癌基因、免疫调节相关基因、细胞凋亡和应激反应蛋白相关基因、信号转导相关基因等. 以通用引物进行PCR扩增, PCR产物长度为1 000-3 000 bp. 靶基因以0.5 g/L溶解于3×SSC溶液中, 用Cartesian公司的Cartesian 7 500点样仪及TeleChem公司的硅烷化玻片进行点样. 玻片经水合(2 h)、室温干燥(30 min), UV交联, 再分别用2 g/L SDS、水及2 g/L的硼氢化钠溶液处理10 min, 晾干备用.
将基因芯片和杂交探针在95 ℃水浴变性5 min, 将混合探针加在基因芯片上, 置于60 ℃杂交15-17 h. 依次以2×SSC+2 g/L SDS、1×SSC+2 g/L SDS、1×SSC洗涤10 min, 室温晾干.
用General Scanning公司的ScanArray 3000扫描芯片. 用预先选定的内参照基因(24条管家基因, 每个基因点2个点, 共48个点)对Cy3和Cy5的原始提取信号进行均衡和修正. 用ImaGene3.0软件分析Cy3、Cy5两种荧光信号的强度, 计算Cy5/Cy3比值.阳性结果判断: Cy5/Cy3>2.0, 红色荧光, 显示表达增强; Cy5/Cy3<0.5, 为绿色荧光, 显示表达减弱.
pcDNA3.1(-)-HBcAg载体经Kpn I/Nhe I双酶切鉴定可见550 bp的HBcAg基因DNA条带, 证明载体构建正确(图1).
总RNA的吸光度A260/A280 = 1.82, 热稳定实验70 ℃保温1 h与-20 ℃ 1 h的电泳条带比较, 显示28 S条带无明显降解, 电泳结果证实已抽提高纯度的总RNA.
在芯片上共有1 152个cDNA. 为了监控芯片杂交体系, 在芯片上设置了阴性对照(8条水稻基因, 共8个点), 这些点的杂交信号均很低, 证实了数据的可靠性. 由于实验组探针标记Cy5荧光素(呈红色), 对照组探针标记Cy3荧光素(呈绿色), 红绿颜色的差异就显示该基因在实验组和对照组中基因表达水平上的差异, 黄色代表表达水平无差异.按阳性标准, 从1 000个基因中筛选出差异表达基因共46条, 其中29条基因表达显著增强(Cy5/Cy3>2.0), 17条基因表达显著降低(Cy5/Cy3<0.5).
表达显著增强的基因有29条(表1). 表达显著降低的基因有17条(表2).
| 序号 | GenBank登录号 | 编码蛋白 | Cy5/Cy3 |
| 1 | NM_005917 | 苹果酸脱氢酶1(MDH1) | 2.010 |
| 2 | NM_013943 | 细胞内氯化物通道蛋白4(CLIC4) | 2.018 |
| 3 | NM_000176 | 核受体亚家族3, C组, 1号(NR3C1) | 2.020 |
| 4 | AF012086 | Ran结合蛋白2(RanBP2alpha) | 2.034 |
| 5 | AF072928 | 肌管蛋白相关蛋白6 | 2.038 |
| 6 | AK055660 | 克隆IMR321000219 | 2.045 |
| 7 | NM_001892 | 酪蛋白激酶1, α1(CSNK1A1) | 2.065 |
| 8 | NM_004661 | 细胞分裂周期蛋白23(CDC23) | 2.068 |
| 9 | NM_005335 | 造血细胞特异性Lyn底物1(HCLS1) | 2.071 |
| 10 | AF070674 | 凋亡蛋白1抑制剂(MIHC) | 2.085 |
| 11 | AB014731 | 平滑肌细胞相关蛋白3(SMAP-3) | 2.113 |
| 12 | NM_003591 | cullin2(CUL2) | 2.122 |
| 13 | NM_005836 | 翻译抑制蛋白p14.5(UK114) | 2.133 |
| 14 | NM_053274 | FK506结合蛋白相关蛋白(FAP48) | 2.145 |
| 15 | AJ243706 | 视网膜母细胞瘤结合蛋白(rbbp2h1a gene) | 2.162 |
| 16 | NM_000817 | 谷氨酸脱羧酶1(GAD1) | 2.170 |
| 17 | NM_007268 | 免疫球蛋白超家族蛋白(Z39IG) | 2.178 |
| 18 | NM_006407 | 维生素A反应性细胞骨架相关蛋白(JWA) | 2.289 |
| 19 | NM_003129 | 鲨烯环氧酶(SQLE) | 2.291 |
| 20 | D50683 | 转化生长因子βⅡ受体α | 2.326 |
| 21 | NM_006995 | Butyrophilin亚家族2成员A2(BTN2A2) | 2.353 |
| 22 | NM_006918 | 固醇C5去饱和酶(SC5DL) | 2.366 |
| 23 | NM_006183 | 神经降压素(NTS) | 2.398 |
| 24 | NM_002736 | cAMP依赖的蛋白激酶调节亚单位2b(PRKAR2B) | 2.429 |
| 25 | NM_001316 | 染色体分离蛋白1(CSE1L) | 2.556 |
| 26 | AF134802 | cofilin亚型1 | 2.614 |
| 27 | BC017169 | 未知基因MGC: 9535 | 2.619 |
| 28 | NM_007034 | DnaJ样热休克蛋白40(HLJ1) | 2.630 |
| 29 | NM_014887 | 假设蛋白(CG005) | 2.813 |
| 序号 | GenBank登录号 | 编码蛋白 | Cy5/Cy3 |
| 1 | NM_015385 | Src同源区3蛋白5(SH3D5) | 0.352 |
| 2 | AB032951 | KIAA1125蛋白 | 0.358 |
| 3 | NM_021138 | TNF受体相关因子2(TRAF2) | 0.365 |
| 4 | AF110908 | TNF受体相关因子3(TRAF-3) | 0.481 |
| 5 | NM_000269 | 表达在NME1的非转移性细胞蛋白1 | 0.373 |
| 6 | NM_006609 | 丝裂原激活蛋白激酶激酶激酶2(MAP3K2) | 0.415 |
| 7 | NM_001910 | 组织蛋白酶E(CTSE) | 0.447 |
| 8 | NM_002388 | 微小染色体维持缺陷蛋白3(MCM3) | 0.450 |
| 9 | NM_006913 | 环指蛋白5(RNF5) | 0.452 |
| 10 | NM_005345 | Mr 70 000热休克蛋白1A(HSPA1A) | 0.461 |
| 11 | AF090094 | 克隆IMAGE 172979 | 0.464 |
| 12 | NM_002421 | 基质金属蛋白酶1(MMP1) | 0.468 |
| 13 | NM_004728 | DEAD/H盒多肽21(DDX21) | 0.367 |
| 14 | NM_013330 | 表达在二磷酸核苷激酶的非转移性细胞蛋白7(NME7) | 0.485 |
| 15 | NM_006077 | 钙结合的过敏相关的自身抗体1(CBARA1) | 0.492 |
| 16 | NM_001343 | 果蝇同源的丝裂原反应磷蛋白2(DAB2) | 0.496 |
| 17 | NM_033138 | 调钙蛋白的结合蛋白1(CALD1) | 0.497 |
HBcAg存在于Dane颗粒的核心, 是HBV的结构蛋白即病毒核壳蛋白, 他反映血清中Dane颗粒的存在及肝内HBV的复制. HBcAg由HBV基因组的C开放读码区(ORF)编码, 从第二个ATG(1 901 nt)起始翻译(终止密码子在2 458 nt). HBcAg有保守的三维结构, 可因血清型不同而有183-185个氨基酸残基(aa), Mr 21 000, 180个HBcAg形成27 nm的正20面体以构成核心颗粒. HBcAg已被证明含有HBV特异性DNA聚合酶, 是有能力合成DNA的病毒特异性酶, 是HBV复制的标志. 存在于乙型肝炎患者血液和肝组织内的HBcAg具有重要的生物学特性和临床病理意义. HBcAg具有高度免疫原性, 在T细胞和B细胞水平上, HBcAg的抗原性较HBeAg强100倍, 几乎所有HBV感染者都产生抗-HBc, HBcAg既可作为T细胞依赖性抗原, 也可作为非T细胞依赖性抗原, 诱导强烈细胞毒T淋巴细胞(CTL)应答[8]. 对HBcAg的免疫应答在病毒清除中可能有重要作用[9-10]. HBcAg有鱼精蛋白样亲胞核性的羧基末端, 可介导细胞核内转运信号, 使大量HBcAg进入细胞核内, 而其他嗜肝DNA病毒的核壳蛋白都不向核内转运. 在病毒成熟过程中, 核壳和外膜相互作用, 形成病毒颗粒分泌的信号[11-12]. HBcAg具有保护病毒mRNA, 防止其被RNA酶降解的作用, 对于乙肝病毒前基因组RNA的装配、基因组DNA的合成具有重要的作用. HBcAg有高免疫原性, HBcAg与HBeAg一同为细胞毒性T细胞(Tc)细胞识别的主要靶抗原. HBcAg在HBV的装配和致病机制中是必不可少的, HBcAg是否还通过其他途径影响免疫应答, 损害肝细胞的功能, 是否参与肝细胞癌的发生, 如何作用, 是研究HBcAg复杂功能中需要解决的关键点. 寻找肝细胞中HBcAg的相互作用的基因, 并进一步探明其机制, 对解答上述问题有着重要意义.
我们用分子生物学技术构建了HBcAg的真核表达载体pcDNA3.1(-)-HBcAg, 并用空载体作为阴性对照, 利用脂质体转染HepG2细胞, 之后从中提取总RNA, 逆转录为cDNA, 进行基因芯片技术分析. 结果表明, 29种基因的表达水平上调, 17种基因的表达水平下调.
在差异表达的基因中包括各种参与氧化还原反应的酶类(苹果酸脱氢酶、谷氨酸脱羧酶1、鲨烯环氧酶、固醇C5去饱和酶)、细胞信号转导和细胞周期相关蛋白基因(HCLS1、翻译抑制蛋白、转化生长因子βⅡ受体α、酪蛋白激酶1、cAMP依赖蛋白激酶、SH3D5)、细胞凋亡(CLIC4、MIHC、TRAF2、TRAF3)、免疫反应调节蛋白基因(FKBP相关蛋白、cullin2)及肿瘤发生相关基因(视网膜母细胞瘤结合蛋白)等及未知基因.
上调的基因中, CLIC4是氯化物细胞内通道蛋白家族中的成员[13], 存在于角化细胞的线粒体和细胞质内, 参与细胞凋亡过程[14-15], 在多重压力作用下, CLIC4从胞质向胞核内迁移, 免疫金电镜和共聚焦分析显示核内CLIC4表达先于细胞凋亡. Ran是真核细胞核运输过程中必需的小GTP酶, 在细胞mRNA加工, 细胞周期调节等方面发挥作用, Ran结合蛋白2即RanBP2是核孔蛋白, 在细胞核浆物质转运过程中起重要作用[16-17]. 细胞分裂周期蛋白CDC23与酿酒酵母的CDC23高度同源, 是促进细胞分裂后期复合物(anaphase-promoting complex, APC)的成员, 对于细胞周期由G2期向M期过渡是必不可少的. APC能够催化细胞周期蛋白B与泛素(cyclin B-ubiquitin)形成共轭复合物, 在泛素介导的细胞周期蛋白B的蛋白水解过程中起重要作用, HBcAg通过上调CDC23的表达对细胞周期有调控作用[18]. HCLS1基因表达于人造血细胞内[19], 编码蛋白-酪氨酸激酶的底物, 这种细胞内蛋白与B和T淋巴细胞抗原受体的信号传导有关. 凋亡蛋白1抑制剂能够抑制细胞凋亡, 促进肿瘤细胞的生长[20]. Cullin2基因为肿瘤抑制基因[21]. FKBP相关蛋白(FAP48)是细胞内与免疫调节有密切关系的蛋白, 可以与肽基脯氨酰异构酶FK506结合蛋白59(FK506-binding protein 59, FKBP59)和FKBP12蛋白结合, 由于其相对分子质量为Mr 48 000, 故命名为FKBP相关蛋白(FAP48), 此三种蛋白均具有大环内酯类分子的结合位点, 可能是这些免疫抑制剂药物受体的天然的共同配体分子. FAP48与FKBP分子之间的结合可被大环内酯类物FK506所阻断, 说明免疫亲和素分子中的结合位点与FK506的结合位点相重叠. FAP48的过表达可以抑制细胞的增生, 促进IL2的合成, 在免疫调节及临床疾病的发生、发展过程中具有十分重要的作用[22-23]. 鲨烯环氧酶(SQLE)与脂类代谢相关, SQLE参与胆固醇的生物合成, 是胆固醇生物合成过程中重要的限速酶, 其升高可以促进血中胆固醇水平升高, 许多药物通过抑制其活性可起到降低血中胆固醇的作用[24-26]. cAMP依赖的蛋白激酶调节亚单位ⅡβPRKAR2B是参与细胞信号转导途径主要因子[27].
在下调的基因中, SH3D5[28]、丝裂素活化的蛋白激酶激酶激酶2参与细胞内信号传导途径. TRAF2、TRAF-3具有重要的介导肿瘤坏死因子信号的作用, 能够活化核因子kappaB、JNK和细胞凋亡[29-30].
微小染色体维持缺陷3蛋白是DNA复制中起重要作用的核蛋白[31]. DEAD/H盒多肽(DDX21)是推定的重要的RNA解旋酶, 参与众多细胞的RNA二级结构加工过程, 如翻译起始、核糖体RNA合成及加工过程. DDX21在肿瘤组织中低表达, 而在正常组织中有显著的高水平表达. HBcAg对DDX21的表达有下调作用, 提示, HBcAg蛋白在一定程度上可能下调细胞核糖体RNA加工与合成[32]. 钙调素结合蛋白1(CALD1)与体内金属代谢有关. 基质金属蛋白酶1(MMP1)[33]具有细胞毒性作用, 其可能的机制是通过分解赖以维持细胞生存的基质蛋白而提高细胞毒性, 也可分解底物产生促使细胞死亡的分子, 另外还可作用于细胞表面受体而影响细胞生存.
总之, 我们利用基因表达谱芯片分析了HBcAg对肝细胞基因表达谱的影响, HBcAg能够上调或下调HepG2细胞中许多不同基因表达的变化, 这些基因变化是复杂的, 有的甚至是矛盾的, 这些基因与细胞信号转导、细胞增生与分化、免疫应答、物质运输、细胞凋亡、肿瘤发生等生物过程密切相关. 我们的实验结果为了解HBcAg在肝细胞中的生物学效应提供了某些线索, 当然, 还需要进一步的实验加以深入研究.
编辑: N/A
| 2. | Pumpens P, Grens E. Hepatitis B core particles as a universal display model: a structure-function basis for development. FEBS Lett. 1999;442:1-6. [DOI] |
| 3. | Le Pogam S, Shih C. Influence of a putative intermolecular interaction between core and the pre-S1 domain of the large envelope protein on hepatitis B virus secretion. J Virol. 2002;76:6510-6517. [PubMed] [DOI] |
| 4. | Yang GP, Ross DT, Kuang WW, Brown PO, Weigel RJ. Combining SSH and cDNA microarrays for rapid identification of differentially expressed genes. Nucleic Acids Res. 1999;27:1517-1523. [PubMed] [DOI] |
| 5. | Zhang MQ. Large-scale gene expression data analysis: a new challenge to computational biologists. Genome Res. 1999;9:681-688. [PubMed] |
| 6. | Golub TR, Slonim DK, Tamayo P, Huard C, Gaasenbeek M, Mesirov JP, Coller H, Loh ML, Downing JR, Caligiuri MA. Molecular classification of cancer: class discovery and class prediction by gene expression monitoring. Science. 1999;286:531-537. [PubMed] [DOI] |
| 8. | Livingston BD, Crimi C, Fikes J, Chesnut RW, Sidney J, Sette A. Immunization with the HBV core 18-27 epitope elicits CTL responses in humans expressing different HLA-A2 supertype molecules. Hum Immunol. 1999;60:1013-1017. [PubMed] [DOI] |
| 9. | Marinos G, Torre F, Chokshi S, Hussain M, Clarke BE, Rowlands DJ, Eddleston AL, Naoumov NV, Williams R. Induction of T-helper cell response to hepatitis B core antigen in chronic hepatitis B: a major factor in activation of the host immune response to the hepatitis B virus. Hepatology. 1995;22:1040-1049. [PubMed] [DOI] |
| 10. | Shimada N, Yamamoto K, Kuroda MJ, Terada R, Hakoda T, Shimomura H, Hata H, Nakayama E, Shiratori Y. HBcAg-specific CD8 T cells play an important role in virus suppression, and acute flare-up is associated with the expansion of activated memory T cells. J Clin Immunol. 2003;23:223-232. [PubMed] [DOI] |
| 11. | Watts NR, Conway JF, Cheng N, Stahl SJ, Belnap DM, Steven AC, Wingfield PT. The morphogenic linker peptide of HBV capsid protein forms a mobile array on the interior surface. EMBO J. 2002;21:876-884. [PubMed] [DOI] |
| 12. | Karpenko LI, Ivanisenko VA, Pika IA, Chikaev NA, Eroshkin AM, Veremeiko TA, Ilyichev AA. Insertion of foreign epitopes in HBcAg: how to make the chimeric particle assemble. Amino Acids. 2000;18:329-337. [PubMed] [DOI] |
| 13. | Berryman MA, Goldenring JR. CLIC4 is enriched at cell-cell junctions and colocalizes with AKAP350 at the centrosome and midbody of cultured mammalian cells. Cell Motil Cytoskeleton. 2003;56:159-172. [PubMed] [DOI] |
| 14. | Suh KS, Mutoh M, Nagashima K, Fernandez-Salas E, Edwards LE, Hayes DD, Crutchley JM, Marin KG, Dumont RA, Levy JM. The organellular chloride channel protein CLIC4/mtCLIC translocates to the nucleus in response to cellular stress and accelerates apoptosis. J Biol Chem. 2004;279:4632-4641. [PubMed] [DOI] |
| 15. | Fernández-Salas E, Suh KS, Speransky VV, Bowers WL, Levy JM, Adams T, Pathak KR, Edwards LE, Hayes DD, Cheng C. mtCLIC/CLIC4, an organellular chloride channel protein, is increased by DNA damage and participates in the apoptotic response to p53. Mol Cell Biol. 2002;22:3610-3620. [PubMed] [DOI] |
| 16. | Miyauchi Y, Yogosawa S, Honda R, Nishida T, Yasuda H. Sumoylation of Mdm2 by protein inhibitor of activated STAT (PIAS) and RanBP2 enzymes. J Biol Chem. 2002;277:50131-50136. [PubMed] [DOI] |
| 17. | Nothwang HG, Rensing C, Kübler M, Denich D, Brandl B, Stubanus M, Haaf T, Kurnit D, Hildebrandt F. Identification of a novel Ran binding protein 2 related gene (RANBP2L1) and detection of a gene cluster on human chromosome 2q11-q12. Genomics. 1998;47:383-392. [PubMed] [DOI] |
| 18. | Zhao N, Lai F, Fernald AA, Eisenbart JD, Espinosa R, Wang PW, Le Beau MM. Human CDC23: cDNA cloning, mapping to 5q31, genomic structure, and evaluation as a candidate tumor suppressor gene in myeloid leukemias. Genomics. 1998;53:184-190. [PubMed] [DOI] |
| 19. | Egashira M, Kitamura D, Watanabe T, Niikawa N. The human HCLS1 gene maps to chromosome 3q13 by fluorescence in situ hybridization. Cytogenet Cell Genet. 1996;72:175-176. [PubMed] [DOI] |
| 20. | Gordon GJ, Appasani K, Parcells JP, Mukhopadhyay NK, Jaklitsch MT, Richards WG, Sugarbaker DJ, Bueno R. Inhibitor of apoptosis protein-1 promotes tumor cell survival in mesothelioma. Carcinogenesis. 2002;23:1017-1024. [PubMed] [DOI] |
| 21. | Duerr EM, Gimm O, Neuberg DS, Kum JB, Clifford SC, Toledo SP, Maher ER, Dahia PL, Eng C. Differences in allelic distribution of two polymorphisms in the VHL-associated gene CUL2 in pheochromocytoma patients without somatic CUL2 mutations. J Clin Endocrinol Metab. 1999;84:3207-3211. [PubMed] [DOI] |
| 23. | Krummrei U, Baulieu EE, Chambraud B. The FKBP-associated protein FAP48 is an antiproliferative molecule and a player in T cell activation that increases IL2 synthesis. Proc Natl Acad Sci USA. 2003;100:2444-2449. [PubMed] [DOI] |
| 24. | Hiyoshi H, Yanagimachi M, Ito M, Yasuda N, Okada T, Ikuta H, Shinmyo D, Tanaka K, Kurusu N, Yoshida I. Squalene synthase inhibitors suppress triglyceride biosynthesis through the farnesol pathway in rat hepatocytes. J Lipid Res. 2003;44:128-135. [PubMed] |
| 25. | Mukherjee PK, Leidich SD, Isham N, Leitner I, Ryder NS, Ghannoum MA. Clinical trichophyton rubrum strain exhibiting primary resistance to terbinafine. Antimicrob Agents Chemother. 2003;47:82-86. [DOI] |
| 26. | Sawada M, Matsuo M, Seki J. Inhibition of cholesterol synthesis causes both hypercholesterolemia and hypocholesterolemia in hamsters. Biol Pharm Bull. 2002;25:1577-1582. [PubMed] [DOI] |
| 27. | Solberg R, Sistonen P, Träskelin AL, Bérubé D, Simard J, Krajci P, Jahnsen T, de la Chapelle A. Mapping of the regulatory subunits RI beta and RII beta of cAMP-dependent protein kinase genes on human chromosome 7. Genomics. 1992;14:63-69. [PubMed] [DOI] |
| 28. | Chamnongpol S, Li X. SH3 domain protein-binding arrays. Methods Mol Biol. 2004;278:183-190. [DOI] |
| 29. | Gotoh Y, Oishi K, Shibata H, Yamagiwa A, Isagawa T, Nishimura T, Goyama E, Takahashi M, Mukai H, Ono Y. Protein kinase PKN1 associates with TRAF2 and is involved in TRAF2-NF-kappaB signaling pathway. Biochem Biophys Res Commun. 2004;314:688-694. [PubMed] [DOI] |
| 30. | Ramalingam P, Chu WS, Tubbs R, Rybicki L, Pettay J, Hsi ED. Latent membrane protein 1, tumor necrosis factor receptor-associated factor (TRAF) 1, TRAF-2, TRAF-3, and nuclear factor kappa B expression in posttransplantation lymphoproliferative disorders. Arch Pathol Lab Med. 2003;127:1335-1339. [PubMed] |
| 31. | Kubota Y, Mimura S, Nishimoto S, Takisawa H, Nojima H. Identification of the yeast MCM3-related protein as a component of Xenopus DNA replication licensing factor. Cell. 1995;81:601-609. [PubMed] [DOI] |
| 32. | Valdez BC, Yang H, Hong E, Sequitin AM. Genomic structure of newly identified paralogue of RNA helicase II/Gu: detection of pseudogenes and multiple alternatively spliced mRNAs. Gene. 2002;284:53-61. [PubMed] [DOI] |
| 33. | Conant K, St Hillaire C, Nagase H, Visse R, Gary D, Haughey N, Anderson C, Turchan J, Nath A. Matrix metalloproteinase 1 interacts with neuronal integrins and stimulates dephosphorylation of Akt. J Biol Chem. 2004;279:8056-8062. [PubMed] [DOI] |