修回日期: 2004-06-09
接受日期: 2004-08-28
在线出版日期: 2004-10-15
目的: 筛选与克隆重组人肝再生增强因子激活基因, 了解其在体内的调节功能线索及机制.
方法: 应用大肠杆菌系统, 表达、纯化重组人肝再生增强因子蛋白. 刺激HepG2细胞, 以溶剂PH7.8的磷酸盐缓冲液为平行对照, 制备转染后的细胞裂解液, 提取mRNA并逆转录为cDNA, 经Rsa I酶切后, 将实验组cDNA分成两组, 分别与两种不同的接头衔接, 再与对照组cDNA进行两次消减杂交及两次抑制性聚合酶链反应(PCR), 将产物与T/A载体连接, 构建cDNA消减文库, 并转染大肠杆菌进行文库扩增, 随机挑选克隆PCR扩增后进行测序及同源性分析.
结果: 成功构建重组人肝再生增强因子激活基因差异表达的cDNA消减文库.文库扩增后得到30个阳性克隆, 进行菌落PCR分析, 均得到200-1000 bp插入片段.对插入片段测序, 并通过生物信息学分析获得其全长基因序列, 结果共获得19种编码基因, 包括15种已知基因和4种未知基因.
结论: 筛选到的cDNA全长序列, 包括一些与细胞生长调节、肿瘤免疫发生及物质代谢密切相关的蛋白编码基因, 推测了重组人肝再生增强因子在体内可能存在的调控机制的线索, 尚需进一步的实验证明.
引文著录: 刘蔚, 成军, 张连峰, 纪冬, 刘妍, 郭江, 张黎颖. 应用抑制性消减杂交技术筛选rhALR调节基因. 世界华人消化杂志 2004; 12(10): 2463-2466
Revised: June 9, 2004
Accepted: August 28, 2004
Published online: October 15, 2004
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2004; 12(10): 2463-2466
- URL: https://www.wjgnet.com/1009-3079/full/v12/i10/2463.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i10.2463
近年来对于肝细胞再生的研究主要集中于几种调节因子上, 从胰岛素开始[1], 至今已发现参与调节肝再生的刺激和抑制因子共20多种. 肝再生增强因子是一种多肽调节因子, 具有重要的生物学功能, 与高等生物线粒体的生成、细胞分裂周期调节及肝脏, 睾丸等重要脏器发育有关, 最主要是能特异的刺激肝源细胞的增生[2-5].对于ALR的产生、分泌、体内转运的过程都存有诸多疑问. 因此为了进一步研究该蛋白质的功能, 我们应用抑制性消减杂交技术(suppression subtractive hybridization, SSH), 构建rhALR激活相关基因差异表达的cDNA消减文库, 筛选相关的靶基因片段, 并应用生物信息学(Bioinformatics)技术获得其基因全长序列, 为研究rhALR在体内的调节功能机制提供一定的线索.
HepG2细胞及感受态大肠杆菌JM109(本室保存); FuGENE6 转染试剂(Roche), mRNA Purification试剂盒(Amersham Pharmacia Biotech), PCR-Select cDNA Subtraction试剂盒, 50×PCR Enzyme Mix、Advantage PCR Cloning试剂盒(Clontech), High Pure PCR Product Purification试剂盒(Boehringer Mannheim).
1.2.1 重组人肝再生增强因子的制备: 应用聚合酶链反应(PCR)技术扩增人ALR的cDNA基因片段, 克隆到大肠杆菌表达载体pBV220中, 转化大肠杆菌JM109, 制备工程菌. 发酵后, 分离纯化包涵体, 经过包涵体的变性和复性, 然后利用阴离子交换层析、阳离子交换层析和分子筛等技术, 纯化重组人肝再生增强因子, 通过聚丙烯酰胺凝胶电泳(PAGE)检测显示, 制备的重组人肝再生增强因子纯度在98%以上, 具有促进肝细胞再生的生物学活性.
1.2.2 细胞刺激及细胞mRNA提取: 用FuGENE6 转染试剂将rhALR终浓度为0.01 g/L及PH7.8的溶剂磷酸盐缓冲液分别刺激35 mm平皿HepG2 细胞, 24 h后收获细胞. 使用mRNA Purification试剂盒, 直接提取经rhALR及磷酸盐缓冲液刺激的HepG2细胞mRNA, 经琼脂糖凝胶电泳及分光光度计进行定性定量分析.
1.2.3 双链cDNA(dscDNA)合成: 用Clontech公司的PCR-SelectTM cDNA Subtraction Kit中的试剂, 以获得的mRNA为模板逆转录合成cDNA.
1.2.4 消减杂交文库的建立: 采用Clontech公司的PCR-SelectTM cDNA Subtraction Kit, 常规SSH方法按说明书进行: 经rhALR及磷酸盐缓冲液刺激的HepG2 细胞cDNA分别标记为Tester和Driver, 经RsaⅠ(一种识别4碱基序列的内切酶)消化, 产生相对较短的平端片段, 纯化酶切产物. 将Tester的cDNA分为两份, 分别连接试剂盒提供的特殊设计的寡核苷酸接头Adapter 1和Adapter 2, 然后与过量的Driver cDNA进行杂交;合并两种杂交产物后再与Driver cDNA作第2次杂交;然后将杂交产物做选择性PCR扩增, 使Tester cDNA中特异性表达或高表达的片段得到特异性扩增.
1.2.5 克隆鉴定分析: 扩增产物与pGEM-Teasy载体连接, 转化JM109感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37℃培养18 h. 挑取白色菌落, 增菌, 以pGEM-Teasy载体多克隆位点两端T7/SP6引物进行菌落PCR扩增, 证明含有插入片段后(200-1 000 bp), 测序(上海博亚公司). 应用生物信息学技术将测得序列与GenBank数据库进行同源性分析.
使用高质量的mRNA是保证cDNA高产量的前提. 紫外分光检测显示, 经rhALR及溶剂磷酸盐缓冲液刺激的HepG2 细胞mRNA分别为6.5 mg和4.6 mg, A260/A 280=1.87. 10 g/L琼脂糖凝胶电泳见 mRNA为大于0.5 kb清晰慧尾片状条带.
dscDNA与接头连接效率的高低是决定抑制性消减杂交成败的最关键步骤. 将连接有adaptor l和adaptor 2的两组dscDNA分别用不同的特异性引物(看家基因甘油三磷酸脱氢酶G3PDH引物)进行28个循环扩增, 产物用20 g/L琼脂糖凝胶电泳鉴定.结果显示两组dscDNA扩增产物浓度相当, 说明dscDNA已与接头高效率连接.
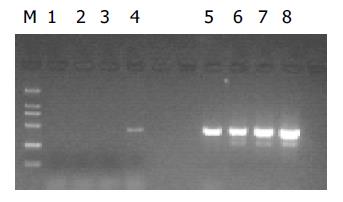
分别取抑制性及非抑制性PCR产物l mL为模板, 用看家基因G3PDH引物进行PCR扩增, 分别在18, 23, 28, 33次循环结束时从体系中吸取5 mL进行电泳鉴定.结果显示, 与非抑制性PCR产物相比, 抑制性PCR产物中G3PDH基因产物大大减少, 说明所构建的消减文库具有很高的消减效率(图1).
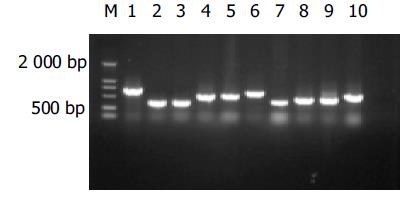
杂交产物经两轮PCR扩增后, 扩增产物与pGEM-Teasy载体连接, 转化JM109感受态细菌后有较多菌落生长, 以pGEM-Teasy载体多克隆位点两端T7/SP6引物进行菌落PCR扩增, 结果显示为200-1 000 bp大小不等的插入片段, 所获得的克隆中几乎均含有插入片段, 这些条带可能代表差异表达的基因片段(图2).
随机选择30个克隆测序, 与GenBank数据库进行初步比较, 发现其中含有4个克隆未检索到任何对应的相似序列, 可能代表了某些新基因, 进一步的分析和功能鉴定正在进行中.有15个与已知基因的部分序列高度同源(96-100%, 表1).
| 同源蛋白基因 | 同源菌株 | 同源性 |
| 线粒体 | 4 | 99-100% |
| 线粒体膜易位酶 | 1 | 100% |
| 肿瘤蛋白翻译调节 | ||
| 因子1 (TPT1) | 1 | 100% |
| 酪氨酸磷酸酶ⅣA | 1 | 97% |
| 核糖体蛋白S21 | 1 | 100% |
| 核糖体蛋白L9 | 1 | 100% |
| 乳酸脱氢酶 | 1 | 100% |
| 硫氧还蛋白 | 1 | 96% |
| 载脂蛋白H | 1 | 99% |
| 细胞周期蛋白依赖激酶1,Cdk1 | 1 | 99% |
| XTP5 | 1 | 97% |
| 蛋白酶体26S亚基 | 1 | 100% |
| 人染色体克隆 | 4 | 99-100% |
人和动物血清中存在着促进肝再生的蛋白质因子, 早期的研究中统称为肝刺激物质(HSS)[6-7]. 研究表明, 肝再生增强因子(ALR)是HSS的主要成分[8]. 关于ALR蛋白的生物学功能, Lisowsky et al[9-10]首先发现的是scERV1对于细胞的存活以及线粒体的生物学生成过程有关, 进一步的研究结果表明scERV1基因对于酵母细胞的细胞周期具有重要的调节功能. Lee et al[11]的研究发现, 酵母细胞的ERV1 的编码产物由189个氨基酸残基组成, 这种蛋白对于线粒体功能和细胞的存活是必不可少的. 这种蛋白具有黄素相关性巯基氧化酶的催化活性, 与二硫键的形成有关.ERV1p蛋白氨基末端序列的缺失研究表明, 这种蛋白质酶学催化活性位点位于羧基末端的15 kDa的部位. ERV1p羧基末端部分保留了与FAD结合和催化二硫键形成的能力, 但不能象完整蛋白那样形成二聚体形式. 这种蛋白的羧基末端具有CXXC结构域, 从酵母到人的同源基因中都有这段保守结构.因此ERV1p代表了酵母细胞中具有FAD结合功能的第一种巯基氧化酶, 并首次证实其与线粒体的形成有关. 酵母细胞scERV1基因的编码产物与其线粒体的发生、细胞生存和细胞周期的调节有密切的联系. 哺乳动物细胞中的同源基因是肝再生增强因子, 与肝再生、精子细胞形成过程有关. 此外, 肝再生增强因子与干扰素g的调节关系密切. 外源性ALR可以通过诱导转录因子和翻译相关因子的表达调节线粒体的功能, 或者通过抑制NK细胞的细胞毒性. Polimeno et al[12]的研究结果表明, ALR对于干扰素g(IFNg)基因表达有影响.IFNg由激活的NK细胞所产生, 对于线粒体转录因子A的表达具有显著的调控作用. 外源性ALR对于NK细胞IFNg的表达具有显著的下调作用, 70%肝切除动物模型如果施以外源性IFNg, 则可以抑制线粒体转录因子A的表达活性, 同时抑制肝再生的过程.这一结果表明ALR是通过生长因子和免疫因子调节的双重调节机制, 影响肝再生的过程.另外, 肝再生增强因子与自然杀伤细胞活性调节有关.Tanigawa et al[13]的研究结果表明, 肝脏中的NK细胞对再生的肝细胞的细胞毒作用可能是爆发性肝炎不能进行有效的肝再生的重要机制. ALR就是通过抑制肝脏内NK细胞的细胞毒活性, 来实现促进肝再生的.对于各种类型的急性肝脏疾病肝脏的ALR mRNA、血清中ALR蛋白以及外周血NK细胞毒活性进行检测, 以阐明肝再生不力与肝内NK细胞活性之间的关系. 结果表明, 肝脏内ALR mRNA的表达水平显著提高, 血清中ALR水平与肝内ALR mRNA表达水平呈正相关. 在急性肝损伤期, 血清中ALR水平与NK细胞活性呈显著负相关的关系. ALR由肝脏产生, 在肝脏损伤时释放.研究结果表明, 肝脏产生的ALR通过抑制肝内NK细胞的细胞毒活性, 促进肝再生. 总之, ALR基因家族从酵母这种低级的真核细胞到哺乳动物都是高度保守的, 甚至在一些双链DNA病毒的基因组中也存在ALR的同源基因序列.不同生物种属的ALR都有重要的生物学功能, 与高等生物的线粒体的生成、细胞分裂周期调节以及肝脏、睾丸等重要脏器的发育有关[14]. 最重要的生物学功能就是促进肝脏的再生.
抑制性消减杂交方法是近年发展起来的一项新的基因克隆技术, 能有效地分离扩增低丰度特异表达的基因, 可以在较短的时间内获得较理想的实验结果.我们将rhALR刺激肝母细胞瘤细胞系HepG2, 并以加入PH7.8磷酸盐缓冲液的相同细胞系作为对照, 以这2种细胞系提取的mRNA为起始材料, 应用SSH方法成功地构建了rhALR激活相关基因差异表达的cDNA消减文库, 随机挑选30个克隆测序分析, 结果主要包括2种类型, 第一种是已知基因的序列, 共15个克隆, 与GenBank中数据高度同源(96-100%). 这些克隆中, 我们筛选到的有细胞周期蛋白依赖激酶1(cyclin-dependent kinase 1, Cdk1), 细胞的复制由细胞周期蛋白依赖激酶(Cdks)调控, Cdks通过调控蛋白底物的磷酸化而实现对细胞周期的控制[15]. 细胞周期蛋白B1-Cdk1复合物是有丝分裂起始的关键, 研究发现, B1-Cdk1复合物首先在细胞质中起作用, 中心体通过对细胞周期蛋白B1的整合, 进而引发有丝分裂[16]. 线粒体、乳酸脱氢酶、蛋白酶体、酪氨酸磷酸酶在体内物质及能量代谢中起重要作用. 载脂蛋白H与脂类代谢、血液凝固密切相关, 载脂蛋白H基因的多态性与高血压以及脑出血的发病机制相关[17].线粒体中含硫氧还蛋白和相关的黄素酶, 硫氧还蛋白是二硫化蛋白, NADP及硫氧还蛋白, 在细胞器中与NADPH相连[18], 在氧化还原反应中起重要作用. 线粒体膜易位酶与蛋白质进入线粒体密切相关, 蛋白质进入线粒体必须经过两个易位接触位点, 包括与线粒体外膜易位酶(translocase of the outer membrane, TOM)形成复合物, 以及与线粒体内膜易位酶(translocase of the inner membrane, TIM)形成复合物[19].肿瘤蛋白翻译调节因子1(TPT1)是一种高度保守, 在大部分真核生物中可以表达的基因, 其功能与细胞的生长发育相关[20]. 近来有研究认为抑制其表达可以使肿瘤的恶性程度逆转, 其表达的增高可能与肿瘤的发生相关. 核糖体蛋白为细胞内结构蛋白. 在已知的15个克隆中, 4个是编码线粒体的基因片段.
通过对上述rhALR激活相关基因差异表达文库部分基因的分析, 我们发现rhALR在细胞内表达后, 与线粒体合成关系密切, 部分涉及细胞生长周期调节、物质和能量代谢、肿瘤发生等方面的基因表达增加, 提示rhALR对体内的多个系统可能有影响. 我们只是对rhALR对人细胞激活相关基因进行了初步的筛选分析, 为研究rhALR在体内的致病机制提供了新的线索. 有关激活基因的具体作用方式, 仍需要进一步的实验来证实.
| 1. | Kogut KA, Nifong LW, Witt MJ, Krusch DA. Hepatic insulin extraction after major hepatectomy. Surgery. 1998;123:415-420. [PubMed] [DOI] |
| 2. | Hagiya M, Francavilla A, Polimeno L, Ihara I, Sakai H, Seki T, Shimonishi M, Porter KA, Starzl TE. Cloning and sequence analysis of the rat augmenter of liver regeneration (ALR) gene: expression of biologically active recombinant ALR and demonstration of tissue distribution. Proc Natl Acad Sci USA. 1995;92:3076. [PubMed] [DOI] |
| 3. | Lisowsky T, Weinstat-Saslow DL, Barton N, Reeders ST, Schneider MC. A new human gene located in the PKD1 region of chromosome 16 is a functional homologue to ERV1 of yeast. Genomics. 1995;29:690-697. [PubMed] [DOI] |
| 6. | Francavilla A, Hagiya M, Porter KA, Polimeno L, Ihara I, Starzl TE. Augmenter of liver regeneration: its place in the universe of hepatic growth factors. Hepatology. 1994;20:747-757. [PubMed] [DOI] |
| 8. | Francavilla A, Vujanovic NL, Polimeno L, Azzarone A, Iacobellis A, Deleo A, Hagiya M, Whiteside TL, Starzl TE. The in vivo effect of hepatotrophic factors augmenter of liver regeneration, hepatocyte growth factor, and insulin-like growth factor-II on liver natural killer cell functions. Hepatology. 1997;25:411-415. [PubMed] [DOI] |
| 9. | Lisowsky T. Dual function of a new nuclear gene for oxidative phosphorylation and vegetative growth in yeast. Mol Gen Genet. 1992;232:58-64. [PubMed] [DOI] |
| 10. | Lisowsky T. ERV1 is involved in the cell-division cycle and the maintenance of mitochondrial genomes in Saccharomyces cerevisiae. Curr Genet. 1994;26:15-20. [PubMed] [DOI] |
| 11. | Lee J, Hofhaus G, Lisowsky T. Erv1p from Saccharomyces cerevisiae is a FAD-linked sulfhydryl oxidase. FEBS Lett. 2000;477:62-66. [PubMed] [DOI] |
| 12. | Polimeno L, Margiotta M, Marangi L, Lisowsky T, Azzarone A, Ierardi E, Frassanito MA, Francavilla R, Francavilla A. Molecular mechanisms of augmenter of liver regeneration as immunoregulator: its effect on interferon-gamma expression in rat liver. Dig Liver Dis. 2000;32:217-225. [PubMed] [DOI] |
| 13. | Tanigawa K, Sakaida I, Masuhara M, Hagiya M, Okita K. Augmenter of liver regeneration (ALR) may promote liver regeneration by reducing natural killer (NK) cell activity in human liver diseases. J Gastroenterol. 2000;35:112-119. [PubMed] [DOI] |
| 14. | Polimeno L, Lisowsky T, Francavilla A. From yeast to man--from mitochondria to liver regeneration: a new essential gene family. Ital J Gastroenterol Hepatol. 1999;31:494-500. [PubMed] |
| 15. | Ubersax JA, Woodbury EL, Quang PN, Paraz M, Blethrow JD, Shah K, Shokat KM, Morgan DO. Targets of the cyclin-dependent kinase Cdk1. Nature. 2003;425:859-864. [PubMed] [DOI] |
| 16. | Jackman M, Lindon C, Nigg EA, Pines J. Active cyclin B1-Cdk1 first appears on centrosomes in prophase. Nat Cell Biol. 2003;5:143-148. [PubMed] [DOI] |
| 17. | Xia J, Yang QD, Yang QM, Xu HW, Liu YH, Zhang L, Zhou YH, Wu ZG, Cao GF. Apolipoprotein H gene polymorphisms and risk of primary cerebral hemorrhage in a Chinese population. Cerebrovasc Dis. 2004;17:197-203. [PubMed] [DOI] |
| 18. | Balmer Y, Vensel WH, Tanaka CK, Hurkman WJ, Gelhaye E, Rouhier N, Jacquot JP, Manieri W, Schürmann P, Droux M. Thioredoxin links redox to the regulation of fundamental processes of plant mitochondria. Proc Natl Acad Sci USA. 2004;101:2642-2647. [PubMed] [DOI] |
| 19. | Chacinska A, Rehling P, Guiard B, Frazier AE, Schulze-Specking A, Pfanner N, Voos W, Meisinger C. Mitochondrial translocation contact sites: separation of dynamic and stabilizing elements in formation of a TOM-TIM-preprotein supercomplex. EMBO J. 2003;22:5370-5381. [PubMed] [DOI] |
| 20. | Thaw P, Baxter NJ, Hounslow AM, Price C, Waltho JP, Craven CJ. Structure of TCTP reveals unexpected relationship with guanine nucleotide-free chaperones. Nat Struct Biol. 2001;8:701-704. [PubMed] [DOI] |