修回日期: 2003-02-20
接受日期: 2003-03-21
在线出版日期: 2003-07-15
探讨HCV核心蛋白结合蛋白6对新生多肽相关复合物α多肽启动子的激活作用.
以基因表达谱芯片技术筛选HCBP6表达质粒转染HepG2细胞后的差异表达基因为基础, 利用生物信息学技术确定NACA的启动子区域(NACA-p), 聚合酶链反应(PCR)扩增NACA-p, 克隆至真核表达载体pCAT3中, 构建pCAT3-NACA-p表达载体; 以该质粒转染COS-7、NIH 3T3细胞系, 用酶联免疫黏附方法(ELISA)检测氯霉素乙酰转移酶(CAT)的表达活性; 与pcDNA3.1(-)-HCBP6共转染NIH 3T3细胞系, 用ELISA法检测CAT的表达活性.
结果: 质粒pCAT3-NACA-p在NIH 3T3、COS-7细胞中能够激活CAT的表达; 共转染实验中pCAT3-NACA-p+pcDNA3.1(-)-HCBP6组CAT的表达活性是pCAT3-NACA-p的4倍.
结论: pCAT3-NACA-p具有启动子活性; HCV核心蛋白结合蛋白6对NACA-p有激活作用.本研究为探讨HCV核心蛋白结合蛋白6的功能提供新的实验及理论基础.
引文著录: 杨倩, 刘妍, 成军, 李克, 王建军, 洪源, 张树林. 丙型肝炎病毒核心蛋白结合蛋白6上调新生多肽相关复合物α多肽基因的表达. 世界华人消化杂志 2003; 11(7): 959-962
Revised: February 20, 2003
Accepted: March 21, 2003
Published online: July 15, 2003
By using yeast-two hybrid technique and bioinformatics analysis, human hepatitis C virus core protein-binding protein 6 (HCBP6) was screened and identified. To investigate activity of HCPB6 on alpha chain of nascent polypeptide-associated complex (NACA) promoter by co-transfection and reporter gene expression methods.
Promoter DNA sequence for a subunit α of nascent polypeptide-associated complex was identified in GenBank by bioinformatics and amplified from HepG2 genome by polymerase chain reaction (PCR). The amplified product was cloned to pCAT3 vector. The NIH 3T3 and COS-7 cell line were tranfected by pCAT3-NACA-p, the NIH 3T3 cell line was co-tranfected by pCAT3-NACA-P and pcDNA3.1(-)-HCBP6. The chloromycin acetyltransferase (CAT) activity was detected by an enzyme-linked immunoassay (ELISA) kit.
In all cell lines, we found pCAT3-NACA-p had higher activity of CAT than pCAT3-basic by ELISA kit. The expression of CAT tested in co-transfection was 4 times as higher as pCAT3-NACA-p plasmid.
HCBP6 has trans-activity on NACA promoter, and this result implicates HCBP6 can affect metabolism.
- Citation: Yang Q, Liu Y, Cheng J, Li K, Wang JJ, Hong Y, Zhang SL. Up-regulating effect of human hepatitis C virus core protein-binding protein 6 on NACA gene promoter. Shijie Huaren Xiaohua Zazhi 2003; 11(7): 959-962
- URL: https://www.wjgnet.com/1009-3079/full/v11/i7/959.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v11.i7.959
丙型肝炎病毒(HCV)核心蛋白是一种多功能蛋白质, 除作为核壳蛋白具有病毒颗粒组装功能外, 还具有多种调控细胞及病毒基因表达的功能, 在HCV引起的急慢性肝炎、肝硬化、肝癌的发病机制中起到重要的作用[1,2].其中核心蛋白与宿主蛋白之间的相互作用可能是病毒持续感染的重要原因[3-5]. 为了进一步探索核心蛋白与肝细胞之间的相互作用, 我室采用酵母双杂交系统3, 以HCV核心蛋白作为"诱饵", 对肝细胞cDNA文库进行筛选, 获得了一些与核心蛋白结合的肝细胞蛋白的编码基因, 其中包括未知功能基因6号, 命名为HCV核心蛋白结合蛋白6(HCBP6)[6,7]. 为进一步研究该基因的功能, 利用基因表达芯片技术筛选HCBP6表达质粒转染HepG2细胞后的差异表达基因, 发现HCBP6可上调一些基因的表达, 其中包括新生多肽相关复合物α多肽(α subunit of nascent polypeptide-associated complex, NACA), 本研究利用生物信息学手段对NACA 5'-上游调控序列进行分析、克隆, 并与报告基因氯霉素乙酰转移酶(CAT)构建表达质粒, 与HCBP6表达载体共转染, 证明HCBP6对NACA启动子(promoter)具有激活作用, 使下游CAT基因的表达增强.
大肠杆菌JM109, 细胞株COS-7、NIH 3T3、pcDNA3.1(-)-HCBP6. 为本室保存. pEGM-T、pCAT3载体购自Promega公司, 脂质体FuGENE6购自Roche公司, Taq DNA聚合酶购自鼎国生物公司, 限制性内切酶Kpn I、Xho I购自Takara公司. 质粒DNA提取试剂盒(Promega公司), 玻璃奶DNA回收试剂盒(博大公司), CAT ELISA试剂盒(Roche公司). 引物合成、核苷酸序列测序由上海博亚公司完成.
1.2.1 NACA启动子结构分析 根据GenBank中NACA基因组序列, 确定NACA的转录起始点, 截取其上游963 bp, 设计并合成引物, 在上下游引物的5'-端分别加上Kpn I和Xho I.上游引物: 5'-GGT ACC CTG GGA AGG AGA ATC TGC AC-3'; 下游引物: 5'-CTC GAG GTG CAG GGA ACG CGG AAC CAA G-3'.
1.2.2 质粒构建 PCR扩增NACA启动子序列, 玻璃奶纯化回收DNA片段, 在T4 DNA连接酶的作用下, 与pGEM-T载体连接, 转化大肠杆菌JM109, 挑取在选择平皿(Amp, X-gal/IPTG)生长的白色菌落提取质粒, 经酶切(Kpn I/Xho I)及测序鉴定. Kpn I/Xho I双酶切重组质粒pEGM-T-NACA-promoter, 玻璃奶纯化回收酶切产物, 定向克隆至pCAT3载体, 构建成重组质粒pCAT3-NACA-promoter. 经双酶切及菌落PCR鉴定连接产物. 试剂盒方法提取质粒以备转染.
1.2.3 细胞培养及转染 COS-7、NIH 3T3细胞系在含10%小牛血清的DMEM培养液中生长. 细胞生长至50-80%时采用脂质体转染法, 具体转染方法参照FuGENE6说明书进行. 质粒pCAT3-NACA-promoter转染30 h后收获细胞.
1.2.4 共转染实验 NIH 3T3细胞生长至50-80%时, 质粒pCAT3-NACA-promoter与pcDNA3.1(-)-HCBP6、质粒pCAT3-NACA-promoter与pcDNA3.1(-)空载体共转染. 因pCAT3-NACA-promoter活性较强, 将共转染剂量减至0.2 μg. 具体转染方法参照FuGENE6说明书进行. 转染30 h后收获细胞.
1.2.5 CAT表达量的检测 将收获的细胞进行裂解, 提取上清液, 取200 μl用于检测CAT的表达量. 具体方法严格按照CAT ELISA试剂盒说明书进行. 在415 nm光波下测吸光度A值
根据GenBank中NACA基因组序列, 确定NACA的转录起始点, 截取其上游963 bp, 其核苷酸序列见图1. 对此序列进行分析发现有多个TATA盒存在, 同时含有多个CCGCCC序列.
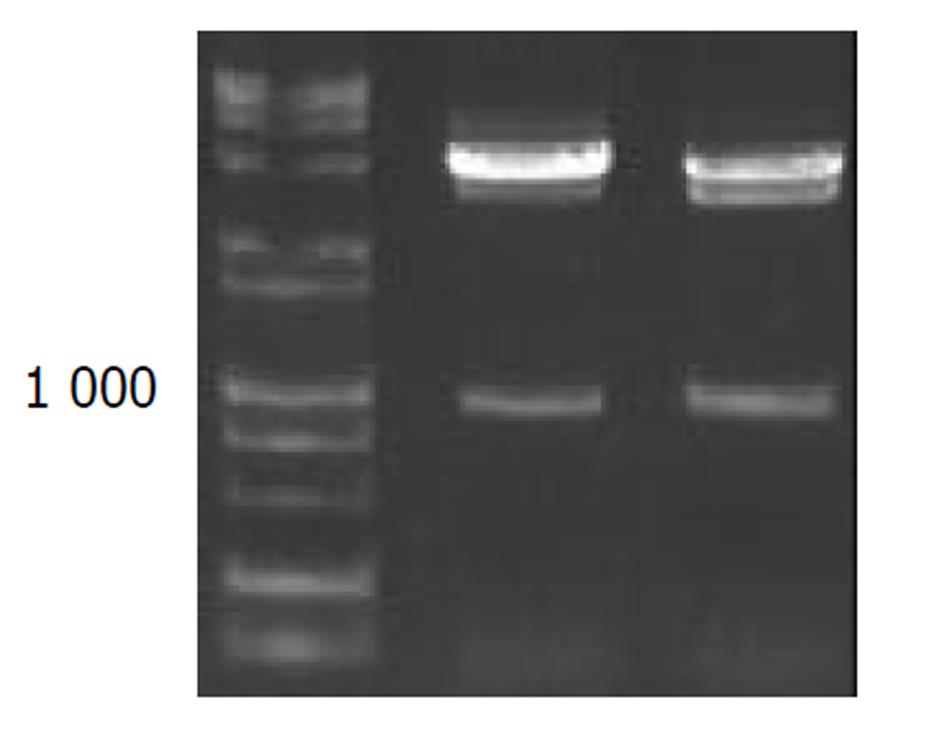
以HepG2基因组为模板, PCR扩增NACA启动子DNA序列, 于963 bp处出现理想条带.将扩增产物与pGEM-T载体连接, 双酶切鉴定, 显示两条带. 并送测序鉴定正确. 双酶切产物与pCAT3载体连接, 连接产物双酶切鉴定如图1所示(4 000 bp的pCAT3-basic空载体和963 bp的NACA-promoter DNA片段)连接转化生长的菌落进行PCR鉴定, 如图2所示, 于963 bp处可见理想条带.
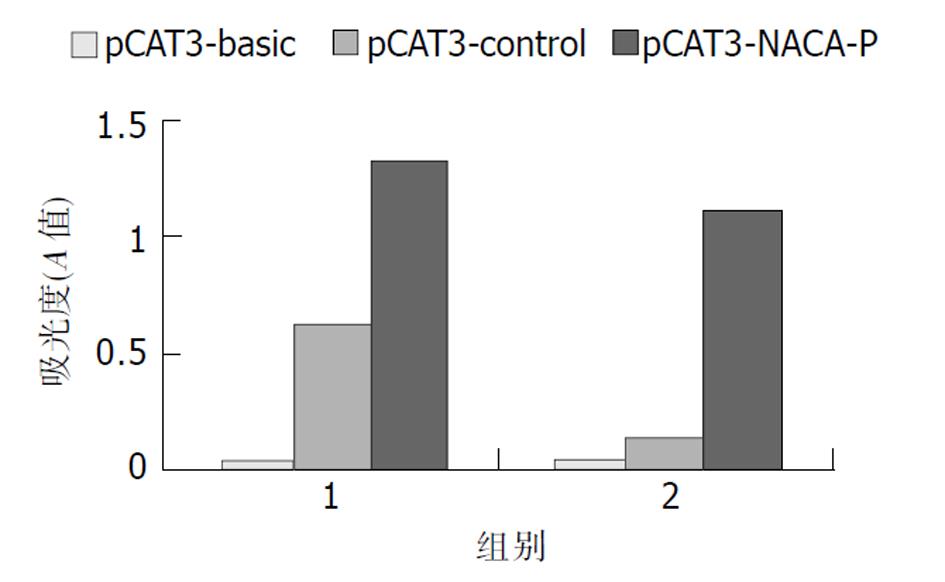
将重组质粒pCAT3-NACA-p转染COS-7、NIH 3T3细胞, 如图3所示pCAT3-NACA-p具有启动子活性. 其中组1为COS-7细胞系: 组2为NIH 3T3细胞系. pCAT3-basic(阴性对照), pCAT3-control(阳性对照).
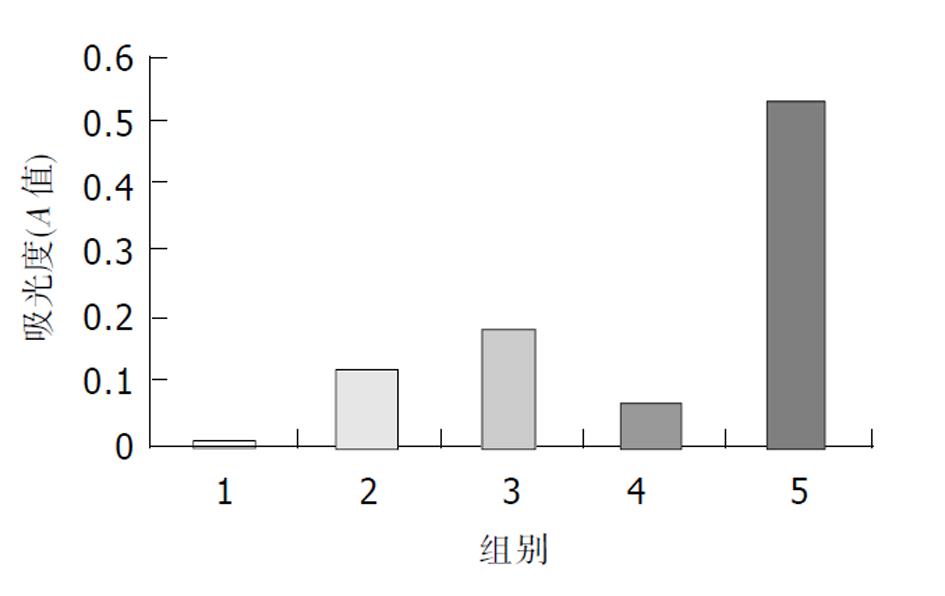
3T3并检测报告基因CAT的表达.共转染pCAT3-NACA-promoter+pcDNA3.1(-)-HCBP6时CAT的表达较对照质粒明显升高, 如图4所示, 说明HCBP6对HCBP6启动子具有激活作用. 其中组1为pCAT3-basic, 组2为pCAT3-control, 组3为pCAT3-NACA, 组4为pcDNA3.1(-)-vector(空载体), 组5为pCAT3-NACA-p.
HCV核心蛋白是一种多功能蛋白, 除了作为核壳蛋白具有病毒颗粒组装功能外, 还结合细胞内的蛋白质, 如载脂蛋白AII、肿瘤坏死因子受体-1(TNFR-1), 淋巴毒素-β受体(LT-βR), 对细胞信号转导、宿主的免疫调节起作用[8,9] . 为研究HCV核心蛋白与宿主肝细胞蛋白的相互作用, 我们将肝细胞cDNA文库与HCV核心蛋白"诱饵"进行酵母双杂交, 筛选并克隆了与HCV核心蛋白相互作用的新的蛋白质基因HCBP6[10,11] . 利用生物信息学技术, 我们确定了HCBP6的染色体定位, 运用在线软件对其蛋白质一级结构序列进行了分析, 为探讨HCBP6蛋白表达的特点及功能奠定了基础[12,13]. 同时在四唑氮蓝(MTT)实验中发现HCBP6可以促进细胞生长, 与HCV核心质粒共转染研究中发现其在一定程度上可抑制HCV核心蛋白对SV40早期启动子的反式激活作用[14].为进一步研究HCBP6的功能, 我们采用基因芯片方法筛选HCBP6基因转染细胞差异表达的基因, 结果表明HCBP6可上一些基因的表达, 其中包括NACA基因[15]. 根据GenBank中NACA基因组序列, 确定NACA的转录起始点[16,17], 截取其上游963 bp核苷酸序列进行克隆, 并构建含报告基因CAT的重组质粒, 结果发现HCBP6对NACA的表达确有激活作用.对HCBP6的功能研究提供了新的依据.
新生多肽相关复合物由α、β两条肽链组成, 主要在胞质中分布[18,19]. 研究发现去除NAC后, 细胞将产生两种缺陷: (1)与新生肽链信号肽链接并将其靶向与内质网连接的信号识别颗粒, 失去信号识别功能, 而与无信号肽的新生肽链交叉连接; (2)核糖体翻译与内质网膜错误连接的非分泌性的蛋白质[20-22]. NACA单独不能与核糖体连接, 但对核酸有较高的亲和力[23,24]. Beatrix et al[25]发现NACA除了能和DNA结合外, 还能与RNA、mRNA、tRNA、7S LRNA相结合, 并且可以进入核内[26,27]. Mossabeb et al[28]发现NACA可以诱导特应性皮炎患者特异性淋巴细胞增生反应, 并且与自发性IgE抗体特异性结合. Yotov et al[29]研究表明富含脯氨酸的不同NACA拼接体可以激活肌球蛋白启动子的转录活性, 在调控肌源性细胞分化时的基因表达发挥重要作用. Moreau et al[30]也证明NACA能够与c-Jun蛋白的1-89位氨基酸结合, 并且作为辅激活蛋白NACA可以与非磷酸化和73位丝氨酸磷酸化的c-Jun蛋白结合, 从而在成骨细胞分化过程中起到转录调节的作用.
HCBP6的生物学功能目前仍不是很明确, 对其反式激活作用的研究有助于探索其在细胞分化、信号传导中的作用. 并对HCV感染慢性化、HCV引起原发性肝癌的致病机制提供新的研究方向.
| 3. | Liu Q, Tackney C, Bhat RA, Prince AM, Zhang P. Regulated processing of hepatitis C virus core protein is linked to subcelluar localization. J Virol. 1997;71:657-662. [PubMed] |
| 4. | Yasui K, Wakita T, Tsukiyama-Kohara K, Funahashi SI, Ichikawa M, Kajita T, Moradpour D, Wands JR, Kohara M. The native form and maturation process of hepatitis C virus core protein. J Virol. 1998;72:6048-6055. [PubMed] |
| 7. | 李 克, 王 琳, 成 军, 张 玲霞, 段 惠娟, 陆 荫英, 杨 继珍, 刘 妍, 洪 源, 夏 小兵. 酵母双杂交技术筛选克隆HCV核心蛋白结合蛋白基因1. 世界华人消化杂志. 2001;9:1379-1383. |
| 9. | 刘 妍, 成 军, 王 刚, 李 克, 段 惠娟, 王 琳, 董 菁, 洪 源, 张 跃新, 李 莉. 应用抑制性消减杂交技术克隆丙型肝炎病毒核心蛋白反式激活基因. 解放军医学杂志. 2001;26:880-883. |
| 10. | 李 克, 王 琳, 成 军, 张 玲霞, 段 惠娟, 陆 荫英, 杨 继珍, 刘 妍, 邵 得志, 夏 小兵. 筛选与克隆丙型肝炎病毒核心蛋白结合蛋白6基因. 中华实验和临床病毒学杂志. 2002;16:51-54. |
| 13. | Husmeier D, Wright F. Probabilistic divergence measures for detecting interspecies recombination. Bioinformatics. 2001;17:S123-131. [PubMed] [DOI] |
| 16. | Yotov WV, St-Arnaud R. Mapping of the human gene for the alpha-NAC/1.9.2 (NACA/1.9.2) transcriptional coactivator to Chromosome 12q23-24.1. Mamm Genome. 1996;7:163-164. [PubMed] [DOI] |
| 17. | Wiedmann B, Sakai H, Davis TA, Wiedmann M. A protein complex required for signal-sequence-specific sorting and translocation. Nature. 1994;370:434-440. [PubMed] [DOI] |
| 18. | Shi X, Parthun MR, Jaehning JA. The yeast EGD2 gene encodes a homologue of the alpha NAC subunit of the human nascent-polypeptide-associated complex. Gene. 1995;165:199-202. [DOI] |
| 19. | Reimann B, Bradsher J, Franke J, Hartmann E, Wiedmann M, Prehn S, Wiedmann B. Initial characterization of the nascent polypeptide-associated complex in yeast. Yeast. 1999;15:397-407. [DOI] |
| 20. | Lauring B, Sakai H, Kreibich G, Wiedmann M. Nascent polypeptide-associated complex protein prevents mistargeting of nascent chains to the endoplasmic reticulum. Proc Natl Acad Sci USA. 1995;92:5411-5415. [DOI] |
| 21. | Wiedmann B, Prehn S. The nascent polypeptide-associated complex (NAC) of yeast functions in the targeting process of ribosomes to the ER membrane. FEBS Lett. 1999;458:51-54. [DOI] |
| 22. | Bacher G, Pool M, Dobberstein B. The ribosome regulates the GTPase of the beta-subunit of the signal recognitionparticle receptor. J Cell Biol. 1999;146:723-730. [DOI] |
| 23. | Whitby MC, Dixon J. Fission yeast nascent polypeptide-associated complex binds to four-way DNA junctions. J Mol Biol. 2001;306:703-716. [PubMed] [DOI] |
| 24. | Franke J, Reimann B, Hartmann E, Kohlerl M, Wiedmann B. Evidence for a nuclear passage of nascent polypeptide-associated complex subunits in yeast. J Cell Sci. 2001;114:(Pt 14)2641-2648. [PubMed] |
| 25. | Beatrix B, Sakai H, Wiedmann M. The alpha and beta subunit of the nascent polypeptide-associated complex have distinct functions. J Biol Chem. 2000;275:37838-37845. [PubMed] [DOI] |
| 26. | St-Arnaud R, Quelo II. Transcriptional coactivators potentiating AP-1 function in bone. Front Biosci. 1998;3:D838-848. [PubMed] [DOI] |
| 27. | Goatley LC, Twigg SR, Miskin JE, Monaghan P, St-Arnaud R, Smith GL, Dixon LK. The African swine fever virus protein j4R binds to the alpha chain of nascent polypeptide -associated complex. J Virol. 2002;76:9991-9999. [DOI] |
| 28. | Mossabeb R, Seiberler S, Mittermann I, Reininger R, Spitzauer S, Natter S, Verdino P, Keller W, Kraft D, Valenta R. Characterization of a novel isoform of alpha-nascent polypeptide-associatedcomplex as IgE-defined autoantigen. J Invest Dermatol. 2002;119:820-829. [PubMed] [DOI] |
| 29. | Yotov WV, St-Arnaud R. Differential splicing-in of a proline-rich exon converts alpha NAC into amuscle-specific transcription factor. Genes Dev. 1996;10:1763-1772. [DOI] |
| 30. | Moreau A, Yotov WV, Glorieux FH, St-Arnaud R. Bone-specific expression of the alpha chain of the nascent polypeptide-associated complex, a coactivator potentiating c-Jun-mediated transcription. Mol Cell Biol. 1998;18:1312-1321. [DOI] |