修回日期: 2025-04-08
接受日期: 2025-05-16
在线出版日期: 2025-05-28
近年来研究发现, 肠道微生物群与人类大脑活动关系密切. 谵妄与肠道菌群及其分泌的多种炎症因子相关, 本研究利用MR方法阐明两者之间的因果关系.
通过孟德尔随机化分析研究肠道菌群与谵妄之间的因果关系, 并分析炎症因子在其中所起的中介作用.
主要采用两样本孟德尔随机化的方法, 利用全基因组关联研究数据, 探究肠道菌群与谵妄发生风险的因果关系. 随后采用乘积法中介分析, 探究炎症因子可能起到的中介作用.
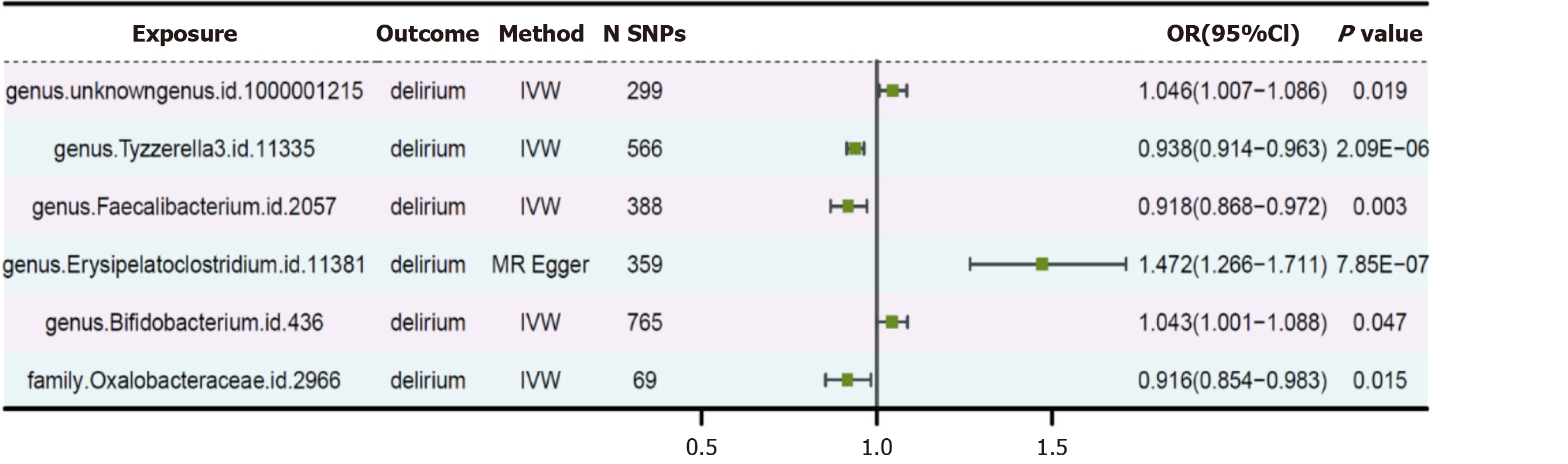
孟德尔随机化分析结果显示, 6种肠道菌群与谵妄发生风险之间存在显著关联. 其中unknown genus属(ID 1000001215)(OR = 1.046, 95%CI: 1.007-1.086, P = 0.019)、Erysipelatoclostridium属(ID 11381)(OR = 1.472, 95%CI: 1.266-1.711, P = 7.85E-07)和Bifidobacterium属(ID 436)(OR = 1.043, 95%CI: 1.001-1.088, P = 0.047)与谵妄风险呈正相关. Tyzzerella3属(ID 11335)(OR = 0.938, 95%CI: 0.914-0.963, P = 2.09E-06)、Faecalibacterium属(ID 2057)(OR = 0.918, 95%CI: 0.868-0.972, P = 0.003)和Oxalobacteraceae科(ID 2966)(OR = 0.916, 95%CI: 0.854-0.983, P = 0.015)与谵妄风险呈负相关. 中介分析结果显示白介素-6(interleukin-6, IL-6)在其中起到部分中介作用.
部分肠道菌群与谵妄存在因果关系, 该关系可能通过IL-6介导.
核心提要: 通过两样本孟德尔分析及中介效应分析全面评估了肠道菌群与谵妄风险之间的因果关系, 并揭示了白介素-6在其中发挥潜在的中介作用, 为肠道菌群与谵妄风险的因果关系提供了新的见解, 也为谵妄的预防和治疗提供新的思路.
引文著录: 周锦航, 徐微懿, 许丹. 肠道菌群、炎症因子与谵妄的因果关系: 孟德尔随机化与中介分析. 世界华人消化杂志 2025; 33(5): 397-403
Revised: April 8, 2025
Accepted: May 16, 2025
Published online: May 28, 2025
Recent studies have found that the gut microbiota is closely related to human brain activity. Delirium is associated with the intestinal microbiota and various inflammatory factors that it secretes. In this study, the Mendelian randomization (MR) method was used to clarify the causal relationship between the two.
To investigate the causal relationship between the gut microbiota and delirium through MR analysis, and to analyze the mediating role of inflammatory factors in this relationship.
The study primarily used a two-sample MR approach, utilizing genome-wide association study data to explore the causal relationship between the gut microbiota and the risk of delirium. Subsequently, mediation analysis using the product method was conducted to examine the potential mediating role of inflammatory factors.
The results of MR analysis showed significant associations between six types of gut microbiota and the risk of delirium. The unknown genus (ID 1000001215) [odds ratio (OR) = 1.046, 95% confidence interval (CI): 1.007-1.086, P = 0.019], genus Erysipelatoclostridium (ID 11381) (OR = 1.472, 95%CI: 1.266-1.711, P = 7.85E-07), and genus Bifidobacterium (ID 436) (OR = 1.043, 95%CI: 1.001-1.088, P = 0.047) were positively associated with delirium risk. Conversely, the genus Tyzzerella3 (ID 11335) (OR = 0.938, 95%CI: 0.914-0.963, P = 2.09E-06), genus Faecalibacterium (ID 2057) (OR = 0.918, 95%CI: 0.868-0.972, P = 0.003), and family Oxalobacteraceae (ID 2966) (OR = 0.916, 95%CI: 0.854-0.983, P = 0.015) were negatively associated with delirium risk. Mediation analysis indicated that interleukin-6 (IL-6) partially mediated this relationship.
Certain gut microbiota species are causally related to delirium, and this relationship may be mediated through IL-6.
- Citation: Zhou JH, Xu WY, Xu D. Causal relationship between gut microbiota, inflammatory factors, and delirium: Mendelian randomization and mediation analysis. Shijie Huaren Xiaohua Zazhi 2025; 33(5): 397-403
- URL: https://www.wjgnet.com/1009-3079/full/v33/i5/397.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v33.i5.397
谵妄是一种急性脑功能障碍, 在危重症患者中发病率高达70%-80%[1]. 谵妄的发生延长患者住院时间, 增加住院费用, 影响患者预后, 降低患者出院后的日常生活能力[2]. 谵妄的发病机制尚不明确, 有研究发现炎症因子会破坏血脑屏障, 导致神经元损伤和神经退行性病变, 进而发生谵妄[3]. 肠道菌群是寄生在肠道内与寄主共生的特定微生物群落的总称, 包括细菌、病毒和真菌, 其中98%是细菌[4]. 肠道菌群与多种神经及精神病学疾病的发生发展密切相关[5].
孟德尔随机化(mendelian randomization, MR)是一种利用遗传变异作为工具变量来评估危险因素和疾病之间因果关系的方法, 该方法的优点是减少潜在混杂因素和逆向因果推断的影响, 提高研究结果的可靠性. MR研究发现肠道微生物群与阿尔茨海默病、重度抑郁症和精神分裂症等疾病有因果关系[6]. 因此, 本研究将肠道菌群作为暴露因素, 谵妄和炎症因子作为结局因素, 采用MR以及中介分析探讨肠道菌群与谵妄和炎症因子的关系, 为该领域的进一步研究提供了新的依据.
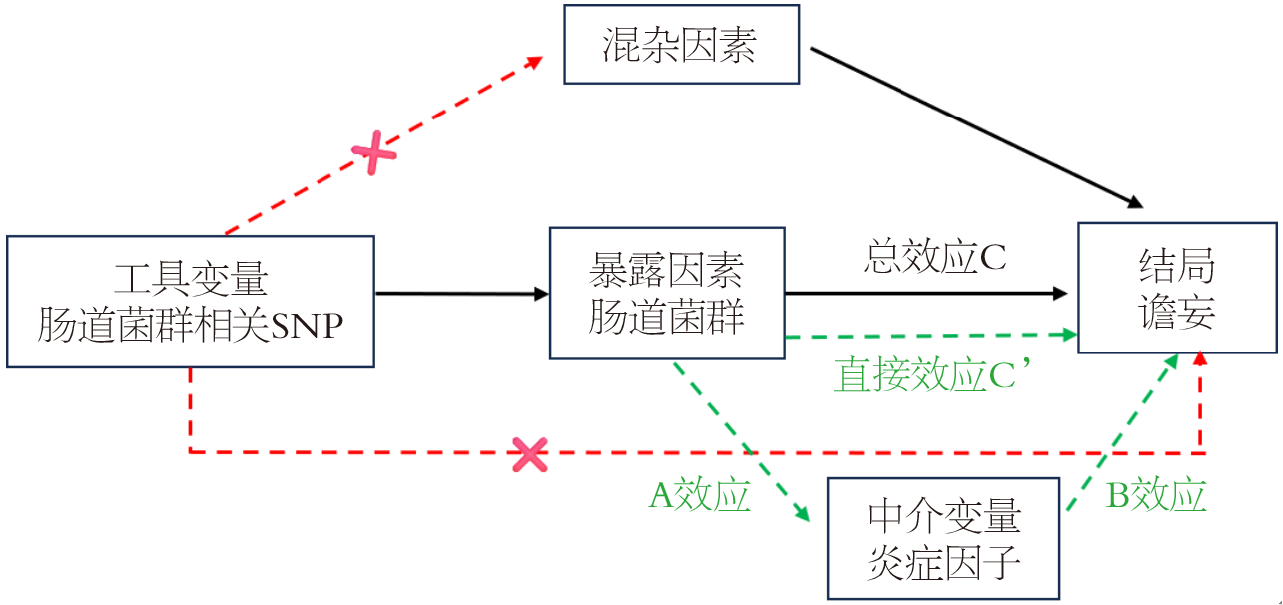
本研究以肠道菌群作为暴露因素, 谵妄作为结局变量, 炎症因子作为潜在中介因素进行MR分析(图1). 进行MR分析的工具变量必须满足以下3个基本条件: (1)工具变量必须与暴露相关; (2)工具变量不能与混杂因素存在关联; (3)工具变量只能通过暴露因素影响结局.
数据来源: 肠道菌群的数据来自公共数据库MiBioGen研究的肠道菌群全基因组关联分析研究(genome-wide association study, GWAS)[7]. 该研究涉及来自11个国家的18340名参与者(24个队列), 包括了211个细菌分群, 含有131种属水平, 35种科水平, 20个目水平, 16个纲水平和9个门水平. 谵妄患者的遗传数据来源于FinnGen R10[8], 其中包含了3371例谵妄患者和388560例对照, 研究对象均为欧洲人群. 炎症因子的GWAS数据分为白介素-1(interleukin-1, IL-1)(研究编号: ebi-a-GCST90012004)、IL-2(研究编号: ebi-a-GCST90019480)、IL-4(研究编号: prot-a-1532)、IL-6(研究编号: ebi-a-GCST90012005)、IL-8(研究编号: ebi-a-GCST90011994)、IL-10(研究编号: prot-a-1464)、IL-12(研究编号: prot-a-1468)、IL-16(研究编号: prot-a-1479)、IL-18(研究编号: ebi-a-GCST90012024)和肿瘤坏死因子α(tumor pnecrosis factor α, TNF-α)(研究编号: ebi-a-GCST004426). 本研究的所有数据都是从公开的数据库中获取, 因此无需经过伦理审批.
工具变量筛选: 为了确保肠道菌群与谵妄间因果关系结论的真实性和准确性, 研究采用如下的筛选条件: (1)从肠道菌群的GWAS数据中选择与肠道菌群最具相关性的单核苷酸多态性(single nucleotide polymorphisms, SNP), 选取P<5×10-8的SNP; (2)基于MR的独立性假设, 各个SNP需相互独立, 为确保各工具变量(instrumental variables, IV)间不存在连锁不平衡(linkage disequilibrium, LD), 设定LD系数r2为0.001, 域宽度为10000 kb; (3)为确保同一SNP在肠道菌群组和谵妄组具有相同的等位基因, 去除了回文SNP; (4)通过计算F值排除弱工具变量. F值的简化计算公式: F = beta2/se2, 其中beta和se分别为该SNP与菌群关联的回归系数和标准误. F值<10的SNP被认为是弱工具变量而被排除, 同时也去除包含混杂因素的SNP.
MR分析: 本研究用R 4.3.3进行统计分析, 其中"TwosampleMR"包用于MR分析, 使用随机效应逆方差加权法(inverse variance weighted, IVW)作为主要方法[9], 采用加权模式、MR-Egger回归、加权中位数、简单模式4种方法来支持和验证主要分析结果, 以P<0.05表示存在相关性. 采用两样本孟德尔随机化方法, 分别计算以肠道菌群为暴露因素, 中介变量为结局变量的效应值和以中介变量为暴露因素, 谵妄为结局变量的效应值, 随后用乘积法来估计中介效应的OR值, 用乘积分布法检验计算中介效应的P值的95%置信区间, 如果置信区间不包括0, 则表明中介效应显著.
统计学处理 为了评价因果效应评估结果的可靠性, 本研究进行了敏感性分析. 本研究采用留一法(1eave-one-out)检验单个IV对整体结果的影响, 从而评估因果关联结果的稳定性, 若未观察到显著的统计学差异, 则表明总效应并不依赖于单一的IV[10]. 异质性检验在两样本MR分析中用于评估IVs之间是否存在差异, 本研究采用Cochran's Q检验异性, 若P>0.05, 则表明不存在异质性; 反之则存在[11]. 本研究采用MR-Egger法来检测水平多效性, 即当截距项与0的差异显著(P<0.05)时, 表明存在水平多效性[9]. 当检测到水平多效性时, 应用MR多效性残差和离群值(MR pleiotropy residual sum and outlier, MR-PRESSO)离群值检验来校正多效性, 提高因果估计的准确性[12].
通过两样本MR分析发现6种菌群与谵妄风险有因果关系(P<0.05), unknowngenus属(ID 1000001215)(OR = 1.046, 95%CI: 1.007-1.086, P = 0.019)、Erysipelatoclostridium属(ID 11381)(OR = 1.472, 95%CI: 1.266-1.711, P = 7.85E-07)和Bifidobacterium属(ID 436)(OR = 1.043, 95%CI: 1.001-1.088, P = 0.047)与谵妄风险增加有关. Tyzzerella3属(ID 11335)(OR = 0.938, 95%CI: 0.914-0.963, P = 2.09E-06)、Faecalibacterium属(ID 2057)(OR = 0.918, 95%CI: 0.868-0.972, P = 0.003)和Oxalobacteraceae科(ID 2966)(OR = 0.916, 95%CI: 0.854-0.983, P = 0.015)与谵妄风险降低有关(图2). 以上P值均用FDR控制进行多重检验校正, 结果表明均有显著性水平.
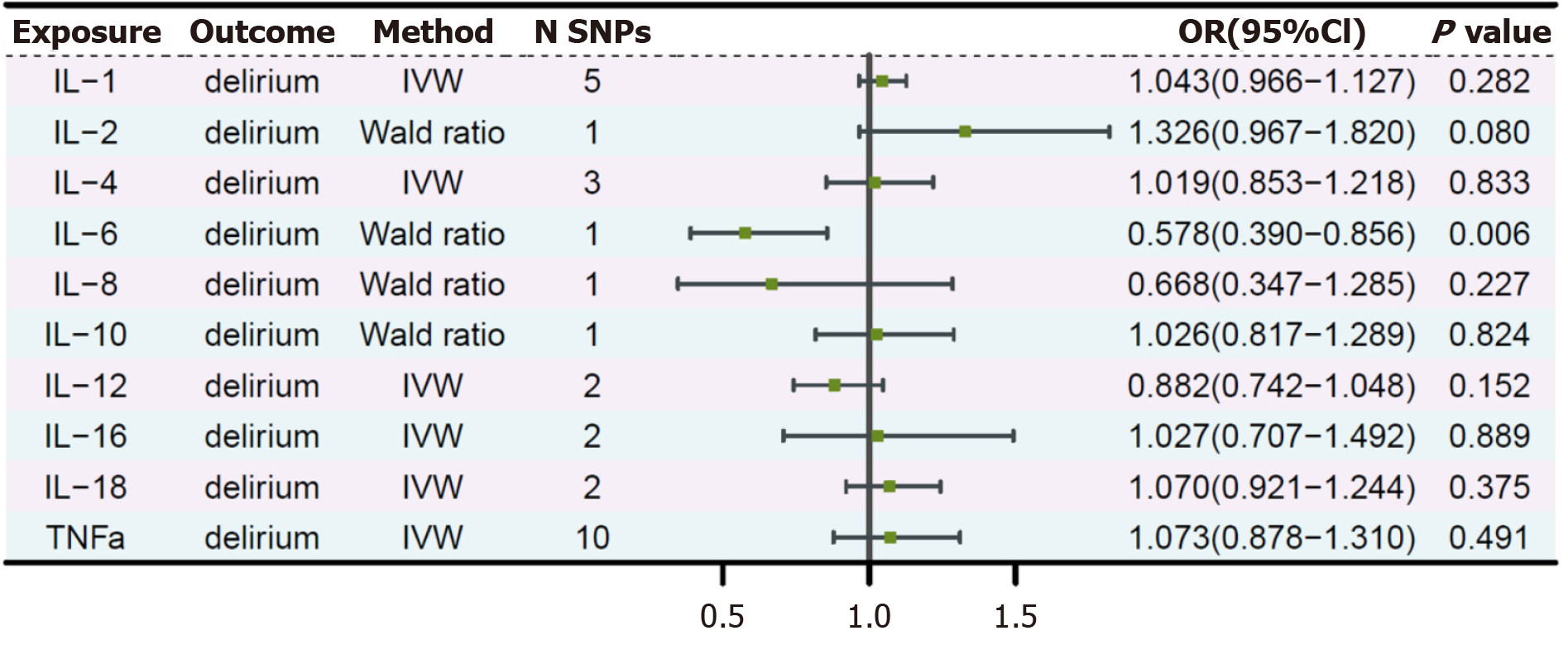
分别以炎症因子IL-1、IL-2、IL-4、IL-6、IL-8、IL-10、IL-12、IL-16、IL-18和TNF-α作为暴露变量, 以谵妄作为结局变量, 进行两样本MR分析. 研究结果显示仅IL-6与谵妄发生风险降低有关(OR = 0.578, 95%CI: 0.390-0.856, P = 0.006)(图3).
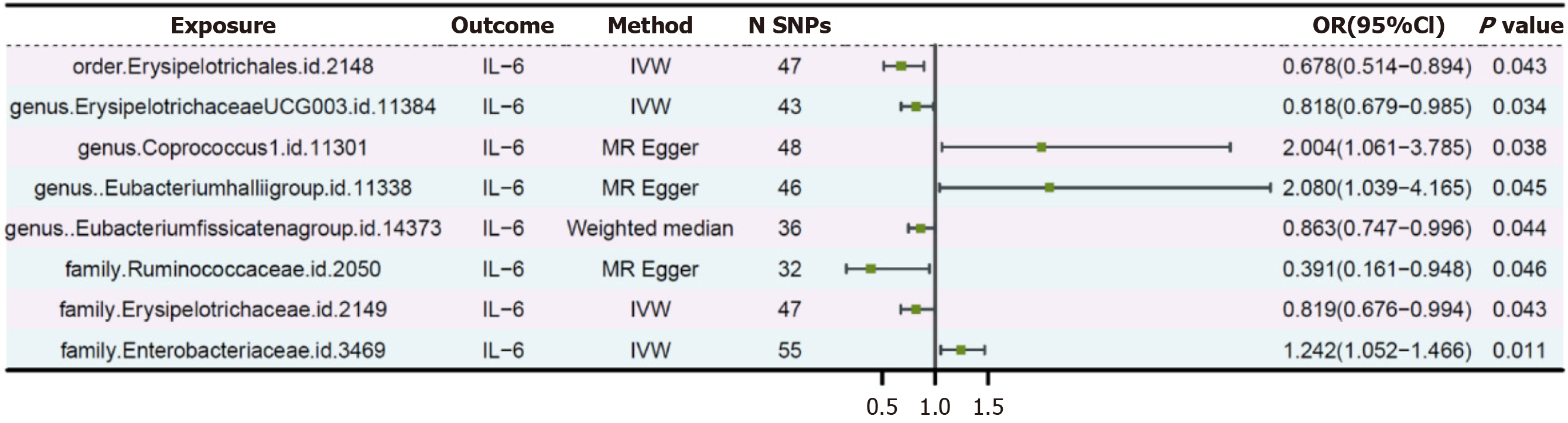
将6种谵妄相关的菌群作为暴露变量, 分别以炎症因子IL-1、IL-2、IL-4、IL-6、IL-8、IL-10、IL-12、IL-16、IL-18和TNF-α作为结局变量, 进行两样本MR分析, 研究结果显示该6种菌群与炎症因子均无因果关系. 故我们调整肠道菌群SNP的P值, 以P<1×10-5为标准纳入更多的SNP, 结果显示Coprococcus1属(ID 11301)(OR = 2.004, 95%CI: 1.061-3.785, P = 0.038)、Eubacteriumhalliigroup属(ID 11338)(OR = 2.080, 95%CI: 1.039-4.165, P = 0.045)和Enterobacteriaceae科(ID 3469)(OR = 1.242, 95%CI: 1.052-1.466, P = 0.011)与IL-6呈正相关. Erysipelotrichales目(ID 2148)(OR = 0.819, 95%CI: 0.676-0.994, P = 0.043)、ErysipelotrichaceaeUCG003属(ID 11384)(OR = 0.818, 95%CI: 0.679-0.985, P = 0.034)、Eubacteriumfissicatenagroup属(ID 14373)(OR = 0.863, 95%CI: 0.747-0.996, P = 0.044)、 Ruminococcaceae科(ID 2050)(OR = 0.391, 95%CI: 0.161-0.948, P = 0.046)和Erysipelotrichaceae科(ID 2149)(OR = 0.819, 95%CI: 0.676-0.994, P = 0.043)与IL-6呈负相关(图4).
对所有的暴露-结局关联进行敏感性分析, 利用留一法检验单个SNP对整体因果关系的影响. 当去除任意一个SNP并重复MR分析时, 整体因果关系差异无统计学意义, 表明所得结果并非由单个SNP所导致, 因果效应稳定. 研究结果显示, Cochran's Q检验的MR-Egger和IVW 2种方法所得均P>0.05, 不存在显著的异质性. MR-Egger截距测试所得截距项均趋近于0且P>0.05, 同时MR-PRESSO全局检验得到的均P>0.05, 提示不存在显著的水平多效性(表1).
| MR Egger (异质性) | IVW (异质性) | MR Egger截距测试 (水平多效性) | MR-PRESSO全局检验 (水平多效性) | ||||||
| Cochran's Q | P值 | Cochran's Q | P值 | 截距项 | P值 | 残差平方和 | P值 | ||
| Unknowngenus属(ID 1000001215) | 谵妄 | 213.370 | 1.000 | 227.713 | 0.999 | 0.027 | 0.184 | 3.577 | 0.872 |
| Tyzzerella3属(ID 11335) | 谵妄 | 474.318 | 0.997 | 538.700 | 0.781 | -0.048 | 0.328 | 9.257 | 0.641 |
| Faecalibacterium属(ID 2057) | 谵妄 | 433.462 | 0.478 | 457.343 | 0.079 | 0.020 | 0.114 | 5.732 | 0.664 |
| Erysipelatoclostridium属(ID 11381) | 谵妄 | 319.764 | 0.922 | 341.567 | 0.725 | -0.027 | 0.529 | 9.040 | 0.592 |
| Bifidobacterium属(ID 436) | 谵妄 | 929.419 | 0.315 | 1001.651 | 0.000 | 0.034 | 0.075 | 8.327 | 0.711 |
| Oxalobacteraceae科(ID 2966) | 谵妄 | 62.367 | 0.638 | 62.613 | 0.662 | 0.007 | 0.621 | 10.438 | 0.320 |
| Erysipelotrichales目(ID 2148) | IL-6 | 35.886 | 0.832 | 36.969 | 0.827 | 0.019 | 0.304 | 18.330 | 0.341 |
| ErysipelotrichaceaeUCG003属(ID 11384) | IL-6 | 42.000 | 0.427 | 42.681 | 0.442 | -0.016 | 0.420 | 3.792 | 0.942 |
| Coprococcus1属(ID 11301) | IL-6 | 46.442 | 0.454 | 51.470 | 0.303 | -0.041 | 0.031 | 12.674 | 0.674 |
| Eubacteriumhalliigroup属(ID 11338) | IL-6 | 54.538 | 0.133 | 60.360 | 0.063 | -0.045 | 0.036 | 3.788 | 0.901 |
| Eubacteriumfissicatenagroup属(ID 14373) | IL-6 | 33.726 | 0.481 | 35.932 | 0.425 | 0.049 | 0.147 | 5.035 | 0.496 |
| Ruminococcaceae科(ID 2050) | IL-6 | 21.774 | 0.862 | 24.592 | 0.786 | 0.041 | 0.104 | 4.882 | 0.499 |
| Erysipelotrichaceae科(ID 2149) | IL-6 | 35.886 | 0.832 | 36.969 | 0.827 | 0.019 | 0.304 | 14.538 | 0.335 |
| Enterobacteriaceae科(ID 3469) | IL-6 | 55.751 | 0.372 | 56.028 | 0.399 | -0.010 | 0.610 | 6.636 | 0.143 |
为了进一步探究炎症因子影响谵妄的具体机制, 根据上述研究结果我们选取了IL-6作为中介变量进行中介分析. 结果显示在Erysipelotrichales目(ID 2148)、ErysipelotrichaceaeUCG003属(ID 11384)、Erysipelotrichaceae科(ID 2149)和Enterobacteriaceae科(ID 3469)与谵妄的因果关系中, IL-6的中介效应分别为56.51%(OR = 0.473, 95%CI: 0.0004-0.2698)、51.2%(OR = 0.473, 95%CI: 0.0033-0.2671)、56.51%(OR = 0.473, 95%CI: 0.0004-0.2698)、79.4%(OR = 0.718, 95%CI: -0.2691至-0.0147)(表2).
| 菌群 | 中介 | 结局 | OR(95%CI) | 中介效应比(%) | |||
| 总效应 | A效应 | B效应 | 中介效应 | ||||
| Erysipelotrichales目(ID 2148) | IL-6 | 谵妄 | 0.837 (0.786-0.89) | 0.819 (0.676-0.994) | 0.578 (0.390-0.856) | 0.473 (0.0004-0.2698) | 56.51 |
| ErysipelotrichaceaeUCG003属(ID 11384) | IL-6 | 谵妄 | 0.923 (0.878-0.97) | 0.818 (0.679-0.985) | 0.578 (0.390-0.856) | 0.473 (0.0033-0.2671) | 51.2 |
| Erysipelotrichaceae科(ID 2149) | IL-6 | 谵妄 | 0.837 (0.786-0.89) | 0.819 (0.676-0.994) | 0.578 (0.390-0.856) | 0.473 (0.0004-0.2698) | 56.51 |
| Enterobacteriaceae科(ID 3469) | IL-6 | 谵妄 | 0.904 (0.859-0.952) | 1.242 (1.052-1.466) | 0.578 (0.390-0.856) | 0.718 (-0.2691至-0.0147) | 79.4 |
本研究采用MR和中介分析了肠道中的211种常见微生物群与谵妄和炎症因子的关系, 结果显示6种菌群与谵妄发生风险有因果关系, 其中Unknowngenus属(ID 1000001215)、Erysipelatoclostridium属(ID 11381)和Bifidobacterium属(ID 436)与谵妄风险增加有关, Tyzzerella3属(ID 11335)、Faecalibacterium属(ID 2057)和Oxalobacteraceae科(ID 2966)与谵妄风险降低有关. IL-6在部分肠道菌群与谵妄因果关系中存在中介效应.
已有研究表明肠道微生物群的改变与谵妄密切相关. 动物模型的研究发现, 腹部手术后肠道微生物群的异常组成可能会增加术后谵妄的风险[13]. 尤其是志贺氏菌被认为能够预测老年人术后谵妄的发生[14]. 此外, 肠道菌群通过"微生物-肠-脑轴"对神经系统的影响发挥着重要作用. 异常的肠道微生物群组成可以通过该轴与神经系统、内分泌系统和免疫系统的双向通路互相联系, 进而调节中枢神经系统的功能[15]. 而肠道菌群失调会导致炎症因子的合成增加, 例如IL-1β、IL-6和TNF-α, 这些因子可以通过血脑屏障激活下丘脑-垂体-肾上腺皮质轴, 进而引发神经炎症、神经损伤等, 直接影响脑功能, 从而导致谵妄的发生[16,17]. 研究表明[18], 焦虑和谵妄与炎症因子有关.
已有研究发现, 脱硫弧菌科与各种神经系统疾病密切相关[19], 这与本研究中Erysipelatoclostridium与谵妄风险增加的结论相一致. 此外, 本研究结果还表明, 某些肠道微生物群可以降低谵妄发生的风险. 其主要机制似乎是加强上皮细胞连接、保护粘膜屏障、降低肠道通透性, 从而减少肠道炎症因子进入血液循环时对神经系统的损伤[20]. 肠道微生物群还通过分解有害物质来减少对机体的损害. 特别是草杆菌科, 它在草酸盐的代谢中起着重要作用, 草酸代谢失衡可能引发肾脏疾病、炎症及心脑血管并发症[21], 本研究中Oxalobacteraceae属于草杆菌科, 它降低谵妄发生风险的机制可能与草酸代谢平衡有关. 在中介分析中, 本研究发现Erysipelotrichaceae等部分厚壁菌门肠道菌群与谵妄风险之间的因果效应由IL-6介导. 这一发现与先前的一项研究结果相符, 该研究显示厚壁菌门与炎症和氧化应激密切相关, 从而可能会导致谵妄的发生[22].
与观察性研究相比, 本研究采用MR分析方法减少混杂因素和反向因果关系的偏倚, 并通过敏感性分析验证了结果的稳健性. 同时进一步中介分析, 阐述了IL-6的中介效应. 然而, 本研究也存在一些局限性. 本研究中的GWAS汇总数据多来自欧洲, 其结论推广到其他种族和地区的个体时应谨慎. 肠道微生物群的丰度可能会受到饮食、性别、药物和采样时间等因素的影响, 可能潜在地减少由遗传表达的变异比例. 本研究仅分析了部分炎症因子与肠道菌群和谵妄的关联, 进行了IL-6的中介分析, 忽略了其他可能的中介途径.
综上所述, 通过两样本孟德尔分析及中介效应分析全面评估了肠道菌群与谵妄风险之间的因果关系, 并揭示了IL-6在其中发挥潜在的中介作用, 为肠道菌群与谵妄风险的因果关系提供了新的见解, 也为谵妄的预防和治疗提供新的思路.
谵妄是住院患者的常见并发症, 估计发病率为20%-50%, 可能导致机械通气和住院时间延长, 再次住院的风险增加, 住院患者的发病率和死亡率增加. 但是谵妄病因尚未明确, 本研究为谵妄与肠道菌群之间的关系提供部分理论依据.
本研究通过MR分析评估肠道菌群与谵妄发生风险的因果关系, 同时利用中介分析评估部分炎症因子在其中所起的中介作用, 为谵妄的防治提供新思路.
基于以往MR研究在评估因果关系方面的极高可靠性, 我们希望利用MR方法阐明肠道微生物群与谵妄之间的关系, 并为该领域的进一步研究提供新的证据.
本研究利用MR分析从211个肠道微生物群和谵妄的大规模GWAS汇总数据中选择工具变量, 主要采用逆方差加权及中介分析方法进行统计分析, 同时评估多效性和异质性确保实验结果的可靠性.
本实验结果发现在211种肠道菌群中, 3种与谵妄发生风险之间呈正相关, 3种呈负相关, 中介分析结果显示白介素-6(interleukin-6, IL-6)在该关系中起部分中介作用.
本实验利用MR及中介分析, 从遗传基因层面分析因果关系, 通过大规模数据分析结果可靠性强, 我们结果发现6种肠道菌群与谵妄发生风险存在因果关系, 且IL-6在部分关系中存在中介效应, 为谵妄的发生机制提供新的见解, 同时为临床防治谵妄提供新思路.
肠道菌群作为人体内重要的微生物生态系统, 对人体健康和疾病的发生发展有着深远影响. 本研究只分析了部分炎症因子的中介作用, 我们希望有更多的研究, 尤其是随机对照试验研究来发现肠道菌群与谵妄发生风险之间的关系, 及其病理生理机制, 从而为临床预防和治疗谵妄提供理论依据.
学科分类: 胃肠病学和肝病学
手稿来源地: 浙江省
同行评议报告学术质量分类
A级 (优秀): A
B级 (非常好): B
C级 (良好): C, C
D级 (一般): D
E级 (差): E
科学编辑: 刘继红 制作编辑:郑晓梅
| 1. | Zhang S, Han Y, Xiao Q, Li H, Wu Y. Effectiveness of Bundle Interventions on ICU Delirium: A Meta-Analysis. Crit Care Med. 2021;49:335-346. [PubMed] [DOI] |
| 4. | Kc D, Sumner R, Lippmann S. Gut microbiota and health. Postgrad Med. 2020;132:274. [PubMed] [DOI] |
| 5. | Vendrik KEW, Ooijevaar RE, de Jong PRC, Laman JD, van Oosten BW, van Hilten JJ, Ducarmon QR, Keller JJ, Kuijper EJ, Contarino MF. Fecal Microbiota Transplantation in Neurological Disorders. Front Cell Infect Microbiol. 2020;10:98. [PubMed] [DOI] |
| 6. | Xiang K, Wang P, Xu Z, Hu YQ, He YS, Chen Y, Feng YT, Yin KJ, Huang JX, Wang J, Wu ZD, Yang XK, Wang DG, Ye DQ, Pan HF. Causal Effects of Gut Microbiome on Systemic Lupus Erythematosus: A Two-Sample Mendelian Randomization Study. Front Immunol. 2021;12:667097. [PubMed] [DOI] |
| 7. | Kurilshikov A, Medina-Gomez C, Bacigalupe R, Radjabzadeh D, Wang J, Demirkan A, Le Roy CI, Raygoza Garay JA, Finnicum CT, Liu X, Zhernakova DV, Bonder MJ, Hansen TH, Frost F, Rühlemann MC, Turpin W, Moon JY, Kim HN, Lüll K, Barkan E, Shah SA, Fornage M, Szopinska-Tokov J, Wallen ZD, Borisevich D, Agreus L, Andreasson A, Bang C, Bedrani L, Bell JT, Bisgaard H, Boehnke M, Boomsma DI, Burk RD, Claringbould A, Croitoru K, Davies GE, van Duijn CM, Duijts L, Falony G, Fu J, van der Graaf A, Hansen T, Homuth G, Hughes DA, Ijzerman RG, Jackson MA, Jaddoe VWV, Joossens M, Jørgensen T, Keszthelyi D, Knight R, Laakso M, Laudes M, Launer LJ, Lieb W, Lusis AJ, Masclee AAM, Moll HA, Mujagic Z, Qibin Q, Rothschild D, Shin H, Sørensen SJ, Steves CJ, Thorsen J, Timpson NJ, Tito RY, Vieira-Silva S, Völker U, Völzke H, Võsa U, Wade KH, Walter S, Watanabe K, Weiss S, Weiss FU, Weissbrod O, Westra HJ, Willemsen G, Payami H, Jonkers DMAE, Arias Vasquez A, de Geus EJC, Meyer KA, Stokholm J, Segal E, Org E, Wijmenga C, Kim HL, Kaplan RC, Spector TD, Uitterlinden AG, Rivadeneira F, Franke A, Lerch MM, Franke L, Sanna S, D'Amato M, Pedersen O, Paterson AD, Kraaij R, Raes J, Zhernakova A. Large-scale association analyses identify host factors influencing human gut microbiome composition. Nat Genet. 2021;53:156-165. [PubMed] [DOI] |
| 8. | Kurki MI, Karjalainen J, Palta P, Sipilä TP, Kristiansson K, Donner KM, Reeve MP, Laivuori H, Aavikko M, Kaunisto MA, Loukola A, Lahtela E, Mattsson H, Laiho P, Della Briotta Parolo P, Lehisto AA, Kanai M, Mars N, Rämö J, Kiiskinen T, Heyne HO, Veerapen K, Rüeger S, Lemmelä S, Zhou W, Ruotsalainen S, Pärn K, Hiekkalinna T, Koskelainen S, Paajanen T, Llorens V, Gracia-Tabuenca J, Siirtola H, Reis K, Elnahas AG, Sun B, Foley CN, Aalto-Setälä K, Alasoo K, Arvas M, Auro K, Biswas S, Bizaki-Vallaskangas A, Carpen O, Chen CY, Dada OA, Ding Z, Ehm MG, Eklund K, Färkkilä M, Finucane H, Ganna A, Ghazal A, Graham RR, Green EM, Hakanen A, Hautalahti M, Hedman ÅK, Hiltunen M, Hinttala R, Hovatta I, Hu X, Huertas-Vazquez A, Huilaja L, Hunkapiller J, Jacob H, Jensen JN, Joensuu H, John S, Julkunen V, Jung M, Junttila J, Kaarniranta K, Kähönen M, Kajanne R, Kallio L, Kälviäinen R, Kaprio J, FinnGen, Kerimov N, Kettunen J, Kilpeläinen E, Kilpi T, Klinger K, Kosma VM, Kuopio T, Kurra V, Laisk T, Laukkanen J, Lawless N, Liu A, Longerich S, Mägi R, Mäkelä J, Mäkitie A, Malarstig A, Mannermaa A, Maranville J, Matakidou A, Meretoja T, Mozaffari SV, Niemi MEK, Niemi M, Niiranen T, O Donnell CJ, Obeidat ME, Okafo G, Ollila HM, Palomäki A, Palotie T, Partanen J, Paul DS, Pelkonen M, Pendergrass RK, Petrovski S, Pitkäranta A, Platt A, Pulford D, Punkka E, Pussinen P, Raghavan N, Rahimov F, Rajpal D, Renaud NA, Riley-Gillis B, Rodosthenous R, Saarentaus E, Salminen A, Salminen E, Salomaa V, Schleutker J, Serpi R, Shen HY, Siegel R, Silander K, Siltanen S, Soini S, Soininen H, Sul JH, Tachmazidou I, Tasanen K, Tienari P, Toppila-Salmi S, Tukiainen T, Tuomi T, Turunen JA, Ulirsch JC, Vaura F, Virolainen P, Waring J, Waterworth D, Yang R, Nelis M, Reigo A, Metspalu A, Milani L, Esko T, Fox C, Havulinna AS, Perola M, Ripatti S, Jalanko A, Laitinen T, Mäkelä TP, Plenge R, McCarthy M, Runz H, Daly MJ, Palotie A. Author Correction: FinnGen provides genetic insights from a well-phenotyped isolated population. Nature. 2023;615:E19. [PubMed] [DOI] |
| 9. | Bowden J, Davey Smith G, Burgess S. Mendelian randomization with invalid instruments: effect estimation and bias detection through Egger regression. Int J Epidemiol. 2015;44:512-525. [PubMed] [DOI] |
| 10. | Gronau QF, Wagenmakers EJ. Limitations of Bayesian Leave-One-Out Cross-Validation for Model Selection. Comput Brain Behav. 2019;2:1-11. [PubMed] [DOI] |
| 11. | Yuan S, Kim JH, Xu P, Wang Z. Causal association between celiac disease and inflammatory bowel disease: A two-sample bidirectional Mendelian randomization study. Front Immunol. 2022;13:1057253. [PubMed] [DOI] |
| 12. | Boehm FJ, Zhou X. Statistical methods for Mendelian randomization in genome-wide association studies: A review. Comput Struct Biotechnol J. 2022;20:2338-2351. [PubMed] [DOI] |
| 13. | Zhang J, Bi JJ, Guo GJ, Yang L, Zhu B, Zhan GF, Li S, Huang NN, Hashimoto K, Yang C, Luo AL. Abnormal composition of gut microbiota contributes to delirium-like behaviors after abdominal surgery in mice. CNS Neurosci Ther. 2019;25:685-696. [PubMed] [DOI] |
| 14. | Liu H, Cheng G, Xu YL, Fang Q, Ye L, Wang CH, Liu XS. Preoperative Status of Gut Microbiota Predicts Postoperative Delirium in Patients With Gastric Cancer. Front Psychiatry. 2022;13:852269. [PubMed] [DOI] |
| 15. | Agirman G, Yu KB, Hsiao EY. Signaling inflammation across the gut-brain axis. Science. 2021;374:1087-1092. [PubMed] [DOI] |
| 17. | Huang X, Hussain B, Chang J. Peripheral inflammation and blood-brain barrier disruption: effects and mechanisms. CNS Neurosci Ther. 2021;27:36-47. [PubMed] [DOI] |
| 18. | Stamenkovic DM, Rancic NK, Latas MB, Neskovic V, Rondovic GM, Wu JD, Cattano D. Preoperative anxiety and implications on postoperative recovery: what can we do to change our history. Minerva Anestesiol. 2018;84:1307-1317. [PubMed] [DOI] |
| 19. | Murros KE, Huynh VA, Takala TM, Saris PEJ. Desulfovibrio Bacteria Are Associated With Parkinson's Disease. Front Cell Infect Microbiol. 2021;11:652617. [PubMed] [DOI] |
| 20. | Yu H, Wan X, Yang M, Xie J, Xu K, Wang J, Wang G, Xu P. A large-scale causal analysis of gut microbiota and delirium: A Mendelian randomization study. J Affect Disord. 2023;329:64-71. [PubMed] [DOI] |
| 21. | Ermer T, Nazzal L, Tio MC, Waikar S, Aronson PS, Knauf F. Oxalate homeostasis. Nat Rev Nephrol. 2023;19:123-138. [PubMed] [DOI] |