修回日期: 2021-05-07
接受日期: 2021-05-25
在线出版日期: 2021-07-08
长基因间非编码RNA 00963(long intergene non-coding RNA 00963, LINC00963)在肿瘤表达上调, 但LINC00963在胃癌中的功能和作用机制并未阐明. Starbase预测显示微小RNA(microRNA, miR)-146a-5p可能是LINC00963的靶基因, 核因子类红细胞2相关因子1(nuclear factor erythroid-2 like 1, NFE2L1)可能是miR-146a-5p的靶基因. 本研究假设LINC00963可能通过调控miR-146a-5p/NFE2L1轴表达影响胃癌细胞增殖、迁移和侵袭.
探讨LINC00963对胃癌细胞增殖、迁移和侵袭的影响及分子机制.
选取42例胃癌患者的癌组织及癌旁组织, 实时定量PCR(RT-qPCR)检测胃癌组织中LINC00963表达水平; RT-qPCR检测胃癌细胞系SNU-1、AGS、HS-746T中LINC00963、miR-146a-5p和NFE2L1 mRNA的表达水平; 将胃癌细胞SNU-1分为正常对照(NC)组、si-LINC00963组、si-NFE2L1组、si-NC组、miR-146a-5p组、miR-NC组、si-LINC00963+pcDNA-NC组、si-LINC00963+pcDNA-NFE2L1组; CCK-8检测细胞活性; Transwell检测细胞迁移和侵袭; 双荧光素酶报告实验检测LINC00963、miR-146a-5p、NFE2L1之间的靶向关系.
胃癌组织及胃癌细胞系SNU-1、AGS、HS-746T中LINC00963表达水平升高, 胃癌细胞系中miR-146a-5p表达水平降低, NFE2L1 mRNA表达水平升高(P<0.05). LINC00963、NFE2L1低表达或miR-146a-5p高表达, 胃癌细胞SNU-1的活性降低, 细胞迁移和侵袭数量减少(P<0.05). 高表达NFE2L1可以逆转LINC00963低表达对SNU-1细胞的作用. LINC00963靶向调控miR-146a-5p; miR-146a-5p靶向调控NFE2L1.
LINC00963低表达通过调控miR-146a-5p/NFE2L1轴抑制胃癌细胞增殖、迁移和侵袭.
核心提要: LINC00963和NFE2L1在胃癌中表达上调, miR-146a-5p表达下调. 双荧光素酶报告实验证实LINC00963能够靶向miR-146a-5p进而上调NFE2L1表达. LINC00963低表达通过调控miR-146a-5p/NFE2L1轴抑制胃癌细胞增殖、迁移和侵袭.
引文著录: 徐万苏, 柯飞, 许怡, 郑艺. LINC00963通过miR-146a-5p/NFE2L1轴调控胃癌细胞增殖、迁移和侵袭的机制研究. 世界华人消化杂志 2021; 29(13): 690-700
Revised: May 7, 2021
Accepted: May 25, 2021
Published online: July 8, 2021
Long intergenic non-coding RNA 00963 (LINC00963) is up-regulated in tumors, but the function and mechanism of LINC00963 in gastric cancer have not been elucidated. It was predicted using Starbase that microRNA (miR)-146a-5p may be the target gene of LINC00963, and nuclear factor erythroid-2 like 1 (NFE2L1) may be the target gene of miR-146a-5p. We hypothesized that LINC00963 may affect the proliferation, migration and invasion of gastric cancer cells by regulating the miR-146a-5p/NFE2L1 axis.
To explore the effect of LINC00963 on the proliferation, migration, and invasion of gastric cancer cells and the underlying molecular mechanism.
The cancer tissues and adjacent tissues of 42 patients with gastric cancer were collected, and real-time quantitative PCR (RT-qPCR) was used to detect the expression level of LINC00963 in these tissues. RT-qPCR was also used to detect the expression of LINC00963, miR-146a-5p, and NFE2L1 mRNA in gastric cancer cell lines SNU-1, AGS, and HS-746T. SNU-1 cells were then divided into a normal control group (NC) group, si-LINC00963 group, si-NFE2L1 group, si-NC group, miR-146a-5p group, miR-NC group, si-LINC00963 + pcDNA-NC Group, and si-LINC00963 + pcDNA-NFE2L1 group; CCK-8 was used to detect cell viability, Transwell assay was used to detect cell migration and invasion, and dual luciferase reporter assay was used to detect the targeting relationship among LINC00963, miR-146a-5p, and NFE2L1.
The expression level of LINC00963 in gastric cancer tissues and gastric cancer cell lines SNU-1, AGS, and HS-746T was increased, the expression level of miR-146a-5p was decreased in gastric cancer cell lines, and the expression level of NFE2L1 mRNA was increased (P < 0.05). With low expression of LINC00963 and NFE2L1 or high expression of miR-146a-5p, the viability of SNU-1 cells was decreased, and the ability of cell migration and invasion was decreased (P < 0.05). High expression of NFE2L1 can reverse the effect of low expression of LINC00963 on SNU-1 cells. LINC00963 targets and regulates miR-146a-5p, while miR-146a-5p targets and regulates NFE2L1.
Low expression of LINC00963 inhibits the proliferation, migration, and invasion of gastric cancer cells by regulating the miR-146a-5p/NFE2L1 axis.
- Citation: Xu WS, Ke F, Xu Y, Zheng Y. LINC00963 regulates gastric cancer cell proliferation, migration, and invasion through miR-146a-5p/NFE2L1 axis. Shijie Huaren Xiaohua Zazhi 2021; 29(13): 690-700
- URL: https://www.wjgnet.com/1009-3079/full/v29/i13/690.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v29.i13.690
胃癌是全球常见癌症, 死亡率高, 靶向治疗是改善晚期和转移性胃癌治疗效果的重要方法, 开发新型靶向药物, 可个性化治疗并改善胃癌患者预后[1-3]. 研究表明长链非编码RNA(long non-coding RNA, lncRNA), 微小RNA(microRNA, miRNA)参与胃癌的发生发展过程, 提示其在胃癌发生中的作用可用于胃癌的诊断、治疗和预后[4,5]. 研究报道敲低长基因间非编码RNA 00963(long intergene non-coding RNA 00963, LINC00963)显著抑制了黑色素瘤细胞的增殖, 迁移和侵袭[6]. LINC00963通过miR-378g/壳多糖酶3样蛋白1(chitosan enzyme 3-like protein 1, CHI3L1)轴促进卵巢癌的增殖, 迁移和上皮间质转化(epithelial mesenchymal transition, EMT)[7]. LINC00963充当miR-625的分子海绵上调高迁移率蛋白家族A1(high mobility protein family A1, HMGA1), 从而促进乳腺癌的进展[8]. 然而LINC00963对胃癌细胞增殖, 迁移侵袭的影响及机制尚不清楚.
研究发现胃癌细胞中miR-146a-5p下调表达, 且胃癌组织低表达的miR-146-5p与静脉血管侵犯侵袭转移有关[9,10]. 且有研究发现高腹膜转移潜能的胃癌细胞中miR-146a-5p也下调表达[11]. 说明miR-146a-5p可能参与胃癌进展过程, 但miR-146a-5p对胃癌细胞增殖, 迁移侵袭的影响尚不清楚. 本实验通过在线软件预测发现LINC00963与miR-146a-5p, miR-146a-5p与核因子类红细胞2相关因子1(nuclear factor erythroid 2-like 1, NFE2L1)均有结合位点. NFE2L1是CNC-bZIP转录因子家族的成员, 研究发现其与癌症发展以及变性和代谢性疾病的发病机理有关[12]. 研究报道线粒体呼吸缺陷通过信号转导与转录激活因子3(signal transduction and transcriptional activator 3, STAT3)/NFE2L1/突触融合蛋白12(synaptic fusion protein 12, STX12)轴增强肝癌细胞的侵袭性[13]. lncRNA SLCO4A1反义RNA1(SLCO4A1 antisense RNA1, SLCO4A1-AS1)通过调节miR-4701-5p/NFE2L1轴激活WNT途径, 促进肺腺癌的细胞生长并诱导其耐药性[14]. 而NFE2L1对胃癌细胞的影响尚未可知. 本实验旨在研究LINC00963对胃癌细胞增殖, 迁移侵袭的影响及机制是否与miR-146a-5p、NFE2L1有关.
1.1.1 临床资料: 选取本院病理科存档且已确诊为胃癌的组织标本42例, 并选取其相应的癌旁组织, 所有患者均知情且同意. 纳入标准: 经病理证实为胃癌患者; 术前未接受抗肿瘤治疗者; 排除标准: 术前接受放疗或化疗等抗肿瘤治疗者; 患有其他脏器重大疾病者. 男性27例, 女性15例, 年龄在40-73岁之间, 中位年龄63岁. 淋巴结转移: 转移18例, 未转移24例; TNM分期: Ⅰ期15例, Ⅱ期13例, Ⅲ期14例.
1.1.2 细胞与主要试剂: 永生化的人胃粘膜上皮细胞系GES1, 胃癌细胞系SNU-1、AGS、HS-746T购自上海冠导生物工程有限公司. RPMI-1640培养基(美国Gibco); Trizol试剂、SYBR Premix ExTaqTM试剂盒(日本Takara); 总蛋白提取试剂盒(美国Invent); NFE2L1、细胞周期素D1(CyclinD1)、基质金属蛋白酶2(matrix metalloproteinase 2, MMP2)、MMP9抗体(英国biorbyt公司); 细胞计数试剂盒8(Cell Counting Kit 8, CCK-8)(日本同仁研究所); 双荧光素酶报告基因检测试剂盒(美国AAT Bioquest); Transwell小室、Matrigel(美国BD).
1.1.3 细胞培养与分组: GES1及SNU-1、AGS、HS-746T细胞均用RPMI-1640培养基培养, 取对数生长期细胞SNU-1, 将LINC00963、NFE2L1的干扰表达载体及其阴性对照, miR-146a-5p的模拟物及阴性对照转染至SNU-1细胞中, 记为si-LINC00963组、si-NFE2L1组、si-NC组、miR-146a-5p组、miR-NC组, 正常培养的SNU-1细胞作为NC组; 将LINC00963干扰表达载体分别与NFE2L1的过表达载体及阴性对照转染至SNU-1细胞中, 记为si-LINC00963+pcDNA-NC组、si-LINC00963+pcDNA-NFE2L1组.
1.2.1实时荧光定量PCR(RT-qPCR)检测LINC00963、miR-146a-5p和NFE2L1 mRNA的表达水平: 取适量胃癌组织、癌旁组织, 研磨粉碎后加入Trizol试剂提取总RNA; 收集GES1及SNU-1、AGS、HS-746T细胞及各组SNU-1细胞, 直接加入Trizol试剂提取细胞总RNA; 合成互补DNA(complementary DNA, cDNA), 用SYBR Premix ExTaqTM试剂盒进行荧光定量PCR, 以β-actin和U6为内参, 用2-△△Ct法计算相对表达量.
1.2.2 Western blot法检测蛋白表达: 提取细胞总蛋白, 定量后取适量变性蛋白进行聚丙烯酰胺凝胶电泳, 转至聚偏二氟乙烯膜, 用5%脱脂奶粉封闭1 h, 加入一抗(1:800)4 ℃孵育过夜, 加入二抗(1:1200)室温孵育2 h, 用ECL发光液显影, 成像后检测蛋白条带灰度值, 蛋白相对表达水平 = 目的条带灰度值/β-actin条带灰度值.
1.2.3 双荧光素酶报告实验: Starbase预测到LINC00963与miR-146a-5p、miR-146a-5p与NFE2L1-3'非翻译区(untranslated regions, UTR)存在结合位点. 根据预测结果构建LINC00963及NFE2L1-3'UTR的野生型和突变型荧光素酶载体, 将其分别与miR-NC和miR-146a-5p共转染至SNU-1细胞中, 转染48 h收集细胞按照说明书检测荧光素酶活性.
将pcDNA-NC、pcDNA-LINC00963、si-NC、si-LINC00963转染至SNU-1细胞中, 按1.4中方法检测miR-146a-5p表达水平. 将miR-NC、miR-146a-5p、anti-miR-NC、anti-miR-146a-5p转染至SNU-1细胞中; miR-146a-5p分别与pcDNA-NC、pcDNA-LINC00963共转染至SNU-1细胞中, anti-miR-146a-5p分别与si-NC、si-LINC00963共转染至SNU-1细胞中, 按1.5中方法检测NFE2L1蛋白表达水平.
1.2.4 CCK-8检测细胞增殖活性: 各组细胞培养48 h后每孔加入10 μL的CCK-8试剂, 孵育2 h后用酶标仪检测490 nm处吸光度值(absorbance, A), 以A值代表细胞活性.
1.2.5 Transwell检测细胞迁移和侵袭: 细胞迁移实验: 将细胞用无血清培养基重悬, 取100 μL细胞悬浮液添加到Transwell上室, 下室加600 μL含血清的培养基, 培养48 h, 用棉签除去膜顶表面上未迁移的细胞, 膜下表面的细胞用多聚甲醛固定, 清洗后用结晶紫染色, 清洗、干燥; 倒置显微镜下计数细胞. 细胞侵袭实验: 按照1:8比例用无血清培养基稀释Matrigel, 包被Transwell小室底部膜的上室面, 放入37 ℃培养箱孵育4 h使Matrigel聚合成胶备用. 其余步骤和迁移实验一致.
统计学处理 用SPSS 20.0软件进行统计学分析, 符合正态分布的计量资料用平均数±标准差(mean±SD)表示, 两组比较行t检验, 多组间比较采用单因素方差分析, 组间两两比较采用LSD-t检验. 以P<0.05为差异有统计学意义.
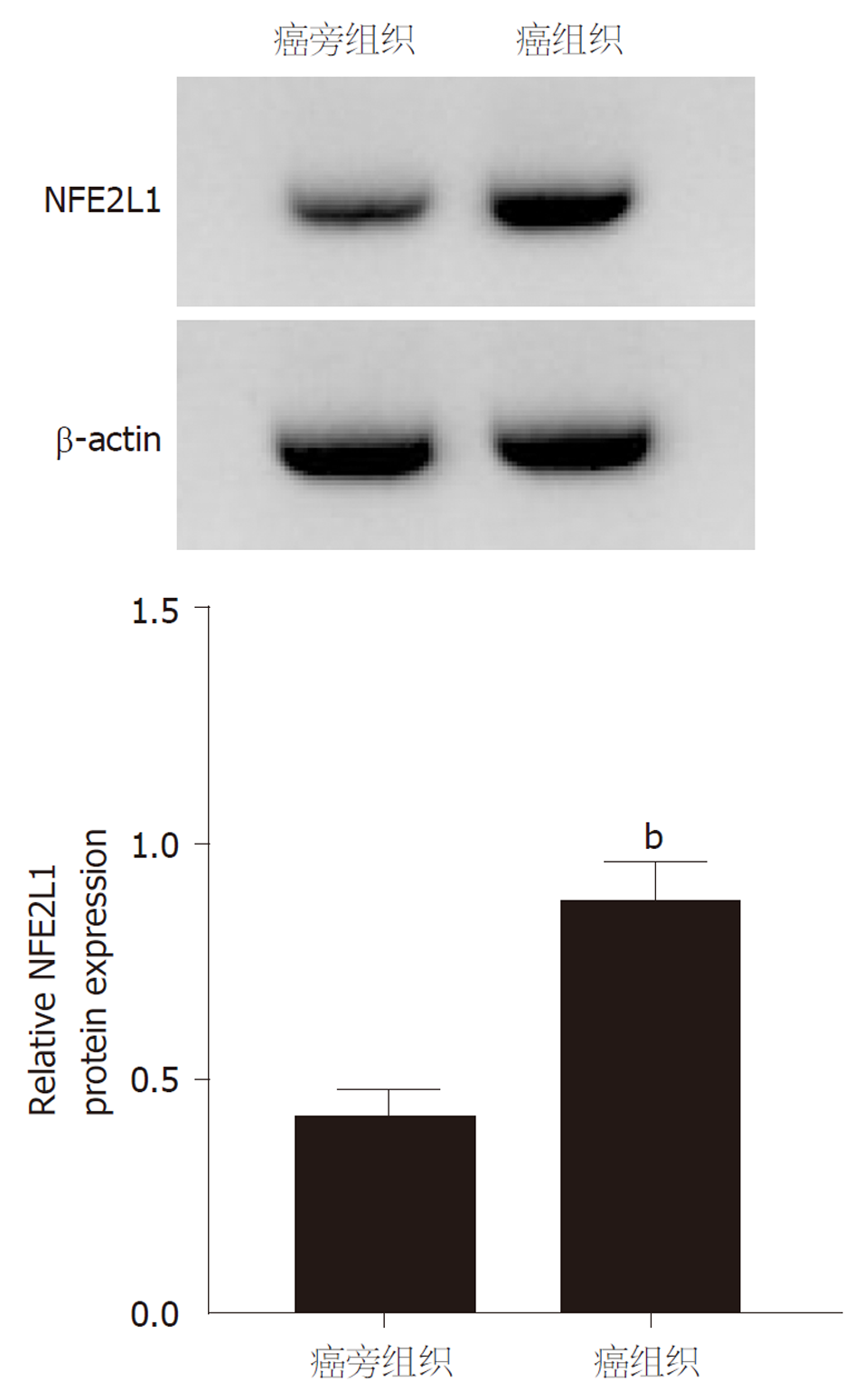
胃癌组织中LINC00963表达水平(3.76±0.24 vs 1.00±0.13, t = 65.532, P<0.05)、NFE2L1 mRNA表达水平(2.16±0.25 vs 1.00±0.12, t = 27.109, P<0.05)、NFE2L1蛋白表达水平(0.87±0.09 vs 0.42±0.05, t = 28.326, P<0.05)高于癌旁组织; 胃癌组织中miR-146a-5p表达水平(0.37±0.07 vs 1.00±0.18, t = 21.140, P<0.05)低于癌旁组织. 见图1.
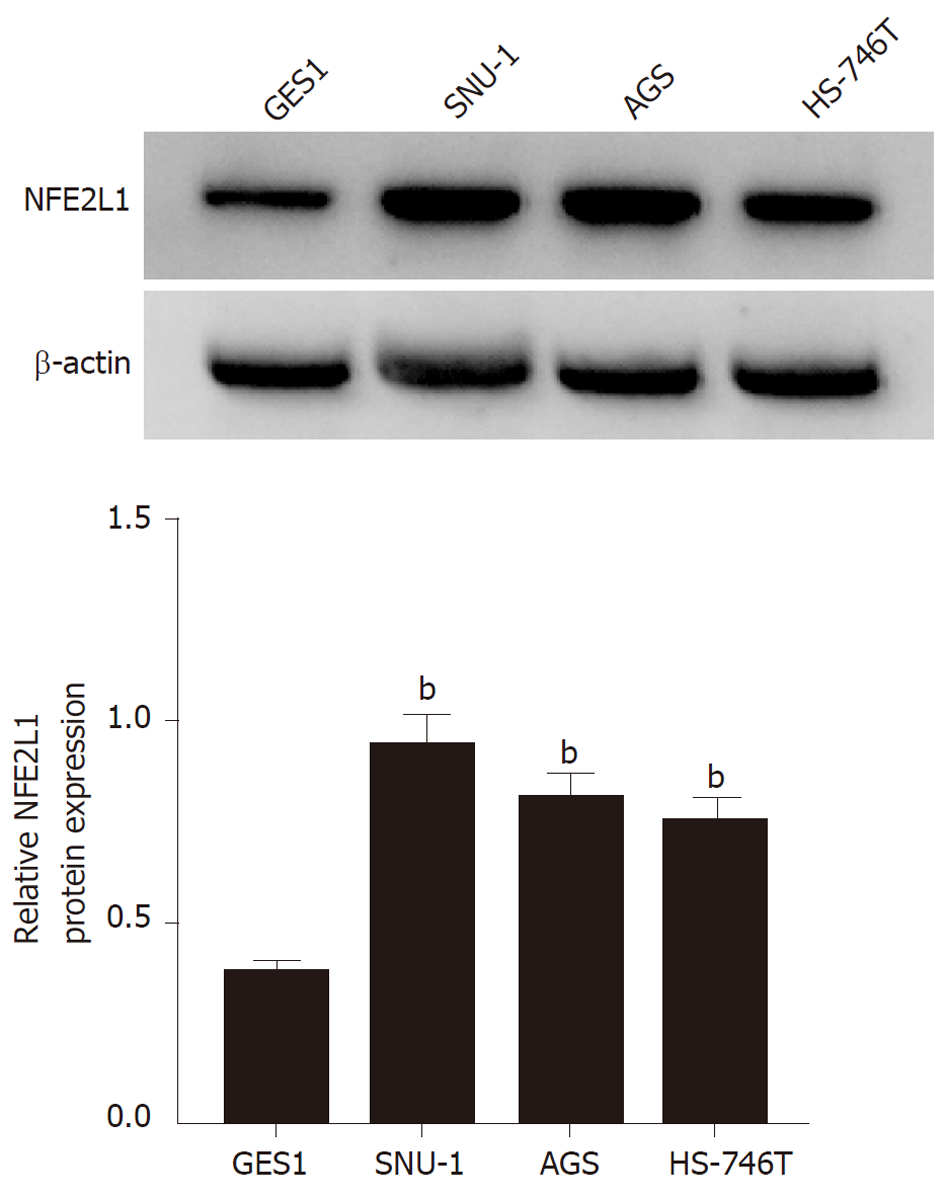
与GES1细胞比较, 胃癌细胞系SNU-1、AGS、HS-746T中LINC00963表达水平升高, miR-146a-5p表达水平降低, NFE2L1 mRNA和蛋白表达水平升高(P<0.05)(图2, 表1).
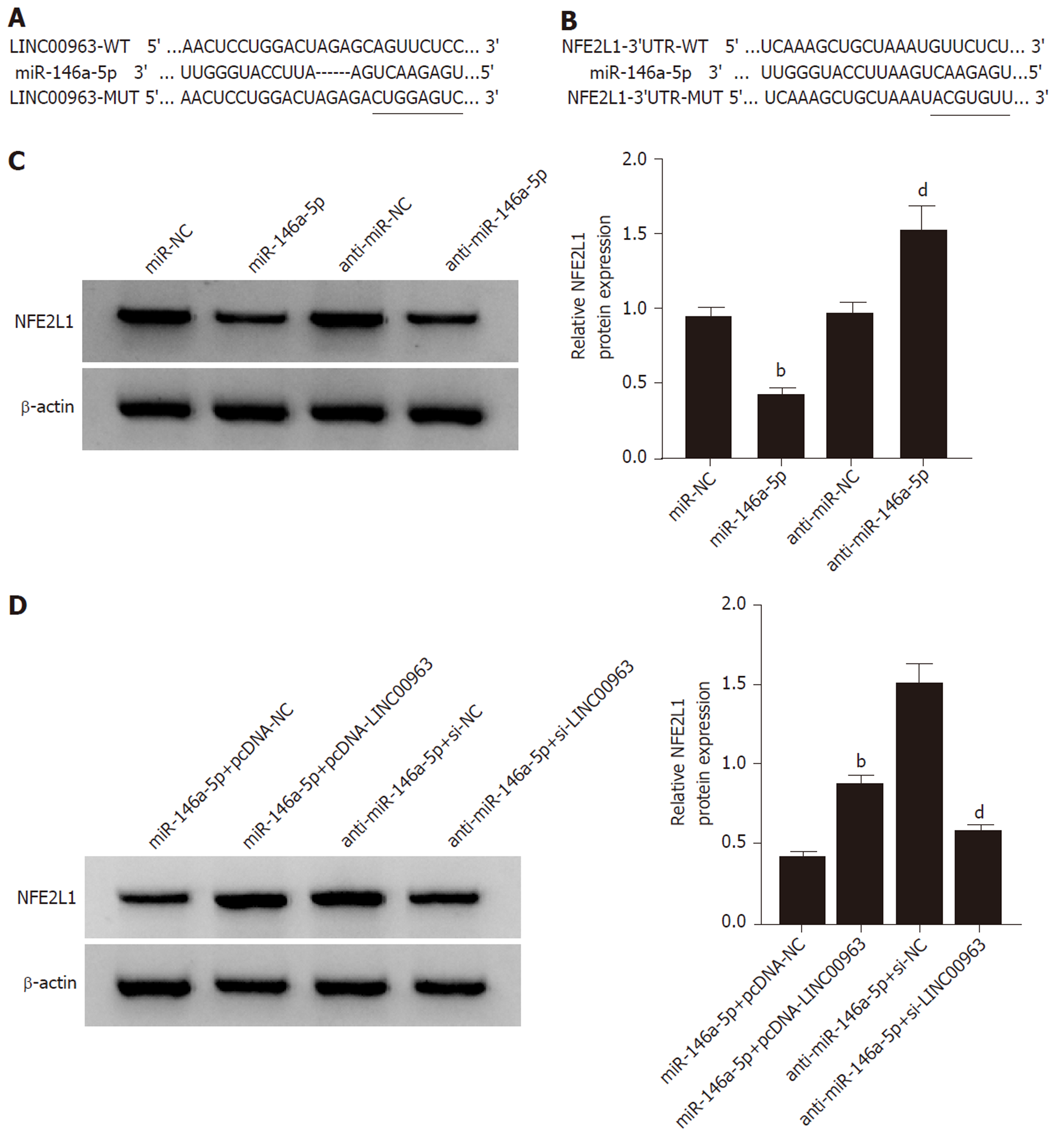
Starbase预测显示, LINC00963与miR-146a-5p存在结合位点(图3A), NFE2L1与miR-146a-5p存在结合位点(图2B); 双荧光素酶实验结果显示, miR-146a-5p与LINC00963、NFE2L1的野生型报告质粒共转染的SNU-1细胞荧光素酶活性降低, 而与LINC00963、NFE2L1的突变型报告质粒共转染的SNU-1细胞荧光素酶活性无显著变化(表2, 表3); 过表达LINC00963后miR-146a-5p表达水平降低; 抑制LINC00963表达后miR-146a-5p表达水平升高(P<0.05)(表4); 过表达miR-146a-5p后NFE2L1蛋白表达水平降低; 抑制miR-146a-5p表达后NFE2L1蛋白表达水平升高(P<0.05)(图3C); 同时过表达miR-146a-5p和LINC00963后NFE2L1蛋白表达水平升高; 而同时抑制miR-146a-5p和LINC00963表达后NFE2L1蛋白表达水平降低(P<0.05)(图3D).
| 组别 | 相对荧光素酶活性 | |
| WT | MUT | |
| miR-NC | 1.00±0.07 | 1.00±0.13 |
| miR-146a-5p | 0.46±0.02b | 1.03±0.07 |
| t | 22.252 | 0.610 |
| P | 0.000 | 0.551 |
| 组别 | 相对荧光素酶活性 | |
| WT | MUT | |
| miR-NC | 1.00±0.11 | 1.00±0.14 |
| miR-146a-5p | 0.42±0.02b | 1.03±0.12 |
| t | 15.563 | 0.488 |
| P | 0.000 | 0.632 |
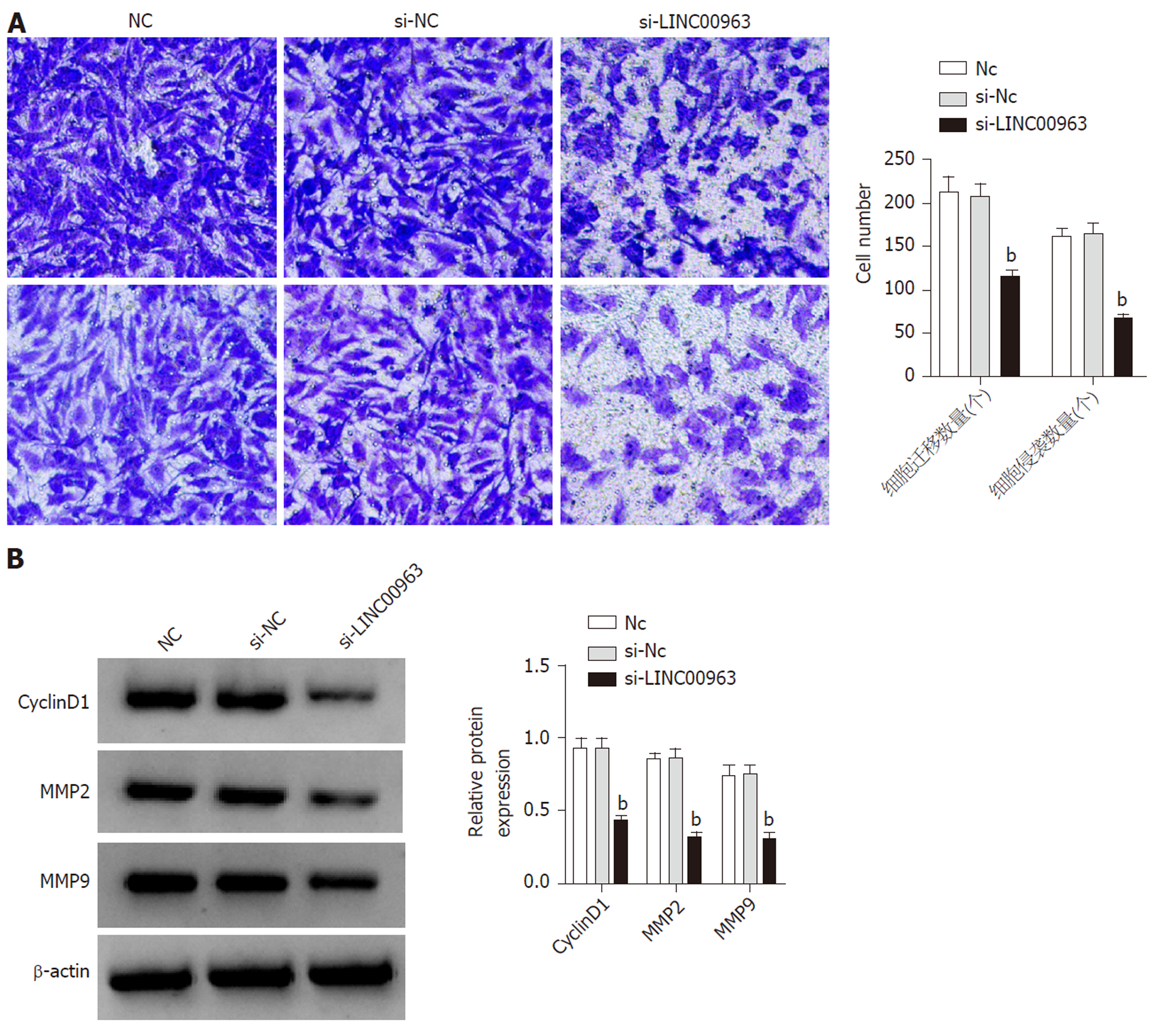
与si-NC组比较, si-LINC00963组胃癌细胞SNU-1中LINC00963表达水平降低, CyclinD1、MMP2、MMP9蛋白表达水平降低, 细胞活性降低, 细胞迁移和侵袭数量减少(P<0.05)(图4, 表5).
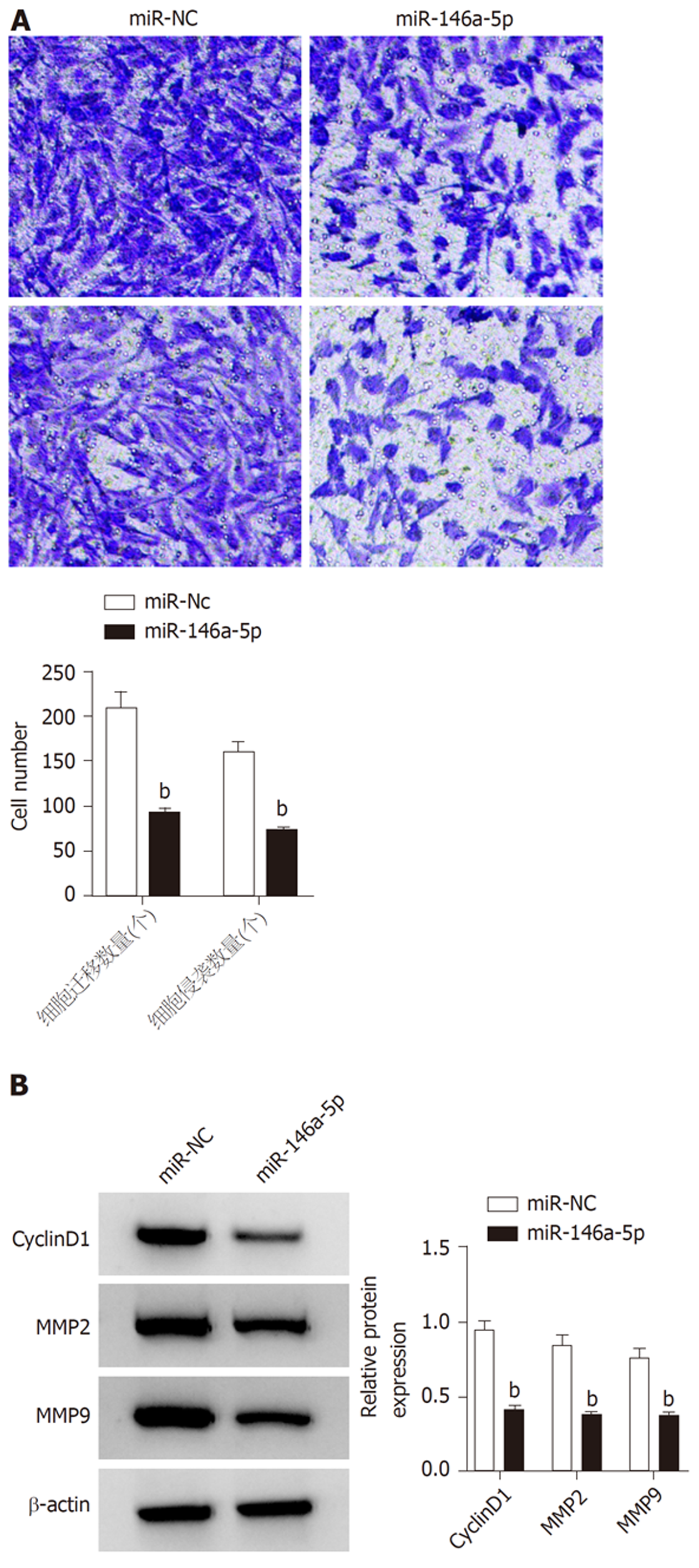
与miR-NC组比较, miR-146a-5p组细胞活性及相关蛋白CyclinD1、MMP2、MMP9的表达水平降低, 细胞迁移和侵袭数量减少(P<0.05)(图5, 表6).
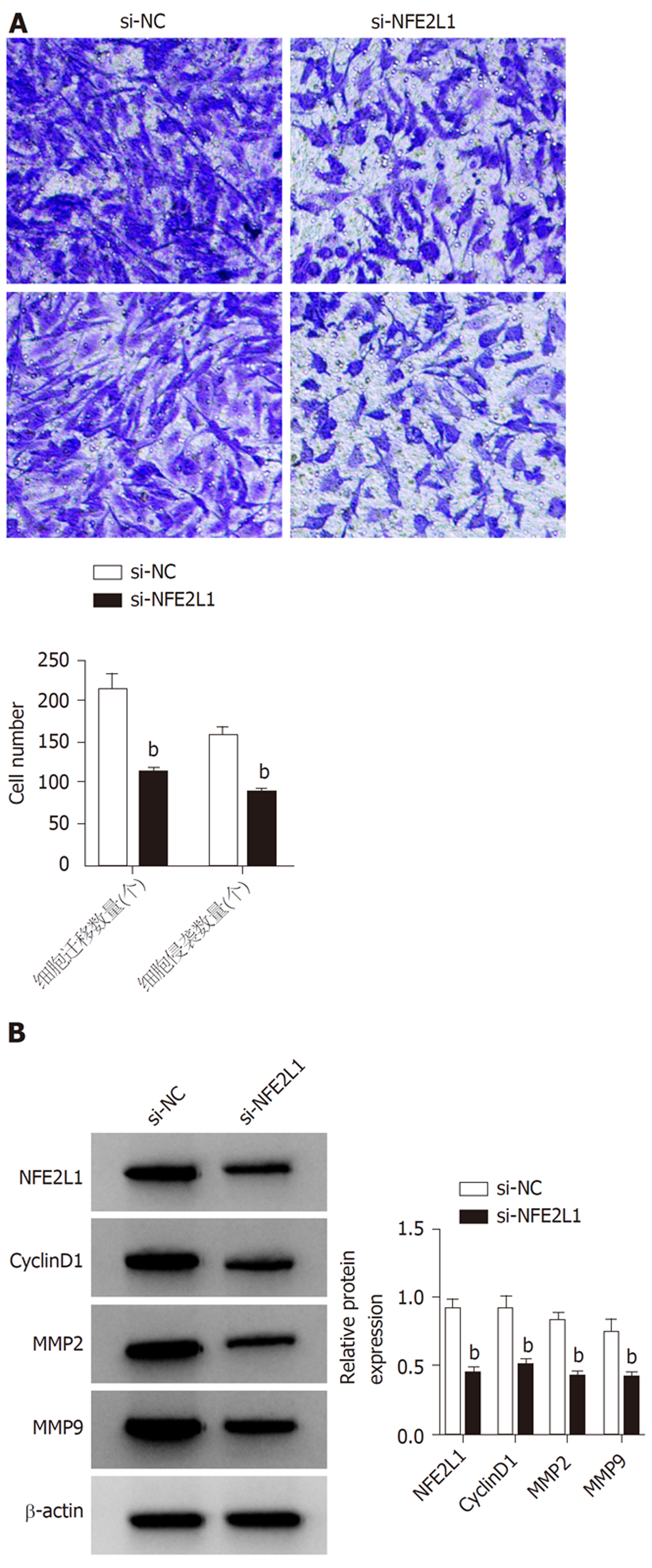
与si-NC组比较, si-NFE2L1组NFE2L1表达按水平降低, CyclinD1、MMP2、MMP9蛋白表达水平降低, 细胞活性降低, 细胞迁移和侵袭数量减少(P<0.05)(图6, 表7).
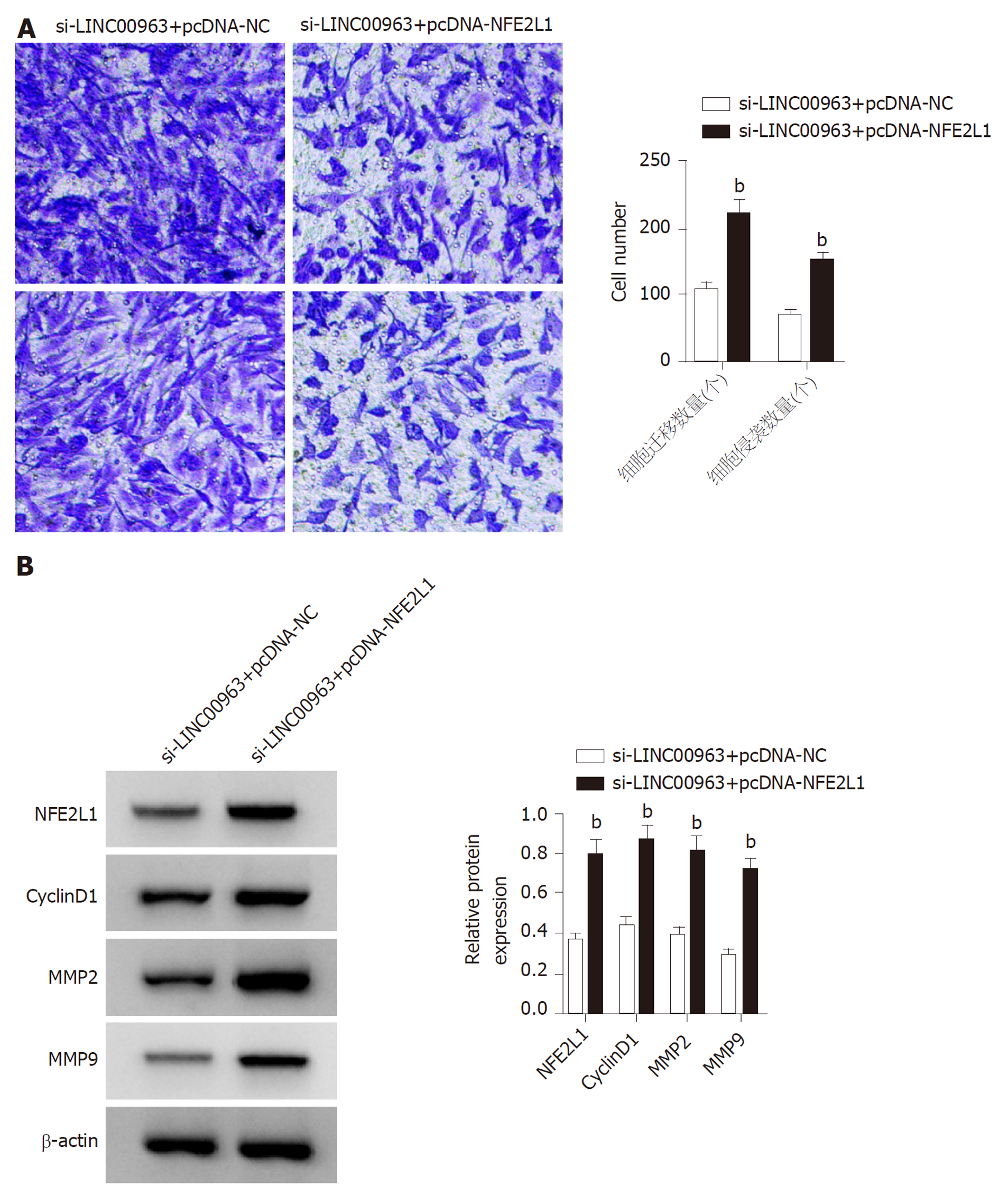
与si-LINC00963+pcDNA-NC组比较, si-LINC00963+pcDNA-NFE2L1组NFE2L1表达水平升高, 细胞活性以及增殖、迁移相关蛋白CyclinD1、MMP2、MMP9的表达水平升高, 且迁移和侵袭细胞数量增加(P<0.05)(图7, 表8).
胃癌是全球高发肿瘤, 研究胃癌的发生及进展机制, 开发相应的靶向药物应用于临床是目前靶向治疗的关键[15]. 研究报道LINC00963在骨肉瘤样品和细胞中高表达, LINC00963通过抑制miR-204-3p/纤维连接蛋白1(fibronectin 1, FN1)轴促进骨肉瘤的增殖和侵袭[16]. LINC00963通过调节miR-506/支链氨基转移酶1(branched-chain amino acid transaminase 1, BCAT1)轴促进了神经胶质瘤细胞增殖, 细胞周期进程, 迁移和体外侵袭以及体内肿瘤发生[17]. LINC00963通过miR-124-3p/卷曲蛋白4(frizzled-4, FZD4)途径促进结直肠癌的增殖和迁移[18]. 以上研究表明LINC00963在多种肿瘤中起促癌作用. 本实验结果显示, LINC00963在胃癌组织和细胞系中高表达, 提示LINC00963可能在胃癌中也起促癌作用. 抑制LINC00963表达后, 细胞活性降低, 迁移和侵袭数量减少; 说明抑制LINC00963表达可抑制胃癌细胞SNU-1增殖、迁移和侵袭.
研究表明miR-146a-5p可用作非侵入性生物标志物和多种癌症的靶向治疗药物[19]. miR-146a-5p通过靶向下调白介素1受体相关激酶1(interleukin-1 receptor-associated kinase 1, IRAK1)的表达可抑制乳腺癌细胞的生长、迁移和侵袭[20]. miR-146a-5p通过调节性别决定区Y框蛋白5(sex determining region Ybox protein 5, SOX5)抑制三阴性乳腺癌细胞的增殖和转移[21]. 本实验结果显示, 胃癌细胞系中miR-146a-5p表达水平降低; 与文献[9,10]中结果一致. 为进一步研究miR-146a-5p对胃癌细胞的影响, 本实验高表达miR-146a-5p, 结果发现高表达miR-146a-5p可抑制胃癌细胞SNU-1的增殖、迁移和侵袭. 研究报道LncRNA钾电压门控通道亚家族Q成员1重叠转录本(KCNQ1oppositestrand/ antisensetranscript1, KCNQ1OT1)通过miR-146a-5p抑制放射敏感性并促进肝细胞癌的发生[22]. 本实验通过在线软件预测LINC00963可能靶向结合的miRNA, 结果发现LINC00963可靶向结合miR-146a-5p; 且本实验证实了LINC00963靶向调控miR-146a-5p, 说明LINC00963可能通过调控miR-146a-5p影响胃癌细胞增殖、迁移和侵袭等过程.
本实验通过在线软件预测发现miR-146a-5p与NFE2L1有结合位点; 研究报道NFE2L1在食管鳞癌中高表达, 与食管鳞癌肿瘤分化程度和淋巴转移相关[23]. 肺泡Ⅱ型上皮细胞中NFE2L1在乌拉坦所致的小鼠肺腺癌中发挥肿瘤抑制作用[24]. 本实验结果显示, 胃癌细胞系中NFE2L1 mRNA和蛋白表达水平升高. 抑制NFE2L1表达可降低细胞活性降低, 减少迁移和侵袭数量; 表明NFE2L1低表达可抑制胃癌细胞增殖、迁移和侵袭. 且本实验表明miR-146a-5p靶向调控NFE2L1; 还发现LINC00963通过miR-146a-5p调控NFE2L1的表达, 高表达NFE2L1逆转了LINC00963低表达对SNU-1增殖, 迁移和侵袭的抑制作用.
综上所述, LINC00963低表达可能通过调控miR-146a-5p/NFE2L1轴抑制胃癌细胞增殖、迁移和侵袭.
胃癌是全球常见癌症, 死亡率高, 但胃癌发生、进展的潜在机制尚未阐明, 已有报道显示lncRNA表达异常可调控胃癌细胞恶性生物学行为, 但仍有部分lncRNA在胃癌进展中作用机制尚未可知.
LINC00963在胃癌中表达上调, 但LINC00963在胃癌进展中的作用机制并不清楚. 既往研究证实LINC00963在肿瘤进展中具有致癌作用. 因此, 探讨LINC00963在胃癌进展中的作用和机制对胃癌诊断和治疗意义重大.
LINC00963低表达通过调控miR-146a-5p/NFE2L1轴可抑制胃癌细胞增殖、迁移和侵袭, 为胃癌治疗提供潜在靶点.
RT-qPCR和western blot检测胃癌组织、细胞系中LINC00963、miR-146a-5p和NFE2L1的表达. 将si-LINC00963、si-NFE2L1、miR-146a-5p mimics分别转染到胃癌细胞SNU-1中, CCK-8法检测细胞增殖; Transwell实验检测细胞迁移和侵袭; 双荧光素酶报告实验验证LINC00963与miR-146a-5p、miR-146a-5p与NFE2L1的靶向关系. 将si-LINC00963和pcDNA-NFE2L1共转染到SNU-1细胞中, 采用CCK-8、Transwell实验检测细胞增殖、迁移和侵袭能力. Western blot检测增殖、迁移和侵袭相关蛋白表达.
LINC00963、NFE2L1在胃癌组织、细胞系中表达上调, miR-146a-5p表达下调. 低表达LINC00963或低表达NFE2L1或高表达miR-146a-5p均可抑制SNU-1细胞增殖、迁移和侵袭, 下调CyclinD1、MMP2、MMP9蛋白表达. 双荧光素酶报告实验证实LINC00963可靶向结合miR-146a-5p, miR-146a-5p可靶向结合NFE2L1. LINC00963可负向调控miR-146a-5p表达, miR-146a-5p可负向调控NFE2L1表达. 高表达NFE2L1可以逆转LINC00963低表达对SNU-1细胞增殖、迁移和侵袭的抑制作用.
胃癌中LINC00963表达增加, LINC00963低表达通过调控miR-146a-5p/NFE2L1轴能够减弱胃癌细胞的增殖、迁移和侵袭能力.
LINC00963下游是否存在其他miRNA、miR-146a-5p下游是否存在其他靶点仍需进一步确认, 还需体内移植瘤实验验证LINC00963的致癌作用, LINC00963/miR-146a-5p/NFE2L1轴可能作为胃癌治疗的靶标基因.
学科分类: 胃肠病学和肝病学
手稿来源地: 浙江省
同行评议报告学术质量分类
A级 (优秀): 0
B级 (非常好): B
C级 (良好): C, C, C, C
D级 (一般): 0
E级 (差): E
科学编辑: 张砚梁 制作编辑:张砚梁
| 1. | Zhao TT, Xu H, Xu HM, Wang ZN, Xu YY, Song YX, Yin SC, Liu XY, Miao ZF. The efficacy and safety of targeted therapy with or without chemotherapy in advanced gastric cancer treatment: a network meta-analysis of well-designed randomized controlled trials. Gastric Cancer. 2018;21:361-371. [PubMed] [DOI] |
| 2. | Pellino A, Riello E, Nappo F, Brignola S, Murgioni S, Djaballah SA, Lonardi S, Zagonel V, Rugge M, Loupakis F, Fassan M. Targeted therapies in metastatic gastric cancer: Current knowledge and future perspectives. World J Gastroenterol. 2019;25:5773-5788. [PubMed] [DOI] |
| 3. | Raimondi A, Nichetti F, Peverelli G, Di Bartolomeo M, De Braud F, Pietrantonio F. Genomic markers of resistance to targeted treatments in gastric cancer: potential new treatment strategies. Pharmacogenomics. 2018;19:1047-1068. [PubMed] [DOI] |
| 4. | Ghafouri-Fard S, Taheri M. Long non-coding RNA signature in gastric cancer. Exp Mol Pathol. 2020;113:104365. [PubMed] [DOI] |
| 5. | Link A, Kupcinskas J. MicroRNAs as non-invasive diagnostic biomarkers for gastric cancer: Current insights and future perspectives. World J Gastroenterol. 2018;24:3313-3329. [PubMed] [DOI] |
| 6. | Jiao H, Jiang S, Wang H, Li Y, Zhang W. Upregulation of LINC00963facilitates melanoma progression through miR-608/NACC1 pathway and predicts poor prognosis. Biochem Biophys Res Commun. 2018;504:34-39. [PubMed] [DOI] |
| 7. | Liu W, Yang YJ, An Q. LINC00963Promotes Ovarian Cancer Proliferation, Migration and EMT via the miR-378g /CHI3L1 Axis. Cancer Manag Res. 2020;12:463-473. [PubMed] [DOI] |
| 8. | Wu Z, Wang W, Wang Y, Wang X, Sun S, Yao Y, Zhang Y, Ren Z. Long noncoding RNA LINC00963promotes breast cancer progression by functioning as a molecular sponge for microRNA-625 and thereby upregulating HMGA1. Cell Cycle. 2020;19:610-624. [PubMed] [DOI] |
| 10. | Li H, Xie S, Liu M, Chen Z, Liu X, Wang L, Li D, Zhou Y. The clinical significance of downregulation of mir-124-3p, mir-146a-5p, mir-155-5p and mir-335-5p in gastric cancer tumorigenesis. Int J Oncol. 2014;45:197-208. [PubMed] [DOI] |
| 11. | Feng Y, Bai F, You Y, Bai F, Wu C, Xin R, Li X, Nie Y. Dysregulated microRNA expression profiles in gastric cancer cells with high peritoneal metastatic potential. Exp Ther Med. 2018;16:4602-4608. [PubMed] [DOI] |
| 12. | Kim HM, Han JW, Chan JY. Nuclear Factor Erythroid-2 Like 1 (NFE2L1): Structure, function and regulation. Gene. 2016;584:17-25. [PubMed] [DOI] |
| 13. | Lee YK, Kwon SM, Lee EB, Kim GH, Min S, Hong SM, Wang HJ, Lee DM, Choi KS, Park TJ, Yoon G. Mitochondrial Respiratory Defect Enhances Hepatoma Cell Invasiveness via STAT3/NFE2L1/STX12 Axis. Cancers (Basel). 2020;12. [PubMed] [DOI] |
| 14. | Wei Y, Wei L, Li J, Ma Z, Zhang Q, Han Z, Li S. SLCO4A1-AS1 promotes cell growth and induces resistance in lung adenocarcinoma by modulating miR-4701-5p/NFE2L1 axis to activate WNT pathway. Cancer Med. 2020;9:7205-7217. [PubMed] [DOI] |
| 16. | Zhou Y, Yin L, Li H, Liu LH, Xiao T. The LncRNA LINC00963 facilitates osteosarcoma proliferation and invasion by suppressing miR-204-3p/FN1 axis. Cancer Biol Ther. 2019;20:1141-1148. [PubMed] [DOI] |
| 17. | Ye F, Xu R, Ge Y, Zheng Y, Liu X, Deng P, Xu X. LINC00963Confers Oncogenic Properties in Glioma by Regulating the miR-506/BCAT1 Axis. Cancer Manag Res. 2020;12:2339-2351. [PubMed] [DOI] |
| 18. | Zheng K, Zhang TK. LncRNA LINC00963promotes proliferation and migration through the miR-124-3p/FZD4 pathway in colorectal cancer. Eur Rev Med Pharmacol Sci. 2020;24:7634-7644. [PubMed] [DOI] |
| 19. | Iacona JR, Lutz CS. miR-146a-5p: Expression, regulation, and functions in cancer. Wiley Interdiscip Rev RNA. 2019;10:e1533. [PubMed] [DOI] |
| 20. | Long JP, Dong LF, Chen FF, Fan YF. miR-146a-5p targets interleukin-1 receptor-associated kinase 1 to inhibit the growth, migration, and invasion of breast cancer cells. Oncol Lett. 2019;17:1573-1580. [PubMed] [DOI] |
| 21. | Si C, Yu Q, Yao Y. Effect of miR-146a-5p on proliferation and metastasis of triple-negative breast cancer via regulation of SOX5. Exp Ther Med. 2018;15:4515-4521. [PubMed] [DOI] |
| 22. | Yang G, Zhou L, Xu Q, Meng F, Wan Y, Meng X, Wang L, Zhang L. LncRNA KCNQ1OT1 inhibits the radiosensitivity and promotes the tumorigenesis of hepatocellular carcinoma via the miR-146a-5p/ACER3 axis. Cell Cycle. 2020;19:2519-2529. [PubMed] [DOI] |