修回日期: 2019-08-15
接受日期: 2019-09-13
在线出版日期: 2019-09-28
大量研究已证实肝硬化患者存在肠道菌群失调的现象, 这为临床防治肝硬化及并发症提供了新的线索与思路. 藏族人群生活环境和饮食习惯有别于汉族人群, 因而藏族肝硬化患者肠道菌群结构及多样性亦可能存在差异.
探讨藏族肝硬化患者肠道菌群结构、丰度与多样性, 并对比汉族肝硬化患者肠道菌群的差异及特点.
前瞻性纳入2017-10/2018-10四川大学华西医院成办分院消化内科36例肝硬化患者, 其中20例藏族设为实验组, 16例汉族肝硬化患者设为对照组; 收集患者基本信息、血常规、生化、凝血功能、影像学等, 并采集大便标本, 提取基因组DNA并对细菌16S rRNA基因V4区进行PCR扩增, 应用Illumina Miseq平台进行高通量测序, 测序原始结果进行Reads拼接、操作分类单元(operational taxonomic units, OTUs)聚类、物种注释、Alpha多样性分析等, 最终得到样品的生物信息学结果.
对照组和实验组分别获得15006和36872条有效序列, 在实验组中Chao1指数显著降低(P<0.01), 实验组的物种丰富度较对照组显著降低. 基于97%的相似性进行物种注释, 对实验组和对照组进行微生物物种丰度比较. 结果显示, 相较于对照组, 在门水平上, 实验组中拟杆菌门显著减少(P<0.001), 厚壁菌门和放线菌门在实验组中增加(P<0.01和P<0.05); 在属水平上, 葡萄球菌属、放线菌属在实验组中显著减少(P<0.01), 乳杆菌属显著增加(P<0.01).
藏族肝硬化患者的肠道微生物丰度明显低于汉族肝硬化患者, 肠道微生物组成存在着差异. 本研究首次展现了藏族肝硬化患者的肠道微生物组成特点, 为肝硬化的个性化治疗奠定了基础.
核心提要: 通过肠道菌群高通量测序, 探讨藏族肝硬化患者肠道菌群结构、丰度与多样性, 并对比汉族肝硬化患者肠道菌群的差异及特点. 结论: 藏族肝硬化患者的肠道微生物丰度明显低于汉族肝硬化患者, 肠道微生物组成存在着差异.
引文著录: 宦徽, 胡红, 陈小红, 高薇娜, 李玲丽, 李骥, 邓凯, 刘超. 基于高通量测序技术的藏族肝硬化患者肠道菌群多样性研究. 世界华人消化杂志 2019; 27(18): 1142-1148
Revised: August 15, 2019
Accepted: September 13, 2019
Published online: September 28, 2019
A large number of studies have confirmed that intestinal flora imbalance exists in patients with liver cirrhosis, which provides new clues and ideas for clinical prevention and treatment of liver cirrhosis and its complications. The living environment and dietary habits of Tibetan people are different from those of Han people, so the structure and diversity of intestinal flora in Tibetan patients with liver cirrhosis may also be different.
To explore the structure, abundance, and diversity of intestinal flora in Tibetan patients with cirrhosis, and to compare the differences and characteristics of intestinal flora between Tibetan and Han patients with cirrhosis.
Sixteen Chinese Han patients with liver cirrhosis (control group) and 20 Tibetans with liver cirrhosis (experimental group) were enrolled from October 2017 to October 2018. Basic information, routine blood examination, biochemical information, coagulation function, and imaging information of the patients were detected and collected. DNA was extracted from fecal samples and the V4 variable region of 16S rDNA was amplified. The amplification products were sequenced using the Illumina Miseq platform, and the sequencing results were subject to cluster analysis of OTUs (operational taxonomic units), species annotation, and alpha diversity analysis.
A total of 15006 and 36872 valid tags were obtained in the control and experimental groups, respectively. Chao1 index of the experimental group decreased significantly when compared to the control group, indicating a lower abundance of gut microbiota in the experimental group. Based on a 97% similarity, species annotation and the comparison of abundance between two groups were carried out. In the experimental group, at the phylum level, the relative abundance level of Bacteroidetes was significantly lower (P < 0.001), while the relative abundance level of Firmicutes and Actinobacteria significantly increased (P < 0.01 and P < 0.05). At the genus level, the relative abundance of Staphylococcus and Actinomycetes decreased (P < 0.01), while the relative abundance of Lactobacillus increased (P < 0.01).
There is a significant difference in the constitution of gut microbiota between Tibetan and Chinese Han patients with liver cirrhosis. This work for the first time reveals the feature of intestinal microbiota in Tibetan patients with liver cirrhosis, which lays a foundation for individual treatment of liver cirrhosis.
- Citation: Huan H, Hu H, Chen XH, Gao WN, Li LL, Li J, Deng K, Liu C. Diversity of intestinal microflora in tibetan patients with liver cirrhosis based on high-throughput sequencing. Shijie Huaren Xiaohua Zazhi 2019; 27(18): 1142-1148
- URL: https://www.wjgnet.com/1009-3079/full/v27/i18/1142.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v27.i18.1142
在我国, 肝硬化占所有疾病的1.39%, 占全部肝病住院病例的51.1%, 是我国最严重的公共卫生问题之一, 而在藏区这一疾病的高发性尤为突出. 大量研究证实在肝硬化患者中明显存在肠道菌群失调的现象. 藏族人群生活环境和饮食习惯有别于汉族人群, 这些差异可能影响肠道菌群, 目前缺乏藏族肝硬化患者肠道菌群的研究, 国内国外均未见报道, 亟待相关研究填补空白. 本研究对比藏族肝硬化与汉族肝硬化患者肠道菌群差异, 为下一步探索个性化治疗提供依据.
前瞻性纳入2017-10/2018-10四川大学华西医院成办分院消化内科41例肝硬化患者. 纳入标准: (1)肝硬化患者(存在可引起肝硬化的病因; 存在门静脉高压和或肝功能障碍表现; 腹部CT或彩超等影像学证据支持); (2)年龄18-75岁; (3)同意参加本临床研究并签署知情同意书(2017研第10号). 排除标准: (1)有外科分流、肝叶切除、肝移植手术等肝脏手术史者; (2)未控制的感染或败血症; (3)合并恶性肿瘤患者; (4)近2 wk内使用抗生素、类固醇等激素和益生菌制剂; (5)恶病质; (6)妊娠期或哺乳期女性; (7)1 mo内有肠道感染病史者; (8)存在重要器官功能衰竭的患者; (9)藏族患者离开高原环境2 wk以上, 且饮食习惯发生明显改变者.
其中, 1例因存在重要器官功能衰竭; 2例藏族患者因离开高原环境2 wk以上且饮食习惯发生明显改变; 1例合并恶性肿瘤; 1例近2 wk内使用抗生素被排除, 最终36例纳入本研究. 将此中20例藏族肝硬化患者设为实验组; 16例汉族肝硬化患者设为对照组. 收集患者基本信息、血常规、生化、凝血功能、影像学等.
1.2.1 粪便样本的收集与DNA提取: 采集36例肝硬化患者的大便标本(在清洁环境中采集研究对象的新鲜粪便(不少于6 g), 采集到的粪便样本立即放入-80 ℃冰箱进行冻存. 采用DNA提取试剂盒(DNeasy PowerSoil Kit, 天根生化科技有限公司, 中国北京)对粪便样本的总DNA进行提取, 提取方法参考试剂盒操作说明书. 提取得到的总DNA用琼脂糖凝胶电泳检测其完整性, 用并用NanoDrop检测DNA的浓度. 检测合格的DNA放入-20 ℃冻存待下一步的操作.
1.2.2 16S rRNA基因V4区扩增与测序结果分析: 使用16S rRNA基因通用引物343F(5'-TACGGRAGGCAGCAG-3')和798R(5'-AGGGTATCTAATCCT-3')对V4区可变区进行PCR扩增, 扩增产物应用Illumina Miseq平台进行高通量测序, 由上海欧易生物医学科技有限公司完成(中国上海). 采用QIIME 1.8.0软件对测序原始结果进行过滤, 过滤得到的clean tags按照97%的相似性进行操作分类单元(operational taxonomic units, OTUs)聚类, 对分类的OTUs进行物种注释分析, 并进行Alpha多样性计算, 包括香农(Shannon)指数、辛普森(Simpson)指数和丰富度指数Chao1. 此外, 根据分类学层次门和属分析微生物的群落结构以及物种丰度.
统计学处理 采用SPSS 19.0软件进行统计学分析. 呈正态分布的计量资料以mean±SD表示, 比较采用方差分析; 非正态分布, 资料以中位数及四分位数表示, 比较采用Kruskal-Wallis检验. 计数资料以例数和百分数表示, 比较采用χ2检验. 基于Illumina Miseq测序平台获得关于肠道细菌16S rDNA的测序结果, 并在对原始数据进行质量控制后, 采用相应的生物数据分析方法比较各组肠道菌群情况. P<0.05认为有统计学差异.
两组在性别、年龄、白蛋白、总胆红素、PT、腹水及Child-Pugh评分均无显著差异(表1).
| 组别 | 例数 | 性别[男/女(例)] | 年龄(岁) | 白蛋白(g/L) | 总胆红素(mmol/L) | PT (s) | 肝性脑病(例) | 腹水(例) | Child-Pugh(分) |
| 实验组 | 20 | 16/4 | 51.20±11.18 | 34.88±6.27 | 21.68±10.70 | 14.26±1.87 | 1 | 11 | 6.9 |
| 对照组 | 16 | 13/3 | 49.18±10.67 | 35.40±6.99 | 19.83±9.82 | 13.79±1.76 | 3 | 9 | 6.5 |
| P值 | 0.261 | 0.583 | 0.798 | 0.558 | 0.410 | 0.472 | 0.940 | 0.473 |
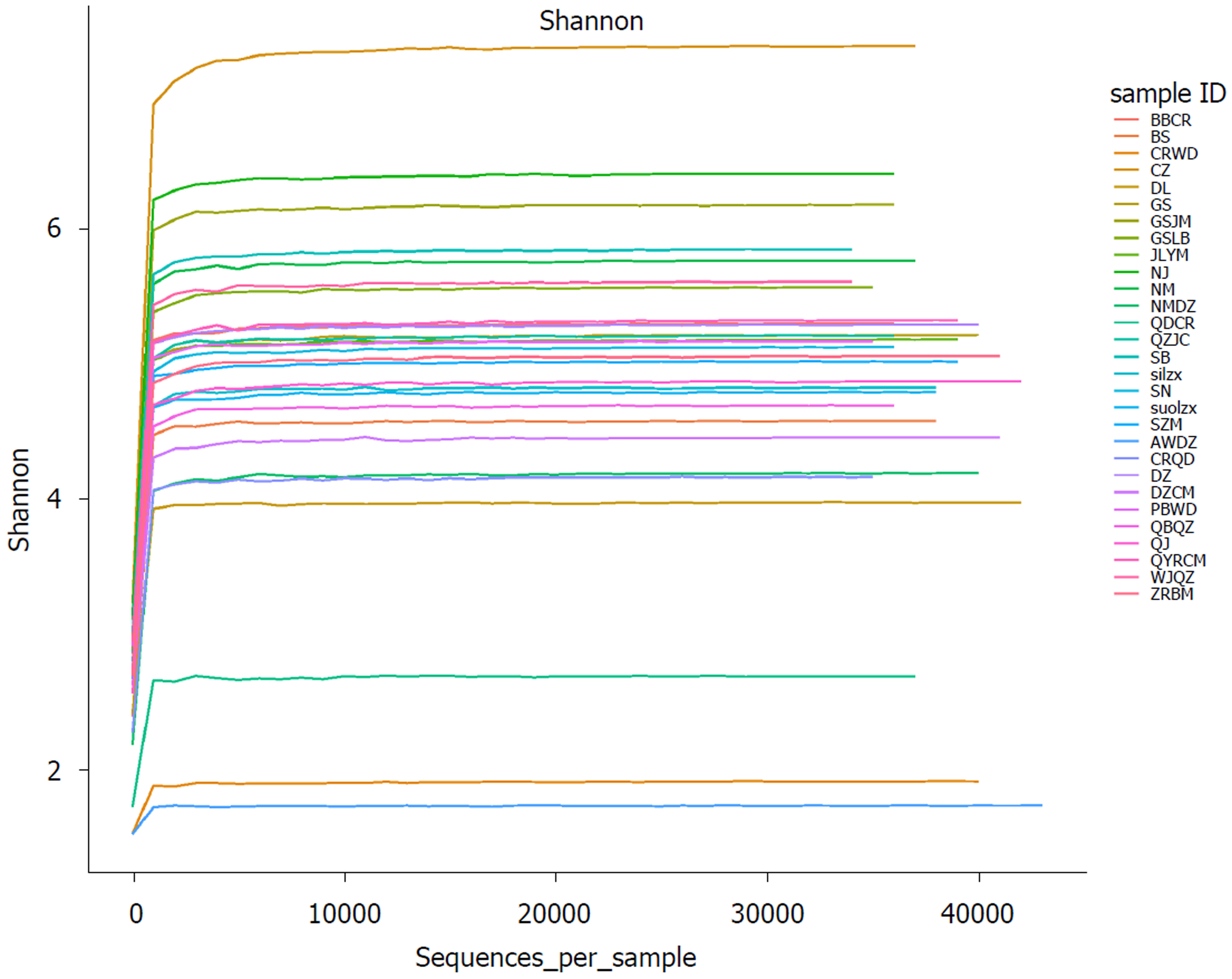
收集到的36个样本进行16S rDNA基因序列测序, 利用QIIME软件对原始序列进行过滤, 最终对照组和实验组分别得到平均19963和48338条clean tags, 其中有效tags分别为15006(75.17%)和36872(76.28%), 有效tags平均读长分别为428 bp和431 bp. Shannon指数稀释曲线显示在Shannon指数为4-6时, 绝大多数样本的稀释曲线趋向平坦, 说明测序深度合理(图1).
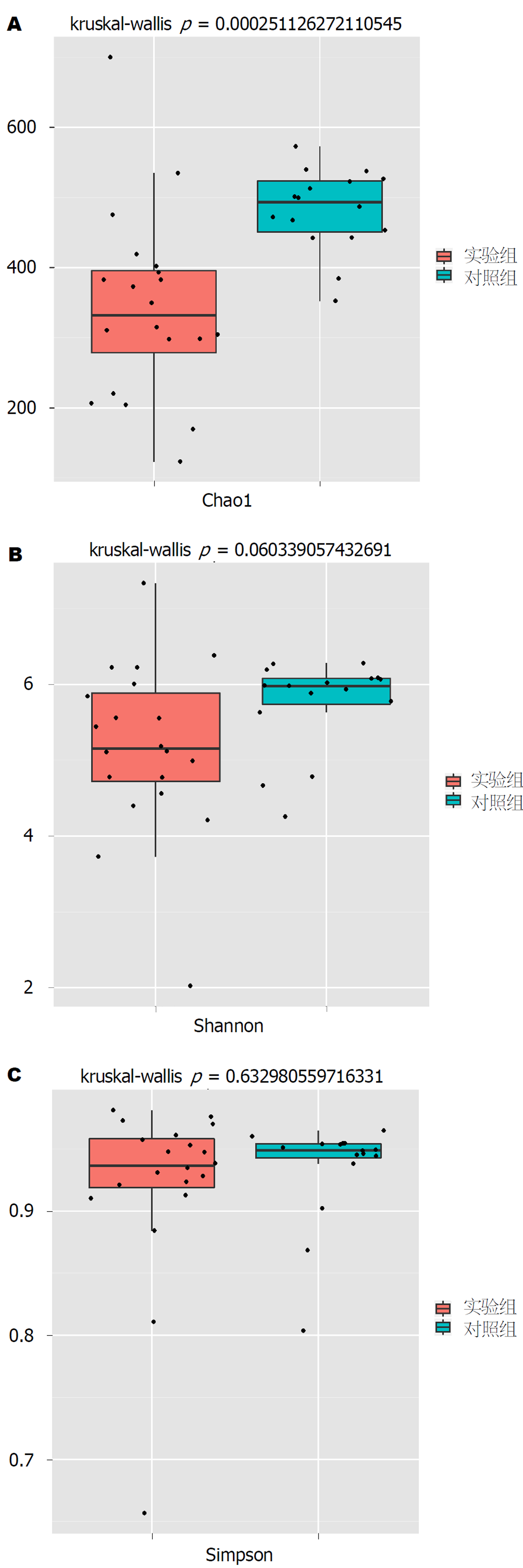
Alpha多样性是对样本中物种多样性的分析, 基于OTU种类和丰度计算Chao1、Shannon指数和Simpson指数. 实验组和对照组的各样本的覆盖度均大于99%, 说明本次测序所检测到的微生物物种能够覆盖样本中大多数的物种, 与Shannon指数稀释曲线的结果一致. Chao1指数反映样本中物种的丰度, 即OTU的数量. Chao1值越大代表物种总数越多, 表明丰富度越高. 如图2A, 实验组的Chao1指数为332.30, 对照组的Chao1指数为493.38, 实验组的物种丰富度较对照组显著降低(P<0.01), 说明藏、汉肝硬化患者肠道微生物物种丰度有显著差异.
Shannon和Simpson指数用于反映群落的多样性, 包括物种丰度和物种均匀度, Shannon越大和Simpson指数越小, 样品中的物种多样性越高. 结果显示, 实验组的Shannon指数中值为5.15略低于对照组的5.98, 但差异并不显著(P = 0.060). Simpson指数中值实验组与对照组分别为0.936与0.949, 差异无统计学意义(P = 0.632)(图2B、C).
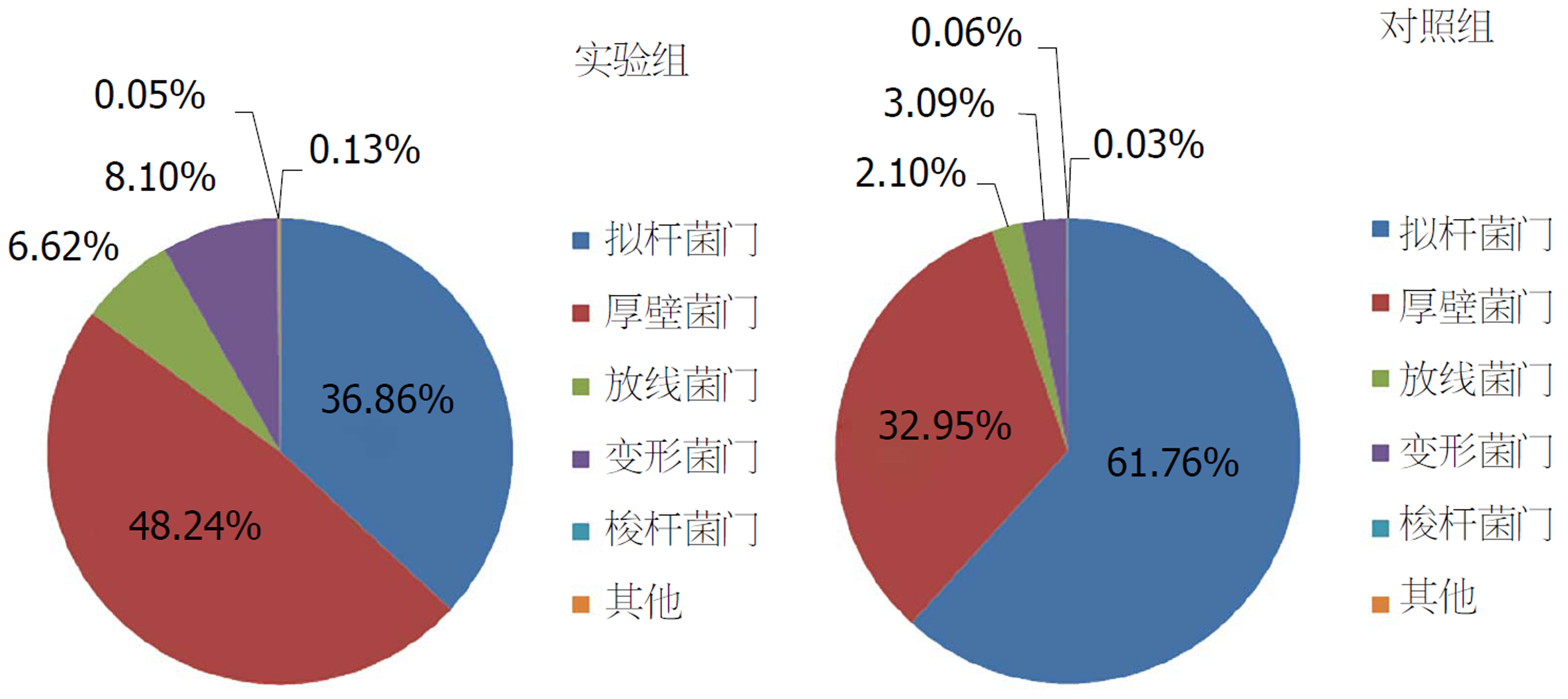
与Ribosomal Database Project数据库进行相似性比对以及物种注释, 在门和属两个分类层次下对各个样品的物种丰度进行统计. 在门水平下, 两组均以拟杆菌门和厚壁菌门的丰度最高, 其中拟杆菌门对照组与实验组丰度百分比分别占到61.76%和36.86%, 实验组显著减少(P<0.001); 厚壁菌门分别占到32.95%和48.24%, 实验组显著增加(P<0.01); 其次, 丰度较高的是放线菌门对照组与实验组分别是2.10%和6.62%, 实验组增加(P<0.05). 同样丰度较高的变形菌门、梭杆菌门实验组分别占比为3.09%和0.06%, 对照组为8%和0.05%, 但两组比较差异均不显著(P>0.05)(图3).
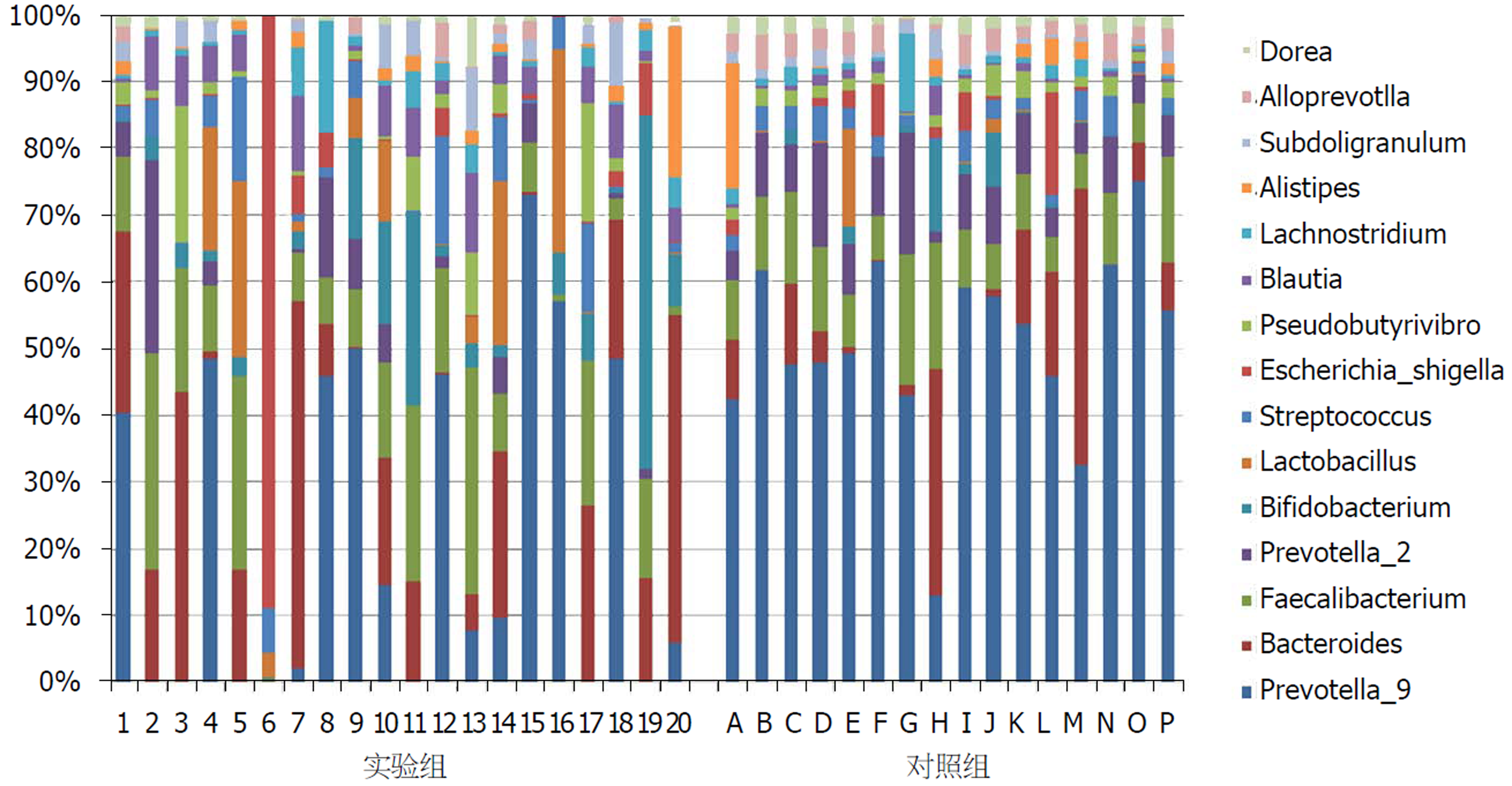
在属水平上, 对照组和实验组的top15优势菌主要有普氏菌属_9、拟杆菌属、粪杆菌属、普氏菌属_2、双歧杆菌属、乳杆菌属、链球菌属、志贺氏埃希菌属、假丁酸弧菌属、布劳特氏菌属、Lachnoclostridium、另枝菌属、Subdoligranulum、拟普雷沃菌属、长奈瑟氏菌. 如图4, 实验组与对照组的肠道微生物组成由明显差异, 普氏菌属_9为对照组的主要优势菌, 而实验组的菌群组成更加复杂. 与对照组相比, 实验组葡萄球菌、放线菌明显减少, 差异显著(P<0.01); 乳杆菌属明显增加, 差异显著(P<0.01).
肝脏与消化道毗邻, 因其特殊的解剖位置, 可形成所谓的肝-肠轴, 肝脏因此成为抵御抗原、细菌和细菌产物等的第一道防线. 大量研究已充分证实在肝硬化患者中明显存在肠道菌群失调的现象, 主要表现为固有定植菌的减少, 非固有定植菌的增多, 其中以厌氧菌及革兰氏阴性菌杆菌增多更为明显[1-4]. 然而, 肠道菌群失调与肝硬化之间是否存在因果关系及其具体病理生理机制目前仍不清楚. 有研究推测肠道菌群失调可通过改变胆汁酸生成, 增加肠道黏膜通透性, 引起细菌移位及小肠细菌过度生长等等方式共同参与肝硬化及其并发症的发病过程[5,6]. 如肠道菌群失调参与细菌移位、内毒素血症、甚至肝性脑病、自发性细菌性腹膜炎等的发生. 有研究报道证实肝性脑病患者中肠道微生物平衡失调可导致细菌抗原移位, 从而促进炎症反应的发生, 损伤宿主肠道黏膜屏障的抵御机制, 参与门静脉高压、肝性脑病等的发病机制[5,7]. 还有研究发现随着肝脏疾病恶化, 肝硬化患者的肠道菌群也会随之改变, 其中微生物基因组中丰度较高的细菌种属与疾病的严重性有明显关系[8].
高通量Illumina测序可检测不可人工培养的肠道细菌, 测序快速且结果准确度高, 可全面反应复杂样品的微生物群落的组成, 是目前微生物多样性研究的重要技术手段之一[9]. 本研究采用该技术对16S rDNA的V4可变区进行测序, 首次开展藏族肝硬化人群肠道细菌菌落组成分析. 研究对20例藏族肝硬化患者(实验组)和16例汉族肝硬化患者(对照组)粪便样本中微生物的多样性和群落结构进行了初步分析, Chao1指数在实验组和对照组之间有显著差异(P<0.05), 说明种族会对肝硬化患者的微生物丰度产生影响, 藏族肝硬化患者的微生物丰度相较于汉族肝硬化患者降低. 物种丰度统计分析结果显示, 在门水平上, 两组的微生物种类主要有拟杆菌门、厚壁菌门、放线菌门、变形菌门、梭杆菌门, 其中拟杆菌门在实验组中显著减少(P<0.001), 而厚壁菌门和放线菌门在实验组中增加(P<0.01和P<0.05); 在属水平上, 两组的前15微生物种类主要有普氏菌属_9、拟杆菌属、粪杆菌属、普氏菌属_2、双歧杆菌属、乳杆菌属、链球菌属、志贺氏埃希菌属、假丁酸弧菌属、布劳特氏菌属、Lachnoclostridium、另枝菌属、Subdoligranulum、拟普雷沃菌属、长奈瑟氏菌, 其中葡萄球菌属、放线菌属在实验组中显著减少(P<0.01), 乳杆菌属显著增加(P<0.01).
实验组中拟杆菌门和厚壁菌门丰度百分比的显著改变将会对F/B值(厚壁菌门/拟杆菌门丰度比值)造成较大的影响, F/B是衡量肠道菌群失调的标准之一, F/B值的改变说明在藏族肝硬化患者和汉族肝硬化患者之间存在不同程度的肠道菌群失调, 将会对肝硬化的进一步发展以及并发症的发生造成不同程度的影响[10]. 藏族人群世居青藏高原, 独特的环境气候也使藏族人群形成了独特的饮食习惯. 在藏族人群的日常饮食中, 肉类(例如牛肉、羊肉等)和发酵类食品(例如酸奶)为主要食材, 而对于碳水化合物、蔬菜和水果的摄入较少[11]. 此外, 也有报道表明人种所带来的基因上的差异也是造成藏汉肠道微生物组成差异的原因之一[12]. 由此可见, 了解藏族肝硬化患者的肠道微生物组成将是建立肝硬化个性化治疗方案的重要基础, 有利于藏族肝硬化患者的治疗及预后的改善. 本研究利用16S rDNA测序技术初步探索了藏族肝硬化患者的肠道微生物组成特点, 为进一步了解藏族肝硬化患者的肝硬化的发展进程奠定了基础, 同时为失代偿性肝硬化的个性化治疗及预后的改善提供方向. 但基于本研究病例数较少, 不可避免地存在偏倚, 因此需要更多大样本、多中心、多层次的随机对照研究进一步证实.
肝硬化是我国最严重的公共卫生问题之一, 而在藏区这一疾病的高发性尤为突出. 大量研究已充分证实在肝硬化患者中明显存在肠道菌群失调的现象, 主要表现为固有定植菌的减少, 非固有定植菌的增多. 这为临床防治肝硬化及并发症提供了新的线索与思路. 揭示肝硬化患者肠道菌群的特征, 将有助于进一步认识肠道微生态与肝硬化的关系, 必将推进肠道微生态在肝硬化综合治疗中的地位. 由于藏族人群所生活的环境和饮食习惯及构成均不同于普通人群, 这些因素均参与人体肠道微生态的形成, 也必将导致藏族人群肠道菌群特征也不同于普通人群, 因此很有必要专门针对藏族人群肠道菌群特征进行针对性研究. 然而目前针对藏族人群肠道菌群特征, 特别是针对藏族肝硬化患者肠道菌群的研究, 国内国外均未见报道, 亟待相关研究填补空白. 因此本研究拟针对藏族肝硬化肠道微生态进行针对性研究, 旨在揭示藏族人群中肠道菌群特征与肝硬化及相关并发症的关系.
进一步认识肠道微生态与肝硬化的关系, 为临床防治藏族肝硬化及并发症提供新的线索与思路.
对比藏、汉族肝硬化患者肠道菌群的差异, 探讨可能的内在联系.
收集纳入受试者新鲜粪便, 迅速于-80 ºC度冰箱保存, 同时收集受试者临床相关资料. 16SRNA测序及数据分析(高通量测序): (1)肠道微生物DNA提取; (2)荧光定量PCR反应; (3)PCR产物凝胶电泳; (4)PCR产物混合及纯化; (5)测序分析: 将纯化的PCR混合产物使用Illumina Miseq测序平台行双端测序, 并根据测序结果行相应的生物数据统计分析. 测序技术可以在不需要培养的条件下进行混合细菌的鉴定, 特别是一些目前还无法培养的细菌可通过测序分析, 极大丰富了肠道微生物的数据库. 特别是高通量是测序技术, 针对大样本的研究, 只要能够提供满足测序要求的DNA, 就可以对样本中可能存在的微生物进行分析.
本研究达到了试验目标, 结果显示实验组的Chao1指数为332.30, 对照组的Chao1指数为493.38, 实验组的物种丰富度较对照组显著降低(P<0.01), 说明藏、汉肝硬化患者肠道微生物物种丰度有显著差异. 在反映群落的多样性的Shannon、Simpson指数上, 两组对比差异无统计学意义. 基于97%的相似性进行物种注释, 对实验组和对照组进行微生物物种丰度及构成比上, 门水平上实验组中拟杆菌门显著减少(P<0.001), 厚壁菌门和放线菌门在实验组中增加(P<0.01和P<0.05); 在属水平上, 葡萄球菌属、放线菌属在实验组中显著减少(P<0.01), 乳杆菌属显著增加(P<0.01). 说明藏、汉族肝硬化患者的肠道微生物肠道微生物构成比存在着差异. 上述发现, 填补了目前有关藏族肝硬化患者肠道菌群特征的空白.
本研究发现藏族肝硬化患者的肠道微生物丰度明显低于汉族肝硬化患者, 肠道微生物组成存在着差异. 推测可能与高原环境、饮食习惯及基因差异等有关. 这些差异可能参与了肝硬化疾病的发展, 如果能进一步明确这些有差异的微生物的功能与代谢机制, 阐明微生物与疾病的内在联系, 可为肝硬化的个性化治疗提供可行方案.
本研究首次展现了藏族肝硬化患者的肠道微生物组成特点, 下一步可探究微生物的功能与代谢机制, 进一步探讨微生物与疾病的内在联系, 为肝硬化的个性化治疗, 提供更详实的理论基础及可行方案.
学科分类: 胃肠病学和肝病学
手稿来源地: 四川省
同行评议报告分类
A级 (优秀): 0
B级 (非常好): B
C级 (良好): C, C
D级 (一般): D, D
E级 (差): 0
编辑: 王禹乔 电编:刘继红
| 1. | Bajaj JS, Hylemon PB, Ridlon JM, Heuman DM, Daita K, White MB, Monteith P, Noble NA, Sikaroodi M, Gillevet PM. Colonic mucosal microbiome differs from stool microbiome in cirrhosis and hepatic encephalopathy and is linked to cognition and inflammation. Am J Physiol Gastrointest Liver Physiol. 2012;303:G675-G685. [PubMed] [DOI] |
| 2. | Bajaj JS, Ridlon JM, Hylemon PB, Thacker LR, Heuman DM, Smith S, Sikaroodi M, Gillevet PM. Linkage of gut microbiome with cognition in hepatic encephalopathy. Am J Physiol Gastrointest Liver Physiol. 2012;302:G168-G175. [PubMed] [DOI] |
| 3. | Qin N, Yang F, Li A, Prifti E, Chen Y, Shao L, Guo J, Le Chatelier E, Yao J, Wu L, Zhou J, Ni S, Liu L, Pons N, Batto JM, Kennedy SP, Leonard P, Yuan C, Ding W, Chen Y, Hu X, Zheng B, Qian G, Xu W, Ehrlich SD, Zheng S, Li L. Alterations of the human gut microbiome in liver cirrhosis. Nature. 2014;513:59-64. [PubMed] [DOI] |
| 4. | Tuomisto S, Pessi T, Collin P, Vuento R, Aittoniemi J, Karhunen PJ. Changes in gut bacterial populations and their translocation into liver and ascites in alcoholic liver cirrhotics. BMC Gastroenterol. 2014;14:40. [PubMed] [DOI] |
| 5. | Macnaughtan J, Jalan R. Clinical and pathophysiological consequences of alterations in the microbiome in cirrhosis. Am J Gastroenterol. 2015;110:1399-410; quiz 1411. [PubMed] [DOI] |
| 6. | Wiest R, Lawson M, Geuking M. Pathological bacterial translocation in liver cirrhosis. J Hepatol. 2014;60:197-209. [PubMed] [DOI] |
| 7. | Gómez-Hurtado I, Such J, Francés R. Microbiome and bacterial translocation in cirrhosis. Gastroenterol Hepatol. 2016;39:687-696. [PubMed] [DOI] |
| 8. | Bajaj JS, Heuman DM, Hylemon PB, Sanyal AJ, White MB, Monteith P, Noble NA, Unser AB, Daita K, Fisher AR, Sikaroodi M, Gillevet PM. Altered profile of human gut microbiome is associated with cirrhosis and its complications. J Hepatol. 2014;60:940-947. [PubMed] [DOI] |
| 9. | Woo PC, Lau SK, Teng JL, Tse H, Yuen KY. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories. Clin Microbiol Infect. 2008;14:908-934. [PubMed] [DOI] |
| 10. | Acharya C, Bajaj JS. Gut Microbiota and Complications of Liver Disease. Gastroenterol Clin North Am. 2017;46:155-169. [PubMed] [DOI] |