修回日期: 2017-01-27
接受日期: 2017-02-13
在线出版日期: 2017-03-18
探讨miR-944在食管鳞癌(esophageal squamous cell carcinoma, ESCC)组织中的表达与临床意义及其对食管癌细胞Eca109增殖和迁移能力的影响.
收集36例经病理确诊的ESCC患者手术切除癌及癌旁组织, 采用实时荧光定量PCR(real-time quantitative PCR, qRT-PCR)检测癌组织和癌旁组织中miR-944表达, 并分析miR-944在不同临床病理资料之间的差异. 脂质体法转染miR-944 mimics、inhibitors、无关对照序列(negative control, NC)到Eca109细胞, qRT-PCR检测各组miR-944表达水平, MTT法检测转染后24、48、72、96 h增殖能力, Transwell侵袭实验检测细胞侵袭能力.
miR-944在ESCC组织表达水平显著高于癌旁组织(P<0.01). 同时, miR-944在癌症组织中的表达水平随患者TNM分期的升高而升高(P<0.01), 而淋巴结转移阳性组miR-944表达水平显著高于阴性组(P<0.01). 与NC组相比, miR-944 mimics和inhibitors转染组miR-944表达水平显著升高和细胞增殖能力明显均增强, 而inhibitors组均受到抑制(P<0.01).
miR-944在ESCC组织表达上调, 并与ESCC TNM分期和淋巴结转移相关, 其可能通过促进癌细胞的增殖和侵袭能力参与ESCC的发生与发展.
核心提要: miR-944是新近发现与肿瘤发展密切相关的微小RNA, 在肺癌、宫颈癌、乳腺癌均检测到高表达. 通过对食管鳞癌组织中miR-944表达的检测及其与不同临床资料关系的分析, 推测miR-944在食管癌进程中发挥促癌基因作用, 可能成为抗食管癌的重要靶点.
引文著录: 邓鑫, 罗茂. miR-944在食管鳞癌中的表达及其对Eca109细胞增殖、侵袭的影响. 世界华人消化杂志 2017; 25(8): 684-690
Revised: January 27, 2017
Accepted: February 13, 2017
Published online: March 18, 2017
To detect the expression of miRNA-944 in esophageal squamous cell carcinoma (ESCC), and to explore its role in cell proliferation and migration in human esophageal cancer cell line Eca109.
ESCC and matched tumor adjacent noncancerous tissue samples were obtained from 36 patients who underwent surgical treatment and were pathologically diagnosed with ESCC. Real-time quantitative PCR (qRT-PCR) was used to detect the expression levels of miRNA-944, and the relationship between miRNA-944 and clinical and pathological parameters were then analyzed. Eca109 cells were transfected with miR-944 mimic, inhibitor and negative control using LipofectamineTM2000, and then the expression level of miR-944 was detected by qRT-PCR. Cell proliferation and invasion were assessed by MTT assay and transwell assay, respectively.
The expression level of miR-944 in ESCC tissues was significantly higher than that in tumor adjacent non-cancerous tissues (P < 0.01).The up-regulation of miR-944 expression in ESCC was correlated with advanced TNM stage (P < 0.01) and lymph node metastasis (P < 0.01). Compared to control cells, transfection of miR-944 mimic and inhibitor up- and down-regulated miR-944 expression in Eca109 cells, respectively (P < 0.01). Furthermore, transfection of miR-944 mimic enhanced cell proliferation and invasion, while transfection of miR-944 inhibitor inhibited cell proliferation and invasion (P < 0.01).
The expression of miR-944 is up-regulated in ESCC and associated with TNM stage and lymph node metastasis, indicating that miR-944 may facilitate ESCC occurrence possibly by promoting the proliferation and invasion of ESCC cells.
- Citation: Deng X, Luo M. Expression of miR-944 in esophageal squamous cell carcinoma and its role in cell proliferation and invasion in human esophageal carcinoma cell line Eca109. Shijie Huaren Xiaohua Zazhi 2017; 25(8): 684-690
- URL: https://www.wjgnet.com/1009-3079/full/v25/i8/684.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v25.i8.684
食管鳞癌(esophageal squamous cell carcinoma, ESCC)是我国高发的消化系恶性肿瘤, 严重威胁人类健康[1,2]. 而肿瘤细胞的侵袭和转移是食管癌术后患者致死的主要原因[3,4]. 微小RNA(microRNA, miRNA)是一类内源性的、19-25个碱基长度的小分子非编码RNA, 他可以通过与靶mRNA的3'-UTR区互补导致其降解或抑制蛋白翻译, 从而在人体生命活动中具有广泛的调控作用[5-7]. 近年研究表明, 部分miRNA广泛参与ESCC的疾病进程, 发挥促进/抑制肿瘤发生的作用. 如He等[8]研究表明miR-143-3p可通过靶向RNA结合蛋白QKI-5在ESCC中发挥抑癌作用; Zhang等[9]研究表明miR-644a在ESCC组织中表达降低, 促进ESCC细胞的侵袭性和干细胞样特性. MiR-944是新近发现与肿瘤相关的miRNA, 首次在人宫颈癌细胞中发现, 位于肿瘤p63基因的内含子, 并定位于人染色体3q28[10]. 目前国内外对其功能的研究还比较少, 至今未见miR-944在ESCC的表达情况、患者临床病理特征相关性、肿瘤细胞生物学功能影响的研究报道. 有研究证实miR-944表达上调与肺癌[11]、宫颈癌[12]、乳腺癌[13]发生发展有密切关系. 本研究通过检测ESCC患者癌组织中的miR-944表达, 分析其在不同临床资料之间的表达差异; 同时利用合成的miR-944 mimic和inhibitor转染食管癌Eca109细胞, 探讨和分析miR-944对食管癌细胞增殖和侵袭能力的影响.
收集2016-06/2016-10西南医科大学附属第一医院心胸外科36例经病理确诊的ESCC患者, 所有病例均经病理确诊为单一鳞状细胞癌, 患者术前未经放疗、化疗, 无其他肿瘤史. 取手术切除的新鲜食管癌组织及相应癌旁组织(距癌组织>5 cm, 经病理证实无肿瘤细胞), 立即用液氮速冻后置于-80 ℃冰箱保存备用. 其中男性20例, 女性16例; 年龄为46-73岁, 平均年龄为59.25岁±7.43岁; 高中分化27例, 低分化9例; 淋巴结转移阳性21例, 淋巴结转移阴性15例; 肿瘤位于胸上段6例, 胸中段20例, 下段10例; TNM分期Ⅰ期5例, Ⅱ期18例, Ⅲ期7例, Ⅳ期6例. 肿瘤分化程度和分期参照国际抗癌联盟第7版食管癌TNM分期标准. 本研究经西南医科大学附院第一医院伦理委员会批准, 全部调查和取样均征得受试者同意并签署知情同意书.
人食管癌细胞株Eca109由西南医科大学附属第一医院医学实验中心保存; RPMI 1640培养基、胎牛血清购自Gibco BRL公司; LipofectamineTM2000、TRIzol购自Invitrogen公司; MTT购自Sigma公司; Matrigel购自BD公司; Transwell板购自Corning公司; Mir-XTM miRNA first-strand synthesis和SYBR® RT-qPCR试剂盒购自Clontech公司; miR-944 mimics(5'-AAAUUAUUGUACAUCGGAUGAG-3'), miR-944 inhibitors(5'-CUCAUCCGAUGUACAAUAAUUU-3'), miRNA NC对照(5'-AAGAUGUCAAUUCGAGUAAUUG-5'), 由上海生工生物有限公司合成.
1.2.1 细胞培养: 液氮罐中取出Eca109细胞, 37 ℃水浴锅复苏, 使用10%FBS RPMI 1640培养基(1×105 U/L青霉素、100 mg/L链霉素)培养, 置于5%CO2、37 ℃饱和湿度培养箱中常规培养.
1.2.2 miR-944转染: 转染前24 h, 取对数生长期Eca109(1×105)铺于6孔板中, 待生长至60%-70%汇合时, 更换无血清培养基后同步进行miR-944 mimics(50 nmol/L)、miR-944 inhibitors(100 nmol/L)及其对应无关对照序列(negative control, NC)的转染. 按照lipofectamine2000转染试剂说明书进行, 转染12 h后更换新鲜培养基, 24 h收集细胞提取总RNA.
1.2.3 RT-qPCR检测食管癌组织和Eca109细胞中miR-944表达: 称取食管癌和癌旁各100 mg, 加入1 ml TRIzol充分裂解; 胰酶消化离心收集各组细胞, 每组加入1 ml TRIzol, 反复吹打裂解. 按TRIzol试剂盒步骤提取组织和各实验组的细胞总RNA, 并分别应用Mir-XTM miRNA first-strand synthesis and SYBR® RT-qPCR试剂盒和PrimeScriptTM RT reagent kit with gDNA eraser (Perfect Real Time)试剂盒制备cDNA模板. 以U6为内参检测miR-944的达水平. miR-944上游引物为: 5'-GCGGCGGAAATTATTGTACATC-3', 下游引物为: 5'-ATCCAGTGCAGGGTCCGAGG-3'; U6上游引物为: 5'-CTCGCTTCGGCAGCACA-3', 下游引物为: 5'-AACGCTTCACGAATTTGCGT-3'(引物序列由上海生工合成). 应用SYBR Premix Ex Taq Ⅱ (TaKaRa)进行qPCR检测. 扩展条件为95 ℃预变性10 min; 95 ℃变性10 s, 50-60 ℃复性20 s, 72 ℃延伸30 s, 共进行40个循环, 72 ℃延伸3 min后, 60 ℃到95 ℃绘制融解曲线. 应用2-△△CT±SE法计算miR-944的表达量, 设置6个复孔.
1.2.4 MTT检测细胞增殖: 取对数生长期Eca109细胞, 按3×103个细胞/孔接种于96孔, 接种12 h后, 根据实验分组用Lipofectamine 2000脂质体转染试剂按说明书步骤进行转染. 转染后24、48、72、96 h, 每孔加入20 μl MTT, 37 ℃孵育4 h后弃上清, 加入150 μl DMSO, 轻微振荡10 min, 490 nm波长下检测各孔吸光度(A)值, 每组设6个复孔, 实验重复3次.
1.2.5 Transwell实验: 实验步骤按照Corning公司Transwell(24孔板)小室说明书进行, Transwell小室加入细胞悬液(200 μl培养液/孔, 1×105细胞). 下室加入500 μl含FBS的培养基, 常规培养48 h, 结晶紫染色, 显微镜观察和拍照, 随机选5个视野, 计数取平均侵袭细胞数.
统计学处理 采用SPSS17.0统计学软件对数据进行分析. 以P<0.05表示差异有统计学意义. 用独立样本的t检验分析比较miR-944在食管癌和癌旁组织中的表达差异; miR-944表达水平与食管癌临床病理参数间的关系用非参数检验-两独立样本的秩和检验进行分析, 多组间比较采用Kruskal-Wallis test分析.
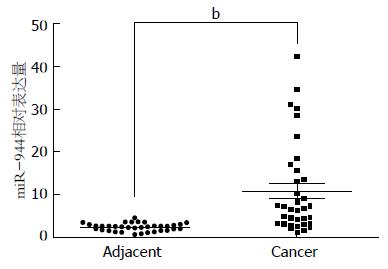
miR-944在36例ESCC组织中的相对表达量显著高于癌旁组织, 差异有统计学意义(P<0.001).
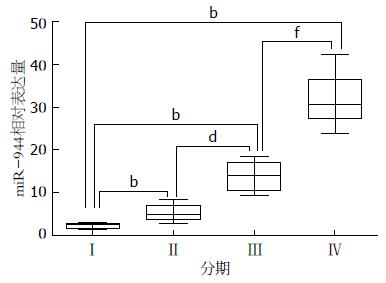
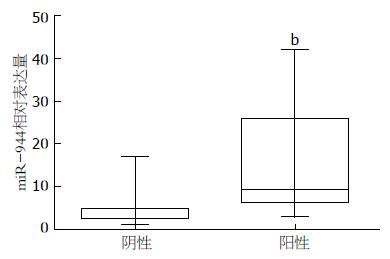
分析ESCC组织中miR-944与临床病理资料关系, 发现miR-944表达与TNM分期和淋巴结转移发生与否相关, 如图1和图2显示: miR-944表达随TNM分期升高(P<0.01); 而图3显示: 淋巴结转移阳性组miR-944表达明显高于淋巴结转移阴性组(P<0.01). miR-944表达和年龄、性别、肿瘤位置、组织分化程度无关(表1).
| 临床病理资料 | n | miR-944相对表达量 | P值 |
| 性别 | 0.858 | ||
| 男 | 20 | 11.841±11.637 | |
| 女 | 16 | 9.482±9.241 | |
| 年龄(岁) | 0.601 | ||
| ≤60 | 18 | 12.239±12.123 | |
| >60 | 18 | 9.609±9.206 | |
| 肿瘤位置 | 0.995 | ||
| 上段 | 6 | 11.385±12.400 | |
| 中段 | 20 | 12.405±12.326 | |
| 下段 | 10 | 7.683±4.661 | |
| 组织分化程度 | 1.000 | ||
| 高中分化 | 27 | 11.372±11.658 | |
| 低分化 | 9 | 9.577±7.464 | |
| TNM分期 | <0.001 | ||
| Ⅰ期 | 5 | 2.115±0.670 | |
| Ⅱ期 | 18 | 5.189±1.809b | |
| Ⅲ期 | 7 | 14.033±3.414 | |
| Ⅳ期 | 6 | 31.840±6.346 | |
| 淋巴结转移 | <0.001 | ||
| 有 | 21 | 15.175±11.944 | |
| 无 | 15 | 4.971±4.114 |
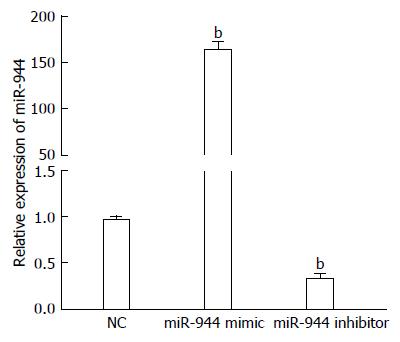
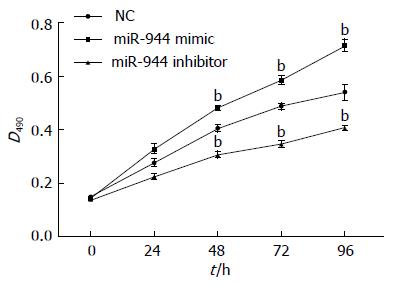
应用miR-944 mimics, inhibitors和NC直接转染Eca109 24 h后收集细胞并提取总RNA. 经逆转录制备各组cDNA模板后qPCR实验检测表明, 与NC组比较, 转染miR-944 mimics和inhibitors能够分别显著促进和抑制miR-944的表达(miR-944 mimics组 or inhibitors组 vs NC, P<0.05; miR-944 mimics组 or inhibitors组 vs NC组, P<0.01), 该步骤转染细胞可进行下一步实验(图4).
MTT法分析细胞增殖能力变化, 以Eca109细胞在不同时间点的光密度值绘制细胞生长曲线, 结果显示, miR-944 mimics组相比NC组显著升高(P<0.01), miR-944 inhibitors组相比NC组显著降低(P<0.01, 图5).
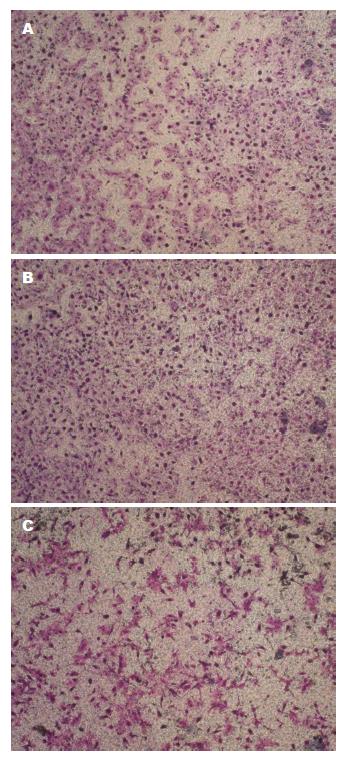
Tanswell实验检测细胞侵袭能力改变, 用Matrigel胶模仿基底膜检测肿瘤细胞侵袭能力, 结果显示miR-944 mimics组视野平均侵袭细胞个数显著高于NC组(156.2±13.6 vs 81.50±11.31), miR-944 inhibitors组视野平均侵袭细胞个数显著低于NC组(51.8±12.5 vs 81.50±11.31), 差异有显著性(P<0.05, 图6).
miRNAs异常表达是ESCC等肿瘤的重要特征, 目前已有诸多研究报道揭示ESCC中的miRNAs异常表达[14-16]. 如Takeshita等[17]研究发现ESCC患者血清中miR-1246、miR-3202、miR-23a、miR-718、miR-3610、miR-4271高表达, 而miR-144、miR-106b、miR-486-5p、miR-93、miR-451低表达; Fu等[18]利用miRNA芯片技术分析34例中国淮安地区ESCC患者癌组织发现miR-1、miR-29c、miR-100、miR-133a、miR-133b、miR-143、miR-145、miR-195低表达, 而miR-7、miR-21、miR-223、miR-1246高表达, 并结合生物信息学分析发现miR-29c、miR-100、miR-133a、miR-133b可能与ESCC侵袭转移相关, miR-7和miR-21可能与ESCC分化相关. 新近研究发现其与多种肿瘤密切相关, 如肺癌[11]、乳腺癌[13]、宫颈癌[12], 并显示其与某些肿瘤的组织学分期和化疗耐药性发生相关. 但尚无相关研究揭示miR-944在ESCC中的表达及其对癌细胞的增殖和侵袭能力的影响.
本研究通过实时荧光定量PCR技术分析了36例ESCC组织及对应癌旁正常组织中miR-944表达情况, 发现癌组织中miR-944表达显著高于癌旁正常组织, 提示miR-944可能参与了ESCC的发生发展过程. 同时, 在其他肿瘤中也有发现miR-944的表达升高. 如Powrózek等[11]研究显示早期肺癌患者联合检测血浆miR-944和miR-3662表达升高水平具有潜在的诊断价值; Xie等[12]研究显示宫颈癌组织和宫颈癌细胞系中miR-944表达显著升高. 这些研究表明miR-944主要在肿瘤疾病进程中发挥促癌作用, 并可能参与ESCC的病理生理过程.
大量研究揭示miRNA表达水平与ESCC患者临床病理资料密切相关[19,20]. 如Chen等[21]分析107例ESCC患者临床资料发现ESCC组织中miR-92a表达升高水平与TNM分期和淋巴结转移发生与否正相关, 并且miR-92a表达上调患者预后往往较差; 而Wu等[22]通过miRNA基因芯片技术发现miR-143和miR-145表达水平在ESCC组织中显著降低, 其表达水平与肿瘤浸润程度相关. 本研究通过进一步分析ESCC患者不同临床病理资料组之间miR-944表达差异, 发现miR-944表达水平与TNM分期和淋巴结转移相关. MiR-944表达水平随ESCC患者TNM分期增加呈逐渐上升趋势, 同时淋巴结转移阳性患者癌组织中的表达高于转移阴性患者. 而在非小细胞癌中Ma等[23]同样发现miR-944表达显著上调, 并与肿瘤分期相关. 以上研究结果提示miR-944可能参与调控ESCC肿瘤细胞的增殖和侵袭转移过程. 此外, 本研究结果显示miR-944表达情况与患者的年龄、性别、肿瘤位置、组织分化程度均无关.
细胞的增殖能力与肿瘤发生密切相关, 而肿瘤的侵袭能力对肿瘤浸润扩散、远处转移又起关键作用[24-27]. 研究[18,28]发现多种miRNAs在肿瘤环境下表达异常, 参与调控肿瘤细胞的增殖和侵袭能力等. 如Xie等[12]研究显示miR-944在宫颈癌中高表达, 可抑制靶基因HECW2和S100PBP表达, 促进宫颈癌细胞增殖和迁移. 相反, 在一些肿瘤中也有报道miR-944的抑癌作用. 如Flores-Pérez等[29]最新研究发现乳腺癌中miR-944低表达, 可上调靶标基因SIAH1和PTP4A1表达, 促进乳腺癌细胞MDA-MB-231的迁移和侵袭; Liu等[30]最近报道miR-944在非小细胞肺癌中低表达, 并通过调控靶基因EPHA7发挥促进癌细胞增殖的作用. 本研究体外实验证明上调/下调Eca109细胞中miR-944表达可分别促进和抑制细胞的增殖与侵袭能力, 表明miR-944可通过调控癌细胞增殖与侵袭从而参与ESCC的发生发展. 因此, 深入研究miR-944在ESCC中的分子机制, 有助于明确miR-944参与肿瘤疾病进程的机制.
本研究发现miR-944在食管癌组织中表达升高, 其表达水平与淋巴结转移、TNM分期等因素相关, 可能用于预测ESCC恶性程度及淋巴转移发生. MiR-944发挥促癌作用可能是通过调控下游靶基因表达从而促进Eca109细胞的增殖和侵袭能力, 但其具体机制需进一步的实验探究和证明.
微小RNA(micro-RNA, miRNA)往往在肿瘤组织表达异常, 同时诸多研究发现异常miRNA对肿瘤细胞的增殖和侵袭能力有重要的调节作用. 多项研究发现, miRNAs在一些肿瘤中异常表达, 提示miRNA可能参与肿瘤的发生发展过程.
miR-944在食管癌中表达下调, 但miR-944在食管癌中的具体作用仍然不明确.
新近研究发现miR-944与部分肿瘤密切相关, 如肺癌、宫颈癌、乳腺癌均呈现高表达. 不仅在肿瘤组织中表达异常, 而且还显示其参与某些肿瘤的生物学行为, 具体调控肿瘤基因表达.
本文首次检测到miR-944在食管癌组织高表达, 并与肿瘤分期和转移发生相关. 利用合成的miR-944mimic和inhibitor转染食管癌Eca109细胞, 发现miR-944可调控食管癌Eca109细胞增殖和侵袭能力.
MiR-944的显著升高与肿瘤分期和淋巴转移相关, 可为临床诊断提供切实的数据支撑. 未来通过更大规模的样本收集和病理数据分析研究, 可进一步明确其在临床实际运用价值.
耿明, 主任医师, 济南军区总医院病理科; 江建新, 教授, 主任医师, 湖北省肿瘤医院肝胆胰腺外科; 李欣, 教授, 承德医学院基础医学院
本文在国内研究中首次探讨了miR-944在食管癌中的表达及其对食管癌细胞增殖和侵袭的影响, 创新性较强, 实验设计基本合理. 此外, 手稿的科学性、创新性和可读性能较好地反映我国临床和基础研究的水平.
手稿来源: 自由投稿
学科分类: 胃肠病学和肝病学
手稿来源地: 四川省
同行评议报告分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): C, C, C
D级 (一般): 0
E级 (差): 0
编辑: 马亚娟 电编:胡珊
| 2. | 陈 洁君, 周 余春, 刘 红建, 陈 甜甜, 袁 子宇, 蒋 跃明, 陈 兴栋, 吕 明. 江苏省泰兴市2003-2010年食管癌发病趋势分析. 复旦学报(医学版). 2014;41:22-26. |
| 4. | Napier KJ, Scheerer M, Misra S. Esophageal cancer: A Review of epidemiology, pathogenesis, staging workup and treatment modalities. World J Gastrointest Oncol. 2014;6:112-120. [PubMed] [DOI] |
| 5. | Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM. Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs. Nature. 2005;433:769-773. [PubMed] [DOI] |
| 6. | Burroughs AM, Ando Y, de Hoon MJ, Tomaru Y, Nishibu T, Ukekawa R, Funakoshi T, Kurokawa T, Suzuki H, Hayashizaki Y. A comprehensive survey of 3' animal miRNA modification events and a possible role for 3' adenylation in modulating miRNA targeting effectiveness. Genome Res. 2010;20:1398-1410. [PubMed] [DOI] |
| 7. | Kreth S, Limbeck E, Hinske LC, Schütz SV, Thon N, Hoefig K, Egensperger R, Kreth FW. In human glioblastomas transcript elongation by alternative polyadenylation and miRNA targeting is a potent mechanism of MGMT silencing. Acta Neuropathol. 2013;125:671-681. [PubMed] [DOI] |
| 8. | He Z, Yi J, Liu X, Chen J, Han S, Jin L, Chen L, Song H. MiR-143-3p functions as a tumor suppressor by regulating cell proliferation, invasion and epithelial-mesenchymal transition by targeting QKI-5 in esophageal squamous cell carcinoma. Mol Cancer. 2016;15:51. [PubMed] [DOI] |
| 9. | Zhang JX, Chen ZH, Xu Y, Chen JW, Weng HW, Yun M, Zheng ZS, Chen C, Wu BL, Li EM. Downregulation of MicroRNA-644a Promotes Esophageal Squamous Cell Carcinoma Aggressiveness and Stem Cell-like Phenotype via Dysregulation of PITX2. Clin Cancer Res. 2017;23:298-310. [PubMed] [DOI] |
| 10. | Lui WO, Pourmand N, Patterson BK, Fire A. Patterns of known and novel small RNAs in human cervical cancer. Cancer Res. 2007;67:6031-6043. [PubMed] [DOI] |
| 11. | Powrózek T, Krawczyk P, Kowalski DM, Winiarczyk K, Olszyna-Serementa M, Milanowski J. Plasma circulating microRNA-944 and microRNA-3662 as potential histologic type-specific early lung cancer biomarkers. Transl Res. 2015;166:315-323. [PubMed] [DOI] |
| 12. | Xie H, Lee L, Scicluna P, Kavak E, Larsson C, Sandberg R, Lui WO. Novel functions and targets of miR-944 in human cervical cancer cells. Int J Cancer. 2015;136:E230-E241. [PubMed] [DOI] |
| 13. | He H, Tian W, Chen H, Jiang K. MiR-944 functions as a novel oncogene and regulates the chemoresistance in breast cancer. Tumour Biol. 2016;37:1599-1607. [PubMed] [DOI] |
| 16. | Harada K, Baba Y, Ishimoto T, Shigaki H, Kosumi K, Yoshida N, Watanabe M, Baba H. The role of microRNA in esophageal squamous cell carcinoma. J Gastroenterol. 2016;51:520-530. [PubMed] [DOI] |
| 17. | Takeshita N, Hoshino I, Mori M, Akutsu Y, Hanari N, Yoneyama Y, Ikeda N, Isozaki Y, Maruyama T, Akanuma N. Serum microRNA expression profile: miR-1246 as a novel diagnostic and prognostic biomarker for oesophageal squamous cell carcinoma. Br J Cancer. 2013;108:644-652. [PubMed] [DOI] |
| 18. | Fu HL, Wu DP, Wang XF, Wang JG, Jiao F, Song LL, Xie H, Wen XY, Shan HS, Du YX. Altered miRNA expression is associated with differentiation, invasion, and metastasis of esophageal squamous cell carcinoma (ESCC) in patients from Huaian, China. Cell Biochem Biophys. 2013;67:657-668. [PubMed] [DOI] |
| 19. | Komatsu S, Ichikawa D, Kawaguchi T, Takeshita H, Miyamae M, Ohashi T, Okajima W, Imamura T, Kiuchi J, Arita T. Plasma microRNA profiles: identification of miR-23a as a novel biomarker for chemoresistance in esophageal squamous cell carcinoma. Oncotarget. 2016;7:62034-62048. [PubMed] [DOI] |
| 20. | Hemmatzadeh M, Mohammadi H, Karimi M, Musavishenas MH, Baradaran B. Differential role of microRNAs in the pathogenesis and treatment of Esophageal cancer. Biomed Pharmacother. 2016;82:509-519. [PubMed] [DOI] |
| 21. | Chen ZL, Zhao XH, Wang JW, Li BZ, Wang Z, Sun J, Tan FW, Ding DP, Xu XH, Zhou F. microRNA-92a promotes lymph node metastasis of human esophageal squamous cell carcinoma via E-cadherin. J Biol Chem. 2011;286:10725-10734. [PubMed] [DOI] |
| 22. | Wu BL, Xu LY, Du ZP, Liao LD, Zhang HF, Huang Q, Fang GQ, Li EM. MiRNA profile in esophageal squamous cell carcinoma: downregulation of miR-143 and miR-145. World J Gastroenterol. 2011;17:79-88. [PubMed] [DOI] |
| 23. | Ma J, Mannoor K, Gao L, Tan A, Guarnera MA, Zhan M, Shetty A, Stass SA, Xing L, Jiang F. Characterization of microRNA transcriptome in lung cancer by next-generation deep sequencing. Mol Oncol. 2014;8:1208-1219. [PubMed] [DOI] |
| 24. | Quail DF, Joyce JA. Microenvironmental regulation of tumor progression and metastasis. Nat Med. 2013;19:1423-1437. [PubMed] [DOI] |
| 25. | Rocha C, Papon L, Cacheux W, Marques Sousa P, Lascano V, Tort O, Giordano T, Vacher S, Lemmers B, Mariani P. Tubulin glycylases are required for primary cilia, control of cell proliferation and tumor development in colon. EMBO J. 2014;33:2247-2260. [PubMed] [DOI] |
| 29. | Flores-Pérez A, Marchat LA, Rodríguez-Cuevas S, Bautista VP, Fuentes-Mera L, Romero-Zamora D, Maciel-Dominguez A, de la Cruz OH, Fonseca-Sánchez M, Ruíz-García E. Suppression of cell migration is promoted by miR-944 through targeting of SIAH1 and PTP4A1 in breast cancer cells. BMC Cancer. 2016;16:379. [PubMed] [DOI] |
| 30. | Liu M, Zhou K, Cao Y. MicroRNA-944 Affects Cell Growth by Targeting EPHA7 in Non-Small Cell Lung Cancer. Int J Mol Sci. 2016;17:pii: E1493. [PubMed] [DOI] |