修回日期: 2017-11-02
接受日期: 2017-11-10
在线出版日期: 2017-12-18
评估烯醇酶超家族成员1(enolase superfamily member1, ENOSF1)基因多态性与卡培他滨治疗安全性的关系, 探讨其预测价值.
采集62例接受卡培他滨单药或联合治疗的结直肠癌患者外周静脉血, 提取基因组DNA, 检测ENOSF1基因单核苷酸多态位点rs2612091、IVS10-61C>T、IVS10-60G>A、rs1059394基因型; 并对62例患者在接受卡培他滨治疗中出现的不良反应进行记录, 比较不同基因型患者之间不良反应的差异.
根据不良反应级别分为0-1级和2-4级两组, 基因型采用两种分类方式, 显性模型和隐性模型. 显性模型中, IVS10-60G>A GG型发生2-4级手足综合征的风险明显高于AG/AA型, 差异有统计学意义(χ2 = 5.421, P = 0.020, OR = 4.364, 95%CI: 1.217-15.641); 隐性模型中, IVS10-61C>T TT型发生2-4级腹泻的风险明显高于CT/CC型, 差异有统计学意义(Fisher精确概率: P = 0.039, OR = 0.108, 95%CI: 0.015-0.788).
ENOSF1基因IVS10-61C>T与IVS10-60G>A位点有望作为遗传标志物, 预测卡培他滨治疗的安全性.
核心提要: 烯醇酶超家族成员1基因多态性与卡培他滨治疗安全性的关系研究不多, 其IVS10-61C>T及IVS10-60G>A位点尚未查阅到相关报道, 本研究表明与卡培他滨不良反应发生有相关性, 有望作为遗传学标志物, 预测卡培他滨治疗的安全性.
引文著录: 王鑫, 谢甲贝, 吴刚, 李修岭, 韩双印. ENOSF1基因多态性预测结直肠癌患者卡培他滨治疗安全性. 世界华人消化杂志 2017; 25(35): 3133-3140
Revised: November 2, 2017
Accepted: November 10, 2017
Published online: December 18, 2017
To evaluate the relationship between single nucleotide polymorphisms of enolase superfamily member 1 (ENOSF1) and capecitabine related toxic reactions.
Peripheral venous blood was collected from 62 patients with colorectal cancer who were treated with capecitabine alone or combined with chemotherapy. Genomic DNA was extracted from the peripheral venous blood to genotype ENOSF1 single nucleotide polymorphisms rs2612091, IVS10-61C>T, IVS10-60G>A, and rs1059394 by sequencing. The toxic reactions of capecitabine were recorded, and their relationship with different genotypes was compared.
According to the level of toxic reactions, the patients were divided into two groups: patients with grade 0-1 toxicities and those with grade 2-4 toxicities. ENOSF1 genotypes were divided into a dominant model and a recessive model. In the dominant model, the risk of grade 2-4 hand-foot syndrome in patients with IVS10-60G>A GG genotype was significantly higher than that in patients with AG/AA genotype (χ2 = 5.421, P = 0.020, OR = 4.364, 95%CI: 1.217-15.641). In the recessive model, the risk of grade 2-4 diarrhea in patients with IVS10-61C>T TT genotype was significantly higher than that in patients with CC/CT genotype (Fisher's exact test: P = 0.817, OR = 0.108, 95%CI: 0.015-0.788).
The IVS10-61C>T and IVS10-60G>A loci of ENOSF1 gene are expected to be used as genetic markers to predict the therapeutic safety of capecitabine treatment.
- Citation: Wang X, Xie JB, Wu G, Li XL, Han SY. Single nucleotide polymorphisms of ENOSF1 are predictors of therapeutic safety of capecitabine in colorectal cancer. Shijie Huaren Xiaohua Zazhi 2017; 25(35): 3133-3140
- URL: https://www.wjgnet.com/1009-3079/full/v25/i35/3133.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v25.i35.3133
卡培他滨是一种口服的5-氟尿嘧啶(5-fluorouridine, 5-Fu)前体药物, 作为一线化疗方案应用于结直肠癌[1,2], 然而其不良反应是临床中普遍存在的问题, 导致治疗过程中药物减量, 甚至终止治疗[3-6]. 腹泻、手足综合征(hand-foot syndrome, HFS)、骨髓抑制则是其最常见的剂量限制性不良反应[7,8]. 预测及抑制不良反应的发生, 是卡培他滨临床应用中亟待解决的问题. 随着药物基因组学的深入研究[9,10]表明, 大多药物不良反应的发生与个体基因变异相关, 如能确定与不良反应相关的基因变异, 预测其治疗安全性, 将为个体化用药方案提供有益的指导. 研究[11]表明, 卡培他滨主要在肝脏及肿瘤组织内代谢为5-Fu, 其主要通过影响胸苷酸合成酶(thymidylate synthetase, TYMS)表达水平, 抑制细胞DNA合成及修复发挥抗肿瘤作用. 烯醇酶超家族成员1(enolase superfamily member1, ENOSF1)酶是卡培他滨代谢关键酶之一, 通过调节TYMS mRNA反转录过程及蛋白表达水平, 在卡培他滨代谢过程中发挥作用[12]. ENOSF1存在多态性位点, 作用可能存在差异性. 本课题研究单药或联合应用卡培他滨治疗的结直肠癌患者的不良反应与ENOSF1基因多态性之间的关系.
纳入2016-06/2017-06在河南省人民医院消化科和胃肠外科原发结直肠癌患者共62例, 所有患者均有可供病理组织学诊断的标本, 并经病理确诊, 无第二原发癌, 未发生转移, 并满足以下入组标准: (1)接受卡培他滨单药(每日2500 mg/m2)或者以卡培他滨为基础的联合方案化疗(每日2000 mg/m2), 连用2 wk, 停药1 wk为1个周期, 患者至少完成2周期的治疗, 在此化疗方案前未接受过化疗; (2)ECOG体力评分≤1分; (3)无重要器官功能严重受损, 血常规、肝肾功能及心电图均处于正常范围内. 排除标准: (1)有以下合并症或并发症: 感染急性期, 严重的心肝肾疾病及血液系统性疾病; (2)处于妊娠期或哺乳期. 研究取得所有患者的知情同意, 并取得我院伦理委员会同意.
1.2.1 标本采集及DNA提取: 在每个化疗周期开始前, 用乙二胺四乙酸抗凝管采集结直肠癌患者外周静脉血2 mL, 根据上海生工公司基因组DNA提取试剂盒说明书提取基因组DNA, -20 ℃储存.
1.2.2 设计及合成引物: 在美国国家生物技术信息中心提供的基因及单核苷酸多态位点(single nucleotide polymorphism, SNP)公共数据库中, 查询ENOSF1基因全序列及2个SNP序列, 设计引物, 由上海生工公司合成引物(表1).
| SNP位点 | 引物(5'→3') |
| rs2612091 | 上游引物: CTAGTGGAGTAGGTGTGCCA |
| 下游引物: ATGTCATCAGGGGAGGTTGG | |
| rs1059394 | 上游引物: TTTACCTCGGCATCCAGCC |
| 下游引物: GCCAGGGACAGATGGGTTAG |
1.2.3 PCR反应: 配制50 μL反应体系: 1 μL(浓度50 ng/μL)模板DNA, 2 μL(浓度10 μmol/μL)上游引物, 2 μL(浓度10 μmol/μL)下游引物, 2xPCRMix(广州东盛科技有限公司)25 μL, 超纯水20 μL; 反应条件: 94 ℃预变性3 min, 94 ℃变性30 s、57 ℃退火30 s、72 ℃延伸1 min, 30个循环, 72 ℃延伸5 min. 2%琼脂糖凝胶电泳, 凝胶成像分析系统检测聚合酶链反应(polymerase chain reaction, PCR)产物. 纯化PCR产物、测序, 测序结果用DNASTAR软件中的Seqman和SeqBuilder进行分析.
1.2.4 不良反应的记录及评价标准: 不良反应的评价是根据美国国家癌症研究所常见不良反应事件评价标准(CTCAE)4.0版, 在每次治疗前进行不良反应的记录, 连续住院期间随访或电话随访大于2个治疗周期.
统计学处理 SPSS19.0统计软件分析, 基因型组间率的比较采用χ2检验或Fisher确切概率法, 以P<0.05为差异具有统计学意义.
本研究共入组62例结直肠癌患者, 人口学资料及临床病理特征如表2. 性别、年龄、肿瘤原发部位、肿瘤的分期以及化疗方案对不良反应的发生率无明显统计学差异(P>0.05).
| 人口学资料及临床病理特征 | n (%) |
| 性别 | |
| 男 | 39 (62.90) |
| 女 | 23 (37.10) |
| 年龄(岁) | |
| <60 | 31 (50.00) |
| ≥60 | 31 (50.00) |
| 民族 | |
| 汉族 | 62 (100) |
| 肿瘤原发部位 | |
| 结肠 | 27 (43.55) |
| 直肠 | 35 (56.45) |
| 肿瘤分期(TNM) | |
| Ⅰ+Ⅱ期 | 40 (64.52) |
| Ⅲ+Ⅳ期 | 22 (35.48) |
| ECOG评分 | |
| 0-1分 | 62 (100) |
| 化疗方法 | |
| 卡培他滨单药化疗 | 3 (4.84) |
| 卡培他滨联合方案化疗 | 59 (95.16) |
| ≥2级不良反应 | |
| HFS | 44 (70.97) |
| 腹泻 | 14 (22.58) |
| 中性粒细胞减少 | 25 (40.32) |
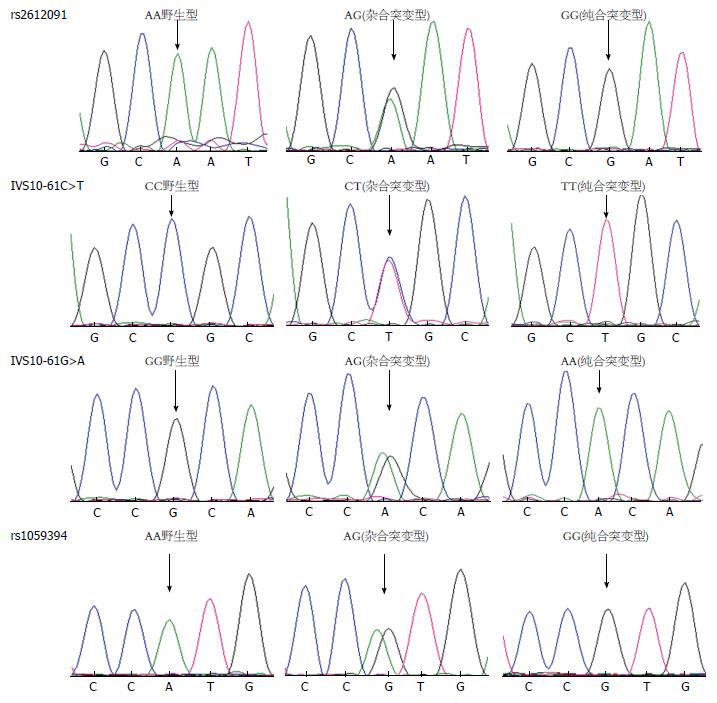
在分析rs2612091测序结果时, 其下游第166 bp、第167 bp存在多态性, 根据人类基因序列变异通用命名法[13], 这两个位点分别命名为IVS10-61C>T、IVS10-60G>A, 因此本研究纳入ENOSF1基因4个SNP位点: rs2612091、IVS10-61C>T、IVS10-60G>A、rs1059394, 各基因型测序代表图如图1. 其中14例rs2612091、IVS10-61C>T、IVS10-60G>A位点因目的序列发生插入突变, 未能检测出基因型. 对ENOSF1基因的4个SNP点进行Hardy-Weinberg遗传平衡检验, 结果如表3. ENOSF1基因SNP位点rs2612091(χ2 = 1.6261, P>0.05), IVS10-61C>T(χ2 = 0.0440, P>0.05), IVS10-60G>A(χ2 = 1.2325, P>0.05), rs1059394(χ2 = 2.5424, P>0.05), P>0.05说明样本人群4个SNP点基因型符合Hardy-Weinberg遗传平衡.
| SNP点 | 实际频数 | 理论频数 | 最小等位基因频率(MAF) | HWE检验(P值) |
| rs2612091 | G = 0.3542 | >0.05 | ||
| AA(野生型) | 18 | 20.0188 | ||
| AG(杂合突变型) | 26 | 21.9592 | ||
| GG(纯合突变型) | 4 | 6.0220 | ||
| IVS10-61C>T | T = 0.3125 | >0.05 | ||
| CC(野生型) | 23 | 22.6875 | ||
| CT(杂合突变型) | 20 | 20.6250 | ||
| TT(纯合突变型) | 5 | 4.6875 | ||
| IVS10-60G>A | A = 0.3021 | >0.05 | ||
| GG(野生型) | 25 | 23.3808 | ||
| AG(杂合突变型) | 17 | 20.2416 | ||
| AA(纯合突变型) | 6 | 4.3776 | ||
| rs1059394 | G = 0.3306 | >0.05 | ||
| AA(野生型) | 25 | 27.7822 | ||
| AG(杂合突变型) | 33 | 27.4412 | ||
| GG(纯合突变型) | 4 | 6.7766 |
观察卡培他滨剂量限制性不良反应HFS、腹泻、中性粒细胞减少分别与各SNP之间的关系. 显性模型即为野生型/杂合突变型+纯合突变型; 隐性模型即为纯合突变型/野生型+杂合突变型. SNP显性模型与不良反应之间的关系如表4. IVS10-60G>A GG型发生2-4级HFS的风险明显高于AG/AA型, 差异有统计学意义(χ2 = 5.421, P = 0.020, OR = 4.364, 95%CI: 1.217-15.641), 而与2-4级腹泻及2-4级HFS的发生无明显相关性. 显性模型中, rs2612091、IVS10-61C>T、rs1059394各基因型与2-4级不良反应的发生均无明显相关性. SNP隐性模型与不良反应之间的关系如表4. IVS10-61C>T TT型发生2-4级腹泻的风险明显高于CT/CC型, 差异统计学意义(Fisher精确概率: P = 0.039, OR = 0.108, 95%CI: 0.015-0.788), 与2-4级HFS及2-4级中性粒细胞减少无明显相关性; 隐性模型中, rs2612091、IVS10-60G>A、rs1059394各基因型与2-4级不良反应的发生均无明显相关性.
| 不良反应SNP | HFS | 腹泻 | 中性粒细胞减少 | |||
| P值 | OR (95%CI) | P值 | OR (95%CI) | P值 | OR (95%CI) | |
| 显性模型 | ||||||
| rs2612091 | 0.311 | 0.536 (0.159-1.804) | 0.215 | 2.500 (0.573-10.914) | 0.762 | 0.832 (0.253-2.739) |
| IVS10-61C>T | 0.085 | 0.344 (0.101-1.178) | 0.817 | 0.842 (0.196-3.615) | 0.130 | 0.404 (0.123-1.321) |
| IVS10-60G>A | 0.020 | 4.364 (1.217-15.641) | 0.331 | 2.105 (0.460-9.640) | 0.353 | 1.731 (0.541-5.536) |
| rs1059394 | 0.672 | 0.787 (0.259-2.389) | 0.402 | 1.667 (0.502-5.531) | 0.373 | 0.618 (0.213-1.787) |
| 隐性模型 | ||||||
| rs2612091 | 1.000 | 0.583 (0.056-6.086) | 1.000 | 1.257 (1.082-1.460) | 1.000 | 0.692 (0.089-5.378) |
| IVS10-61C>T | 0.643 | 0.422 (0.043-4.112) | 0.039 | 0.108 (0.015-0.788) | 0.385 | 3.167 (0.326-30.727) |
| IVS10-60G>A | 0.167 | 4.462 (0.724-27.511) | 1.000 | 1.176 (0.120-11.513) | 1.000 | 1.500 (0.247-9.111) |
| rs1059394 | 1.000 | 0.804 (0.078-8.285) | 0.566 | 1.318 (1.140-1.524) | 0.637 | 0.611 (0.080-4.655) |
结直肠癌是我国发病率及死亡率均位居前5的肿瘤, 且呈逐年递增趋势[14,15]. 卡培他滨是结直肠癌化疗一线用5-Fu类抗肿瘤药物[16], 口服后经小肠黏膜吸收, 在肝脏及肿瘤组织内经羟羧酯酶、胞苷脱氨酶(cytidine deaminase, CDA)代谢为5-Fu, 主要通过抑制TYMS活性, 阻断肿瘤细胞DNA合成及修复而发挥作用, 最终经双氢嘧啶脱氢酶(dihydropyrimidine dehydrogenase, DPYD)分解代谢[17,18]. ENOSF1因其可调节TYMS mRNA反转录及蛋白表达水平而在卡培他滨代谢过程中起到关键作用[12]. ENOSF1基因位于18p11.32, 有17个外显子, mRNA总长42345 bp, rs2612091、IVS10-61C>T、IVS10-60G>A位于第10内含子上, rs1059394位于第17外显子上.
近年来, 关于卡培他滨代谢酶与不良反应关系的研究[19-25]较多, Caronia等[19]对CDA预测卡培他滨相关严重的HFS的研究表明, 由于CDA基因rs3215400 C等位基因的缺失突变出现了一个转录调控因子E2F新的结合位点, 影响CDA基因表达, 可明显增加卡培他滨相关HFS的发生率; DPYD基因作为卡培他滨代谢关键酶之一, 近些年的研究尤其多, DPYD酶的缺失或活性降低[24,25], 可明显增加氟尿嘧啶相关不良反应的发生. Deenen等[20]的研究表明, DPYD基因IVS14+1 G>A、2846 A>T的突变可导致DPYD活性降低, 5-Fu代谢减慢引起体内蓄积, 从而增加卡培他滨相关严重不良反应的发生率; 关于DPYD基因与5-Fu相关不良反应关系的荟萃分析[26]表明, 除了DPYD基因DPYD*2A和2846 A>T与氟尿嘧啶相关不良反应有关, DPYD多态位点1679 T>G、1236 G>A也与氟尿嘧啶相关胃肠道毒性以及血液系统毒性相关, 但与氟尿嘧啶相关HFS发生无关. Pellicer等[27]对与卡培他滨严重不良反应相关的多个新多态位点的研究表明, CDA rs2072671与卡培他滨全部的不良反应均相关, ENOSF1/TYMS rs699517与恶习、呕吐、厌食症、乏力相关, 而ENOSF1 rs3744962、rs2612086、rs1059384则与卡培他滨不良反应无明显相关性.
对ENOSF1基因多态性与卡培他滨治疗不良反应发生之间的关系的其他研究中, Rosmarin等[11]关于结直肠癌患者卡培他滨相关毒性候选基因的研究表明, ENOSF1 rs2612091 GG型可明显减少ENOSF1在脂肪及淋巴细胞中的表达, 其3-4级HFS发生率显著增加; 一个对西班牙结直肠癌人群关于CDA和ABCB1基因多态位点可预测卡培他滨相关毒性的研究[28]提出, ENOSF1 rs2612091纯合突变型GG型明显地提高2-4级HFS发生率, 此结果与我们研究结果不符, 此研究的隐性模型中, ENOSF1 rs2612091纯合突变型GG型与2-4级HFS发生率并无明显相关性; Shen等[29]关于TYMS基因多态性位点预测胃癌易感性及生存率的研究表明: ENOSF1 rs1059394纯合突变TT型较CC型/CT型胃癌的患病率显著增加, 并且生存率显著降低, 未对治疗不良反应做出评价. 本研究中提及的ENOSF1基因rs2612091及rs1059394位点预测卡培他滨相关毒性的结果与既往研究结果不相符, 考虑与多种原因相关, 首先是化疗方案不同, 本研究是卡培他滨单药口服或联合奥沙利铂, 既往研究[11,27,28]是卡培他滨联合贝伐单抗、伊立替康等, 不同化疗方案, 不良反应存在差异; 其次是研究对象不同, 本研究人群为汉族人, 而既往研究[11,27,28]以高加索人及西班牙人为主, 不同人种基因多态性可能存在有差异; 再者, CDA、DPYD基因对卡培他滨不良反应有影响[19,20], 因此可能造成部分研究结论受到影响; 最后, 本研究样本量不足, 得到的阴性结果也可能与检验效率低下相关.
ENOSF1基因IVS10-61C>T及IVS10-60G>A位点尚未查阅到相关报道, 并且其不良反应发生率有显著统计学差异, 即IVS10-61C>T TT型发生2-4级腹泻的风险明显高于CT/CC型, IVS10-60G>A GG型发生2-4级HFS的风险明显高于AG/AA型, 考虑ENOSF1基因IVS10-61C>T及IVS10-60G>A位点突变导致ENOSF1在组织中表达异常, 进而改变了TYMS基因反转录以及蛋白表达水平, 最终影响肿瘤细胞DNA的合成、修复等功能[30], 具体作用机制尚需实验证实. IVS10-61C>T以及IVS10-60G>A位点有望作为预测卡培他滨治疗安全性的遗传学标志物. ENOSF1基因多态位点IVS10-61C>T以及IVS10-60G>A的提出及现有的阳性结果, 为以后对ENOSF1基因的研究提供了一个新的方向.
本研究尚且存在不足, 样本量小, 且尚未对生存期以及总缓解率作出总结分析, 需继续随访以提高本研究总体质量, 并需要在机制及功能层面进一步验证ENOSF1基因多态性与卡培他滨代谢的关系. 本研究为卡培他滨治疗的安全性、个体化治疗方案的实施提供了研究基础.
卡培他滨是结直肠癌一线化疗方案药物, 其不良反应是临床亟待解决的问题, 而药物不良反应的发生与个体基因变异相关, 烯醇酶超家族成员1(enolase superfamily member1, ENOSF1)酶是卡培他滨代谢关键酶之一, ENOSF1基因存在多态性, 本研究初步探讨ENOSF1基因多态性与卡培他滨不良反应之间的关系.
在肿瘤化疗中, 患者不良反应差异明显, 通过检测患者基因多态性位点预测治疗安全性、制定个体化用药方案, 是目前研究热点之一. 卡培他滨广泛应用于临床, 个体差异显著, 成为亟待研究的对象.
Rosmarin等研究表明, ENOSF1 rs2612091 GG型可明显减少ENOSF1在脂肪及淋巴细胞中的表达, 其3-4级手足综合征发生率显著增加. 临床数据的分析结果, 需要基础实验在机制层面进一步验证.
本研究中ENOSF1基因IVS10-61C>T及IVS10-60G>A位点尚未见到相关报道, 其不良反应发生率有显著统计学差异, 为ENOSF1基因的研究提供了一个新的方向, 具有明显的创新性.
通过阐明ENOSF1基因IVS10-61C>T及IVS10-60G>A位点多态性与不良反应的关系, 制定个体化用药方案, 提高卡培他滨治疗安全性.
药物基因组学: 研究基因变异所致的不同疾病对药物的不同反应, 并在此基础上研制出新药或新的用药方法, 这一新概念被称为药物基因组学.
本文有较好的创新性, 对ENOSF1基因多态性与卡培他滨临床治疗的反应性和安全性进行了有益的探索, 为卡培他滨的临床应用提供了研究基础和依据.
冯继红, 副教授, 遵义医学院附属医院肿瘤医院; 牛伟新, 教授, 主任医师, 复旦大学附属中山医院普外科; 宋武, 教授, 主任医师, 中山大学附属第一医院胃肠外科; 张录顺, 副教授, 成都医学院病理教研室
手稿来源: 自由投稿
学科分类: 胃肠病学和肝病学
手稿来源地: 河南省
同行评议报告分类
A级 (优秀): 0
B级 (非常好): B, B
C级 (良好): C
D级 (一般): D
E级 (差): 0
编辑: 闫晋利 电编:杜冉冉
| 2. | Twelves C, Wong A, Nowacki MP, Abt M, Burris H 3rd, Carrato A, Cassidy J, Cervantes A, Fagerberg J, Georgoulias V, Husseini F, Jodrell D, Koralewski P, Kröning H, Maroun J, Marschner N, McKendrick J, Pawlicki M, Rosso R, Schüller J, Seitz JF, Stabuc B, Tujakowski J, Van Hazel G, Zaluski J, Scheithauer W. Capecitabine as adjuvant treatment for stage III colon cancer. N Engl J Med. 2005;352:2696-2704. [PubMed] [DOI] |
| 3. | Kadoyama K, Miki I, Tamura T, Brown JB, Sakaeda T, Okuno Y. Adverse event profiles of 5-fluorouracil and capecitabine: data mining of the public version of the FDA Adverse Event Reporting System, AERS, and reproducibility of clinical observations. Int J Med Sci. 2012;9:33-39. [PubMed] [DOI] |
| 4. | Saif MW, Katirtzoglou NA, Syrigos KN. Capecitabine: an overview of the side effects and their management. Anticancer Drugs. 2008;19:447-464. [PubMed] [DOI] |
| 5. | Kelly H, Goldberg RM. Systemic therapy for metastatic colorectal cancer: current options, current evidence. J Clin Oncol. 2005;23:4553-4560. [PubMed] [DOI] |
| 6. | Wheeler HE, González-Neira A, Pita G, de la Torre-Montero JC, Alonso R, Lopez-Fernandez LA, Alba E, Martín M, Dolan ME. Identification of genetic variants associated with capecitabine-induced hand-foot syndrome through integration of patient and cell line genomic analyses. Pharmacogenet Genomics. 2014;24:231-237. [PubMed] [DOI] |
| 7. | Webster-Gandy JD, How C, Harrold K. Palmar-plantar erythrodysesthesia (PPE): a literature review with commentary on experience in a cancer centre. Eur J Oncol Nurs. 2007;11:238-246. [PubMed] [DOI] |
| 8. | Nagore E, Insa A, Sanmartín O. Antineoplastic therapy-induced palmar plantar erythrodysesthesia ('hand-foot') syndrome. Incidence, recognition and management. Am J Clin Dermatol. 2000;1:225-234. [PubMed] [DOI] |
| 9. | Jennings BA, Kwok CS, Willis G, Matthews V, Wawruch P, Loke YK. Functional polymorphisms of folate metabolism and response to chemotherapy for colorectal cancer, a systematic review and meta-analysis. Pharmacogenet Genomics. 2012;22:290-304. [PubMed] [DOI] |
| 11. | Rosmarin D, Palles C, Pagnamenta A, Kaur K, Pita G, Martin M, Domingo E, Jones A, Howarth K, Freeman-Mills L. A candidate gene study of capecitabine-related toxicity in colorectal cancer identifies new toxicity variants at DPYD and a putative role for ENOSF1 rather than TYMS. Gut. 2015;64:111-120. [PubMed] [DOI] |
| 12. | Dolnick BJ, Angelino NJ, Dolnick R, Sufrin JR. A novel function for the rTS gene. Cancer Biol Ther. 2003;2:364-369. [PubMed] [DOI] |
| 13. | den Dunnen JT, Antonarakis SE. Nomenclature for the description of human sequence variations. Hum Genet. 2001;109:121-124. [PubMed] [DOI] |
| 14. | Chen W, Zheng R, Baade PD, Zhang S, Zeng H, Bray F, Jemal A, Yu XQ, He J. Cancer statistics in China, 2015. CA Cancer J Clin. 2016;66:115-132. [PubMed] [DOI] |
| 15. | Dai Z, Zheng RS, Zou XN, Zhang SW, Zeng HM, Li N, Chen WQ. [Analysis and prediction of colorectal cancer incidence trend in China]. Zhonghua Yufang Yixue Zazhi. 2012;46:598-603. [PubMed] [DOI] |
| 16. | Cassidy J, Twelves C, Van Cutsem E, Hoff P, Bajetta E, Boyer M, Bugat R, Burger U, Garin A, Graeven U. First-line oral capecitabine therapy in metastatic colorectal cancer: a favorable safety profile compared with intravenous 5-fluorouracil/leucovorin. Ann Oncol. 2002;13:566-575. [PubMed] |
| 17. | Thorn CF, Marsh S, Carrillo MW, McLeod HL, Klein TE, Altman RB. PharmGKB summary: fluoropyrimidine pathways. Pharmacogenet Genomics. 2011;21:237-242. [PubMed] [DOI] |
| 18. | Lam SW, Guchelaar HJ, Boven E. The role of pharmacogenetics in capecitabine efficacy and toxicity. Cancer Treat Rev. 2016;50:9-22. [PubMed] [DOI] |
| 19. | Caronia D, Martin M, Sastre J, de la Torre J, García-Sáenz JA, Alonso MR, Moreno LT, Pita G, Díaz-Rubio E, Benítez J. A polymorphism in the cytidine deaminase promoter predicts severe capecitabine-induced hand-foot syndrome. Clin Cancer Res. 2011;17:2006-2013. [PubMed] |
| 20. | Deenen MJ, Tol J, Burylo AM, Doodeman VD, de Boer A, Vincent A, Guchelaar HJ, Smits PH, Beijnen JH, Punt CJ. Relationship between single nucleotide polymorphisms and haplotypes in DPYD and toxicity and efficacy of capecitabine in advanced colorectal cancer. Clin Cancer Res. 2011;17:3455-3468. [PubMed] |
| 21. | Hamzic S, Kummer D, Milesi S, Mueller D, Joerger M, Aebi S, Amstutz U, Largiader CR. Novel Genetic Variants in Carboxylesterase 1 Predict Severe Early-Onset Capecitabine-Related Toxicity. Clin Pharmacol Ther. 2017;102:796-804. [PubMed] [DOI] |
| 22. | Afzal S, Jensen SA, Vainer B, Vogel U, Matsen JP, Sørensen JB, Andersen PK, Poulsen HE. MTHFR polymorphisms and 5-FU-based adjuvant chemotherapy in colorectal cancer. Ann Oncol. 2009;20:1660-1666. [PubMed] [DOI] |
| 23. | Salgado J, Zabalegui N, Gil C, Monreal I, Rodríguez J, García-Foncillas J. Polymorphisms in the thymidylate synthase and dihydropyrimidine dehydrogenase genes predict response and toxicity to capecitabine-raltitrexed in colorectal cancer. Oncol Rep. 2007;17:325-328. [PubMed] [DOI] |
| 24. | Thomas F, Hennebelle I, Delmas C, Lochon I, Dhelens C, Garnier Tixidre C, Bonadona A, Penel N, Goncalves A, Delord JP. Genotyping of a family with a novel deleterious DPYD mutation supports the pretherapeutic screening of DPD deficiency with dihydrouracil/uracil ratio. Clin Pharmacol Ther. 2016;99:235-242. [PubMed] [DOI] |
| 25. | Amstutz U, Farese S, Aebi S, Largiadèr CR. Dihydropyrimidine dehydrogenase gene variation and severe 5-fluorouracil toxicity: a haplotype assessment. Pharmacogenomics. 2009;10:931-944. [PubMed] [DOI] |
| 26. | Meulendijks D, Henricks LM, Sonke GS, Deenen MJ, Froehlich TK, Amstutz U, Largiadèr CR, Jennings BA, Marinaki AM, Sanderson JD. Clinical relevance of DPYD variants c.1679T>G, c.1236G>A/HapB3, and c.1601G>A as predictors of severe fluoropyrimidine-associated toxicity: a systematic review and meta-analysis of individual patient data. Lancet Oncol. 2015;16:1639-1650. [PubMed] [DOI] |
| 27. | Pellicer M, García-González X, García MI, Robles L, Grávalos C, García-Alfonso P, Pachón V, Longo F, Martínez V, Blanco C. Identification of new SNPs associated with severe toxicity to capecitabine. Pharmacol Res. 2017;120:133-137. [PubMed] [DOI] |
| 28. | García-González X, Cortejoso L, García MI, García-Alfonso P, Robles L, Grávalos C, González-Haba E, Marta P, Sanjurjo M, López-Fernández LA. Variants in CDA and ABCB1 are predictors of capecitabine-related adverse reactions in colorectal cancer. Oncotarget. 2015;6:6422-6430. [PubMed] [DOI] |
| 29. | Shen R, Liu H, Wen J, Liu Z, Wang LE, Wang Q, Tan D, Ajani JA, Wei Q. Genetic polymorphisms in the microRNA binding-sites of the thymidylate synthase gene predict risk and survival in gastric cancer. Mol Carcinog. 2015;54:880-888. [PubMed] [DOI] |
| 30. | Ulrich CM, Bigler J, Velicer CM, Greene EA, Farin FM, Potter JD. Searching expressed sequence tag databases: discovery and confirmation of a common polymorphism in the thymidylate synthase gene. Cancer Epidemiol Biomarkers Prev. 2000;9:1381-1385. [PubMed] |