修回日期: 2013-12-16
接受日期: 2013-12-19
在线出版日期: 2014-02-18
目的: 研究结肠癌大鼠与正常大鼠粪便菌群结构性变化.
方法: 通过1, 2-二甲肼(dimethyl hydrazine, DMH)腹腔注射构建大肠癌的动物模型, 造模成功后收集实验组和对照组大鼠肠道粪便, 利用PCR-DGGE(polymerase chain reaction- denaturing gradient electrophoresis)技术分离菌群, 对两组菌群相似性(聚类分析)和多态性(丰富度、均一性、Shannon-Wiener指数、Simpson指数)进行分析, 并对目的菌群与GenBank进行比对, 确定菌群种属, 研究其变化规律.
结果: 相较于对照组, 实验组中毛螺菌科(Lachnospiraceae)、Ruminococcaceae科菌、肠乳杆菌(Lactobacillus intestinalis)、帕拉普氏菌属(Paraprevotella)、鼠乳杆菌(Lactobacillus murinus)、乳杆菌属(Lactobacillus)、普氏菌属(Prevotella)、卷曲乳杆菌(Lactobacillus crispatus)和Lachnospiracea incertae sedis等菌群丰度显著降低, 而粪芽孢菌属(Coprobacillus)丰度则明显增加. 尽管两组菌群多样性均较丰富, 但菌群结构相似性存在明显差异.
结论: 肠癌大鼠与正常对照组相比出现粪便菌群结构的偏移, 有益菌减少, 潜在致病菌增加.
核心提示: 与正常大鼠相比, 肠癌大鼠粪便中菌群发生明显偏移, 有益菌数量及种类明显下降, 而致病菌却增加.
引文著录: 高仁元, 朱庆超, 伍雯, 秦环龙. 肠癌大鼠与正常大鼠粪便菌群的结构性差异. 世界华人消化杂志 2014; 22(5): 661-667
Revised: December 16, 2013
Accepted: December 19, 2013
Published online: February 18, 2014
AIM: To investigate the compositional differences in fecal flora between rats with colorectal cancer and normal rats.
METHODS: A rat model of colorectal cancer was developed by intraperitoneal injection of 1, 2-dimethyl hydrazine (DMH). Fecal samples were collected from rats with colorectal cancer and normal controls, and the microbiota was isolated by PCR-DGGE technique to perform flora similarity analysis (cluster analysis) and polymorphism analysis (richness, uniformity, Shannon-Wiener index, Simpson index) and to compare with the GenBank to identify the genus so as to study the variation.
RESULTS: Compared with normal rats, the abundance of Lachnospiraceae, Ruminococcaceae, Lactobacillus intestinalis, Paraprevotella, Lactobacillus murinus, Lactobacillus, Prevotella, Lactobacillus crispatus and Lachnospiracea incertae sedis was significantly reduced and that of Coprobacillus was significantly increased in rats with colorectal cancer. Although the flora diversity between the two groups showed no statistical difference, there was a significant difference in flora composition.
CONCLUSION: The composition of fecal microflora changes in rats with colorectal cancer compared with normal rats, with the number of beneficial bacteria reduced and that of potential pathogens increased.
- Citation: Gao RY, Zhu QC, Wu W, Qin HL. Compositional differences in fecal microbiota between rats with colorectal cancer and normal rats. Shijie Huaren Xiaohua Zazhi 2014; 22(5): 661-667
- URL: https://www.wjgnet.com/1009-3079/full/v22/i5/661.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v22.i5.661
大肠癌的发生发展是伴随着一系列基因突变的多步骤病变过程, 其发病率在全球范围内高居第3位, 全球每年新发病例数超过120万例[1]. 随着经济的发展, 在发展中国家, 大肠癌发生率呈现快速升高的趋势. 如在中国上海地区, 大肠癌发病率已猛增至56/10万人, 达到欧美发达国家发病率水平[2]. 目前, 遗传因素和环境因素被认为是大肠癌发生发展的重要原因, 而环境因素中的肠道微生态异常改变及其与肠癌相关性的研究近几年称为研究的热点, 菌群结构和数量的异常促进肠癌发生的报道也逐渐增多[3]. 在哺乳类动物肠道内也存在着种类繁多和数量庞大的微生物, 他们在宿主肠道营养摄取、维生素合成、多酚类物质代谢、保护肠上皮细胞、脂肪贮存、刺激肠道组织血管生成以及肠道免疫功能形成等多方面具有不可或缺的作用[4,5]. 因此, 这些微生物构成了维持整个肠道微生态稳定的重要基础. 而在这些微生物中, 占据了绝大部分数量的是细菌, 他们的基因组大小是宿主的150-160倍[6,7]. 因此, 对其肠道菌群结构多样性及种群动态变化进行研究具有至关重要的意义.
传统的微生物检测方法主要依赖从宿主新鲜粪便中分离微生物进行培养, 进而明确菌属性质. 但是由于技术条件、培养条件的严苛性, 肠道中可以被培养的菌群较少(不超过20%-30%), 且并不能代表全部肠道菌群, 更不能明确物种之间的系统发育学关系[7]. 随着微生态学检测技术的迅速发展, 特别是应用了PCR-变性梯度凝胶电泳(polymerase chain reaction- denaturing gradient electrophoresis, DGGE)等较先进的技术后, 菌群变化和相关疾病之间的关系研究取得了巨大的进步. DGGE技术主要原理是基于DNA碱基配对的基础, 将具有相同长度但不同组成的双链PCR产物分离, 使具有不同序列的DNA片段停留在凝胶的不同位置, 形成相互分开的条带图谱, 从而对菌群进行分离[8]. 该技术不依赖于培养技术且最低可检测到只占总群数量1%的菌群[9]. 因此, 本研究我们利用DGGE技术来比较大鼠肠癌模型与正常对照组之间粪便中菌群的变化, 并阐述肠道菌群在肠癌发生发展中的结构变化及潜在作用.
♂Wistar大鼠共计12只, 体质量150-180 g. 购自上海史莱克实验动物有限公司, 生产许可证号为SCXK(沪)2007-0005, 使用许可证号为SYXK(沪)2006-0010. Taq酶等试剂(生工生物工程股份有限公司); PCR反应扩增仪(加拿大, BBI公司); DYY-8型稳压稳流电泳仪(上海琪特分析仪器有限公司); H6-1微型电泳槽(上海精益有机玻璃制品仪器厂); 凝胶成像系统(Gene Genius公司); U-3010紫外-可见分光光度计(Hitachi公司); DGGE仪(美国Bio-Rad公司); DMH(Sigma-aldrich上海贸易有限公司).
1.2.1 构建动物模型及收集标本: 所有大鼠共计12只被随机分为两组, 一组为正常饮食组, 即对照组(n1 = 6). 另一组为1, 2-二甲肼(1, 2-dimethylhydrazine, DMH)诱导肠癌组[10], 即实验组(n2 = 6). 两组均给予啮齿类动物基础饲料. 所有大鼠自由饮食、水1 wk, 以适应周围环境. 随后, 对实验组大鼠腹腔注射DMH(剂量为40 mL/kg, 1次/wk), 连续注射10 wk. 对照组大鼠腹腔注射生理盐水和乙二胺四乙酸(ethylene diamine tetraacetic acid, EDTA). 12 wk肿瘤形成后(肿瘤组织由病理切片证实), 取其肠道内粪便, 无菌操作收集于冻存管中, 置于-80 ℃冰箱中保存(实验组标记为T1-T6, 对照组标记为N1-N6). 该实验已获得上海市第六人民医院医学伦理委员会批准.
1.2.2 样品DNA的制备: 离心收集菌体, 溶于5 mL提取缓冲液中, 37 ℃振荡45 min. 加入0.75 mL 20%SDS, 65 ℃水浴1 h. 12000 r/min, 离心10 min, 收集上清. 上清用等体积的酚: 氯仿: 异戊醇(25:24:1)抽提2次, 加入终浓度0.3 mol/L醋酸钠(pH 5.2)及2倍体积的无水乙醇, 室温沉淀1 h. 4 ℃, 12000 r/min, 离心20 min, 收集沉淀, 并用700 mL/L乙醇漂洗2次, 晾干后溶于50 µL TE中.
1.2.3 16S rRNA gene V3区扩增: 所用引物为细菌16S rRNA V3高变区F357和R518(表1), 反应体系总体积为50 µL, ddH2O 41.25 µL, 10×Buffer(含2.0 mmol/L MgCl2)5 µL, dNTP(10 mmol/L)1 µL, F357-GC(10 µmol/L)1 µL, R518(10 µmol/L)1 µL, Taq酶(5 U/µL)0.25 µL, 模板DNA 0.5 µL, 设置阴性对照. 反应程序按照以下进行: 94 ℃预变性4 min; 94 ℃ 0.5 min; 56 ℃ 1 min; 72 ℃ 0.5 min; 30个循环, 72 ℃延伸7 min. 取PCR产物各3 µL, 1.5%琼脂糖凝胶加5%Goldview染液, 120 V稳压电泳30 min, 用UVI凝胶成像系统拍照.
| 引物 | 序列 | 扩增片段大小 |
| F357-GC | 5'-CCCGCCGCGCCCCGCGCCCGGCCCGCCGCCCCCGCCCCCCTACGGGAGGCAGCAG-3' | 230 bp |
| R518 | 5'-ATTACCGCGGCTGCTGG-3' |
1.2.4 变性梯度凝胶电泳(DGGE): 取样品各400 ng的V3区PCR产物, 采用D-Code突变检测系统对样品进行DGGE分析. 所用的聚丙烯酰胺凝胶浓度为8%(丙稀酰胺: 双丙稀酰胺 = 37.5:1), 变性剂浓度从30%-60%(100%的变性剂为7 mol/L尿素+40%甲酰胺). 在180 V电压下, 60 ℃恒温, 1×TAE中电泳4.0 h. 电泳完毕后, 用超纯水冲洗胶, 然后将胶放进染液中, 置于摇床上染色后, 在UVI成像系统拍照.
1.2.5 PCR扩增及产物回收及克隆: 选取较有代表性的条带, 用洁净的手术刀片将目标DGGE条带完整的切下并装入1.5 mL离心管中, 按UNIQ-10柱式DNA胶回收试剂盒回收. 对目的片段进行克隆测序, 与已知GenBank比对获得菌群的种属名称.
统计学处理 对DGGE凝胶图进行相似性和多样性分析. 相似性分析采用聚类分析, 使用非加权组平均法(unweighted pair-group method with arithmetic means, UPGMA)计算系统发育图[11]; 多样性分析采用丰富度和生物多样性分析. 丰富度(S)用DGGE图谱每条泳道的条带数表示; Shannon-Wiener指数(H')的计算方法为H' = -∑si = 1(Pi)(lnPi) , Pi为第i条带的吸光度与该泳道所有条带吸光度总和的比值, S为总的条带数目; 均一性(E)表示菌群种属分布的一致性, 计算方法为E = H'/㏑S[12]. Simpson多样性指数(D)计算方法为D = 1-∑si = 1 (Pi)2 , Pi及S计算方法同上, 该指数意义与Shannon指数相似[13]. 分析所得数据使用SPSS19.0进行Student-t检验, 计量资料结果以mean±SD表示. P<0.05为差异有统计学意义.
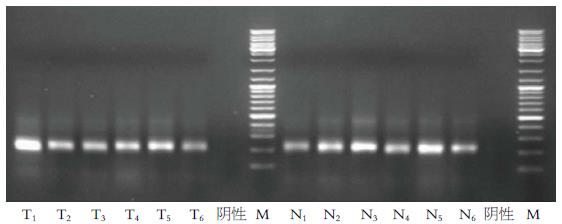
PCR扩增结果示各组样本目标条带及大小合适(图2). 在DGGE指纹图谱中每一条泳道代表一个样本中的菌群(图3), 不同位置的条带代表不同的优势菌群. PCR-DGGE电泳结果显示对照组大鼠粪便中菌群与罹患肠癌的大鼠粪便中的菌群均有较好的多样性, 且两者的均一性均较好. Shannon-Wiener指数(H')和Simpson指数(D)分析实验组菌群多样性高于对照组, 但二者并无统计学上的差异(P>0.05)(表2).
| 多样性指数 | 实验组 | 对照组 | P值 |
| 丰富度(S) | 25.833±5.845 | 21.500±2.665 | 0.142 |
| 均一性(E) | 1.000 | 1.000 | - |
| Shannon-Wiener指数(H') | 3.231±0.222 | 3.062±0.122 | 0.133 |
| Simpson指数(D) | 0.960±0.089 | 0.953±0.060 | 0.149 |
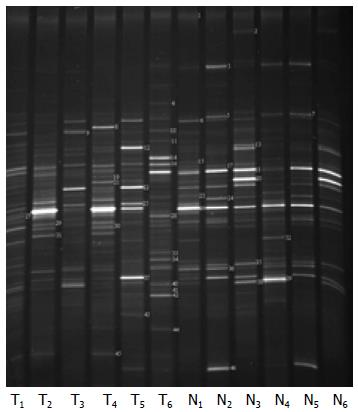
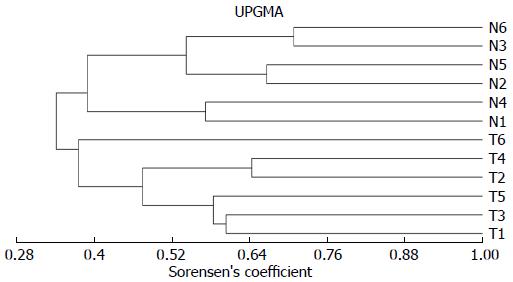
根据DGGE指纹图谱条带的亮度不同, 说明代表该样本中菌群的相对含量也不相同. 由此, 我们筛选了10组处于同一位置但亮度明显有差异(甚至消失)的条带进行菌群鉴定, 这些菌群主要分布在厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)(表3). 其中, 实验组中毛螺菌科(Lachnospiraceae)、Ruminococcaceae科菌、肠乳杆菌(Lactobacillus intestinalis)、帕拉普氏菌属(Paraprevotella)、鼠乳杆菌(Lactobacillus murinus)、乳杆菌属(Lactobacillus)、普氏菌属(Prevotella)、卷曲乳杆菌(Lactobacillus crispatus)等菌群丰度显著降低, 而粪芽孢菌属(Coprobacillus)丰度则明显增加. 此外, 对两组大鼠粪便菌群DGGE的图谱进行聚类分析, 结果表明实验组和对照组之间菌群相似度较低(图4).
| 条带编号 | 菌群(门) | 菌群(属) | 种属相似性(%) | V3区大小(bp) |
| 3 | Spirochaetes | Treponema amylovorum | 82 | 194 |
| 7 | Firmicutes | unclassified_Ruminococcaceae | 90 | 172 |
| 12 | Firmicutes | Lactobacillus crispatus | 93 | 193 |
| 16 | Firmicutes | Coprobacillus1 | 95 | 193 |
| 18 | Firmicutes | Lachnospiracea incertae sedis | 97 | 168 |
| 20 | Bacteroidetes | Prevotella | 91 | 189 |
| 22 | Firmicutes | Lactobacillus intestinalis | 100 | 194 |
| 26 | Bacteroidetes | Paraprevotella | 100 | 188 |
| 27 | Firmicutes | Lactobacillus murinus | 100 | 194 |
| 35 | Firmicutes | Lactobacillus | 96 | 194 |
人体肠道含有复杂的微生态系统, 他是由细菌、古细菌、真菌以及病毒等一系列微生物组成[14,15]. 肠道内微生物可以看作是人体这个"大器官"内的一个"小器官", 他与人体共同进化, 逐渐形成共栖关系并维持肠道内环境的稳态[16]. 最近的研究揭示, 肠道菌群的确在肠癌的发生发展中具有重要的影响. Boleij等[17]研究显示感染牛链球菌的患者肠道中伴生腺瘤或癌的病例中位数高达60%, 而感染该链球菌Ⅰ型的患者罹患大肠癌的风险非常高(合并OR值为7.26). 检测结果也证实牛链球菌抗原RpL7/L12浓度在早期(Ⅰ/Ⅱ)肠癌患者中明显增高[18]. Kostic等[19]发现梭形杆菌属细菌(F. nucleatum、F. mortiferum和F. necrophorum)在肿瘤组织内数量明显多于正常组织, 且组织学和肿瘤转移灶梭形杆菌属DNA分析也证实了该结果. Tjalsma等[20]提出了一种大肠癌发生过程中细菌动态变化的模式, 其认为肠道内有一部分菌群属于"启动(driver)"菌群, 如肠杆菌科、梭状芽孢杆菌等, 他们能够损害肠上皮细胞DNA, 诱导大肠癌的发生. 还有一部分菌群属于"过路(passenger)"菌群, 他们能够适应肠道微环境的改变而大量增殖, 成为肠道内优势菌群, 包括梭形杆菌属、链球菌科等. 这些有害菌群通过刺激宿主免疫功能, 引起肠道炎症反应, 并通过诱导树突细胞和辅助性TH17细胞释放白介素(interleukin, IL)如IL-23、IL-17, 引起肠上皮细胞分泌β-防御素(βdefensins)、钙卫蛋白(calprotectin)和脂质运载蛋白-2(lipocalin 2)等抗菌物质, 或提供黏附界面, 帮助某些细菌定植于肿瘤微环境, 促进肿瘤生长.
本研究通过对正常组和实验组大鼠粪便菌群检测, 发现PCR-DGGE技术能够较清晰地分离出肠道内的主要菌群. 本试验两份样本均显示了良好的菌群多样性. 但聚类分析发现, 实验组大鼠粪便的菌群组成结构与正常组有明显的差异性. 分析可见, 两组之间出现最显著差异的为四种菌, 其中毛螺旋菌科、帕拉普氏菌属、乳杆菌属在对照组中均有明显表达, 而在实验组中出现不表达. 毛螺旋菌科主要包括丁酸弧菌属(Butyrivibrio)、毛螺菌属(Lachnospira)和罗氏菌属(Roseburia)[21]. 在炎症性肠病(innammatory bowel diseases, IBD)患者的肠道组织样本中, 毛螺旋菌科含量可降至正常含量的1/300[22]. 研究发现, 该科细菌具有水解多种植物多糖、脱乙酰基作用以降解毒素、拮抗艰难梭菌感染等作用[21,23]. 不过, 也有研究认为, 某些产丁酸盐的细菌(如毛螺菌科)丰度下降, 导致丁酸盐诱导凋亡和分化、抑制增生及血管形成等功能下降, 也可能间接促进了肿瘤的进展[24]. 帕拉普氏菌属为革兰染色阴性厌氧菌, 一般在动物的瘤胃内易分离出该菌[25,26]. 近期研究显示该菌也常存在于人群肠道中. 研究人员对人肠癌组织及癌旁组织对比显示, 帕拉普氏菌属在癌旁组织(距离癌组织10-20 cm)中丰度更高[27]. 这种丰度差异提示其在预防肿瘤发生发展方面具有一定作用. 乳杆菌属属于厚壁菌门, 是存在于人体内的益生菌. 研究显示乳杆菌属能够上调结肠癌细胞中肿瘤抑制基因, 抑制核因子κB抑制蛋白(nuclear factor of kappa light polypeptide gene enhancer in B-cells inhibitor, IκB)的降解, 降低β-葡萄糖醛酸酶和硝基还原酶的活力, 并且还能抵抗诱变剂突变的作用, 从而抑制炎症、阻碍病原微生物定植, 拮抗肿瘤的发生[28-31]. 而在本实验中, 由于这3种有益菌群数量大幅降低, 导致菌群明显失调, 可见其丰度降低与肿瘤的发生发展密切相关. 另外, 实验组还发现粪芽孢菌属在6组样本中均见明显条带, 而在对照组中条带消失. 该菌属于厚壁菌门, 既往研究发现该细菌常在肠道炎症组织和高脂饮食大鼠体内出现, 而这两者均为肠癌的高危因素, 因此根据本实验结果, 提示该菌其与肠癌的发生的相关性较高[3,32]. 因此, 本实验揭示了在正常组织中丰富较高的菌群(特别是乳杆菌属一般可达总数的10%-15%), 在肠癌组织中出现了不同程度的比例降低, 这与文献报道结果一致[33-35]. 实验组大鼠肠道内菌群谱明显紊乱, 除了受到大鼠个体的遗传性状的影响外, 还可能与肿瘤生长的微环境及肿瘤的进展阶段有关.
总之, 尽管本次实验样本数量较少, 但已基本阐明肠癌模型大鼠肠道内菌群与正常相比, 已发生了明显的偏移. 实验结果表明大量有益菌的下降, 有害菌的增殖, 通过多种途径直接或间接地促进了肿瘤的发生发展. 但是, 对肠道内肿瘤组织的侵犯程度与菌群的相关性、肠癌发生不同时期菌群的结构和数量等的动态变化以及微生态致肠癌的最终生物学机制仍需要进一步探索.
随着肠癌发生率的逐年增加, 人们对肠道内环境的研究愈加重视. 研究发现, 肠道微生态(microbiota)在肠癌的发生发展中具有重要作用.
白雪, 副主任医师, 中国人民解放军北京军区总医院普通外科; 侯风刚, 副主任医师, 副教授, 上海中医药大学附属市中医医院肿瘤科
基于PCR-DGGE技术对两种不同肠道微生态环境的大鼠菌群进行比较, 可以筛选出在肠癌发生发展过程中特异性的肠道菌群, 便于进一步研究其可能促进肠癌的机制.
目前已有研究发现肠道菌群与肥胖、糖尿病以及心血管疾病等多种疾病密切相关甚至是起到关键作用, 其可能机制与炎症及肠屏障功能损伤相关. 通过调节肠道菌群, 可以帮助恢复肠道内环境的稳态, 减少疾病的发生.
本文采用新型检测技术, 对肠道菌群进行分离、鉴定和对比, 初步揭示了大鼠肠癌模型中粪便菌群较正常菌群产生的偏移, 帮助理解肠癌发生发展过程中内环境的改变.
随着对肠道微生态及其致病机制的深入研究, 相关研究成果可以为预防和治疗肠癌提供新理论依据.
本文内容通过对正常大鼠肠道和结肠癌大鼠肠道内菌群的比较分析来研究结直肠癌肠内微环境的改变, 有较好的研究价值及临床指导意义.
编辑: 郭鹏 电编:鲁亚静
| 1. | Ferlay J, Shin HR, Bray F, Forman D, Mathers C, Parkin DM. Estimates of worldwide burden of cancer in 2008: GLOBOCAN 2008. Int J Cancer. 2010;127:2893-2917. [PubMed] [DOI] |
| 2. | Siegel R, Naishadham D, Jemal A. Cancer statistics, 2012. CA Cancer J Clin. 2012;62:10-29. [PubMed] [DOI] |
| 3. | Castellarin M, Warren RL, Freeman JD, Dreolini L, Krzywinski M, Strauss J, Barnes R, Watson P, Allen-Vercoe E, Moore RA. Fusobacterium nucleatum infection is prevalent in human colorectal carcinoma. Genome Res. 2012;22:299-306. [PubMed] [DOI] |
| 4. | Hamer HM, De Preter V, Windey K, Verbeke K. Functional analysis of colonic bacterial metabolism: relevant to health? Am J Physiol Gastrointest Liver Physiol. 2012;302:G1-G9. [PubMed] [DOI] |
| 5. | Zwielehner J, Lassl C, Hippe B, Pointner A, Switzeny OJ, Remely M, Kitzweger E, Ruckser R, Haslberger AG. Changes in human fecal microbiota due to chemotherapy analyzed by TaqMan-PCR, 454 sequencing and PCR-DGGE fingerprinting. PLoS One. 2011;6:e28654. [PubMed] [DOI] |
| 6. | Zoetendal EG, Rajilic-Stojanovic M, de Vos WM. High-throughput diversity and functionality analysis of the gastrointestinal tract microbiota. Gut. 2008;57:1605-1615. [PubMed] [DOI] |
| 7. | Maccaferri S, Biagi E, Brigidi P. Metagenomics: key to human gut microbiota. Dig Dis. 2011;29:525-530. [PubMed] [DOI] |
| 8. | Green SJ, Leigh MB, Neufeld JD. Denaturing Gradient Gel Electrophoresis (DGGE) for Microbial Community Analysis. Handbook of Hydrocarbon and Lipid Microbiology. 1st ed. Germany: Springer Berlin Heidelberg 2010; 4137-4158. |
| 9. | Dar SA, Kuenen JG, Muyzer G. Nested PCR-denaturing gradient gel electrophoresis approach to determine the diversity of sulfate-reducing bacteria in complex microbial communities. Appl Environ Microbiol. 2005;71:2325-2330. [PubMed] [DOI] |
| 11. | Fromin N, Hamelin J, Tarnawski S, Roesti D, Jourdain-Miserez K, Forestier N, Teyssier-Cuvelle S, Gillet F, Aragno M, Rossi P. Statistical analysis of denaturing gel electrophoresis (DGE) fingerprinting patterns. Environ Microbiol. 2002;4:634-643. [PubMed] [DOI] |
| 12. | Muyzer G, de Waal EC, Uitterlinden AG. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA. Appl Environ Microbiol. 1993;59:695-700. [PubMed] |
| 14. | Bäckhed F, Ley RE, Sonnenburg JL, Peterson DA, Gordon JI. Host-bacterial mutualism in the human intestine. Science. 2005;307:1915-1920. [PubMed] [DOI] |
| 15. | Reyes A, Haynes M, Hanson N, Angly FE, Heath AC, Rohwer F, Gordon JI. Viruses in the faecal microbiota of monozygotic twins and their mothers. Nature. 2010;466:334-338. [PubMed] [DOI] |
| 16. | Lepage P, Leclerc MC, Joossens M, Mondot S, Blottière HM, Raes J, Ehrlich D, Doré J. A metagenomic insight into our gut's microbiome. Gut. 2013;62:146-158. [PubMed] [DOI] |
| 17. | Boleij A, van Gelder MM, Swinkels DW, Tjalsma H. Clinical Importance of Streptococcus gallolyticus infection among colorectal cancer patients: systematic review and meta-analysis. Clin Infect Dis. 2011;53:870-878. [PubMed] [DOI] |
| 18. | Boleij A, Roelofs R, Schaeps RM, Schülin T, Glaser P, Swinkels DW, Kato I, Tjalsma H. Increased exposure to bacterial antigen RpL7/L12 in early stage colorectal cancer patients. Cancer. 2010;116:4014-4022. [PubMed] [DOI] |
| 19. | Kostic AD, Gevers D, Pedamallu CS, Michaud M, Duke F, Earl AM, Ojesina AI, Jung J, Bass AJ, Tabernero J. Genomic analysis identifies association of Fusobacterium with colorectal carcinoma. Genome Res. 2012;22:292-298. [PubMed] [DOI] |
| 20. | Tjalsma H, Boleij A, Marchesi JR, Dutilh BE. A bacterial driver-passenger model for colorectal cancer: beyond the usual suspects. Nat Rev Microbiol. 2012;10:575-582. [PubMed] [DOI] |
| 21. | Cotta M, Forster R. The Family Lachnospiraceae, Including the Genera Butyrivibrio, Lachnospira and Roseburia. The prokaryotes. 3th ed. USA: Springer US 2006; 1002-1021. |
| 22. | Frank DN, St Amand AL, Feldman RA, Boedeker EC, Harpaz N, Pace NR. Molecular-phylogenetic characterization of microbial community imbalances in human inflammatory bowel diseases. Proc Natl Acad Sci U S A. 2007;104:13780-13785. [PubMed] |
| 23. | Reeves AE, Koenigsknecht MJ, Bergin IL, Young VB. Suppression of Clostridium difficile in the gastrointestinal tracts of germfree mice inoculated with a murine isolate from the family Lachnospiraceae. Infect Immun. 2012;80:3786-3794. [PubMed] [DOI] |
| 24. | Fung KY, Cosgrove L, Lockett T, Head R, Topping DL. A review of the potential mechanisms for the lowering of colorectal oncogenesis by butyrate. Br J Nutr. 2012;108:820-831. [PubMed] [DOI] |
| 25. | Sakamoto M, Ohkuma M. Identification and classification of the genus Bacteroides by multilocus sequence analysis. Microbiology. 2011;157:3388-3397. [PubMed] [DOI] |
| 26. | Kim M, Morrison M, Yu Z. Status of the phylogenetic diversity census of ruminal microbiomes. FEMS Microbiol Ecol. 2011;76:49-63. [PubMed] [DOI] |
| 27. | Chen W, Liu F, Ling Z, Tong X, Xiang C. Human intestinal lumen and mucosa-associated microbiota in patients with colorectal cancer. PLoS One. 2012;7:e39743. [PubMed] [DOI] |
| 28. | Yang T, Owen JL, Lightfoot YL, Kladde MP, Mohamadzadeh M. Microbiota impact on the epigenetic regulation of colorectal cancer. Trends Mol Med. 2013;19:714-725. [PubMed] [DOI] |
| 29. | Candela M, Guidotti M, Fabbri A, Brigidi P, Franceschi C, Fiorentini C. Human intestinal microbiota: cross-talk with the host and its potential role in colorectal cancer. Crit Rev Microbiol. 2011;37:1-14. [PubMed] [DOI] |
| 30. | Grivennikov SI. Inflammation and colorectal cancer: colitis-associated neoplasia. Semin Immunopathol. 2013;35:229-244. [PubMed] [DOI] |
| 31. | Chong ES. A potential role of probiotics in colorectal cancer prevention: review of possible mechanisms of action. World J Microbiol Biotechnol. 2013; Sep 26. [Epub ahead of print]. [PubMed] [DOI] |
| 32. | Daniel H, Gholami AM, Berry D, Desmarchelier C, Hahne H, Loh G, Mondot S, Lepage P, Rothballer M, Walker A. High-fat diet alters gut microbiota physiology in mice. ISME J. 2013; Sep 12. [Epub ahead of print]. [PubMed] [DOI] |
| 33. | Greiner T, Bäckhed F. Effects of the gut microbiota on obesity and glucose homeostasis. Trends Endocrinol Metab. 2011;22:117-123. [PubMed] [DOI] |
| 34. | Tomas J, Langella P, Cherbuy C. The intestinal microbiota in the rat model: major breakthroughs from new technologies. Anim Health Res Rev. 2012;13:54-63. [PubMed] [DOI] |