修回日期: 2014-09-18
接受日期: 2014-09-21
在线出版日期: 2014-10-28
目的: 探讨结直肠癌(colorectal cancer, CRC)组织中microRNA(miRNA)的差异表达谱及其在CRC临床生物学特点判定中的检测意义.
方法: 应用AFFX miRNA芯片实验初步筛查手术切除的11例新鲜CRC及癌旁正常组织中miRNAs的差异表达, 并选取CRC中显著高表达的miR-31、miR-105和miR-196b, 以53例新鲜的CRC及其15例癌旁正常组织作为研究对象, 应用qRT-PCR技术对比分析其与临床生物学之间的关系.
结果: CRC和癌旁正常组织中miRNAs表达有明显差异, 与癌旁正常组织相比, CRC中有25个miRNAs表达显著不同(P<0.001), 包括15个上调和10个下调的miRNAs. miR-31与CRC的病理分型(P<0.01)和临床分期(P<0.05)显著相关, 与淋巴转移及远处转移不相关(P>0.05); miR-105与病理分型、临床分期、淋巴结转移及远处转移均呈正相关(P<0.05); miR-196b与病理分型有关(P<0.05), 与淋巴结转移和远处转移及临床分期均无关(P>0.05).
结论: CRC中存在着明显的miRNAs差异表达, miRNAs特异表达可能成为CRC的潜在的诊断和治疗的分子靶点. miR-31、miR-105和miR-196b高表达与CRC的病理分型密切相关.
核心提示: 本文应用AFFX microRNA(miRNA)芯片实验初步筛查结直肠癌(colorectal cancer, CRC)及癌旁正常组织中miRNAs的差异表达, 应用qRT-PCR技术对比分析其与临床生物学之间的关系. 显示CRC中存在着明显的miRNAs差异表达, miRNAs特异表达可能成为CRC的潜在的诊断和治疗的分子靶点. miR-31、miR-105和miR-196b高表达与CRC病理分型密切相关.
引文著录: 张媛, 林贞花, 金铁峰, 金仁顺. 结直肠癌中miRNAs差异表达谱的筛查. 世界华人消化杂志 2014; 22(30): 4578-4587
Revised: September 18, 2014
Accepted: September 21, 2014
Published online: October 28, 2014
AIM: To examine the expression of microRNAs (miRNAs) in colorectal cancer (CRC) and adjacent tissues by miRNA gene microarray, determine some cellular functions and molecular pathways targeted by these differentially expressed miRNAs, and discuss the clinical significance of differential miRNA expression profile in CRC.
METHODS: Surgical CRC and adjacent tissues were collected from 11 CRC patients and frozen immediately. For microarray analysis, the AFFX miRNA biochip was used. Total RNA was isolated from samples and expression of miRNAs was analyzed. Of the screened differentially expressed miRNAs, miR-31, miR-105 and miR-196b, which were strongly up-regulated in CRC, were selected for validation by real-time PCR using 53 CRC and 15 adjacent tissues. The relationships between the differential expression of these miRNAs and clinical and pathological characteristics were analyzed.
RESULTS: MiRNA expression in CRC and adjacent normal tissues had obvious differences. A total of 25 miRNAs that were significantly differentially expressed in CRC were screened (P < 0.001), of which 15 were up-regulated and 10 down-regulated. MiR-31 expression was significantly correlated with CRC pathological type (P < 0.01) and clinical stage (P < 0.05), but not with lymph node metastasis or distant metastasis (P > 0.05). MiR-105 was positively correlated with pathologic classification, clinical stage, lymph node metastasis and distant metastasis (P < 0.05). MiR-196b was significantly correlated with pathological classification (P < 0.05), but not with lymph node metastasis, distant metastasis or clinical stage (P > 0.05).
CONCLUSION: Differentially expressed miRNAs exist in CRC, and these miRNAs may become potential molecular targets for diagnosis and treatment of CRC. High expression of miR-31, miR-105 and miR-196b is related to the pathological classification of CRC.
- Citation: Zhang Y, Lin ZH, Jin TF, Jin RS. MicroRNA expression profile in colorectal cancer. Shijie Huaren Xiaohua Zazhi 2014; 22(30): 4578-4587
- URL: https://www.wjgnet.com/1009-3079/full/v22/i30/4578.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v22.i30.4578
结直肠癌(colorectal cancer, CRC)是一种常见的消化系恶性肿瘤. 其发病与高脂肪和缺少膳食纤维的不良饮食结构有关. 随着我国人民群众生活水平的大幅提高和生活方式的西化, CRC的发病率逐年提高. 近年, CRC在我国发病率已经上升到第5位[1], 死亡率也居第5位[2]. 根据世界其他国家CRC发生发展的变化规律, 结合我国各地区近年来癌症发病率动态, CRC将成为21世纪我国发病率上升速度最快的恶性肿瘤之一. 因此, 寻找能够早期诊断和判断预后的标志物, 对CRC的诊断及治疗具有重大意义. microRNAs(miRNAs)属于内源性非蛋白质编码小RNA(small non-protein-coding RNA).
miRNA最早是在分析线虫遗传学信息时发现[3,4], 并命名为lin-14. 在细胞的生长、组织分化、各种疾病的发生、甚至在肿瘤的发生发展中miRNA都发挥着极为重要的作用. miRNA的研究尚处于起步阶段, miRNA的表达与结肠癌的发生发展以及浸润和转移之间到底存在着什么样的具体关系, 目前还不是十分明了. 本研究拟采用miRNA芯片及RT-PCR等研究方法, 通过对比研究CRC及其癌旁正常组织中miRNAs的差异表达, 初步探讨CRC组织中miRNA的差异表达谱及其在CRC临床生物学特点判定中的检测意义.
收集的64例的CRC组织及其癌旁正常组织均来源于延边大学医院普外科手术切除的具备完整临床资料的标本. 手术切除的CRC组织及其癌旁正常组织(距离癌组织10 cm以上)于5 min内切取大小约0.5 cm3, 立即送入液氮冰冻, 于-80 ℃冰箱长期保存待用. 所用CRC病理组织标本另取石蜡包埋及常规HE染色后, 由两位经验丰富的病理医生参照2003年世界卫生组织肿瘤分类标准进行诊断和分类(表1). 总RNA提取试剂盒购自美国Invitrogen公司; miRNA提取试剂盒购自美国Ambion公司; 基因芯片购自美国Affymetrix公司; 反转录试剂盒及PCR酶、ExTaq酶购自日本TaKaRa公司; GeneChip® Command ConsoleTM1.1分析软件购自美国Affymetrix公司.
| 临床资料 | 基因芯片用标本 | RT-PCR用标本 |
| n | 11 | 53 |
| 性别 | ||
| 男 | 5 | 29 |
| 女 | 6 | 24 |
| 年龄(岁) | ||
| 中位(范围) | 57(47-82) | 61(41-83) |
| 病理分级 | ||
| 高 | 2 | 23 |
| 中 | 8 | 12 |
| 低 | 1 | 18 |
| 临床分期 | ||
| Ⅰ-Ⅱ | 3 | 26 |
| Ⅲ-Ⅳ | 8 | 27 |
| 淋巴转移 | ||
| 有 | 7 | 31 |
| 无 | 4 | 22 |
| 远处转移 | ||
| 有 | 1 | 11 |
| 无 | 10 | 42 |
| 部位 | ||
| 结肠 | 2 | 20 |
| 直肠 | 9 | 33 |
1.2.1 AFFX miRNA芯片实验: 手术切除的CRC组织及其癌旁正常组织(距离癌组织10 cm以上)于5 min内切取大小约0.5 cm3, 立即送入液氮冰冻, 于-80 ℃冰箱长期保存待用. 64例手术切除标本包括用于基因芯片筛查的11例和用于Real-time PCR检测的53例. 总RNA的提取: (1)从-80 ℃冰箱取出标本, 切成50-100 mg的小块, 立即投入到放置在冰上研钵中研磨, 研成粉末; (2)加入1/10体积的TRIzol RNA提取液裂解细胞, 转入离心管, 室温静置5 min, 4 ℃, 12000 r/min离心10 min, 取上清; (3)加1/5体积(200 μL)氯仿振荡15 s混匀后, 室温静置15 min, 4 ℃, 12000 r/min离心15 min, 离心后TRIzol抽提混合液分成3层, 上层为无色透明水相, 中层为薄层蛋白, 下层为粉红色有机相, 取上清; (4)加1/5体积(500 μL)异丙醇上下颠倒3-4次混匀, 室温静置10 min, 4 ℃, 12000 r/min离心10 min, 弃上清, RNA沉于离心管底部; (5)加1体积(1 mL)750 mL/L乙醇悬浮沉淀, 4 ℃, 1000 r/min离心5 min, 弃上清, 自然干燥; (6)加50 μL TE Buffer超纯净水充分溶解, 利用NanoDrop超微量分光光度仪测RNA浓度及质量. 总RNA的质量分析: 利用NanoDrop超微量分光光度仪测RNA浓度及质量. 提取的每份样品总RNA含量≥5 μg, 测定A260/A280>1.9, 且电泳结果显示总RNA有清晰的28S和18S条带, 28S:18S条带亮度大于或近2:1者, 用于本实验的基因芯片检测及Real-time PCR检测. 稀释ATP Mix; Poly(A)加尾; 生物素标记miRNA; 芯片杂交; 芯片清洗染色、扫描、应用GeneChip® Command ConsoleTM1.1软件对芯片图像进行数据分析.
1.2.2 荧光定量实时聚合酶链反应验证hsa-miR-31, hsa-miR-105和hsa-miR-196b芯片结果: 以逆转录获得的cDNA为模版, 反应条件: 95 ℃ 30 s, 95 ℃ 5 s, 60 ℃ 20 s, 72 ℃ 20 s, 共循环45次, 在11例CRC标本中共检测在芯片结果中表现为上调的6种以及下调的4种miRNAs的表达水平进行验证. 计算方法为2-△△Ct相对定量法. 从中选择hsa-miR-31、hsa-miR-105和hsa-miR-196b共3种miRNAs作为进一步研究的对象, 在53例CRC及其15例癌旁正常组织中进行Real-time PCR检测. 其引物序列如表2. 反应体系(反应液配制在冰上进行): PCR反应五要素: 参加PCR反应的物质主要有5种即引物、酶、dNTP、模板和Mg2+ 10倍浓缩扩增缓冲液10 μL, 4种dNTP混合物各200 μmol/L 800 μL, 引物各10-100 pmol, 模板DNA 0.1-2.0 μg, Taq DNA聚合酶2.5 U, Mg2+ 1.5 mmol/L, 加双或三蒸水至100 μL. 反应条件: (1)95 ℃ 30 s, 起始模板预变性, 循环1次; (2)95 ℃ 5 s, 循环中模板变性; (3)60 ℃ 20 s, 退火; (4)72 ℃ 20 s, 延伸, 反应共循环45次; (5)溶解曲线分析, 由55 ℃以0.1 ℃/s上升至95 ℃, 最后冷却至40 ℃. 反应结果判断: (1)确认Real-time PCR反应的扩增曲线及溶解曲线, 判断扩增效率及特异性; 调节基线至适宜处(指数扩增的起始处), 各扩增曲线与基线的交叉点对应的横坐标即为Ct值; (2)miRNA计算方法为2-△△Ct相对定量法.
| 基因名称 | 引物 | 序列 5'-3' | |
| hsa-miR-31 | RT-Primer | GTCGTATCCAGTGCAGGGTCCGAGG | |
| TATTCGCACTGGATACGACAGCTAT | |||
| PCR-Primer | Forward Primer | AGGCAAGATGCTGGCATAGCT | |
| Reverse Primer | CAAGGCAAGATGCTGGCATA | ||
| hsa-miR-105 | RT-Primer | GTCGTATCCAGTGCAGGGTCCGAGG | |
| TATTCGCACTGGATACGACACAGGA | |||
| PCR-Primer | Forward Primer | TCAAATGCTCAGACTCCTGT | |
| Reverse Primer | GCGTCAAATGCTCAGACTCC | ||
| hsa-miR-196b | RT-Primer | GTCGTATCCAGTGCAGGGTCCGAGG | |
| TATTCGCACTGGATACGACGAAGGC | |||
| PCR-Primer | Forward Primer | TCGACAGCACGACACTGCCTTC | |
| Reverse Primer | TCGACAGCACGACACTGCC | ||
| U6 | RT-Primer | AACGCTTCACGAATTTGCGT | |
| PCR-Primer | Forward Primer | CTCGCTTCGGCAGCACA | |
| Reverse Primer | GTGCAGGGTCCGAGGT | ||
统计学处理 实验数据采用SPSS16.0软件进行统计学分析. 计量资料采用秩和检验, 以中位数(四分位数间距)来描述, 计数资料采用两独立样本的χ2检验和Fisher确切概率法分析. 以α = 0.05的双尾概率作为统计学检验的显著性标准, P<0.05为差异有统计学意义.
选取经质检合格的11例CRC和其配对癌旁正常组织抽提的RNA寡核苷酸基因芯片杂交结果显示, 有25种miRNAs在CRC与癌旁正常组织相比存在明显差异表达. 其中表达上调的15个, 表达下调的10个(P<0.001). 相对于癌旁正常组织, CRC组织中表达上调的有hsa-miR-31、hsa-miR-767-5p、hsa-miR-105、hsa-miR-196b、hsa-miR-224、hsa-miR-483-5p、hsa-miR-1246、hsa-miR-196b、hsa-miR-663b、hsa-miR-584、hsa-miR-141、hsa-miR-18a、hsa-miR-19a、hsa-miR-452、hsa-miR-622. 表达下调的是hsa-miR-215、hsa-miR-4298、hsa-miR-139-5p、hsa-miR-490-5p、hsa-miR-3201、hsa-miR-363、hsa-miR-187、hsa-miR-133a、 hsa-miR-1825、hsa-miR-30a(表3, 4).
| miRNA | log2Ratio |
| hsa-miR-31 | 21.0559 |
| hsa-miR-767-5p | 13.9791 |
| hsa-miR-105 | 11.7936 |
| hsa-miR-196b | 9.288 |
| hsa-miR-224 | 9.1227 |
| hsa-miR-483-5p | 9.0639 |
| hsa-miR-1246 | 7.6698 |
| hsa-miR-196b | 6.815 |
| hsa-miR-663b | 6.5095 |
| hsa-miR-141 | 6.1256 |
| hsa-miR-584 | 5.6565 |
| hsa-miR-18a | 5.65 |
| hsa-miR-19a | 5.3913 |
| hsa-miR-452 | 5.2654 |
| hsa-miR-622 | 5.2073 |
| miRNA | log2Ratio |
| hsa-miR-215 | -15.87301587 |
| hsa-miR-4298 | -6.075334143 |
| hsa-miR-139-5p | -6.042296073 |
| hsa-miR-490-5p | -5.586592179 |
| hsa-miR-3201 | -4.518752824 |
| hsa-miR-363 | -3.976143141 |
| hsa-miR-187 | -3.755163335 |
| hsa-miR-133a | -3.946329913 |
| hsa-miR-1825 | -3.6900369 |
| hsa-miR-30a | -3.584229391 |
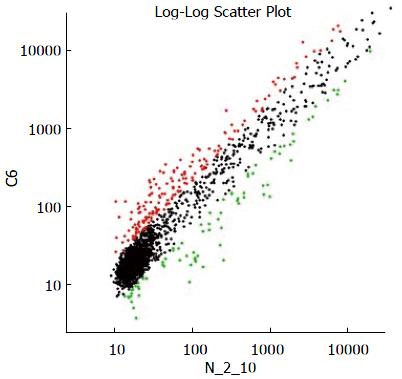
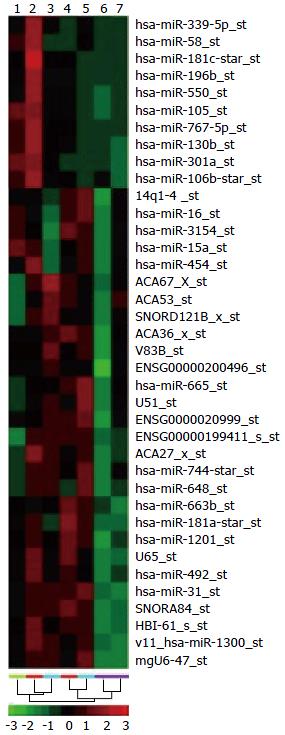
从聚类图及log-log scatter plot(图1, 2)可以看出与癌旁正常组织比较, CRC组织中的miRNA表达有显著性差异; 与癌旁正常组织相比, 其中hsa-miR-31、hsa-miR-105、hsa-miR-196b的表达在CRC组织中显著增高.
图1中可以直观地看出在两个样品之间基因表达的差异情况. 其中X轴和Y轴分别以两个样品的荧光信号强度值为坐标, 图中每一个数据点代表芯片上一个基因点的杂交信号, 红色标记和绿色标记的数据点分别表示C6/N_2_10(B/A)的比值≥2和≤0.5, 属于表达有差异的基因(红点表示CRC中miRNA表达升高, 绿点表示CRC中miRNA表达降低), 黑色标记表示B/A的Ratio值在0.5和2之间, 表达基本无差异.
从聚类图中可以看出相比于癌旁正常组织, 在CRC组织中miR-31、miR-105和miR-196b均为显著高表达, 差异有统计学意义(P<0.05).
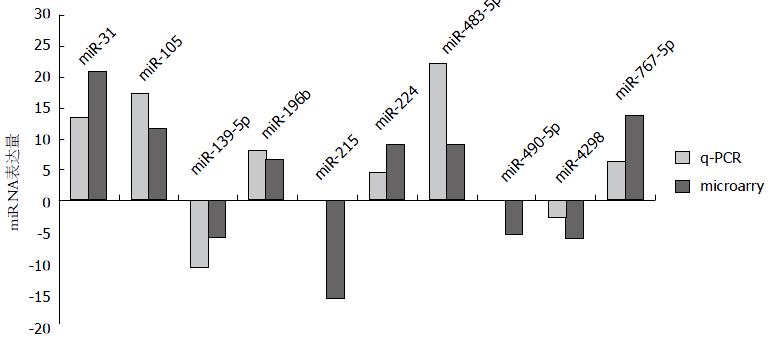
本实验进一步采用荧光定量PCR检测了芯片中明显异常表达的10个miRNAs包括表达上调的6个miRNAs, 分别为miR-31、miR-105、miR-196b、miR-224、miR-483-5p和miR-767-5p, 表达下调的4个miRNAs是miR-139-5p、miR-215、miR-490-5p和miR-4298(图3). 发现与miRNA芯片检查的结果基本吻合, 表明miRNA芯片结果真实可靠. q-PCR与miRNA microarry比较分析结果说明, 在miR-31、miR-105、miR-196b均显示出几乎相等的miRNA表达增高.
在53例新鲜的CRC及其15例癌旁正常组织中提取总RNA, 应用Real- time quantitative PCR(q-PCR)方法比较分析了miR-31、miR-105和miR-196b表达与CRC临床生物学特点之间的关系. 发现在癌组织中miR-31、miR-105和miR-196b表达均较癌旁正常组织明显升高, 差异具有统计学意义(P<0.01).
在53例CRC患者中, 高分化23例, 中分化12例, 低分化18例. q-PCR结果显示, 分化越低miR-31、miR-105、miR-196b的mRNA表达水平越高. 低分化组mRNA平均表达量较高分化组的及中分化组mRNA平均表达量明显升高, 差异具有统计学意义(P<0.05). 但中分化组与高分化组中上述3种miRNAs的mRNA表达量比较分析发现没有统计学差异(P>0.05).
在53例CRC中, 有淋巴结转移的22例, 无淋巴结转移的31例. miR-105在有淋巴转移组中与在无淋巴转移组中的mRNA平均表达量相比, 差异有统计学意义(P<0.01). 而miR-31、miR-196b在有淋巴转移组中与无淋巴转移组中mRNA平均表达量相比, 差异没有统计学意义(P>0.05).
53例CRC中有远处转移的11例, 没有远处转移的42例. miR-105在有转移组中与无转移组的mRNA平均表达量相比, 差异有统计学意义(P<0.01). 而miR-31、miR-196b在有远处转移和没有转移组中的mRNA平均表达量相比, 差异无统计学意义(P>0.05).
在53例CRC中, 临床分期为Ⅰ期的有11例, Ⅱ期15例, Ⅲ期16例, Ⅳ期11例. miR-31在Ⅲ期患者中mRNA平均表达量较Ⅰ期患者的明显升高; Ⅳ期患者中的mRNA平均表达量较Ⅰ期患者的明显升高, 差异有统计学意义(P<0.05). miR-105在Ⅲ期患者中的mRNA平均表达量较Ⅰ期患者的明显升高, 差异有统计学意义(P<0.05); 在Ⅳ期患者中的mRNA平均表达量较Ⅰ期患者中的明显升高, 差异有统计学意义(P<0.01). 而miR-196b在Ⅲ期、Ⅳ期患者中的mRNA平均表达量与Ⅰ期相比, 均无统计学差异(P>0.05)(表5).
| 分组 | n | miR-31 | miR-105 | miR-196b |
| 结直肠癌组织 | 53 | 18.40(14.8)b | 30.56(16.08)b | 19.38(12.42)b |
| 癌旁正常组织 | 15 | 5.73(3.21) | 6.62(4.12) | 4.20(2.51) |
| 病理分级 | ||||
| 高 | 23 | 10.32(11.86)d | 19.35(7.16)c | 11.32(3.31)c |
| 中 | 12 | 11.20(4.84)c | 22.30(9.73)c | 12.30(4.42)c |
| 低 | 18 | 20.83(6.8) | 32.04(4.22) | 22.07(4.3) |
| 淋巴结转移 | ||||
| 阴性 | 22 | 15.21(8.86) | 18.66(3.4) | 15.02(7.62) |
| 阳性 | 31 | 17.05(9.79) | 32.03(7.14)f | 19.10(7.69) |
| 远处转移 | ||||
| 无 | 42 | 13.02(8.01) | 19.23(2.3) | 14.90(6.63) |
| 有 | 11 | 17.23(13.79) | 30.07(11.99)h | 18.93(9.73) |
| 临床分期 | ||||
| Ⅰ | 11 | 10.07(7.81) | 17.02(4.38) | 13.16(4.82) |
| Ⅱ | 15 | 13.23(7.01) | 18.52(5.08) | 12.05(4.93) |
| Ⅲ | 16 | 18.06(9.74)i | 25.37(5.17)i | 17.83(7.78) |
| Ⅳ | 11 | 17.23(5.96)i | 32.20(13.87)j | 19.42(11.73) |
CRC在不同地区发病率不同. 由世界肿瘤流行病学调查统计显示, CRC在美洲的北部、欧洲西部、澳大利亚及新西兰等地区的发病率最高, 美洲南部、非洲等地则比较低(http://baike.baidu.com/view/595143.htm). 通常CRC的发病率与人群的生活水平呈正相关. 虽然我国仍属于发展中国家, 但随着经济的高速发展, , 近年来CRC呈明显的上升趋势, 且其分布表现为以东部沿海地区高发, 西部经济欠发达地区发病率较低. 在研究中发现CRC的发病, 男女比约为1.6:1, 从发病年龄看, 在我国40-50岁发病率较高, 欧美发达国家CRC高发年龄为60-70岁. 资料显示我国CRC好发年龄比国外CRC好发年龄提前10-15岁, 11%-13%是年龄为30岁以下的患者, CRC好发年龄较小是我国CRC的一个主要特点.
针对CRC的早期诊断、早期筛查目前仍没有比较有效的方法. Levin等[5]指出大便潜血试验(fecal occult blood test, FOBT)和结肠镜检查在CRC的早期诊断中最敏感, 最常用. 黄彦钦等[6]在杭州地区对40-74岁之间人群共43713名在籍人口, 进行了包括问卷调查与大便潜血试验检测组成的初筛, 阳性者进行结肠镜诊断性复筛, 发现息肉样病变后取活检进行病理诊断. 分析结果高危人群为14.85%, 总体样本中癌的检出率为1.11%. 初筛结果令人满意. 但此方案也存在一些问题, 比如由于问卷调查的主观性较强, 出现了腺瘤和癌的检出率问卷调查明显低于FOBT者, 结肠镜检操作者操作水平参差不齐, 容易出现漏诊等情况. 此外此方案在鉴别不同年龄不同性别人群CRC风险的判定上仍有欠缺. 加之肿瘤检出的敏感性、特异性以及患者的经济负担等均需审慎考虑. 有学者提出[7], 为了早期诊断预防CRC, 50岁以上健康人群每10年要做一次结肠镜检查. 这对于人口庞大的中国来说, 显然每年要产生一个庞大的筛查群体. 考虑其工作量, 目前国内的医疗资源及经费问题, 其执行难度可想而知. 一种针对CRC的迅速、准确、非侵入性、价格低廉的筛查方法呼之欲出.
以往研究发现蛋白编码基因的异常会导致肿瘤的发生、发展. 自2002年Croce研究组首次报道miRNA表达异常与肿瘤相关以来, miRNA的研究日新月异, 越来越多的研究发现miRNA的表达异常几乎与所有人类肿瘤相关.
内源性非蛋白质编码小RNA(small non-protein-coding RNA, 19-25 nt)广泛存在于高等和低等生物体内, 通过对靶标miRNA直接切除或抑制其翻译在转录及转录后水平对基因表达起调节作用. 已知的小RNA主要分为两大类: 一类是miRNA, 一类是小干扰RNA(small interfering RNA, siRNA). 在1993年Lee等[3]在对线虫进行遗传分析时发现了第一个miRNA, 即Lin-4. 有学者指出[8,9], 在CRC的早期检查中, miRNA的筛查可能成为可靠的诊断和预后指标.
在已确认的人类miRNA中, 50%以上miRNAs位于或靠近基因组中肿瘤相关的脆性区域(fragile sites), 即经常发生缺失、扩增、易位的染色体片段, 而这些脆性区域与肿瘤的发生有着密切的关系[10]. 如在慢性淋巴细胞性白血病中miR-15a/miR-16-1的表达下调, 且常常存在于易于发生缺失的13q14; miR-21位于染色体的脆性区域17q23在淋巴瘤中上调的miR-17-92表达簇, 位于肿瘤中经常发生扩增的13q31[11].
miRNA主要是通过干扰靶mRNA的翻译及转录, 使其降解或被抑制, 表现为mRNA的"沉默"及相应靶蛋白的表达下调. miRNA通过调控靶蛋白在生物体的生命活动以及各种疾病的发生中行使着相应的功能. 在肿瘤的发生发展中, 根据靶蛋白的性质不同, miRNA也可以成为癌基因或抑癌基因. 众多学者发表了关于miRNA在CRC的差异表达[12-16]. miRNA表达谱的建立不仅有助于识别正常组织和早期新发恶性肿瘤, 在对晚期的恶性肿瘤进行临床分期, 判定肿瘤类型, 甚至对治疗及预后均有着重要的参考价值和指导意义. Lu等[17]报道, 通过miRNA表达谱诊断肿瘤的准确率高达70%[18]. 基于不同miRNA在不同肿瘤之中的特异性差异表达, 通过高通量筛查miRNA, 建立人类miRNA表达谱, 以miRNA为指标的肿瘤诊断及评判预后的新方法成为全世界肿瘤学者高度关注的热点问题.
miRNA微阵列(miRNA microarrway)技术是一种快速有效的检测miRNA表达谱的方法. Liu等[19]首次应用这一方法检测了不同肿瘤细胞中245个miRNA的表达谱, 结果重复性很好, 并被Northern blot所验证, 目前已得到大范围的推广.
本研究采用基因芯片微阵列技术, 高通量筛查11例CRC和其11例癌旁对照组织抽提的RNA表达谱, 寡核苷酸基因芯片杂交结果显示: 有25种miRNAs在CRC和其癌旁正常对照组织中存在显著性差异表达. 其中表达增高15个, 表达下调10个(P<0.001). 相对于癌旁正常对照组织, 在CRC组织中表达上调的有hsa-miR-31、hsa-miR-767-5p、hsa-miR-105、hsa-miR-196b、hsa-miR-224、hsa-miR-483-5p、hsa-miR-1246、hsa-miR-196b、hsa-miR-663b、hsa-miR-584、hsa-miR-141、hsa-miR-18a、hsa-miR-19a、hsa-miR-452、hsa-miR-622. 表达下调的是hsa-miR-215、hsa-miR-4298、hsa-miR-139-5p、hsa-miR-490-5p、hsa-miR-3201、hsa-miR-363、hsa-miR-187、hsa-miR-133a、hsa-miR-1825和hsa-miR-30a.
在上述表达增高的miRNA中发现, miR-31是CRC组织与癌旁正常对照组织相比表达差异最大的miRNA. 据研究表明miR-31的表达与肿瘤的关系存在着相当大的组织学差异性. 如在CRC中miR-31的表达明显升高, 与我们的研究基本一致. 其他如头颈部鳞癌[20]、口腔鳞癌[21]、肺癌[22]的表达也是升高的. 但同时在乳腺癌[23]、胃癌[24,25]、浆液性卵巢癌[26]、前列腺癌[27]等组织中的表达却是明显下调. 类似的现象也出现在miR-196b的表达之中. 本实验发现, miR-196b在人CRC中具有显著的表达升高, 证明其高表达与肿瘤的发生呈正相关. 而Liao等[28]研究证明miR-196b作为抑癌基因, 可以抑制胃癌的发生. Bhatia等[29]报道miR-196b的高表达具有显著的抑制T细胞急性淋巴细胞白血病发生的作用. Bhatia等[30]同时报道, miR-196b在B细胞急性淋巴细胞白血病的抑制作用. Li等[31]发现, miR-196b可以有效抑制乳腺癌细胞株的转移. 更为有趣的是Li等[32]在白血病的最新研究中发现miR-196b的高表达不仅可以抑制作为癌基因的HOXA9/MEIS1也可以抑制抑癌基因Fas的表达. 本研究证明miR-196b在CRC中是癌基因, 而在白血病及胃癌中却是抑癌基因. miR-196b在肿瘤中的表达有着较为明确的组织特异性, 这种特异性的来源目前尚不清楚, 还有待于进一步研究.
迄今为止, 有关miR-105与肿瘤发生的关系报道甚少, 与CRC的相关性则未见报道. Benakanakere等[33]报道在口腔角化上皮组织的研究中发现miR-105可以调节TLR2(Toll-like receptor 2)的mRNA的表达, 从而起到抵抗炎症的作用. 本研究的结果表明, miR-105的高表达与CRC发生密切相关(P<0.01).
Karaayvaz等[34]研究表明, 在CRC组织当中miR-215的表达明显低于癌旁正常组织, miR-215主要是通过调节DTL(denticleless protein homolog)的表达从而达到抑制肿瘤的目的. 在本研究中CRC组织内miR-215的表达明显低于周围正常组织, 提示miR-215在结肠癌组织内为抑癌基因, 与上述研究结果相符.
早在2003年就有学者报道miRNA在CRC中的异常表达, Michael等[35]在对CRC的研究中发现, 有两个miRNA即miR-143和miR-145, 出现明显下调. Asangani等[36]在筛查84例美国CRC患者miRNA表达谱时发现有37种miRNA异常表达, 其中miR-21的高表达被证实与肿瘤的临床分期以及预后呈高度相关. Sarver等[37]研究发现与正常结肠组织相比较, CRC中有39个miRNAs异常表达, 包括miR-135b、miR-96、miR-182、miR-183、miR-1和miR-13等. Motoyama等[38]通过4组RNA芯片, 比较分析CRC与正常结直肠组织miRNAs的表达, 发现有21个miRNAs上调, miR-31也包含在其中, 这与本研究结果相一致. 从以上文献报道得知, 多数研究者的miRNAs表达谱不尽相同, 这其中也许存在着样本量等客观因素的存在, 当然也可能存在人为操作的差异等原因.
本研究发现, 与癌旁正常组织比较, CRC组织中有包括15种表达上调的和10种表达下调的共25种显著差异性表达的miRNAs, 其中除了miR-31和miR-215以外, 其他miRNAs在CRC的组织中的差异表达均未见报道. 这些新发现的miRNAs的差异性表达, 有望成为今后在诊断早期CRC及对CRC预后的判断上的可靠指标.
选取53例新鲜的CRC及其15例癌旁正常组织为研究对象, 比较分析疑为癌基因的表达升高的miRNAs, 包括miR-31、miR-105和miR-196b, 发现相对于癌旁正常组织, 他们在CRC组织中有明显的高表达(P<0.01). miR-196b与分化程度具有相关性, 即分化越差其miRANs的表达越高(P<0.05), 而在miR-31这种差异更明显(P<0.01), 且miR-31(P<0.05)与临床分期有关. 有人研究发现[39], miR-31通过控制肿瘤的淋巴道及远处转移来抑制肿瘤. 我们对CRC miR-31高表达组进行临床病理分析结果表明, miR-31高表达组与淋巴道转移及远处转移无相关性, 支持Valastyan等[39]的研究结果. 另外, 在本研究中发现miR-105与CRC的发生、分化、临床分期、有无淋巴道转移及远处转移均呈现出密切的联系(P<0.01). miR-105在CRC的发生及浸润, 侵袭性的增强, 乃至转移的过程所起的作用, 有必要在今后的研究过程中去一一阐明.
感谢金元哲教授的悉心关怀与指导, 感谢车敬昌教授的指导与帮助, 感谢北京博奥生物科技有限公司在基因芯片的生物分析方面给予的大力支持与帮助.
近年来microRNAs(miRNAs)与消化系肿瘤发生的研究取得了较大的进步, 为消化系肿瘤的基因诊断及治疗也提供了新靶点, 然而目前仍有许多miRNA在消化系肿瘤中作用机制尚未阐明. 结直肠癌(colorectal cancer, CRC)也是消化系最常见的恶性肿瘤之一, CRC发生发展, 经历由不典型增生、腺瘤、癌及肿瘤转移等一系列变化, 都有相应的基因发生变化, 同时也有调控这些基因的miRNA发生改变.
许剑民, 教授, 复旦大学附属中山医院普外科
研究已经证明, miRNA几乎参与了肿瘤发生发展中的每一步, 在肿瘤发生、发展及临床演变过程中发挥重要作用, 因此miRNA在肿瘤的诊断和预后及对化疗药物的应用以及治疗的新靶点选择方面均有很好的应用前途. 然而目前仍有许多miRNA在CRC中作用机制尚未阐明.
本研究发现, 与癌旁正常组织比较, CRC组织中有包括15种表达上调的和10种表达下调的共25种显著差异性表达的miRNAs存在, 其中除了miR-31和miR-215以外, 其他miRNAs在CRC的组织中的差异表达均未见报道.
CRC具有明显的miRNAs差异表达谱, miRNAs特异表达可能成为CRC的潜在的诊断和治疗的分子靶点. miR-31、miR-105和miR-196b高表达与CRC的病理分型密切相关.
本文采用AFFX miRNA芯片方法初步筛选了和CRC明显相关的miRNAs, 探讨CRC组织中miRNA的差异表达谱和临床病理因素之间的关系. 有一定临床意义.
编辑: 田滢 电编:闫晋利
| 1. | 2010世界癌症宣传日消化道肿瘤会在京召开. 中国医药导刊 2010; 5: 770. |
| 3. | Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 1993;75:843-854. [PubMed] [DOI] |
| 4. | Wightman B, Ha I, Ruvkun G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 1993;75:855-862. [PubMed] [DOI] |
| 5. | Levin B, Lieberman DA, McFarland B, Smith RA, Brooks D, Andrews KS, Dash C, Giardiello FM, Glick S, Levin TR. Screening and surveillance for the early detection of colorectal cancer and adenomatous polyps, 2008: a joint guideline from the American Cancer Society, the US Multi-Society Task Force on Colorectal Cancer, and the American College of Radiology. CA Cancer J Clin. 2008;58:130-160. [PubMed] |
| 8. | Luo X, Burwinkel B, Tao S, Brenner H. MicroRNA signatures: novel biomarker for colorectal cancer? Cancer Epidemiol Biomarkers Prev. 2011;20:1272-1286. [PubMed] [DOI] |
| 9. | Calin GA, Sevignani C, Dumitru CD, Hyslop T, Noch E, Yendamuri S, Shimizu M, Rattan S, Bullrich F, Negrini M. Human microRNA genes are frequently located at fragile sites and genomic regions involved in cancers. Proc Natl Acad Sci U S A. 2004;101:2999-3004. [PubMed] [DOI] |
| 10. | Calin GA, Dumitru CD, Shimizu M, Bichi R, Zupo S, Noch E, Aldler H, Rattan S, Keating M, Rai K. Frequent deletions and down-regulation of micro- RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia. Proc Natl Acad Sci U S A. 2002;99:15524-15529. [PubMed] [DOI] |
| 11. | Kameoka Y, Tagawa H, Tsuzuki S, Karnan S, Ota A, Suguro M, Suzuki R, Yamaguchi M, Morishima Y, Nakamura S. Contig array CGH at 3p14.2 points to the FRA3B/FHIT common fragile region as the target gene in diffuse large B-cell lymphoma. Oncogene. 2004;23:9148-9154. [PubMed] [DOI] |
| 12. | Zhang W, Dahlberg JE, Tam W. MicroRNAs in tumorigenesis: a primer. Am J Pathol. 2007;171:728-738. [PubMed] [DOI] |
| 13. | Volinia S, Calin GA, Liu CG, Ambs S, Cimmino A, Petrocca F, Visone R, Iorio M, Roldo C, Ferracin M. A microRNA expression signature of human solid tumors defines cancer gene targets. Proc Natl Acad Sci U S A. 2006;103:2257-2261. [PubMed] [DOI] |
| 14. | Cummins JM, He Y, Leary RJ, Pagliarini R, Diaz LA, Sjoblom T, Barad O, Bentwich Z, Szafranska AE, Labourier E. The colorectal microRNAome. Proc Natl Acad Sci U S A. 2006;103:3687-3692. [PubMed] [DOI] |
| 15. | Xi Y, Formentini A, Chien M, Weir DB, Russo JJ, Ju J, Kornmann M, Ju J. Prognostic Values of microRNAs in Colorectal Cancer. Biomark Insights. 2006;2:113-121. [PubMed] |
| 16. | Monzo M, Navarro A, Bandres E, Artells R, Moreno I, Gel B, Ibeas R, Moreno J, Martinez F, Diaz T. Overlapping expression of microRNAs in human embryonic colon and colorectal cancer. Cell Res. 2008;18:823-833. [PubMed] [DOI] |
| 17. | Lu J, Getz G, Miska EA, Alvarez-Saavedra E, Lamb J, Peck D, Sweet-Cordero A, Ebert BL, Mak RH, Ferrando AA. MicroRNA expression profiles classify human cancers. Nature. 2005;435:834-838. [PubMed] [DOI] |
| 18. | Bresalier RS, Kim YS. Diet and colonic cancer: putting the puzzle together. N Engl J Med. 1985;313:1413-1414. [PubMed] [DOI] |
| 19. | Liu CG, Calin GA, Meloon B, Gamliel N, Sevignani C, Ferracin M, Dumitru CD, Shimizu M, Zupo S, Dono M. An oligonucleotide microchip for genome-wide microRNA profiling in human and mouse tissues. Proc Natl Acad Sci U S A. 2004;101:9740-9744. [PubMed] [DOI] |
| 20. | Wong TS, Liu XB, Wong BY, Ng RW, Yuen AP, Wei WI. Mature miR-184 as Potential Oncogenic microRNA of Squamous Cell Carcinoma of Tongue. Clin Cancer Res. 2008;14:2588-2592. [PubMed] [DOI] |
| 21. | Kozaki K, Imoto I, Mogi S, Omura K, Inazawa J. Exploration of tumor-suppressive microRNAs silenced by DNA hypermethylation in oral cancer. Cancer Res. 2008;68:2094-2105. [PubMed] [DOI] |
| 22. | Liu X, Sempere LF, Ouyang H, Memoli VA, Andrew AS, Luo Y, Demidenko E, Korc M, Shi W, Preis M. MicroRNA-31 functions as an oncogenic microRNA in mouse and human lung cancer cells by repressing specific tumor suppressors. J Clin Invest. 2010;120:1298-1309. [PubMed] [DOI] |
| 23. | Valastyan S, Benaich N, Chang A, Reinhardt F, Weinberg RA. Concomitant suppression of three target genes can explain the impact of a microRNA on metastasis. Genes Dev. 2009;23:2592-2597. [PubMed] [DOI] |
| 24. | Zhang Y, Guo J, Li D, Xiao B, Miao Y, Jiang Z, Zhuo H. Down-regulation of miR-31 expression in gastric cancer tissues and its clinical significance. Med Oncol. 2010;27:685-689. [PubMed] [DOI] |
| 25. | Guo J, Miao Y, Xiao B, Huan R, Jiang Z, Meng D, Wang Y. Differential expression of microRNA species in human gastric cancer versus non-tumorous tissues. J Gastroenterol Hepatol. 2009;24:652-657. [PubMed] [DOI] |
| 26. | Creighton CJ, Fountain MD, Yu Z, Nagaraja AK, Zhu H, Khan M, Olokpa E, Zariff A, Gunaratne PH, Matzuk MM. Molecular profiling uncovers a p53-associated role for microRNA-31 in inhibiting the proliferation of serous ovarian carcinomas and other cancers. Cancer Res. 2010;70:1906-1915. [PubMed] |
| 27. | Schaefer A, Jung M, Mollenkopf HJ, Wagner I, Stephan C, Jentzmik F, Miller K, Lein M, Kristiansen G, Jung K. Diagnostic and prognostic implications of microRNA profiling in prostate carcinoma. Int J Cancer. 2010;126:1166-1176. [PubMed] [DOI] |
| 28. | Liao YL, Hu LY, Tsai KW, Wu CW, Chan WC, Li SC, Lai CH, Ho MR, Fang WL, Huang KH. Transcriptional regulation of miR-196b by ETS2 in gastric cancer cells. Carcinogenesis. 2012;33:760-769. [PubMed] [DOI] |
| 29. | Bhatia S, Kaul D, Varma N. Functional genomics of tumor suppressor miR-196b in T-cell acute lymphoblastic leukemia. Mol Cell Biochem. 2011;346:103-116. [PubMed] [DOI] |
| 30. | Bhatia S, Kaul D, Varma N. Potential tumor suppressive function of miR-196b in B-cell lineage acute lymphoblastic leukemia. Mol Cell Biochem. 2010;340:97-106. [PubMed] [DOI] |
| 31. | Li Y, Zhang M, Chen H, Dong Z, Ganapathy V, Thangaraju M, Huang S. Ratio of miR-196s to HOXC8 messenger RNA correlates with breast cancer cell migration and metastasis. Cancer Res. 2010;70:7894-7904. [PubMed] [DOI] |
| 32. | Li Z, Huang H, Chen P, He M, Li Y, Arnovitz S, Jiang X, He C, Hyjek E, Zhang J. miR-196b directly targets both HOXA9/MEIS1 oncogenes and FAS tumour suppressor in MLL-rearranged leukaemia. Nat Commun. 2012;3:688. [PubMed] [DOI] |
| 33. | Benakanakere MR, Li Q, Eskan MA, Singh AV, Zhao J, Galicia JC, Stathopoulou P, Knudsen TB, Kinane DF. Modulation of TLR2 protein expression by miR-105 in human oral keratinocytes. J Biol Chem. 2009;284:23107-23115. [PubMed] [DOI] |
| 34. | Karaayvaz M, Pal T, Song B, Zhang C, Georgakopoulos P, Mehmood S, Burke S, Shroyer K, Ju J. Prognostic significance of miR-215 in colon cancer. Clin Colorectal Cancer. 2011;10:340-347. [PubMed] [DOI] |
| 35. | Michael MZ, O' Connor SM, van Holst Pellekaan NG, Young GP, James RJ. Reduced accumulation of specific microRNAs in colorectal neoplasia. Mol Cancer Res. 2003;1:882-891. [PubMed] |
| 36. | Asangani IA, Rasheed SA, Nikolova DA, Leupold JH, Colburn NH, Post S, Allgayer H. MicroRNA-21 (miR-21) post-transcriptionally downregulates tumor suppressor Pdcd4 and stimulates invasion, intravasation and metastasis in colorectal cancer. Oncogene. 2008;27:2128-2136. [PubMed] [DOI] |
| 37. | Sarver AL, French AJ, Borralho PM, Thayanithy V, Oberg AL, Silverstein KA, Morlan BW, Riska SM, Boardman LA, Cunningham JM. Human colon cancer profiles show differential microRNA expression depending on mismatch repair status and are characteristic of undifferentiated proliferative states. BMC Cancer. 2009;9:401. [PubMed] [DOI] |
| 38. | Motoyama K, Inoue H, Mimori K, Tanaka F, Kojima K, Uetake H, Sugihara K, Mori M. Clinicopathological and prognostic significance of PDCD4 and microRNA-21 in human gastric cancer. Int J Oncol. 2010;36:1089-1095. [PubMed] [DOI] |
| 39. | Valastyan S, Reinhardt F, Benaich N, Calogrias D, Szász AM, Wang ZC, Brock JE, Richardson AL, Weinberg RA. A pleiotropically acting microRNA, miR-31, inhibits breast cancer metastasis. Cell. 2009;137:1032-1046. [PubMed] [DOI] |
| 40. | Bandrés E, Cubedo E, Agirre X, Malumbres R, Zárate R, Ramirez N, Abajo A, Navarro A, Moreno I, Monzó M. Identification by Real-time PCR of 13 mature microRNAs differentially expressed in colorectal cancer and non-tumoral tissues. Mol Cancer. 2006;5:29. [PubMed] |
| 41. | Wang CJ, Zhou ZG, Wang L, Yang L, Zhou B, Gu J, Chen HY, Sun XF. Clinicopathological significance of microRNA-31, -143 and -145 expression in colorectal cancer. Dis Markers. 2009;26:27-34. [PubMed] [DOI] |