修回日期: 2014-07-04
接受日期: 2014-07-15
在线出版日期: 2014-09-08
目的: 获得colo205细胞株不同细胞群肿瘤代谢基因及代谢调控基因的差异表达变化信息, 为深入理解大肠癌肿瘤的发生机制、药物治疗潜在靶点的发现及大肠癌的临床治疗提供实验依据.
方法: 利用无血清体外培养、磁珠分选colo205细胞株来源的大肠癌CD133+细胞群、CD133-细胞群结合裸鼠成瘤实验验证后, 表达谱测序结合生物信息学分析筛选不同细胞群差异表达肿瘤代谢基因及代谢调控基因, 并用定量PCR技术进行验证.
结果: 与CD133-细胞群比较, CD133+细胞群有9个癌症代谢基因表达发生上调, 3个癌症代谢基因表达发生下调, colo205未分选细胞群有12个癌症代谢基因表达发生上调, 3个癌症代谢基因表达发生下调. 与CD133+细胞群比较, colo205细胞有5个癌症代谢基因表达发生上调, 3个癌症代谢基因表达发生下调. 与CD133-细胞群相比, CD133+细胞群中糖酵解相关的基因有6个表达上调, colo205未分选细胞群的糖酵解相关的基因有7个表达轻微上调. 葡糖转运蛋白1(glucose transporter 1, GLUT1)在CD133+细胞群中高表达, GLUT3在CD133-细胞群中高表达, GLUT4在未分选colo205细胞群高表达. 参与三羧酸循环(tricarboxylic acid cycle)的6种癌症代谢基因中, 谷氨酰胺酶1(glutaminase1, GLS1)在CD133-细胞群中高表达; 异柠檬酸脱氢酶2(isocitrate dehydrogenase 2)和细胞色素氧化酶组装因子2(cytochromec oxidase assembly factors 2)在CD133-细胞群中低表达. 共有20种癌症代谢调控基因在不同细胞群中发生一定程度表达变化. 其中与colo205细胞群和CD133+细胞群相比, p53、RAS、细胞外调节蛋白激酶(extracellular regulated protein kinases)、肝脏激酶B1(liver kinase B1)、蛋白激酶B (protein kinase B)、TP53诱导的糖酵解及细胞凋亡调节蛋白(TP53-induced glycolysis and apoptosis regulator)在CD133-细胞群中均表达下调, 而缺氧诱导因子1α(hypoxia inducible factor 1α)、IκB激酶(IκB kinase)和表皮生长因子受体(epidermal growth factor receptor)则表现上调. AMP激活的蛋白激酶(AMP-activated protein kinase)和生长因子受体酪氨酸激酶6(receptor tyrosine kinase)则在未分选细胞群中表达下调.
结论: 大肠癌colo205细胞株来源的CD133+、CD133-及colo205代谢基因及其调控基因表达存在着异质性, 这可能对大肠癌的分子诊断、治疗靶点的选择和治疗手段的确定以及预后的判断提供参考.
核心提示: 本文分析了大肠癌不同细胞群肿瘤代谢基因和参与癌症代谢途径表达调控表达变化, 发现同一细胞来源不同细胞群的代谢基因及代谢调控基因表达存在着异质性, 提示癌细胞的代谢模式可能也存在着异质性.
引文著录: 岳昌武, 吕玉红, 周昕, 王苗, 李园园, 曾庆良, 邵美云. 表达谱测序筛选大肠癌colo205细胞及其来源的CD133+、CD133-细胞群肿瘤代谢相关基因. 世界华人消化杂志 2014; 22(25): 3801-3810
Revised: July 4, 2014
Accepted: July 15, 2014
Published online: September 8, 2014
AIM: To obtain differential gene expression information on cancer metabolic reprogramming from colo205 derived colorectal cancer cells.
METHODS: Colo205 cell spheres were cultured in serum-free medium with cell factors, and CD133+/CD133- cells were sorted using an indirect CD133 microbead kit. In vitro differentiation and nude mouse tumorigenicity assay were carried out to test whether CD133+ cells have stem cell characteristics or not together with colo205 cells and CD133- cells. RNA-seq was employed for analysis of differential genes related to cancer metabolic reprogramming and metabolism regulatory genes, followed by verification by qRT-PCR.
RESULTS: Compared with CD133- cells, 9 cancer metabolism related genes and 6 glycolysis related genes were up-regulated, and 3 cancer metabolism related genes down-regulated in CD133+ cells, while 12 cancer metabolism related genes and 6 glycolysis related genes were up-regulated, and 3 cancer metabolism related genes down-regulated in colo205 cells. For glucose transporters, glucose transporter 1 (GLUT1) was up-regulated in CD133+ cells, GLUT3 was up-regulated in CD133- cells, and GLUT4 was up-regulated in colo205 cells. Among the 6 cancer metabolism related genes involved in the tricarboxylic acid (TCA) cycle, glutaminase1 (GLS1) was up-regulated and isocitrate dehydrogenase 2 (IDH2) was down-regulated along with cytochromec oxidase assembly factors 2 (SCO2) in CD133- cells. The mRNA expression levels of 20 cancer metabolism regulatory genes were changed, including p53, RAS, extracellular regulated protein kinases (ERK), liver kinase B1 (LKB1), protein kinase B (AKT), and TP53-induced glycolysis and apoptosis regulator (TIGAR) that were down-regulated, and hypoxia inducible factor 1α (HIF-1α), IκB kinase (IKK) and epidermal growth factor receptor (EGFR) that were up-regulated in CD133- cells, and AMP-activated protein kinase (AMPK) and receptor tyrosine kinase (RTK6) that were down-regulated in colo205 cells.
CONCLUSION: Heterogeneity of gene expression profile exists in colo205 cells and colo205 derived CD133- cells and CD133+ cells, which may provide a reference for molecular diagnosis, therapeutic target selection, treatment evaluation and prognosis judgment in colorectal cancer.
- Citation: Yue CW, Lv YH, Zhou X, Wang M, Li YY, Zeng QL, Shao MY. Gene expression profiling for screening of cancer metabolism related genes in CD133+/CD133- colorectal cancer cells derived from colo205 cells. Shijie Huaren Xiaohua Zazhi 2014; 22(25): 3801-3810
- URL: https://www.wjgnet.com/1009-3079/full/v22/i25/3801.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v22.i25.3801
癌细胞代谢的一个重要特征就是葡萄糖的摄入和利用能力提高, 80多年前, Warburg等[1,2]发现即使在有氧条件下, 癌细胞仍通过糖酵解途径产生ATP, 即"瓦氏效应"(Warburg effect). 根据瓦氏效应学说, 癌细胞中有氧糖酵解过程的激活及持续进行与癌基因的激活或肿瘤抑制基因的功能丧失密切相关, 癌细胞能量代谢调控失常[即癌细胞代谢重编程(cancer metabolic reprogramming)]有可能导致了许多代谢信号发生改变, 影响了细胞增殖和凋亡等各种生物学过程[3]. 癌细胞代谢过程的根本性变化, 为肿瘤细胞提供了快速生长所需的大分子前体和能量, 满足了癌细胞恶性增殖过程中对能量和各种次级代谢物的需求, 为癌细胞的生存、增殖、侵袭及转移等提供能量和物质基础, 从而从根本上影响着癌症的发生、发展过程[4-6]. 随着分子生物学的深入发展和高通量的分子分析仪器的投入使用, 越来越多的研究结果证明[7,8]癌细胞生长环境的改变可能是导致其代谢改变的重要原因, 即癌细胞密集式生长导致其微环境氧浓度和营养的利用性改变, 癌组织的缺氧和营养物利用性改变可能是导致癌细胞代谢改变的重要原因.
癌细胞的代谢转换涉及有氧糖酵解、三羧酸循环、磷酸戊糖途径、线粒体重组, 氨基酸代谢及脂类代谢等多条代谢途径的改变. 近年来对癌症代谢改变机制的解析的进展表明[9], 癌细胞增殖需要通过癌基因和肿瘤抑制基因突变改变多种信号转导通路, 进而引起癌细胞代谢途径的转变从而促进了癌症的发生、发展. p53、c-Myc、缺氧诱导因子(hypoxia inducible factor, HIF)、RAS、核因子-κB(nuclear factor-κB, NF-κB)、CD44等数十种基因可能在这个转变中发挥重要调控作用. 作为癌细胞的一个共同特征, 代谢异常发生机制的解析将有助于阐明癌症发病机制, 为临床治疗癌症提供有潜力的靶点. 例如癌细胞环境氧浓度降低可能诱导的低氧诱导因子HIF的表达改变, HIF调控的基因涉及许多病理过程, 如代谢转变、药物释放、血管生成、细胞增殖、抗凋亡, 最终导致肿瘤生长和耐药性增加; 异柠檬酸脱氢酶1(isocitrate dehydrogenase 1, IDH1)和IDH2的突变导致细胞内2-羟戊二酸浓度升高, 从而导致α-酮戊二酸依赖的双加氧酶的酶活性的变化, 使恶性肿瘤的风险增加, 近期的研究发现通过设计能抑制这两种酶基因的变异的小分子, 可分别减缓白血病和脑肿瘤细胞的生长[10].
基因表达谱测序等组学技术的进展为研究人员快速、准确的了解特定生理状态下细胞内各种基因的动态变化提供有力工具和可能. 大肠癌是目前国内发病率次仅于肺癌、乳腺癌的严重危害人类健康的恶性肿瘤, 近年发病率呈逐年上升趋势[11]. 大肠癌干细胞是消化系恶性肿瘤中第1个被发现的癌症始动细胞, 对其代谢基因的研究对大肠癌诊断及综合防治措施的制定等具有极为重要的意义. 本研究采用无血清体外球培养[12-17]富集肠癌细胞株colo205来源的细胞球, 通过CD133+标记的免疫磁珠分选、富集大肠癌始动细胞, 利用体外分化实验和裸鼠成瘤实验对分选的大肠癌始动细胞的干细胞特性进行鉴定和验证, 并用RNA-seq研究了大肠癌代谢基因及其调控基因表达变化, 并利用qRT-PCR技术对测序结果进行验证. 可为大肠癌深入理解大肠癌的发生机制、药物治疗潜在靶点的发现及大肠癌的临床治疗提供实验依据.
人大肠癌colo205细胞株购自中国科学院上海生命科学院细胞库, colo205来源的大肠癌CD133+细胞群/CD133-细胞群为本课题组利用无血清培养基富集、磁珠分选后经体外分化及裸鼠成瘤实验验证干性特征后用于本研究. Truseq™ RNA sample prep kit, Dynabeads® mRNA Purification kit, TBS380 (Picogreen), Certified Low Range Ultra Agarose, cBot Truseq PE Cluster Kit v3-cBot-HS, Hiseq2000 Truseq SBS Kit v3-HS(200 cycles)等RNA纯化、无差异表达谱文库构建、高通量测序试剂盒及磁力架及HiSeq 2500高通量测序平台等购自Illumina(上海)公司. TRIzol® Reagent、B27添加剂(B27)、重组人碱性成纤维生长因子(recombinant human basic fibroblast growth factor, bFGF)、重组人表皮细胞生长因子(recombinant human epidermal growth factor, rEGF)及重组人自血病抑制因子(recombinant human leukemia inhibitory factor, rLIF)购自Invitrogen(上海)公司; 二甲基亚砜(DMSO)、胰蛋白酶粉购自Sigma(上海)公司; DMEM/F12培养基和RPMI 1640培养基购自Gibco(上海)公司; 胎牛血清购自杭州四季青公司; CD133细胞分选试剂盒(CD133 Cell Isolation Kit)购自Miltenyi(上海)公司; 常规细胞培养等无酶耗材均购自爱思进(杭州)公司. 无血清培养基(serum free medium, SFM)为本实验新鲜配制(98 mL DMEM/F12培养基含2.0 mL B27, 0.2 μg rbFGF, 0.2 μg rLIF, 0.2 μg rEGF, 加超纯水定容到100 mL, 过滤灭菌, -20 ℃保存, 备用). 倒置荧光显微镜、低温离心机、二氧化碳细胞培养箱等仪器由遵义医学院医学与生物研究中心提供, 磁珠细胞分选仪由遵义医学院邓飞教授课题组提供.
1.2.1 colo205来源细胞群分选及鉴定: 取SSM中处于对数增长期的colo205细胞, 以105/mL的细胞密度接种至SFM中进行培养成结肠癌干细胞球, 10 d后用酶消化法及机械分离法将colo205干细胞球分离成单细胞悬液并观察单细胞悬液传代后重新形成结肠癌干细胞球的过程. 将形态稳定的colo205干细胞球分离成单细胞悬液, 悬浮细胞中加入100 μL FcR blocking及100 μL CD133免疫磁珠, 选择MS柱及匹配的分选器, 继续收集流过柱子的CD133-细胞和CD133+阳性细胞. 将分选所得CD133+与CD133-细胞密度调整至105/mL分别接种SFM中, 37 ℃、5%CO2恒温箱中培养, 每2-3 d加入2 mL SFM培养基, 每天观察细胞在SFM中形态变化. 10 d后向SFM中加入胎牛血清(SFM与胎牛血清的体积比为9:1), 继续37 ℃、5%CO2恒温箱中培养, 每天观察细胞形态变化. 将分选得到CD133+、CD133-细胞及未分选的colo205细胞分别以生理盐水稀释并以1×103、1×104、1×105细胞数量接种至同1只Babl/c裸鼠左右腋下和右侧腹股沟并以生理盐水作对照后养于SPF动物房, 观察并记录肿瘤生长情况.
1.2.2 测序文库构建及高通量测序: 利用TRIzol®总RNA提取试剂盒提取总RNA, 利用Dynabeads® mRNA试剂盒利用磁珠法分离纯化mRNA, 离子打断mRNA后利用Truseq™ RNA纯化试剂盒合成双链cDNA合成、补平、3'端加A、连接index接头构建文库. 利用接头引物PCR扩增15个循环富集文库后用2%琼脂糖胶回收扩增的目的条带, 利用TBS380(Picogreen)定量试剂盒定量回收的富集文库cDNA, 按数据比例混合上, cBot上进行桥式PCR扩增, 生成Clusters后利用Hiseq2000测序平台, 进行1×50 bp测序.
原始fastq数据后利用SeqPrep(https://github.com/jstjohn/SeqPrep)、sickle(https://github.com/najoshi/sickle)等软件进行质控分析得到clean data序列进行数据量统计后与人基因组mapping. 利用tophate(http://tophat.cbcb.umd.edu/)构建转录本, 利用cuffcompare对不同转录本进行相似情况的比较, 利用cuffmerge合并转录本集合, 利用cuffdiff(http://cufflinks.cbcb.umd.edu/)衡量样本间差异表达基因来. 根据样本与人基因组比对的结果, 计算每个基因/转录本在样本中的FPKM(每1百万个map上的reads中map到外显子的每kb碱基上的reads个数)值, 以该值作为基因/转录本在样本中的表达量. 以错误发现率(false discovery rate, FDR)<0.05作为显著差异基因/转录本筛选条件对所有基因/转录本在各组样本中的表达进行差异显著性分析, 找出相对差异表达的基因/转录本. 将有显著差异的基因/转录本进行表达模式聚类分析, 采用距离计算算法: 样本间为spearman, 基因间为pearson, 采用的聚类方法为hcluster(complete算法). 利用R软件中的"noiseq"进行差异基因聚类并可视化输出.
1.2.3 差异基因定量PCR验证: 为了进一步验证表达谱测序获得基因表达变化数据的可靠性, 我们随机选取了p53和BCL11A基因, 并以β-actin基因作为内参, 进行表达变化的定量PCR验证. P53 F: 5'-CCCTCCTCAGCATCTTATCCG-3'; P53 R: 5'-CAACCTCAGGCGGCTCATAG-3'; BCL11A F: 5'-GACAGGGTGCTGCGGTTGA-3', BCL11A R: 5'-GGCTTGCTACCTGGCTGGAA-3'; β-actin引物: β-actin F: 5'-TGGCACCCAGCCAATGAA-3', β-actin R: 5'-CTAAGTCATAGTCCGCCTAGAAGCA-3'. 定量PCR反应条件为: 95 ℃ 预变性2 min, 40个热循环扩增(95 ℃变性10 s, 60 ℃退火扩增20 s). 每轮热循环结束后采集荧光信号, 扩增完全结束后采集荧光信号绘制熔解曲线, 获得Ct值, 以2-△△Ct法[18]进行表达变化分析.
统计学处理 定量PCR结果利用SPSS13.0统计软件, 采用One-way ANOVA方法进行组间比较, P<0.05为差异有统计学意义.
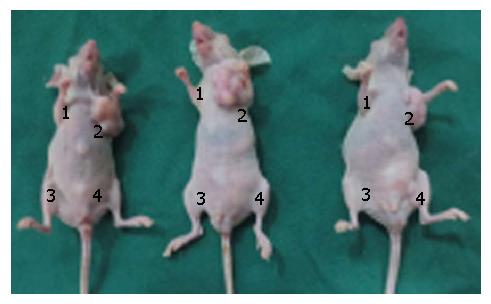
SFM中培养1 d后, 分选的CD133+与CD133-细胞表型出现明显区别: CD133+细胞成团悬浮生长, 4 d可见典型肿瘤细胞球, 而CD133-细胞则始终未见肿瘤细胞球生成. 添加胎牛血清后, 1 d后CD133+细胞球贴壁生长, 3 d后细胞球分化. 接种裸鼠后第15天可见接种105个CD133+细胞处始见肿瘤生长(图1), 第20天接种104个CD133+细胞处见肿瘤生长, 第30天可见接种103个CD133+细胞处见肿瘤生长; 接种第35天可见接种105个colo205细胞处见肿瘤生长, 第40天接种104个和接种103个colo205细胞及CD133-细胞处均未见肿瘤生长. 到42 d观察期结束, 仅需接种103个CD133+细胞就可以在裸鼠皮下形成肿瘤, 所有CD133+细胞接种处均成瘤; 而colo205细胞需接种105个才能在第35天形成肿瘤; 所有CD133-细胞接种处均未见肿瘤生长. CD133+细胞内成瘤能力强于CD133-细胞及colo205细胞, 有明显差异(P<0.05); 而CD133-及colo205细胞间成瘤能力无明显差异.
测序得到的原始图像数据经过Base Calling转化为序列数据, 对获得测序reads的序列信息以及reads的测序质量信息进行评估, 生物信息分析运用统计学的方法, 对所有测序reads的每个circle进行碱基分布和质量波动的统计, 可以从宏观上直观地反映出样本的测序质量和文库构建质量. 本次文库构建和高通量测序结果表明(表1)CD133-组、CD133+组和未分选colo205组得到原始序列分别为17722146条、23174152条和27260265条; 累计碱基数分别达到88107300、1158707600和136013250 bp; 其中长度≥20 nt的reads占分别总reads的96.84%、95.76%和95.92%; 人基因组比对, mapping上的序列分别为12915738条、16118258条和19304605条, 分别占整个高质量的reads的87.07%、85.93%和84.11%, 表明所获得测序数据达到后续分析的要求.
| 样本 | 原始数据统计结果 | 基因组mapping比率 | |||
| 序列数(条) | 碱基数(bp) | ≥Q20(%) | 序列数(条) | 百分比(%) | |
| CD133- | 17722146 | 886107300 | 96.84 | 12915738 | 87.07 |
| CD133+ | 23174152 | 1158707600 | 95.76 | 16118258 | 85.93 |
| colo205 | 27260265 | 1363013250 | 95.92 | 19304605 | 84.11 |
以FDR<0.05为显著差异基因/转录本筛选条件, 利用分析软件Tophat及Cuffdiff分别转录本测序常规数据分析结果表明本次测序所有样品的FPKM分布差异不显著. 对样品测序结果两两进行两个样本中基因或转录本的表达量变化差异的统计学分析结果表明(图2)CD133+组/colo205组/CD133-组等3组样品间两两样品的转录本的表达量变化差异显著, 以P<0.05和q<0.05和表达变化差异为0.75和1.33倍为差异表达基因筛选标准将这些差异基因进行差异基因表达模式聚类, 得到异变化显著基因, 这些差异基因共涉及32个代谢途径, 其中抗原加工递呈、细胞黏附、趋化因子途径、大肠癌途径、细胞因子受体、药物代谢、ErbB信号通路、促性腺激素释放激素(gonadotropin-releasing hormone, GnRH)信号通路、造血细胞谱系、Hippo信号通路、HTLV-I感染、Legionellosis、MAPK通路、NF-kappa B通路、NOD-like受体、Notch信号通路、癌症代谢、PI3K-Akt途径、PPAR途径、主要免疫缺陷、癌症相关聚糖、丙酮酸代谢、肌动蛋白骨架等可能与癌症发生、发展及侵袭、转移相关的代谢途径[6-8].
利用KEEG数据库结合最新文献报道[9]的将表达谱测序结果中的癌症代谢功能催化基因(表2)的表达变化进行统计分析, 结果表明在阳性组与阴性组比较(CD133+/CD133-), 有9个癌症代谢基因表达发生上调, 占分析基因总数的34.6%(9/26), 3个癌症代谢基因表达发生下调, 占分析基因总数的11.5%(3/26); 未分选组与阴性组比较(colo205/CD133-), 有12个癌症代谢基因表达发生上调, 占分析基因总数的46.1%(9/26), 3个癌症代谢基因表达发生下调, 占分析基因总数的11.5%(3/26); 未分选组与阳性组比较阳性(colo205/CD133+), 仅有5个癌症代谢基因表达发生上调, 占分析基因总数的19.2%(5/26), 3个癌症代谢基因表达发生下调, 占分析基因总数的11.5%(3/26). 在糖酵解相关的基因中, 与CD133-细胞群相比, CD133+细胞群中糖酵解相关的基因有6个表达上调, 占整个分析的糖酵解基因的66.6%(6/9); 同样的, 与CD133-细胞群相比, 未分选细胞群的糖酵解相关的基因有7个表达轻微上调, 占整个分析的糖酵解基因的77.8%(7/9), 无基因表达下调发生(0/9). 参与糖基转运4种葡糖转运蛋白中, 除了葡糖转运蛋白2(glucose transporter 2, GLUT2)在3种细胞群中表达变化不大外, GLUT1在CD133+细胞群中高表达(比CD133-高1.37倍, 比未分选colo205细胞群高4.17倍), GLUT3在CD133-细胞群中高表达(比未分选colo205细胞群高1.96倍, 比CD133+细胞群高1.79倍), GLUT4在未分选colo205细胞群高表达(比CD133+细胞群高1.92倍, 比CD133-高1.75倍). 参与磷酸戊糖途径的TKT和TKTL2则在3种细胞群中表达差异不明显. 参与三羧酸循环(tricarboxylic acid cycle, TCA)循环的6种癌症代谢基因中, 谷氨酰胺酶2(glutaminase1, GLS2)、IDH1和PDK1在3种细胞群中表达变化不大, 而GLS1在CD133-细胞群中高表达(比未分选colo205细胞群高1.89倍, 比CD133+细胞群高1.85倍); 与GLS1相反, IDH2和细胞色素氧化酶组装因子2(cytochromec oxidase assembly factors 2, SCO2)则在CD133-细胞群中低表达(分别比未分选colo205细胞群低2.54倍和1.80倍, 分别比CD133+细胞群低1.80倍和1.90倍). 而参与合成代谢的相关癌症代谢基因则表达变化很少(仅SHMT和ODC在未分选细胞群中表达略有变化).
| 代谢途径 | 靶基因 | 功能 | 基因表达变化 | 转录因子 | ||
| 阳性/阴性 | 未分选/阴性 | 未分选/阳性 | ||||
| 糖基转运 | GLUT1 | 糖基转运 | 1.37 | 0.32 | 0.24 | HIF/c-Myc/p53 |
| GLUT2 | 糖基转运 | 1.00 | 1.18 | 1.18 | c-Myc | |
| GLUT3 | 糖基转运 | 0.56 | 0.51 | 0.90 | HIF/p53 | |
| GLUT4 | 糖基转运 | 0.92 | 1.75 | 1.92 | c-Myc/p53 | |
| 糖酵解 | HK2 | 己糖磷酸化 | 0.91 | 1.39 | 1.52 | HIF/c-Myc/p53 |
| ALDOA | 醛缩酶 | 2.11 | 1.44 | 0.68 | HIF/c-Myc | |
| GAPDH | 3-P-甘油醛脱氢 | 1.84 | 1.86 | 1.01 | HIF/c-Myc | |
| PGK1 | 磷酸甘油酸激酶 | 1.58 | 1.37 | 0.87 | HIF/c-Myc | |
| PGM | 磷酸甘油酸变位酶 | 1.72 | 1.20 | 0.70 | p53 | |
| ENO1 | 烯醇化酶 | 1.79 | 2.01 | 1.12 | HIF/c-Myc | |
| PKM2 | 丙酮酸激酶 | 1.15 | 1.15 | 1.00 | HIF/c-Myc | |
| LDHA | 乳酸脱氢酶 | 2.42 | 1.97 | 0.82 | HIF/c-Myc | |
| G6PDH | 6-磷酸葡萄糖脱氢酶 | 1.16 | 1.62 | 1.39 | p53 | |
| 磷酸戊糖 | TKT | 转酮醇酶 | 1.20 | 0.90 | 0.75 | HIF |
| TKTL2 | 转酮醇酶样蛋白2 | 1.15 | 1.15 | 1.00 | HIF | |
| TCA循环 | PDK1 | 丙酮酸脱氢酶激酶1 | 1.43 | 1.30 | 0.91 | HIF/c-Myc |
| GLS1 | 谷氨酰胺酶 | 0.54 | 0.53 | 0.99 | c-Myc | |
| GLS2 | 谷氨酰胺酶 | 0.83 | 0.97 | 1.19 | p53 | |
| IDH1 | 异柠檬酸脱氢酶1 | 1.02 | 1.29 | 1.27 | 未知 | |
| IDH2 | 异柠檬酸脱氢酶2 | 1.80 | 2.54 | 1.42 | 未知 | |
| SCO2 | 细胞色素氧化酶 | 1.90 | 1.80 | 0.94 | p53 | |
| 合成代谢 | CAD | 氨甲酰磷酸合成酶 | 0.87 | 0.80 | 0.92 | c-Myc |
| SHMT | 丝氨酸羟甲基转移酶 | 1.08 | 1.98 | 1.83 | c-Myc | |
| FAS | 脂肪酸合成 | 1.12 | 1.17 | 1.04 | c-Myc | |
| ODC | 鸟氨酸脱羧 | 0.81 | 1.43 | 1.75 | c-Myc | |
| ACLY | ATP-柠檬酸裂解酶 | 0.88 | 1.18 | 1.33 | 未知 | |
将文献报道[9]中可能参与癌症代谢途径表达调控表达情况进行分析, 结果表明(表3)除了HIF-2α、RAF、MEK、PHD2、NF-κB、PTEN和HER2等基因在3种细胞群中表达变化不大外, 共有20种癌症代谢调控基因在不同细胞群中发生一定程度的表达变化. 其中与colo205细胞群和CD133+细胞群相比, p53、RAS、细胞外调节蛋白激酶(extracellular regulated protein kinases, ERK)、肝脏激酶B1(liver kinase B1, LKB1)、蛋白激酶B(protein kinase B, AKT)、TP53诱导的糖酵解及细胞凋亡调节蛋白(TP53-induced glycolysis and apoptosis regulator, TIGAR)在CD133-细胞群中均表达下调, HIF-1α、IκB激酶(IκB kinase, IKK)和表皮生长因子受体(epidermal growth factor receptor, EGFR)则表现上调. AMP激活的蛋白激酶(AMP-activated protein kinase, AMPK)和生长因子受体酪氨酸激酶6(receptor tyrosine kinase, RTK6)则在未分选细胞群中表达下调.
| 靶基因 | 参与调控代谢功能 | 基因表达变化 | ||
| 阳性/阴性 | 未分选/阴性 | 未分选/阳性 | ||
| p53 | 糖酵解/TCA循环/糖基转运 | 2.36 | 2.17 | 0.92 |
| c-Myc | 糖酵解/TCA循环/合成/糖基转运 | 1.43 | 1.27 | 0.87 |
| HIF-1α | 磷酸戊糖/TCA循环/糖酵解/糖基转运 | 0.51 | 0.67 | 1.30 |
| HIF-2α | 磷酸戊糖/TCA循环/糖酵解/糖基转运 | 1.08 | 0.89 | 0.82 |
| RAS | 糖酵解 | 1.49 | 1.78 | 1.19 |
| RAF | 糖酵解 | 0.81 | 0.85 | 1.04 |
| MEK | 糖酵解 | 1.36 | 1.27 | 0.93 |
| ERK | 糖酵解 | 1.39 | 1.99 | 1.44 |
| LKB1 | 糖酵解 | 1.53 | 1.77 | 1.16 |
| AMPK | 糖酵解 | 0.94 | 0.38 | 0.40 |
| mTOR | 糖酵解 | 0.64 | 0.82 | 1.28 |
| PI3K | 糖酵解 | 0.71 | 1.12 | 1.57 |
| AKT | 糖酵解 | 1.52 | 1.68 | 1.11 |
| VHL | 糖酵解 | 0.61 | 0.88 | 1.44 |
| TIGAR | 糖酵解 | 1.40 | 1.38 | 0.98 |
| CD44 | 糖酵解 | 1.78 | 1.20 | 0.67 |
| PHD2 | TCA循环 | 1.01 | 1.35 | 1.34 |
| IKK | 糖基转运 | 0.43 | 0.48 | 1.12 |
| NF-κB | 糖基转运 | 0.90 | 1.21 | 1.34 |
| EGFR | 糖基转运/糖酵解 | 0.73 | 0.54 | 0.74 |
| PTEN | 糖基转运/糖酵解 | 1.17 | 1.04 | 0.88 |
| HER2 | 糖基转运/糖酵解 | 1.33 | 0.92 | 0.69 |
| RTK6 | 糖酵解 | 1.07 | 0.60 | 0.56 |
自上个世纪20年代Warburg发现即使是在氧气供应充足的情况下癌细胞也主要是利用糖酵解途径而不是像正常细胞那样主要通过三羧酸循环-氧化磷酸化产生ATP以来, 大量的研究人员试图揭开这个现象背后隐藏的分子机制[19]. 近年来, 得利与组学研究技术的进步, 大量的高通量测序数据支持肿瘤突变、异质理论[20-22]. 测序数据的生物信息学深度挖掘分析结果表明癌基因的突变可能会影响细胞内数十种与肿瘤相关代谢网络. 癌细胞通过代谢途径的改变来运输核酸、氨基酸、脂类及谷胱甘肽等抗氧化剂进入细胞来满足等细胞快速增殖必须的营养物质需要. 尽管肿瘤代谢基因及代谢调控基因的研究尚还有很长路要走, 但已有的研究表明HIF-1、c-Myc、p53、PKM2、IDH、GLS等基因在肿瘤代谢改变中发挥重要作用[23-25].
本文利用高通量表达谱测序技术分析了肿瘤代谢基因及肿瘤代谢调控基因在colo205细胞株来源不同细胞群的表达变化, 结果表明即使同一细胞株来源的不同细胞群这些代谢相关的基因表达谱也存在着很多改变. 例如以p53为例, 和CD133-细胞群相比, CD133+细胞群中抑癌基因p53的高表达直接抑制了IKK的表达, 后者又通过NF-κB进而降低了代谢基因糖基转运蛋白GLUT3表达, 从而在源头上降低了癌细胞利用糖酵解获得ATP的机会, 这可能也是CD133+细胞群具有干性特征因素之一; p53的高表达对直接抑制低氧诱导因子HIF途径中HIF-1α和mTOR等基因的表达, 从而可能导致HIF途径中羟基化HIF-1α的泛素化降解而不能促进糖酵解等癌症代谢相关的基因的表达; 与此同时p53的高表达促进了其下游SCO2、TIGAR基因的高表达, 进而分别通过促进TCA循环和抑制糖酵解来调控CD133+细胞群ATP产生模式. 有意思的是CD133+细胞群ATP产生模式似乎处于一种动态平衡过程中, 一方面通过抑制基因p53高表达来试图"扭转"ATP通过糖酵解产生, 回归TCA循环为主的正常细胞代谢模式, 另一方面细胞通过高表达RAS、MEK和ERK促进HIF和c-Myc的高表达, 同时通过降低VHL的表达水平来减少HIF的泛素化降解, 从而维持癌细胞糖酵解为主的ATP产生模式[26-29]. 此外, c-Myc还通过抑制GLS1表达水平, 进而减少α-酮戊二酸进入TCA循环, 从而减少TCA产生ATP的机会[30]. 这些结果既进一步佐证了肿瘤基因表达异质理论, 也表明癌细胞的代谢模式可能也存在着异质性.
为了筛选与大肠癌诊断密切且相关的基因, 本研究对裸鼠成立验证的磁珠筛选的colo205来源的不同细胞群进行表达谱测序分析, 并利用定量PCR技术对随机选取的两个锌指蛋白表达进行验证, 结果这两个基因的表达变化趋势与表达谱测序结果一致(如基因p53在CD133+/CD133-分别为2.366和1.61, 在colo205/CD133-分别为2.17和1.6; 基因BCL11A在CD133+/CD133-分别为2.951067和2.25012, 在CD133+/colo205分别为0.149702和0.25174).
总之, 本研究通过对大肠癌colo205细胞株来源的细胞群癌症代谢差异表达谱分析, 我们发现CD133+、CD133-及colo205代谢基因及其调控基因表达存在着异质性, 这可能对大肠癌的分子诊断、治疗靶点的选择和治疗手段的确定以及预后的判断提供参考[31,32].
癌细胞代谢调控失常与癌基因的激活或肿瘤抑制基因的功能丧失密切相关, 研究癌细胞代谢基因表达变化可为其代谢异常发生机制的解析、癌症发病机制的阐明提供依据, 也可为为临床治疗癌症提供有潜力的靶点.
李革, 副教授, 延边大学附属医院
癌始动细胞的干细胞特性可能是癌症复发或转移的重要原因, 其相关生物标志物的发现可为临床癌症的早期诊断、分子分型、治疗手段的选择及预后提供有力工具, 寻找为临床诊疗提供可靠依据的癌症分子标志物尚待加强.
本研究通过对大肠癌colo205细胞株不同细胞群癌症代谢差异表达谱分析, 发现同一细胞株来源不同细胞群代谢基因及代谢调控基因表达存在着异质性, 提示癌细胞的代谢模式可能也存在着异质性.
本文分析了大肠癌不同细胞群肿瘤代谢基因和参与癌症代谢途径表达调控表达变化, 可为深入理解大肠癌代谢转化发生分子机制以及大肠癌发生机制、药物治疗潜在靶点的发现及大肠癌的临床诊疗提供实验依据.
癌细胞代谢重编程: 即使在有氧条件下, 癌细胞仍通过糖酵解途径产生ATP, 癌细胞中有氧糖酵解过程的激活涉及众多代谢基因及代谢调控基因表达模式的改变, 而这种改变可能正是肿瘤发生的基础.
本论文发现同一细胞株来源的不同细胞群代谢相关基因表达谱存在着很多改变. 设计合理, 实验方法先进, 结论可信, 论述条理清晰, 结果有意义. 本论文有一定的科学性和创新性, 结论对大肠癌的基础研究有一定的参考价值.
编辑: 郭鹏 电编:都珍珍
| 1. | Warburg O. über den Stoffwechsel der Carcinomzelle. Klinische Wochenschrift. 1925;12:534-536 [DOI.10.1007/BF01726151]. |
| 3. | Vander Heiden MG, Cantley LC, Thompson CB. Understanding the Warburg effect: the metabolic requirements of cell proliferation. Science. 2009;324:1029-1033. [PubMed] [DOI] |
| 4. | Cairns RA, Harris IS, Mak TW. Regulation of cancer cell metabolism. Nat Rev Cancer. 2011;11:85-95. [PubMed] [DOI] |
| 5. | Stratton MR, Campbell PJ, Futreal PA. The cancer genome. Nature. 2009;458:719-724. [PubMed] [DOI] |
| 6. | Bustamante E, Morris HP, Pedersen PL. Energy metabolism of tumor cells. Requirement for a form of hexokinase with a propensity for mitochondrial binding. J Biol Chem. 1981;256:8699-8704. [PubMed] |
| 7. | Ward PS, Thompson CB. Metabolic reprogramming: a cancer hallmark even warburg did not anticipate. Cancer Cell. 2012;21:297-308. [PubMed] [DOI] |
| 8. | Yeung SJ, Pan J, Lee MH. Roles of p53, MYC and HIF-1 in regulating glycolysis - the seventh hallmark of cancer. Cell Mol Life Sci. 2008;65:3981-3999. [PubMed] [DOI] |
| 9. | Soga T. Cancer metabolism: key players in metabolic reprogramming. Cancer Sci. 2013;104:275-281. [PubMed] |
| 10. | Longo VD, Fontana L. Calorie restriction and cancer prevention: metabolic and molecular mechanisms. Trends Pharmacol Sci. 2010;31:89-98. [PubMed] |
| 12. | Maher CA, Kumar-Sinha C, Cao X, Kalyana-Sundaram S, Han B, Jing X, Sam L, Barrette T, Palanisamy N, Chinnaiyan AM. Transcriptome sequencing to detect gene fusions in cancer. Nature. 2009;458:97-101. [PubMed] [DOI] |
| 13. | Galli R, Binda E, Orfanelli U, Cipelletti B, Gritti A, De Vitis S, Fiocco R, Foroni C, Dimeco F, Vescovi A. Isolation and characterization of tumorigenic, stem-like neural precursors from human glioblastoma. Cancer Res. 2004;64:7011-7021. [PubMed] |
| 14. | Yin S, Li J, Hu C, Chen X, Yao M, Yan M, Jiang G, Ge C, Xie H, Wan D. CD133 positive hepatocellular carcinoma cells possess high capacity for tumorigenicity. Int J Cancer. 2007;120:1444-1450. [PubMed] |
| 15. | O'Brien CA, Pollett A, Gallinger S, Dick JE. A human colon cancer cell capable of initiating tumour growth in immunodeficient mice. Nature. 2007;445:106-110. [PubMed] |
| 16. | Willis ND, Przyborski SA, Hutchison CJ, Wilson RG. Colonic and colorectal cancer stem cells: progress in the search for putative biomarkers. J Anat. 2008;213:59-65. [PubMed] [DOI] |
| 17. | Handra-Luca A, Taconet S. CD133 expression in colorectal adenomas. J Clin Pathol. 2013;66:1097-1098. [PubMed] [DOI] |
| 18. | Schmittgen TD, Livak KJ. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 2008;3:1101-1108. [PubMed] |
| 19. | Kim J, DeBerardinis RJ. Cancer. Silencing a metabolic oncogene. Science. 2013;340:558-559. [PubMed] [DOI] |
| 20. | Iurlaro R, León-Annicchiarico CL, Muñoz-Pinedo C. Regulation of cancer metabolism by oncogenes and tumor suppressors. Methods Enzymol. 2014;542:59-80. [PubMed] [DOI] |
| 21. | Sciacovelli M, Gaude E, Hilvo M, Frezza C. The metabolic alterations of cancer cells. Methods Enzymol. 2014;542:1-23. [PubMed] [DOI] |
| 22. | Jang M, Kim SS, Lee J. Cancer cell metabolism: implications for therapeutic targets. Exp Mol Med. 2013;45:e45. [PubMed] [DOI] |
| 23. | Dória ML, Ribeiro AS, Wang J, Cotrim CZ, Domingues P, Williams C, Domingues MR, Helguero LA. Fatty acid and phospholipid biosynthetic pathways are regulated throughout mammary epithelial cell differentiation and correlate to breast cancer survival. FASEB J. 2014; Jun 26. [Epub ahead of print]. [PubMed] |
| 24. | Chen L, Shi Y, Liu S, Cao Y, Wang X, Tao Y. PKM2: the thread linking energy metabolism reprogramming with epigenetics in cancer. Int J Mol Sci. 2014;15:11435-11445. [PubMed] [DOI] |
| 25. | Lynam-Lennon N, Maher SG, Maguire A, Phelan J, Muldoon C, Reynolds JV, O'Sullivan J. Altered mitochondrial function and energy metabolism is associated with a radioresistant phenotype in oesophageal adenocarcinoma. PLoS One. 2014;9:e100738. [PubMed] [DOI] |
| 26. | Hu ZY, Xiao L, Bode AM, Dong Z, Cao Y. Glycolytic genes in cancer cells are more than glucose metabolic regulators. J Mol Med (Berl). 2014;92:837-845. [PubMed] |
| 27. | Liu S, Yan B, Lai W, Chen L, Xiao D, Xi S, Jiang Y, Dong X, An J, Chen X. As a novel p53 direct target, bidirectional gene HspB2/αB-crystallin regulates the ROS level and Warburg effect. Biochim Biophys Acta. 2014;1839:592-603. [PubMed] [DOI] |
| 28. | Reyes RK, Motiwala T, Jacob ST. Regulation of glucose metabolism in hepatocarcinogenesis by microRNAs. Gene Expr. 2014;16:85-92. [PubMed] [DOI] |
| 29. | Yang C, Jiang L, Zhang H, Shimoda LA, DeBerardinis RJ, Semenza GL. Analysis of hypoxia-induced metabolic reprogramming. Methods Enzymol. 2014;542:425-455. [PubMed] [DOI] |
| 30. | Yuneva MO, Fan TW, Allen TD, Higashi RM, Ferraris DV, Tsukamoto T, Matés JM, Alonso FJ, Wang C, Seo Y. The metabolic profile of tumors depends on both the responsible genetic lesion and tissue type. Cell Metab. 2012;15:157-170. [PubMed] [DOI] |
| 31. | Pate KT, Stringari C, Sprowl-Tanio S, Wang K, TeSlaa T, Hoverter NP, McQuade MM, Garner C, Digman MA, Teitell MA. Wnt signaling directs a metabolic program of glycolysis and angiogenesis in colon cancer. EMBO J. 2014;33:1454-1473. [PubMed] [DOI] |
| 32. | Yang L, Moss T, Mangala LS, Marini J, Zhao H, Wahlig S, Armaiz-Pena G, Jiang D, Achreja A, Win J. Metabolic shifts toward glutamine regulate tumor growth, invasion and bioenergetics in ovarian cancer. Mol Syst Biol. 2014;10:728. [PubMed] [DOI] |