修回日期: 2014-04-18
接受日期: 2014-04-28
在线出版日期: 2014-06-18
目的: 探讨不同胆固醇含量结石的微生物群落差异.
方法: 应用末端标记限制性片段长度多态性(terminal restriction fragment length polymorphism, T-RFLP)和克隆文库分析, 以微生物群落16S rRNA基因(16S rDNA)为目标, 对62例细菌常规培养为阴性的胆囊结石患者中的结石成分及微生物群落结构进行了解析和比较.
结果: (1)根据结石所含胆固醇的不同, 分为胆固醇结石组(胆固醇含量≥90%)11例、胆色素结石组(胆固醇含量<70%)26例和混合性结石组(胆固醇含量70%-89%)25例; (2)胆固醇组结石表面细菌16S rDNA阳性率为18.1%(2/11), 结石核心细菌16S rDNA阳性率为24.3%(3/11). 胆色素组结石表面细菌16S rDNA阳性率为42.3%(11/26), 结石核心细菌16S rDNA阳性率为53.8%(14/26). 混合性结石组结石表面细菌16S rDNA阳性率为36.0%(9/25), 结石核心细菌16S rDNA阳性率为64.0%(16/25).
结论: 胆固醇含量不同的结石中优势菌有所差别, 但以肠杆菌科和微球菌科为主. 其中肠杆菌科(Enterobacteriaceac)的沙门氏菌属(Salmonella)伤寒沙门氏菌可能在结石形成过程中有重要作用.
核心提示: 本研究中在常规细菌培养阴性的胆囊结石患者标本中确有较高的细菌16S rDNA检出率, 随着结石中胆固醇含量不同, 细菌微生物群落16S rDNA基因的检出率也不同, 这些微生物群落在较高的分类学水平(例如科)的群落多样性并不很高, 而在属水平表现出了不同的多样性以肠杆菌科和微球菌科为主.
引文著录: 黄洁, 丁绍晖, 孙敏, 许丁伟, 张捷. 胆囊结石患者结石成分及微生物群落结构的多样性. 世界华人消化杂志 2014; 22(17): 2467-2472
Revised: April 18, 2014
Accepted: April 28, 2014
Published online: June 18, 2014
AIM: To analyze the gallstone composition and microbial community structure in gallstones.
METHODS: The composition of gallstones and microbial community structure in gallstones from patients with cholecystolithiasis were investigated by terminal restriction fragment length polymorphism and clone library approaches. Small subunit rRNA gene (16S rDNA) was analyzed in 62 patients with cholecystolithiasis and negative bile culture.
RESULTS: According to the percentage of cholesterol, the 62 patients with cholecystolithiasis were divided into three groups: 11 in the cholesterol group (cholesterol content ≥ 90%), 26 in the pigment group (cholesterol content < 70%) and 25 in the mixed group (cholesterol content between 70% and 89%). The positive rates of 16S rDNA on stone surface in the cholesterol group, pigment group and mixed group were 18.1% (2/11), 42.3% (11/26) and 36.0% (9/25), respectively. The positive rates of 16S rDNA in stone core in the cholesterol group, pigment group and mixed group were 24.3% (3/11), 53.8% (14/26) and 64.0% (16/25), respectively.
CONCLUSION: The structure of the bacterial community is different in gallstones with different cholesterol contents.
- Citation: Huang J, Ding SH, Sun M, Xu DW, Zhang J. Gallstone composition and microbial community structure in gallstones. Shijie Huaren Xiaohua Zazhi 2014; 22(17): 2467-2472
- URL: https://www.wjgnet.com/1009-3079/full/v22/i17/2467.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v22.i17.2467
自建立Small三角学说以来, 医学界始终认为胆囊结石的发病机制以非生理性胆固醇过饱和、胆汁促抗成核失调以及胆囊局部因素为基础, 但随着近年来大量的研究[1-3]表明, 在胆囊结石患者的胆汁和结石中发现并确定幽门螺杆菌DNA的存在, 细菌参与结石形成的研究逐步开展起来. 研究的关键是首先揭示胆囊结石中细菌的成分和多样性以及他们确切的起源. 目前的研究受限于研究的条件[4]和培养的灵敏度[5,6], 使培养得到的微生物在种类、数量和功能上都无法反映自然状态下微生物群落的真实情况, 所以, 很有必要研究不依赖微生物培养来进行环境微生物群落分析的方法. 本研究为探讨胆囊结石成分与细菌微生物群落之间的关系, 采用T-RFLP技术对胆囊结石微生物群落16S rDNA基因序列进行分析研究.
选取2012-09/2013-03在我科接受择期胆囊切除术的胆囊结石患者, 术前3 mo内无急性胆道感染症状, HBsAg阴性. 完成腹腔镜胆囊切除术, 无菌注射器穿刺取胆囊内胆汁3-5 mL置于无菌试管内, 同时收集胆囊结石, 记录其大小、数目, 用生理盐水冲洗干净. 标本放在无菌容器内-20 ℃保存. 应用红外光谱法分析胆石化学成分(溴化钾压片法), 并对其分类. Ⅰ纯胆固醇结石(cholesterol stone, CS), 胆固醇含量≥90%; Ⅱ胆色素结石: 胆固醇含量<70%; Ⅲ混合性结石: 胆固醇含量70%-89%. 该步骤委托云南省昆明市西山区环境监测站完成. 胆汁细菌常规培养和厌氧培养阳性的病例被剔除.
1.2.1 结石微生物群落DNA提取: 从每位患者取出的结石(直径≥1.0 cm), 收集核心部分(核心部分距结石表面≥0.5 cm)和结石表面, 37 ℃烘干至恒重后粉碎取150 mg结石粉末, 加入1%SDS 700 μL在5 mL聚丙烯试管内, 室温培养过夜. 依据标准方法[7]提取, -20 ℃保存.
1.2.2 结石微生物16S rDNA基因扩增: 对样品DNA进行适当稀释之后, 利用TaKaRa公司的微生物16S rDNA的PCR试剂盒进行扩增. 微生物的扩增引物采用8F(5'-AGA GTT TGA TCC TGG CTC AG-3')和1492R(5'-GGT TAC CTT GTT ACG ACT T-3'), 在引物8 F的5'端标记荧光物质(6-FAM). 用于构建克隆文库的PCR 则采用不带荧光标记的引物. PCR产物利用ChargeSwitch纯化试剂盒(Invitrogen, USA)纯化.
1.2.3 克隆文库的构建、测序和系统发育分析: 利用非荧光标记的引物对微生物的16S rDNA片段进行PCR扩增, 采用pGEM-T easy载体(Promega, USA)和大肠杆菌(Escherichia coli, E. coli) DH5α菌株建立微生物的16S rDNA文库. 利用PCR-T-RFLP分析对微生物克隆文库中随机挑选的46个阳性克隆进行分型筛选, 挑选代表克隆进行测序(北方诺塞基因公司), 获得微生物的16S rDNA部分序列. 对这些序列利用Blast分析与GenBank中核酸数据进行序列比对(http://www.ncbi.nlm.nih.gov/blast).
统计学处理 应用SPSS17.0软件包分析, 统计学方法采用χ2和Fisher的精确检验. P<0.05为差异有统计学意义.
胆固醇含量≥90%者11例, 为胆固醇结石组. 胆固醇含量<70%者26例, 为胆色素结石组. 胆固醇含量70%-89%者25例, 为混合性结石组.
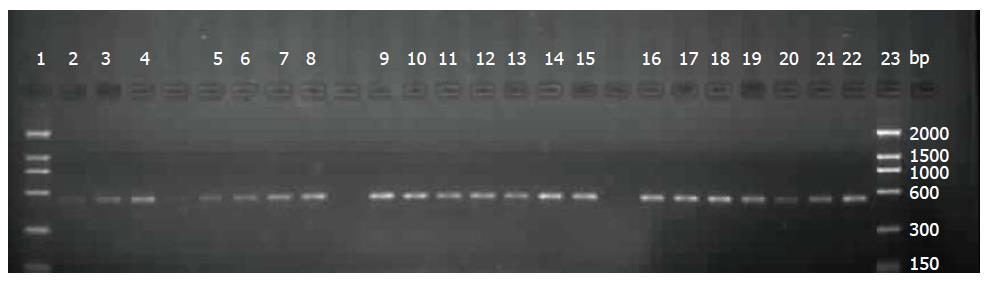
PCR扩增结石微生物16S rDNA的条带灰度均匀一致, 扩增片段长度相等(图1). 胆固醇组结石表面细菌16S rDNA阳性率为18.1%(2/11), 结石核心细菌16S rDNA阳性率为24.3%(3/11); 胆色素组结石表面细菌16S rDNA阳性率为42.3%(11/26), 结石核心细菌16S rDNA阳性率为53.8%(14/26); 混合性结石组结石表面细菌16S rDNA阳性率为36.0%(9/25), 结石核心细菌16S rDNA阳性率为64.0%(16/25)(表1). 胆固醇组结石表面与核心细菌16S rDNA阳性率比较(18.1% vs 24.3%, P = 0.500)无统计学差异; 胆色素组结石表面与核心细菌16S rDNA阳性率比较(42.3% vs 53.8%, P = 0.405)无统计学差异; 混合性组结石表面与核心细菌16S rDNA阳性率比较(36.0% vs 64.0%, P = 0.048)差异有统计学意义; 胆固醇组结石表面与胆色素组结石表面细菌16S rDNA阳性率比较(18.1% vs 42.3%, P = 0.152)无统计学差异; 胆固醇组结石表面与混合性组结石表面细菌16S rDNA阳性率比较(18.1% vs 36.0%, P = 0.254)无统计学差异; 胆色素组结石表面细菌与混合性组结石表面细菌16S rDNA阳性率比较(42.3% vs 36.0%, P = 0.645)无统计学差异; 胆固醇组结石核心与胆色素组结石核心细菌16S rDNA阳性率比较(24.3% vs 53.8%, P = 0.138) 无统计学差异; 胆固醇组结石核心与混合性组结石核心细菌16S rDNA阳性率比较(24.3% vs 64.0%, P = 0.042)差异有统计学意义; 胆色素组结石核心细菌与混合性组结石核心细菌16S rDNA阳性率比较(53.8% vs 64.0%, P = 0.461)无统计学差异.
| 分组 | 结石表面细菌16SrDNA检出率 | 结石核心细菌16S rDNA检出率 |
| 胆固醇组 | 18.1 | 24.3 |
| 胆色素组 | 42.3 | 53.8 |
| 混合性结石组 | 36.0 | 64.0 |
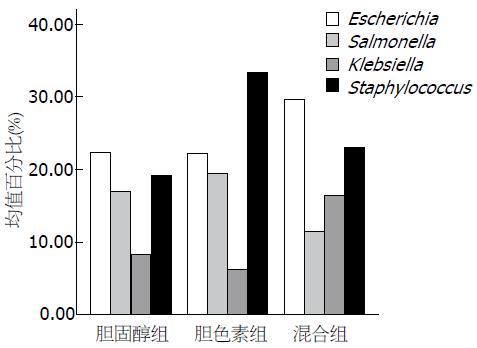
利用PCR-T-RFLP分析对微生物克隆文库中随机挑选的的27个阳性克隆进行分型筛选, 挑选代表克隆进行测序(北方诺塞基因公司), 获得微生物的16S rDNA部分序列. 对这些序列利用Blast分析与GenBank中核酸数据进行序列比对. 测序结果表明胆固醇结石中细菌主要为Enterobacteriaceac科和Mircrococcaccas科的微生物, 分布在Enterobacteriaceac的Escherichia属、Salmonella属和Klebsiella属, Mircrococcaccs科的Staphylococcus属. 胆色素结石中细菌分布较胆固醇结石更为广泛, 主要为Enterobacteriaceac科、Mircrococcaccas科、Streptococcuceac科、Peptococcaceac科和Propinibactericeac科的微生物, 分布在Enterobacteriaceac的Proteus属、Klebsiella属、Escherichia属和Salmonella属; Peptococcaceac的Peptococcus属和Peptostreptoroccus属; Streptococcuceac的Streptococcus属、Neisseriaceac的Acinetobacter属、Mircrococcaccas的Staphylococcus属和Propinibactericeac的Propinibacterium属. 混合性结石中细菌分布与胆色素结石中细菌分布相类似, 主要为Enterobacteriaceac、Mircrococcaccas、Streptococcuceac、Spirillaccac、Peptococcaceac、Corynebacterium和Propinibactericeac的微生物. 这些微生物群落在较高的分类学水平(例如科)的群落多样性并不很高, 而在属水平表现出了不同的多样性如图2. 特别指出本研究在三组胆固醇含量不同的结石中均检测到肠杆菌科(Enterobacteriaceac)的沙门氏菌属(Salmonella).
16S rDNA是编码原核生物核糖体小亚基rRNA (16S rDNA)的基因, 长度约为1500 bp, 是细菌分类学研究中最常用、最有用的"分子钟", 其序列包含10个可变区(variable region)和与之相间的11个恒定区(constant region), 可变区因细菌而异, 且变异程度与细菌的系统发育密切相关. 因此, 16S rDNA可以作为细菌菌落结构分析最常用的系统进化标记分子[8]. 利用16S rDNA可变区与恒定区的特性, 可以进行不同种属细菌的分类和鉴定[8,9]. 本研究中在常规细菌培养阴性的胆囊结石患者标本中确有较高的细菌16S rDNA检出率, 特别是在纯胆固醇结石、胆色素结石及混合性结石中均可检测到丰富的细菌16S rDNA, 表明无论特异性还是灵敏性, 16S rDNA PCR扩增技术均优于细菌培养, 并且其耗时仅为几小时.
本研究表明随着结石中胆固醇含量不同, 细菌微生物群落16S rDNA基因的检出率也不同, 纯胆固醇组细菌16S rDNA阳性率为30.0%(3/10), 胆色素组细菌16S rDNA阳性率为40.0%(4/10), 混合性结石组细菌16S rDNA阳性率为70.0%(7/10). 以利用T-RFLP技术对胆囊结石患者的结石细菌微生物群落16S rDNA基因序列进行分析表明, 胆囊结石的细菌群落主要为肠杆菌科(Enterobacteriaceac)、微球菌科(Mircrococcaccas)、螺菌科(Spirillaccac)、链球菌科(Streptococcus)、棒状菌群(Corynebacterium)、消化球菌科(Peptococcaceae)和丙酸杆菌科(Propinibactericeac)的微生物, 这些微生物群落在较高的分类学水平(例如科)的群落多样性并不很高, 而在属水平表现出了不同的多样性. 各组的优势菌群也有所不同, 但以肠杆菌科(Enterobacteriaceac)和微球菌科(Mircrococcaceae)为主. 这与已知的肠道菌群有较高的相似性, 由此推断定植于肠道的正常菌群或条件致病菌细菌在某种因素的影响下可以突破机体的防御系统通过Oddi氏括约肌进入胆道到达胆囊定植, 通过细菌本身和/或其代谢产物参与胆囊结石的形成. 细菌群落随胆固醇变化有较为显著的改变, 胆色素结石组和混合性结石组的细菌群落比纯胆固醇结石组中的细菌群落复杂, 并且胆色素结石组和混合性结石组的优势菌群与纯胆固醇结石组相比也发生了明显的变化, 胆色素结石组和混合性结石组的优势菌群与链球菌科和消化球菌科的细菌同源性最高, 而纯胆固醇结石组的优势菌群则是肠杆菌科的细菌同源性最高, 但是胆色素结石组和混合性结石组的细菌群落变化却并不显著, 能否由此推断由于结石胆固醇含量的不同而导致细菌群落发生多样性的改变这一结论有待进一步证实, 而目前唯一能够肯定的是由于胆囊对胆汁的浓缩功能, 胆汁中含有一些物质, 如胆汁酸盐、胆固醇和微量元素, 可作为电子受体或营养物质支持细菌生长代谢, 并使结石细菌群落表现出多样性.
特别指出本研究在三组胆固醇含量不同的结石中均检测到肠杆菌科(Enterobacteriaceac)的沙门氏菌属(Salmonella). 目前在体外研究发现[10-12], Salmonella能够形成生物被膜, 抵御胆盐的侵袭, 通过生物被膜与其他定植于胆囊的细菌相连接, 共同构成一个相对稳定的细菌群落, 将其生物被膜不断扩展以保护整个群落不受外界物质侵袭, 而胆汁恰恰是生物被膜形成的最佳刺激信号. 同时发现, Salmonella必须首先要在结石表面定植才能形成被膜, 而且成膜时间平均为10-14 d, 而其他能够成膜的细菌平均成膜时间为几秒钟至几小时不等. 本研究在三组实验组中均发现该细菌的16S rDNA片段绝非偶然, 但在体内Salmonella能否也能形成生物被膜有待证实, 并且胆囊内环境极为复杂, 胆囊的排空功能在体外难以模拟, 对此结论应审慎.
总之, 对于胆囊结石的形成, 我们可以提出一个综合作用假说. 首先, 必须存在胆固醇的高分泌和胆汁胆固醇过饱和状态. 当机体受到感染时, 细菌可通过胆道逆行扩散或通过门静脉系统侵入胆囊, 若其浓度不足以引起胆囊炎症时, 细菌便以低浓度的形式隐伏下来, 一方面产生促成核的代谢产物, 同其他促成核因子一起导致促/抗成核因子失衡[13-15], 并形成一个色素核心, 吸附沉积的钙盐和胆固醇; 另一方面, 细菌感染破坏胆囊功能, 造成胆囊动力减低, 粘液分泌增加, 促进成石, 而胆囊功能受损又利于细菌的侵袭, 上述几方面综合作用的结果导致形成胆囊结石.
本研究表明三组中混合性组的结石表面与结石核心细菌的16S rDNA检出率有统计学差异, 而胆固醇组、胆色素组结石表面与结石核心细菌的16S rDNA检出率无统计学意义, 胆固醇组与混合性结石组的结石核心细菌的16S rDNA检出率有统计学差异, 根据以上结果能否说明由于混合性结石组胆固醇含量为70%-89%, 在这个范围内细菌容易以低浓度的形式隐伏下来形成结石核心, 在结石形成中不断地参与结石的增大尚未肯定, 但能够说明细菌在混合性结石的形成中有重大的意义. 然而本研究由于种种原因, 并未涉及到对细菌代谢产物、胆囊结石患者胆汁、胆囊黏膜以及整个胆道系统细菌群落的同步研究. 推测对可疑致病菌的筛选、研究其生物学行为以及建立细菌的胆结石模型是今后的研究方向. 通过对胆道系统细菌的进一步研究, 深层次地探讨细菌在胆结石的发生、发展所起的作用, 有可能为胆石病的预防和治疗提供新的思路和方法.
自建立Small三角学说以来, 医学界始终认为胆囊结石的发病机制以非生理性胆固醇过饱和、胆汁促抗成核失调以及胆囊局部因素为基础, 但随着近年来大量的研究表明, 在胆囊结石患者的胆汁和结石中发现并确定幽门螺杆菌DNA的存在, 细菌参与结石形成的研究逐步开展起来. 研究的关键是首先揭示胆囊结石中细菌的成分和多样性以及它们确切的起源. 目前的研究受限于研究的条件和培养的灵敏度, 使培养得到的微生物在种类、数量和功能上都无法反映自然状态下微生物群落的真实情况. 所以, 很有必要研究不依赖微生物培养来进行环境微生物群落分析的方法.
陈海龙, 教授, 大连医科大学附属第一医院院办
目前, 在国外已有学者利用T-RFLP方法对人体阴道的微生物群落进行了分析, 但是, 尚未见利用分子微生物生态学方法对胆结石患者胆囊微生物群落进行分析的报道. 在国内, 对于分子微生物生态学的研究刚刚起步.
本论文属于微生态学、分子生物学技术和医学的新型交叉学科. 以分子微生物生态学的原理和手段分析人体胆囊结石微生物群落, 采用T-RFLP技术进行结石形成的细菌学研究, 与传统的16S rRNA基因克隆文库构建和Sanger测序法相结合的方法相比, 通量增加2-4个数量级, 成本降低约50-100倍.
本研究将首次较为全面地探明胆囊结石的微生物群落特征为更深刻地揭示胆结石的病因, 研发具有自主知识产权的防治胆囊炎、胆囊结石的微生物制剂, 制备出具有自主知识产权可快速检测胆囊微生物的一系列产品奠定重要的研究基础.
本研究采用T-RFLP技术对胆囊结石微生物群落16S rDNA基因序列进行分析来探讨胆囊结石成分与细菌微生物群落之间的关系, 探讨细菌在胆结石的发生发展所起的作用, 对胆结石形成机制的研究具有较好的科学意义和实际应用价值.
编辑: 田滢 电编:鲁亚静
| 1. | Toller IM, Neelsen KJ, Steger M, Hartung ML, Hottiger MO, Stucki M, Kalali B, Gerhard M, Sartori AA, Lopes M. Carcinogenic bacterial pathogen Helicobacter pylori triggers DNA double-strand breaks and a DNA damage response in its host cells. Proc Natl Acad Sci U S A. 2011;108:14944-14949. [PubMed] [DOI] |
| 2. | Salama NR, Hartung ML, Müller A. Life in the human stomach: persistence strategies of the bacterial pathogen Helicobacter pylori. Nat Rev Microbiol. 2013;11:385-399. [PubMed] [DOI] |
| 3. | Polk DB, Peek RM. Helicobacter pylori: gastric cancer and beyond. Nat Rev Cancer. 2010;10:403-414. [PubMed] [DOI] |
| 4. | Masella AP, Bartram AK, Truszkowski JM, Brown DG, Neufeld JD. PANDAseq: paired-end assembler for illumina sequences. BMC Bioinformatics. 2012;13:31. [PubMed] [DOI] |
| 5. | Cariati A, Cetta F. Re: Kawai et al.--Bacteria are not important in the formation of pure cholesterol stones. Am J Gastroenterol. 2002;97:2921-292; author reply 2921-292;. [PubMed] |
| 6. | Denaro R, D'Auria G, Di Marco G, Genovese M, Troussellier M, Yakimov MM, Giuliano L. Assessing terminal restriction fragment length polymorphism suitability for the description of bacterial community structure and dynamics in hydrocarbon-polluted marine environments. Environ Microbiol. 2005;7:78-87. [PubMed] |
| 7. | McGuinness LM, Salganik M, Vega L, Pickering KD, Kerkhof LJ. Replicability of bacterial communities in denitrifying bioreactors as measured by PCR/T-RFLP analysis. Environ Sci Technol. 2006;40:509-515. [PubMed] |
| 8. | Hayashi H, Takahashi R, Nishi T, Sakamoto M, Benno Y. Molecular analysis of jejunal, ileal, caecal and recto-sigmoidal human colonic microbiota using 16S rRNA gene libraries and terminal restriction fragment length polymorphism. J Med Microbiol. 2005;54:1093-1101. [PubMed] [DOI] |
| 9. | Thies FL, König W, König B. Rapid characterization of the normal and disturbed vaginal microbiota by application of 16S rRNA gene terminal RFLP fingerprinting. J Med Microbiol. 2007;56:755-761. [PubMed] [DOI] |
| 10. | Prouty AM, Schwesinger WH, Gunn JS. Biofilm formation and interaction with the surfaces of gallstones by Salmonella spp. Infect Immun. 2002;70:2640-2649. [PubMed] |
| 11. | Crawford RW, Gibson DL, Kay WW, Gunn JS. Identification of a bile-induced exopolysaccharide required for Salmonella biofilm formation on gallstone surfaces. Infect Immun. 2008;76:5341-5349. [PubMed] [DOI] |
| 12. | Nordentoft S, Mølbak L, Bjerrum L, De Vylder J, Van Immerseel F, Pedersen K. The influence of the cage system and colonisation of Salmonella Enteritidis on the microbial gut flora of laying hens studied by T-RFLP and 454 pyrosequencing. BMC Microbiol. 2011;11:187. [PubMed] [DOI] |
| 13. | Safaeian M, Gao YT, Sakoda LC, Quraishi SM, Rashid A, Wang BS, Chen J, Pruckler J, Mintz E, Hsing AW. Chronic typhoid infection and the risk of biliary tract cancer and stones in Shanghai, China. Infect Agent Cancer. 2011;6:6. [PubMed] [DOI] |
| 14. | Di Ciaula A, Wang DQ, Bonfrate L, Portincasa P. Current views on genetics and epigenetics of cholesterol gallstone disease. Cholesterol. 2013;2013:298421. [PubMed] [DOI] |