修回日期: 2014-03-20
接受日期: 2014-04-04
在线出版日期: 2014-05-18
目的: 探讨针对丙型肝炎病毒(hepatitis C virus, HCV)C基因的锁核酸核酶对病毒RNA复制与表达的特异性抑制作用.
方法: 设计合成能切割HCV C基因位点的DNAzyme、硫代DNAzyme和LNAzyme. 实验设对照组和实验组. 对照组包括空白对照组、脂质体对照组和脂质体-无关LNAzyme对照组. 实验组包括脂质体-DNAzyme、脂质体-硫代DNAzyme组和脂质体-LNAzyme组. 以阳离子脂质体介导转染HepG2.9706细胞. 采用荧光定量PCR和化学发光技术分别监测24、48、96 h细胞培养上清液中HCV RNA含量及荧光素酶基因表达; 四甲基偶氮唑蓝(MTT)法监测细胞代谢.
结果: 加入药物后, 脱氧核酶、硫代脱氧核酶及锁核酸核酶组对HCV RNA复制和荧光素酶基因表达的抑制作用均较对照组强(P<0.01), 其中, 锁核酸核酶的抑制作用均较脱氧核酶及硫代脱氧核酶明显(P<0.05), 平均抑制率分别达47.55%和52.44%, 且随用药时间延长, 抑制率呈增高趋势, 96 h后, HCV RNA复制和荧光蛋白表达的下降率均较用药前明显(P<0.01), 其中, 锁核酸核酶的抑制作用均较脱氧核酶及硫代脱氧核酶明显(P<0.05), 平均下降率分别达79.40%和80.05%. 而LNAzyme对细胞活性基本无影响.
结论: LNAzyme能特异性抑制HCV C基因的复制与表达, 且优于硫代修饰的DNAzyme.
核心提示: 针对丙型肝炎病毒(hepatitis C virus, HCV)C基因位点的锁核酸核酶, 体外能特异性抑制病毒基因调控, 为抗HCV基因治疗提供理论和实验依据, 有望成为RNA病毒基因治疗的新型反义核酸药物.
引文著录: 邓益斌, 农乐根, 梁祚仁, 张梁, 覃羽华, 何平. LNAzyme设计及其体外特异性抑制丙型肝炎病毒C基因的表达. 世界华人消化杂志 2014; 22(14): 1992-1997
Revised: March 20, 2014
Accepted: April 4, 2014
Published online: May 18, 2014
AIM: To investigate the inhibitory effects of locked nucleic acid enzyme targeting the hepatitis C virus (HCV) C gene on HCV RNA replication and expression in HepG2.9706 cells.
METHODS: The sequences encoding DNAzyme, thiolmodificated DNAzyme and LNAzyme targeting the HCV C gene were designed and synthesized. The following experimental groups were set up: lipo-DNAzyme, lipo-S-DNAzyme, lipo-LNAzyme, blank control, empty liposomes, and lipo-random-LNAzyme. Transfection was performed using cationic liposomes. The level of HCV RNA and luciferase gene expression in supernatants were tested by real-time fluorescent quantitative PCR and chemiluminescence technique 24, 48 and 96 h after treatment, respectively. Cytotoxicity of LNAzyme was evaluated by MTT assay.
RESULTS: Significant down-regulation of HCV RNA replication and luciferase gene expression was noted in the lipo-LNAzyme group, lipo-DNAzyme group and lipo-S-DNAzyme group compared with the control group (P < 0.05 for all). Relative to the lipo-DNAzyme group and lipo-S-DNAzyme group, the average inhibition rates in the lipo-LNAzyme group were 47.55% and 52.44%, respectively. With the prolongation of the treatment time, the inhibition rate increased. At 96 h, HCR RNA replication and fluorescent protein expression were significantly lower than those before treatment in the lipo-LNAzyme group (P < 0.01 for both), and the average inhibition rates were 79.40% and 84.05%, respectively. No obvious toxicity was observed.
CONCLUSION: LNAzyme has a significant inhibitory effect on HCV C gene replication and expression in vitro, which is stronger than that of the thiolmodificated DNAzyme.
- Citation: Deng YB, Nong LG, Liang ZR, Zhang L, Qin YH, He P. Hepatitis C virus gene-specific locked nucleic acid enzyme significantly inhibits C gene expression in vitro. Shijie Huaren Xiaohua Zazhi 2014; 22(14): 1992-1997
- URL: https://www.wjgnet.com/1009-3079/full/v22/i14/1992.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v22.i14.1992
丙型肝炎病毒(hepatitis C virus, HCV)是单股正链RNA病毒, 含5'非编码区、结构区(C、E区)、非结构区(NS区)、和3'非编码区, 其中, C区是丙型肝炎病毒复制所必需的元件, 是HCV十分保守的序列之一. C区开放读码框架(open reading frame, ORF)的编码蛋白是大分子多聚蛋白前体, 在宿主信号肽酶及NS3蛋白激酶的作用下, 裂解成各种小分子病毒蛋白, 很显然, C区在病毒基因表达调控中发挥着重要的作用. 因此, 推测切割破坏该位点RNA序列可能有效阻断病毒的基因表达. 锁核酸(locked nucleic acid, LNA)是新发现的一种带环状结构的核苷酸衍生物, 与其他寡核苷酸相比, 具有热稳定性好[1-5]、分子杂交能力强[6-10]、抗酶解能力强[11-15]、脂溶性好[16-20]和低毒性[21-24]等优势. 锁核酸核酶(locked nucleic acid enzyme, LNAzyme)是在脱氧核酶的2条结合臂上引入1个或多个锁核酸单体构建而成的新型核酸酶, 与其他核酸酶相比, 具有更强的分子杂交能力、更高的酶切活性和切割效率[25-28]. 因此, 在前期研究基础上[29-31], 进一步针对HCV C区ORF的AUG位点设计合成两侧臂长约7-10 nt, 环结构由GGCTAGCTACAACGA碱基组成的LNAzyme, 以阳离子脂质体介导转染HepG2.9706细胞株, 观察其对病毒复制与表达的抑制作用, 以期为抗HCV治疗寻找专一、高效的新型分子药物.
HepG2.9706细胞是一种转染有质粒pHCV-neo的转基因HepG2细胞, 含荧光素酶基因, 由中国人民解放军广州军区空军医院刘光泽博士惠赠, 本室常规培养于含G418(380 ITI1)、10%胎牛血清的DMEM培养基中, 37 ℃、5%CO2条件下5-6 d传代1次; DMEM培养基、G418等购自GIBCO公司; 胎牛血清购自杭州四季清公司; LipofectamineTM 2000购自Invitrogen公司; TRIzol RNA提取试剂盒购自GIBCO公司; 荧光定量PCR检测HCV RNA试剂盒购自深圳匹基公司; 化学发光试剂盒(Bright-GloTM荧光素酶分析系统)购自Promega公司; VictorTM Wallac 1420多标记检测仪购自Promega公司; EVOS® FL Auto全自动荧光倒置显微镜系统购自美国LIFE公司.
1.2.1 核酶分子设计与合成: 针对HCV C区ORF的AUG位点(341-356 nt)分别设计以下几段序列: (1)DNAzyme: 5'-UAGGAUUGGCTAGCTACAACGACGAGUGCUCAU-3'; (2)硫代DNAzyme: 5'-UASGGASUSUGGCTAGCTACAACGACGAGUSGCUSCASU-3', 即在DNAzyme基础上对5'端前5个碱基及3'端前5个碱基进行硫代修饰, 以上标"S"表示; (3)LNAzyme: 5'-UALGGALULUGGCTAGCTACAACGACGAGULGCULCALU-3', 即在DNAzyme的2个结合臂碱基A或T分别引入2个LNA单体, 以上标"L"表示; (4)无关DNAzyme: 5'-TCCAGAGGGGCTAGCTACAACGACATACAGC-3'. 其中, GGCTAGCTACAACGA为DNAzyme的催化活性序列, 其2侧为靶位特异性识别序列. 以上各序列经BLAST排除与人同源后由美国GENELINK公司合成修饰.
1.2.2 实验分组与脂质体转染: 本实验设对照组和实验组. 对照组包括空白对照组、脂质体对照组、脂质体-无关DNAzyme对照组. 实验组包括脂质体-DNAzyme组、脂质体-硫代DNAzyme组、脂质体-LNAzyme组. 将HepG2.9706细胞按1×105个/mL接种于16孔培养板, 每孔100 μL, 共设定6个组, 每组各设6个复孔, 待细胞贴壁后吸取培养上清液(-20 ℃保存), 分别在各组每孔中一次性加入含核酶量为10 μmol/L的DMEM混合液1 mL, 分别于24、48、96 h收集各孔培养上清液500 μL, 保存于-20 ℃待测. 脂质体转染严格按说明书操作.
1.2.3 培养上清液HCV RNA含量检测: 采用荧光定量PCR试剂盒法. (1)在预先准备好的1.5 mL灭菌离心管中加入RNA酶抑制剂20 μL, 然后分别加入待测液, 阴、阳性对照和标准品各100 μL, 再加入裂解液100 μL, 振荡混匀; (2)将上述混合液全部转移至核酸提取柱中, 开启负压装置, 抽干液体, 然后分别用洗涤液A和B液各500 μL, 各洗涤1次, 抽干; (3)关闭负压装置, 将提取柱转入2 mL离心管中, 15000 r/min, 离心1 min; (4)将提取转入新2 mL离心管中, 加入50 μL洗脱液, 静置1 min, 10000 r/min, 离心1 min, 取12.5 μL洗脱液加样, 余下步骤和PCR扩增条件严格按说明书操作. 结果采用计算对数平均值的方法获得cDNA平均拷贝数.
1.2.4 培养细胞内荧光蛋白表达检测: 分别于基因转染后24、48、96 h终止培养, 将培养板平衡至室温, 向每孔中加入经室温平衡后的荧光素酶发光试剂100 μL, 轻轻摇匀2 min以使细胞完全裂解, 立即应用VictorTM Wallac 1420多标记检测仪检测1 s发光强度, 输出值以每秒计算值(counts per second, CPS)表示.
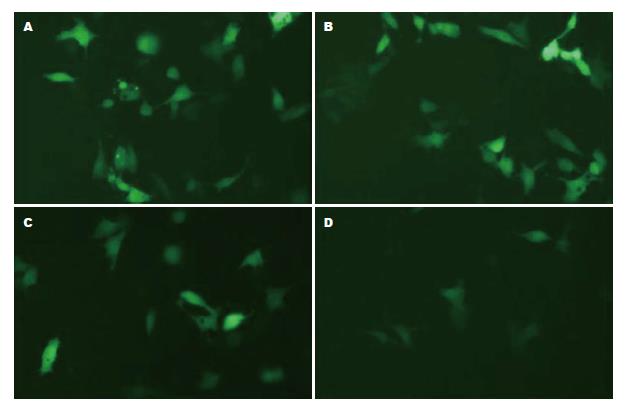
1.2.5 荧光显微镜系统观察细胞荧光蛋白表达: 培养细胞转染96 h后, 用倒转荧光显微镜系统在488 nm激发波长下观察绿色荧光的表达情况, 并拍照.
1.2.6 LNAzyme对细胞的毒性检测: 采用MTT比色法检测LNAzyme对细胞活性的影响. 加入LNAzyme后, 5%CO2, 37℃培养96 h, 每孔加入20 μL 0.5%MTT液, 继续培养4 h后, 终止培养, 小心吸弃培养上清液; 每孔加入150 μL二甲基亚砜, 低速振荡孵育10 min, 使结晶物充分融解; 在酶联免疫监测仪上检测各孔吸光度值, 波长490 nm/570 nm, 同时设置调零孔和对照孔.
统计学处理 所有数据均采用mean±SD表示, 应用SPSS13.0统计软件处理, 组间比较采用重复测量设计方差分析, P<0.05为差异具有统计学意义. 抑制率(%) = (用药前N-用药后N)/用药前N×100%, 或(对照组N-实验组N)/对照组N×100%, 其中, N表示测定值.
加入药物后, 各实验组对HCV RNA的复制均显示出较强的抑制作用(F = 227.50, P<0.01), 其中, 脂质体-LNAzyme组的抑制作用最强(F = 67.50, P<0.05), 平均抑制率为47.55%. 与用药前比较, HCV RNA的复制量也明显下降(F = 156.27, P<0.01), 96 h后, 脂质体-LNAzyme组的平均下降率达79.40%(表1).
| 分组 | 0 h | 24 h | 48 h | 96 h |
| 空白对照组 | 1.52±0.44 | 1.55±0.47 | 1.61±0.52 | 1.65±0.44 |
| 脂质体对照组 | 1.62±0.45 | 1.58±0.46 | 1.67±0.44 | 1.65±0.43 |
| 脂质体-无关LNAzyme对照组 | 1.55±0.45 | 1.57±0.43 | 1.62±0.48 | 1.68±0.42 |
| 脂质体-DNAzyme组 | 1.65±0.58 | 1.23±0.49 | 1.02±0.55 | 0.83±0.53 |
| 脂质体-硫代DNAzyme组 | 1.62±0.48 | 1.02±0.53 | 0.81±0.58 | 0.75±0.42 |
| 脂质体-LNAzyme组 | 1.65±0.44 | 0.83±0.31 | 0.50±0.22 | 0.34±0.15bceh |
加入药物后, 各实验组对细胞荧光素酶基因的表达均显示出较强的抑制作用(F = 78.65, P<0.01), 其中, 脂质体-LNAzyme组的抑制作用最强(F = 72.35, P<0.05), 平均抑制率为52.44%. 与用药前比较, HCV RNA的表达量也明显下降(F = 186.21, P<0.01), 96 h后, 脂质体-LNAzyme组的平均下降率达84.05%(表2).
| 分组 | 0 h | 24 h | 48 h | 96 h |
| 空白对照组 | 4.97±2.92 | 4.95±2.89 | 4.96±2.81 | 4.97±2.79 |
| 脂质体对照组 | 4.89±2.91 | 4.91±2.85 | 4.89±2.75 | 4.91±2.87 |
| 脂质体-无关LNAzyme对照组 | 4.93±2.88 | 4.89±2.78 | 4.91±2.83 | 4.89±2.78 |
| 脂质体-DNAzyme组 | 4.87±2.66 | 3.57±2.66 | 3.02±2.69 | 2.41±2.44 |
| 脂质体-硫代DNAzyme组 | 4.91±2.87 | 3.04±2.61 | 2.44±2.15 | 2.21±1.88 |
| 脂质体-LNAzyme组 | 4.89±2.45 | 2.11±1.52 | 1.66±1.23 | 0.78±0.25bceh |
转染96 h后在荧光电子显微镜下观察, 脂质体-LNAzyme组细胞内荧光蛋白的表达明显少于对照组、脱氧核酶组及硫代脱氧核酶组(图1).
用药7 d后, 采用MTT比色法测定各组A值, 脂质体-DNAzyme、脂质体-硫代DNAzyme和脂质体-LNAzyme组的A值分别为1.14±0.04、1.15±0.04和1.15±0.04, 与空白对照组的1.37±0.04比较均无差异, 表明锁核酸脂质体混合物对细胞活性基本无影响.
本研究针对丙型肝炎病毒5'非编码区内源性核糖体进入位点(internal ribosome entry site, IRES)位点设计合成LNAzyme, 并以阳离子脂质体介导转染HepG2.9706细胞, 采用分子生物学技术分别检测培养上清液中HCV RNA含量及荧光素酶基因表达, 观察锁核酸核酶对病毒基因表达的影响, 结果显示, 脱氧核酶、硫代脱氧核酶及锁核酸核酶对丙型肝炎病毒RNA的复制及细胞内荧光素酶基因的表达均显示出较强的抑制作用(P<0.01), 其中, 锁核酸核酶的抑制作用均较脱氧核酶及硫代脱氧核酶强(P<0.05), 平均抑制率分别达48.02%和53.05%, 而且, 随用药时间延长, 抑制率呈增高趋势, 用药后96 h, HCV RNA复制和荧光蛋白表达的下降率均较用药前明显(P<0.01), 其中, 锁核酸核酶的抑制作用均较脱氧核酶及硫代脱氧核酶明显(P<0.05), 平均抑制率分别达81.21%和84.25%. 此外, MTT比色结果表明, LNAzyme脂质体混合物对细胞活性基本无影响.
总之, 针对HCV 5'非编码区IRES位点的锁核酸核酶, 体外能特异性抑制病毒基因调控, 为抗丙型肝炎病毒基因治疗提供理论和实验依据, 有望成为RNA病毒基因治疗的新型反义核酸药物.
反义核酸技术, 包括反义寡核苷酸、反义寡聚脱氧核苷酸、核酶等技术, 其中, 核酶技术又包括核酶和脱氧核酶技术, 已被证实是研究、治疗病毒感染性疾病、遗传性疾病、恶性肿瘤等多种疾病的有效手段之一, 主要通过针对在疾病发生、发展过程中发挥重要作用的生长因子、受体、关键酶、原癌基因、抑癌基因或凋亡相关基因, 通过导入外源治疗基因(DNA、RNA、核酶等)短序列, 特异性识别、结合、封闭或调控其功能及产物表达, 从而达到治疗目的.
唐世刚, 教授, 湖南省人民医院
核酶技术作为分子生物学的新型基因治疗技术, 目前不仅广泛应用于生物学、病理学、药理学等学科的基础研究, 而且已成为药物发展的新兴策略. 在核酶技术基础上发展起来的锁核酸核酶技术, 是目前研究的新热点. 当前抗病毒基因治疗亟待解决的问题一是没有一种满意的肝靶向性药物载体, 另外就是缺乏特异性抗病毒核酸药物.
邓益斌等针对乙肝病毒S保守区S基因mRNA的翻译起始位点, 设计合成反义LNA片段, 进行体内外实验研究, 结果发现, 反义LNA对靶mRNA基因表达的抑制作用明显强于其他寡核苷酸, 并且存在剂量和时间效应, 当反义LNA片段长度在20-25个核苷酸时, 更容易进入细胞内, 与靶mRNA形成LNA-RNA杂交分子, 封闭靶mRNA基团翻译的同时, 激活RNase H酶活性, 引起靶mRNA的降解, 从翻译水平阻断病毒靶基因表达, 降低病毒蛋白的合成, 抑制病毒装配、成熟和分泌过程, 从而抑制HBV病毒的复制.
锁核酸(locked nucleic acid, LNA)是一种新兴环状核苷酸衍生物, 具有热稳定性好、分子杂交能力强、抗核酸酶解能力高等特性, 本研究在以寡聚脱氧核酶分子的双侧臂上各引入2-4个LNA分子构建成锁核酸核酶(LNAzyme), 从而增强核酶分子与底物RNA的杂交亲和力, 同时, 增强其在细胞内稳定性和酶切能力.
锁核酸核酶在以RNA序列为切割底物的基因治疗研究中具有潜在研究价值, 特别是RNA病毒, 如丙型肝炎病毒、艾滋病病毒等.
该文设计了针对丙型肝炎病毒C基因的LNAzyme, 体外特异性抑制病毒基因调控, 具有一定研究价值.
编辑 郭鹏 电编 鲁亚静
| 1. | Crouzier L, Dubois C, Edwards SL, Lauridsen LH, Wengel J, Veedu RN. Efficient reverse transcription using locked nucleic acid nucleotides towards the evolution of nuclease resistant RNA aptamers. PLoS One. 2012;7:e35990. [PubMed] [DOI] |
| 2. | Hildebrandt-Eriksen ES, Aarup V, Persson R, Hansen HF, Munk ME, Ørum H. A locked nucleic acid oligonucleotide targeting microRNA 122 is well-tolerated in cynomolgus monkeys. Nucleic Acid Ther. 2012;22:152-161. [PubMed] [DOI] |
| 3. | Mishra S, Ghosh S, Mukhopadhyay R. Ordered self-assembled locked nucleic acid (LNA) structures on gold(111) surface with enhanced single base mismatch recognition capability. Langmuir. 2012;28:4325-4333. [PubMed] [DOI] |
| 4. | Robertson KL, Vora GJ. Locked nucleic acid flow cytometry-fluorescence in situ hybridization (LNA flow-FISH): a method for bacterial small RNA detection. J Vis Exp. 2012;e3655. [PubMed] [DOI] |
| 5. | Hull C, Szewcyk C, St John PM. Effects of locked nucleic acid substitutions on the stability of oligonucleotide hairpins. Nucleosides Nucleotides Nucleic Acids. 2012;31:28-41. [PubMed] [DOI] |
| 6. | Owczarzy R, You Y, Groth CL, Tataurov AV. Stability and mismatch discrimination of locked nucleic acid-DNA duplexes. Biochemistry. 2011;50:9352-9367. [PubMed] [DOI] |
| 7. | Bhattacharyya J, Maiti S, Muhuri S, Nakano S, Miyoshi D, Sugimoto N. Effect of locked nucleic acid modifications on the thermal stability of noncanonical DNA structure. Biochemistry. 2011;50:7414-7425. [PubMed] [DOI] |
| 8. | Doessing H, Vester B. Locked and unlocked nucleosides in functional nucleic acids. Molecules. 2011;16:4511-4526. [PubMed] [DOI] |
| 9. | Zhang Y, Qu Z, Kim S, Shi V, Liao B, Kraft P, Bandaru R, Wu Y, Greenberger LM, Horak ID. Down-modulation of cancer targets using locked nucleic acid (LNA)-based antisense oligonucleotides without transfection. Gene Ther. 2011;18:326-333. [PubMed] |
| 10. | Chabot S, Orio J, Castanier R, Bellard E, Nielsen SJ, Golzio M, Teissié J. LNA-based oligonucleotide electrotransfer for miRNA inhibition. Mol Ther. 2012;20:1590-1598. [PubMed] [DOI] |
| 11. | Johannsen MW, Veedu RN, Madsen AS, Wengel J. Enzymatic polymerisation involving 2'-amino-LNA nucleotides. Bioorg Med Chem Lett. 2012;22:3522-3526. [PubMed] [DOI] |
| 12. | Chaubey AK, Dubey KD, Ojha RP. Stability and free energy calculation of LNA modified quadruplex: a molecular dynamics study. J Comput Aided Mol Des. 2012;26:289-299. [PubMed] [DOI] |
| 13. | Sun Z, Xiang W, Guo Y, Chen Z, Liu W, Lu D. Inhibition of hepatitis B virus (HBV) by LNA-mediated nuclear interference with HBV DNA transcription. Biochem Biophys Res Commun. 2011;409:430-435. [PubMed] [DOI] |
| 14. | Moschos SA, Frick M, Taylor B, Turnpenny P, Graves H, Spink KG, Brady K, Lamb D, Collins D, Rockel TD. Uptake, efficacy, and systemic distribution of naked, inhaled short interfering RNA (siRNA) and locked nucleic acid (LNA) antisense. Mol Ther. 2011;19:2163-2168. [PubMed] [DOI] |
| 15. | Ivanova MI, Shivarov VS, Hadjiev EA, Naumova EJ. Novel multiplex bead-based assay with LNA-modified probes for detection of MPL exon 10 mutations. Leuk Res. 2011;35:1120-1123. [PubMed] [DOI] |
| 16. | Seth PP, Allerson CR, Siwkowski A, Vasquez G, Berdeja A, Migawa MT, Gaus H, Prakash TP, Bhat B, Swayze EE. Configuration of the 5'-methyl group modulates the biophysical and biological properties of locked nucleic acid (LNA) oligonucleotides. J Med Chem. 2010;53:8309-8318. [PubMed] [DOI] |
| 17. | Swayze EE, Siwkowski AM, Wancewicz EV, Migawa MT, Wyrzykiewicz TK, Hung G, Monia BP, Bennett CF. Antisense oligonucleotides containing locked nucleic acid improve potency but cause significant hepatotoxicity in animals. Nucleic Acids Res. 2007;35:687-700. [PubMed] [DOI] |
| 18. | Yang CJ, Wang L, Wu Y, Kim Y, Medley CD, Lin H, Tan W. Synthesis and investigation of deoxyribonucleic acid/locked nucleic acid chimeric molecular beacons. Nucleic Acids Res. 2007;35:4030-4041. [PubMed] [DOI] |
| 19. | Brunet E, Corgnali M, Cannata F, Perrouault L, Giovannangeli C. Targeting chromosomal sites with locked nucleic acid-modified triplex-forming oligonucleotides: study of efficiency dependence on DNA nuclear environment. Nucleic Acids Res. 2006;34:4546-4553. [PubMed] [DOI] |
| 20. | Levin JD, Fiala D, Samala MF, Kahn JD, Peterson RJ. Position-dependent effects of locked nucleic acid (LNA) on DNA sequencing and PCR primers. Nucleic Acids Res. 2006;34:e142. [PubMed] [DOI] |
| 21. | Johnson MP, Haupt LM, Griffiths LR. Locked nucleic acid (LNA) single nucleotide polymorphism (SNP) genotype analysis and validation using real-time PCR. Nucleic Acids Res. 2004;32:e55. [PubMed] [DOI] |
| 22. | Crinelli R, Bianchi M, Gentilini L, Palma L, Sørensen MD, Bryld T, Babu RB, Arar K, Wengel J, Magnani M. Transcription factor decoy oligonucleotides modified with locked nucleic acids: an in vitro study to reconcile biostability with binding affinity. Nucleic Acids Res. 2004;32:1874-1885. [PubMed] [DOI] |
| 23. | Sørensen JJ, Nielsen JT, Petersen M. Solution structure of a dsDNA: LNA triplex. Nucleic Acids Res. 2004;32:6078-6085. [PubMed] [DOI] |
| 24. | Hertoghs KM, Ellis JH, Catchpole IR. Use of locked nucleic acid oligonucleotides to add functionality to plasmid DNA. Nucleic Acids Res. 2003;31:5817-5830. [PubMed] [DOI] |
| 25. | Dolinsek J, Dorninger C, Lagkouvardos I, Wagner M, Daims H. Depletion of unwanted nucleic acid templates by selective cleavage: LNAzymes, catalytically active oligonucleotides containing locked nucleic acids, open a new window for detecting rare microbial community members. Appl Environ Microbiol. 2013;79:1534-1544. [PubMed] [DOI] |
| 26. | Suryawanshi H, Lalwani MK, Ramasamy S, Rana R, Scaria V, Sivasubbu S, Maiti S. Antagonism of microRNA function in zebrafish embryos by using locked nucleic acid enzymes (LNAzymes). C. hembiochem. 2012;13:584-589. [PubMed] [DOI] |
| 27. | Kaur H, Scaria V, Maiti S. "Locked onto the target": increasing the efficiency of antagomirzymes using locked nucleic acid modifications. Biochemistry. 2010;49:9449-9456. [PubMed] [DOI] |
| 28. | Donini S, Clerici M, Wengel J, Vester B, Peracchi A. The advantages of being locked. Assessing the cleavage of short and long RNAs by locked nucleic acid-containing 8-17 deoxyribozymes. J Biol Chem. 2007;282:35510-35518. [PubMed] [DOI] |