修回日期: 2013-01-20
接受日期: 2013-01-25
在线出版日期: 2013-02-28
目的: 分析黑龙江地区丙型肝炎病毒(hepatitis C virus, HCV)基因型的流行特征和相关的影响因素, 以便对HCV感染的控制和个体化治疗提供依据.
方法: 采用型特异性巢式多重PCR法对外周血血浆中HCV基因亚型1a、1b、2a和2b进行检测, 分析HCV基因亚型的组成及其与性别、年龄、HCV RNA定量和丙氨酸氨基转移酶(alanine aminotransferase, ALT)水平的关系.
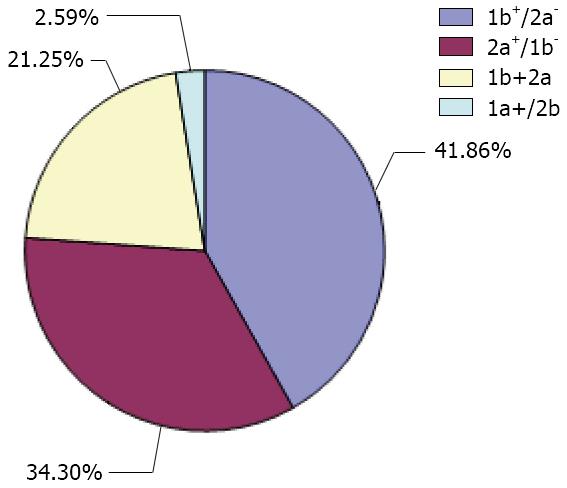
结果: 通过对1 313例HCV感染者外周血血浆标本的检测和分析, HCV基因亚型检出率为70.60%(927/1 313), 其中以1b、2a和1b与2a混合感染为主, 尚包括少量分别与1a和2b的混合感染, 即1b+/2a-为388例(41.86%)、2a+/1b-为318例(34.30%)和1b+2a为197例(21.25%), 单纯1a或2b以及二者混合(1a+/2b)仅检出24例, 占2.59%. 2a+/1b-在女性组(37.50%)、HCV RNA(1.000-9.999)×104组(54.55%)、ALT水平>200组(60.87%)和≤40组(41.02%)比率均较相应的其他各组有意义的增高(P<0.05), 而1b+/2a-在HCV RNA(1.000-9.999)×104组(24.24%)和ALT水平>200组(13.04%)均较相应的其他各组有意义的降低(P<0.05), 未发现与年龄有关.
结论: 本地区HCV主要流行株是1b和2a, 其分布可能与性别、HCV RNA水平和ALT水平有关, 随着医疗条件的改善和联合治疗的开展, 本地区HCV感染有望得到较好的控制.
引文著录: 杜博, 金茜, 刘伟, 李兴库, 于雪源, 张淑云. 黑龙江地区丙型肝炎病毒的基因亚型. 世界华人消化杂志 2013; 21(6): 531-536
Revised: January 20, 2013
Accepted: January 25, 2013
Published online: February 28, 2013
AIM: To analyze the distribution of different hepatitis C virus (HCV) subgenotypes in Heilongjiang province, China.
METHODS: Serum samples from 1 313 patients with HCV infection, including 627 (47.75%) males and 686 (52.25%) females, were subgenotyped using multiplex nested PCR assay. The associations of HCV subgenotypes with gender, age, viral load, and ALT level were analyzed.
RESULTS: Of 1 313 patients, 927 (70.60%) were successfully subgenotyped. Among typable subgenotypes, 1b+/2a- was found in 388 (41.86%) cases, 2a+/1b- in 318 (34.30%) cases, 1b+2a in 197 (21.25%) cases, and 1a+/2b in 24 (2.59%) cases. The percentage of patients with 2a+/1b- subgenotype was significantly higher in females (37.50%) and patients with HCVRNA of (1.000-9.999) × 104 (54.55%), ALT level >200 (60.87%) or ≤ 40 (41.02%) (all P < 0.05). The percentage of patients with 1b+/2a- subgenotype was significantly lower in patients with HCVRNA of (1.000-9.999) × 104 (24.24%) or ALT level >200 (13.04%) (both P < 0.05).
CONCLUSION: Genotypes 1b and 2a are the most prevalent genotypes in Heilongjiang province. The distribution of different HCV subgenotypes is associated with gender, viral load, and ALT level.
- Citation: Du B, Jin X, Liu W, Li XK, Yu XY, Zhang SY. Analysis of hepatitis C virus subgenotypes in patients in Heilongjiang province. Shijie Huaren Xiaohua Zazhi 2013; 21(6): 531-536
- URL: https://www.wjgnet.com/1009-3079/full/v21/i6/531.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i6.531
丙型肝炎病毒(hepatitis C virus, HCV)感染是造成慢性肝炎、肝硬化及肝癌的重要原因之一. 目前HCV感染仍然是全球性健康问题. HCV RNA全长约9.6 kb, 易发生变异, 在长期的进化中形成了不同的基因型(genotype)、基因亚型(subgenotype)、病毒株(isolate)和准种(quasispecies), 核酸序列差异率>30%为不同基因型, 差异率在15%-30%之间为不同基因亚型[1-3]. 1974年Goldfield[4]首次报道了非甲非乙型肝炎, 启动了对其病因的探索. 尤其是在1982-1988年期间美国Chiron公司Houghton实验室的研究人员Choo等[5]用分子杂交和克隆等技术, 在感染的黑猩猩血液标本中获得了一种能导致非甲非乙型肝炎的病毒片断, 并正式将其命名为"丙型肝炎病毒(HCV)". 1991年国际病毒命名委员会将HCV归入了黄病毒科(Flavivirus)丙型肝炎病毒属[5,6]. 1993年Simmonds等[7]通过对NS5B区扩增测序后进行系统进化树分析, 确定了6种基因型和一系列基因亚型. 2005年将其作为国际通用的命名方法, 即以阿拉伯数字1-6表示基因型, 以小写的英文字母(abc等)表示基因亚型[8]. 目前已报道的较公认的基因型有6种, 基因亚型已超过100种[9]. 国内外研究表明, HCV不同基因(亚)型在地区分布、传播途径、疾病进展和抗病毒治疗上均有差异, 基因分型及动态变化已成为制定HCV感染防控策略的重要依据[3,10-13]. 为了解黑龙江地区HCV基因型分布状况, 探讨HCV基因型流行特征, 以便更好地指导HCV感染的治疗和预防, 我们对本地区1 313例HCV感染患者外周血血浆标本进行了HCV基因亚型的检测和分析.
2009-06/2011-12就诊于哈尔滨医科大学附属第二医院的HCV感染患者1 313例, 均为抗-HCV阳性和拟进行抗病毒治疗. 其中男627例, 女686例, 年龄12-87岁, 平均50.68岁±11.14岁. HCV感染诊断符合《丙型肝炎防治指南》(2004). TRIzol购自北京博迈德科技发展有限公司; AMV反转录试剂盒购自ABI公司; Taq DNA聚合酶购自Fermentas公司; HCV RNA型特异性引物引自文献[14], 由南京金思特科技有限公司合成. 引物序列如下: P1: 5'-ATGTACCCCATGAGGTCGGC-3'; P2: 5'-CGCGCGACTAGGAAGACTTC-3'; P共用: 5'-AGGAAGACTTCCGAGCGGTC-3'; P1a型: 5'-TGCCTTGGGGATAGGCTGAC-3'; P1b型: 5'-GAGCCATCCTGCCCACCCCA-3'; P2a型: 5'-CCAAGAGGGACGGGAACCTC-3'; P2b型: 5'-ACCCTCGTTTCCGTACAGAG-3'.
根据试剂使用说明书采用TRIzol法提取HCV感染患者外周血血浆中病毒RNA和进行AMV反转录; 参考文献[14]进行巢式多重PCR扩增; 取第二轮PCR扩增产物10 μL和MarkerDL2000 10 μL, 经3%琼脂糖凝胶电泳, Tanon凝胶成像系统成像、保存和观察结果.
统计学处理 数据处理采用统计学软件SPSS17.0, 计数资料的比较均采用χ2检验(chi-square test), 计量资料比较采用秩和检验(rank-sum test), 其中基因型在年龄以及HCV RNA和ALT水平上总体比较使用Kruskal-Wallis检验, 组间比较使用Mann-Whitney检验, P<0.05有统计学意义.
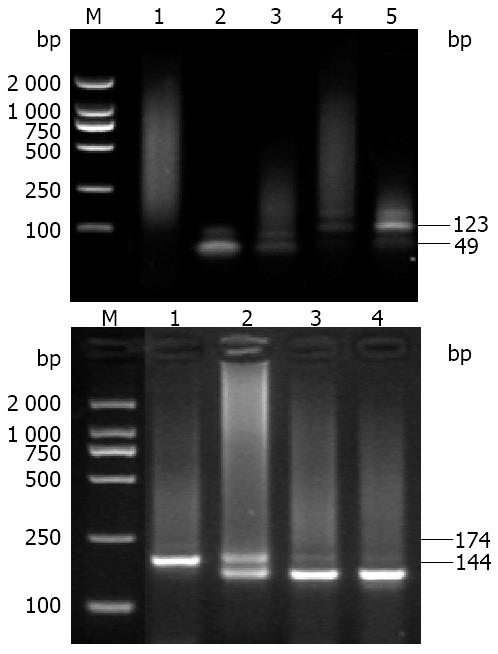
采用型特异性巢式多重PCR法对HCV RNA进行1a、1b、2a和2b基因亚型检测, 电泳结果见图1, 1a: 49 bp; 1b: 144 bp; 2a: 174 bp; 2b: 123 bp.
在1 313例患者HCV RNA基因亚型检测中, 927例检出基因型, 检出率为70.60%, 其中以1b、2a和1b与2a混合感染为主, 尚包括少量分别与其他型(1a/2b)的混合感染, 即1b+/2a-为388例(41.86%)、2a+/1b-为318例(34.30%)和1b+2a为197例(21.25%), 单纯1a或2b以及二者混合(1a+/2b)仅检出24例, 占2.59%(图2).
在1 313例患者中, 男627例, 占47.75%, 检出HCV基因亚型439例, 检出率为70.02%; 女686例, 占52.25%, 检出HCV基因亚型488例, 检出率为71.14%. 男女比例匹配. 在两组中基因亚型分布总体未见差异(χ2 = 5.383, P = 0.146), 只是2a+/1b-在女性组中比例明显高于男性组, 有统计学意义(χ2 = 4.670, P = 0.031, 表1).
在1 313例患者中1 283份有年龄记录, 其中906例检出HCV基因亚型, 检出率70.62%, 与总检出率70.60%未有明显差异(χ2 = 0.000, P = 0.994). 通过进一步的分层分析, 各基因亚型组在不同年龄组中的比率也均未见差异(χ2 = 11.754, P = 0.860). 又通过秩检验1b+/2a-、2a+/1b-、1b+2a和1a+/2b 4组年龄秩均值分别为 459.71、449.50、447.28和457.46, 总体上也未见统计学差异(χ2 = 0.400, P = 0.940). 基因亚型分布可能与年龄无关. 在不同年龄段HCV基因亚型分布见表2.
| 年龄(岁) | n | 检出(率) | 1b+/2a- | 2a+/1b- | 1b+2a | 1a+/2b |
| <20 | 8 | 8(100.00) | 4(50.00) | 3(37.50) | 1(12.50) | 0(0) |
| 20-29 | 49 | 36(73.47) | 10(27.78) | 18(50.00) | 7(19.44) | 1(2.78) |
| 30-39 | 122 | 91(74.59) | 35(38.46) | 29(31.87) | 24(26.37) | 3(3.30) |
| 40-49 | 365 | 243(66.58) | 109(44.86) | 83(34.16) | 44(18.10) | 7(2.88) |
| 50-59 | 471 | 343(72.82) | 140(40.82) | 118(34.40) | 78(22.74) | 7(2.04) |
| 60-69 | 226 | 155(68.58) | 68(43.87) | 51(32.90) | 32(20.65) | 4(2.58) |
| ≥70 | 42 | 30(71.43) | 12(40.00) | 11(36.66) | 5(16.67) | 2(6.67) |
| 合计 | 1 283 | 906(70.62) | 378(41.72) | 313(34.55) | 191(21.08) | 24(2.65) |
在1 313例患者中548例有HCV RNA定量记录, 其中407例检出HCV基因亚型, 检出率74.27%, 与总检出率70.60%未有明显差异(χ2 = 2.563, P = 0.109). 在不同HCV RNA水平上HCV基因亚型总体分布见表3, 差异显著(χ2 = 34.001, P = 0.003), 主要体现在(1.000-9.999)×104组中1b+/2a-比率较其他各组显著降低(24.24% vs 48.28%, χ2 = 5.382, P = 0.020; 24.24% vs 40.00%, χ2 = 4.797, P = 0.029; 24.24% vs 38.93%, χ2 =4.219, P = 0.040; 24.24% vs 57.14%, χ2 = 7.901, P = 0.005), 而2a+/1b-比率较其他各组则显著增高(54.55% vs 26.67%, χ2 = 6.457, P = 0.011; 54.55% vs 31.54%, χ2 = 9.715, P = 0.002; 54.55% vs 35.88%, χ2 = 6.273, P = 0.012; 54.55% vs 14.29%, χ2 = 10.440, P = 0.001). 又通过秩检验1b+/2a-、2a+/1b-、1b+2a和1a+/2b 4组的HCV RNA水平秩均值分别为214.26、192.52、213.10和131.27, 总体上未见统计学差异(χ2 = 7.342, P = 0.062).
| HCV RNA(copies/mL) | n | 检出(率) | 1b+/2a- | 2a+/1b- | 1b+2a | 1a+/2b |
| ≤1.000×103 | 65 | 29(44.62) | 14(48.28) | 10(34.48) | 2(6.90) | 3(10.34) |
| (1.001-9.999)×103 | 47 | 30(63.83) | 10(33.33) | 8(26.67) | 10(33.33) | 2(6.67) |
| (1.000-9.999)×104 | 92 | 66(71.74) | 16(24.24)a | 36(54.55)c | 12(18.18) | 2(3.03) |
| (1.000-9.999)×105 | 170 | 130(76.47) | 52(40.00) | 41(31.54) | 35(26.92) | 2(1.54) |
| (1.000-9.999)×106 | 150 | 131(87.33) | 51(38.93) | 47(35.88) | 32(24.43) | 1(0.76) |
| ≥1.000×107 | 24 | 21(87.50) | 12(57.14) | 3(14.29) | 5(23.81) | 1(4.76) |
| 合计 | 548 | 407(74.27) | 155(38.08) | 145(35.63) | 96(23.59) | 11(2.70) |
在1 313例患者中535例有ALT水平记录, 其中401例检出HCV基因亚型, 检出率74.95%, 与总检出率70.60%未有明显差异(χ2 = 3.560, P = 0.059). 在不同ALT水平上HCV基因亚型总体分布见表4, 差异显著(χ2 = 20.612, P = 0.014), 主要体现在ALT水平>200组中1b+/2a-比率较其他各组均显著降低(≤40组: 13.04% vs 35.39%, χ2 = 4.613, P = 0.032; 41-80组: 13.04% vs 45.24%, χ2 = 8.381, P = 0.004; 81-200组: 13.04% vs 41.89%, χ2 = 6.414, P = 0.011); 而2a+/1b-比率较41-80组和81-200组均显著增高(60.87% vs 27.78%, χ2 = 9.650, P = 0.002; 60.87% vs 29.73%, χ2 = 7.290, P = 0.007). 在ALT水平≤40组2a+/1b-比率较81-200组有意义的增高(41.02% vs 29.73%, χ2 = 5.641, P = 0.018). 又通过秩检验1b+/2a-、2a+/1b-、1b+2a和1a+/2b 4组的ALT水平秩均值分别为203.05、192.39、220.20和103.17, 在总体上也有统计学差异(χ2 = 9.835, P = 0.020), 但组间(1b+/2a-、2a+/1b-和1b+2a)两两比较并无差异, 只是分别与1a+/2b组之间比较有差异(u = -2.565, P = 0.010; u = -2.241, P = 0.025; u = -2.786, P = 0.005). 说明主要HCV基因型分布在ALT水平秩均值上无差异, 但在不同ALT水平上有差异.
我们对本地区1 313例慢性HCV感染患者外周血中HCV基因型1a、1b、2a和2b检测结果的回顾性分析发现, 在检出的基因型中以1b(41.86%)、2a(34.30%)及二者的混合型(21.25%)为主, 1a和2b少见, 说明该地区流行的HCV基因型是1b和2a, 高达97%, 与我国HCV基因型的分布相符[3,15], 反映了该地区HCV基因型的流行特征和趋势. 根据报道1b和2a的感染比率已逐年下降[16,17], 2a在巴基斯坦等国家已根除[15]. 一方面原因是1b和2a的传播主要与输血、血液制品的应用、手术和拔牙或牙处理等传统传播途径有关, 随着医疗政策的完善和医疗条件的改善, 二者的感染比率已逐年下降; 另外一个重要原因是2a对抗病毒治疗有持续的高反应性[18,19], 尤其是目前的联合治疗又大大提高了1b对抗病毒治疗的反应性, 由原来的40%升高到70%以上[20-22]. 所以本地区HCV感染有望得到较好的控制.
我们也分析了基因型分布与性别、年龄、HCV RNA水平和ALT水平等的关系, 发现了一些有意义的差异, 如2a在女性组、HCV RNA(1.000-9.999)×104组、ALT水平>200组和≤40组比率均有意义的增高, 而1b在HCV RNA(1.000-9.999)×104组和ALT水平>200组均有意义的降低, 但未发现与年龄有关. 在Yan等[16]对中国东南地区1 208例患者的基因型分布情况和变化的报道中, 2a在女性中比例较高, Roman等[23]也有类似的报道. Yan等[16]认为主要基因型分布与HCV RNA平均水平无关, 但我们与Ali和Rong等[24,25]的研究均发现主要基因型分布在不同HCV RNA水平上是有差异的, 这可能与资料的选择和统计学方法有关, 本研究资料的秩和检验的秩均值也未发现主要基因型分布与HCV RNA水平有关. 同样的原因也可能说明主要基因型分布在不同ALT水平上有差异, 但在秩和检验的秩均值中未发现差异的现象[26]. 据报道基因型分布在年龄上的差异主要是由于当地优势传播途径的变化, 使得年轻人中以3型和6型为主, 而30或40岁以上感染的HCV仍以亚型1b和2a为主[16]. 在本研究中, 我们只检测了1型和2型, 但检出率达70%以上, 且以1b和2a为主, 说明本地区未发生优势传播途径的变化, 所以未发现基因型分布与年龄有关. 但70.60%的检出率略显低, 分析其原因: (1)未严格控制HCV RNA的含量, 有较低或未检出者影响了检出率; (2)可能存在其他基因型或本地区特征性基因型, 有必要开展进一步研究; (3)方法学需要改进[27].
总之, 通过本研究了解了本地区HCV主要流行株是1b和2a. 2a在女性组、HCV RNA (1.000-9.999)×104组、ALT水平>200组和≤40组比率均有意义的增高, 而1b在HCV RNA(1.000-9.999)×104组和ALT水平>200组均有意义的降低, 未发现与年龄有关. 随着医疗条件的改善和联合治疗的开展, 本地区HCV感染有望得到较好的控制, 但也面临着巨大挑战[28-30].
丙型肝炎病毒(HCV)感染是造成慢性肝炎、肝硬化及肝癌的重要原因之一. 目前HCV感染仍然是全球性健康问题. HCV在长期进化过程中已形成1-6个基因型和100多个基因亚型, HCV 1型和2型呈世界流行, 但其也在不断演变. 通过采用综合和个体化治疗方案, 已使持续病毒反应(SVR)率大大提高, HCV 2/3型达95%, 1型达79%, 在巴基斯坦HCV 2型已消失. HCV基因型的检测和动态分析备受关注.
刘正稳, 教授, 西安交通大学医学院第一附属医院
为了更好地指导HCV感染的预防和治疗, 除了对感染者进行基因型的检测外, 更要全面检测和动态分析不同人群中HCV基因型的变化趋势, 同时要积极开展个体遗传相关基因的检测和研究, 制定有效的预防和个体化治疗方案.
赵璐等较全面地综述了HCV基因型在我国和世界上的流行情况; I-Shyan等较系统地分析了HCV感染治疗的影响因素.
本研究在检测本地区流行的HCV基因型的同时, 更用不同的统计学方法分析了HCV基因型与不同HCV RNA和ALT水平的相关性.
准种: 感染者体内形成的以一个优势株为主的相关突变株病毒群.
本文思路清晰, 设计合理, 能与临床密切结合, 有一定实用意义.
编辑: 李军亮 电编: 闫晋利
| 1. | Negro F, Alberti A. The global health burden of hepatitis C virus infection. Liver Int. 2011;31 Suppl 2:1-3. [PubMed] [DOI] |
| 2. | Alghamdi AS, Sanai FM, Ismail M, Alghamdi H, Alswat K, Alqutub A, Altraif I, Shah H, Alfaleh FZ. SASLT practice guidelines for the management of hepatitis C virus infection: summary of recommendations. Saudi J Gastroenterol. 2012;18:293-298. [PubMed] [DOI] |
| 4. | Goldfield M. Some epidemiologic studies of transfusion-associated hepatitis. Infusionstherapie. 1974;1:645-649. [PubMed] |
| 5. | Choo QL, Kuo G, Weiner AJ, Overby LR, Bradley DW, Houghton M. Isolation of a cDNA clone derived from a blood-borne non-A, non-B viral hepatitis genome. Science. 1989;244:359-362. [PubMed] |
| 7. | Simmonds P, Holmes EC, Cha TA, Chan SW, McOmish F, Irvine B, Beall E, Yap PL, Kolberg J, Urdea MS. Classification of hepatitis C virus into six major genotypes and a series of subtypes by phylogenetic analysis of the NS-5 region. J Gen Virol. 1993;74:2391-2399. [PubMed] [DOI] |
| 8. | Simmonds P, Bukh J, Combet C, Deléage G, Enomoto N, Feinstone S, Halfon P, Inchauspé G, Kuiken C, Maertens G. Consensus proposals for a unified system of nomenclature of hepatitis C virus genotypes. Hepatology. 2005;42:962-973. [PubMed] [DOI] |
| 9. | Attaullah S, Khan S, Ali I. Hepatitis C virus genotypes in Pakistan: a systemic review. Virol J. 2011;8:433. [PubMed] [DOI] |
| 10. | Alexopoulou A, Papatheodoridis GV. Current progress in the treatment of chronic hepatitis C. World J Gastroenterol. 2012;18:6060-6069. [PubMed] [DOI] |
| 11. | Mangia A, Mottola L. What's new in HCV genotype 2 treatment. Liver Int. 2012;32 Suppl 1:135-140. [PubMed] [DOI] |
| 12. | Tian D, Li L, Liu Y, Li H, Xu X, Li J. Different HCV genotype distributions of HIV-infected individuals in Henan and Guangxi, China. PLoS One. 2012;7:e50343. [PubMed] [DOI] |
| 13. | Cho EJ, Jeong SH, Han BH, Lee SU, Yun BC, Park ET. Hepatitis C virus (HCV) genotypes and the influence of HCV subtype 1b on the progression of chronic hepatitis C in Korea: a single center experience. Clin Mol Hepatol. 2012;18:219-224. [PubMed] [DOI] |
| 15. | Sievert W, Altraif I, Razavi HA, Abdo A, Ahmed EA, Alomair A, Amarapurkar D, Chen CH, Dou X, El Khayat H. A systematic review of hepatitis C virus epidemiology in Asia, Australia and Egypt. Liver Int. 2011;31 Suppl 2:61-80. [PubMed] [DOI] |
| 16. | Yan Z, Fan K, Wang Y, Fan Y, Tan Z, Deng G. Changing pattern of clinical epidemiology on hepatitis C virus infection in southwest china. Hepat Mon. 2012;12:196-204. [PubMed] [DOI] |
| 17. | Butt S, Idrees M, Shahid M, Amin I, Younas S, Afzal S, Akbar H, Ur Rehman I. Change in hepatitis C virus clades: a cross-sectional study of chronic HCV patients in Pakistan from 2000-2010. Eur J Clin Microbiol Infect Dis. 2011;30:669-672. [PubMed] [DOI] |
| 18. | Idrees M, Riazuddin S. A study of best positive predictors for sustained virologic response to interferon alpha plus ribavirin therapy in naive chronic hepatitis C patients. BMC Gastroenterol. 2009;9:5. [PubMed] [DOI] |
| 19. | Grassi E, Aghemo A. How to optimize HCV therapy in genotype 2 patients. Liver Int. 2013;33 Suppl 1:35-40. [PubMed] [DOI] |
| 20. | Asselah T. Triple therapy with boceprevir or telaprevir for prior HCV non-responders. Best Pract Res Clin Gastroenterol. 2012;26:455-462. [PubMed] [DOI] |
| 21. | Jesudian AB, Jacobson IM. Optimal treatment with telaprevir for chronic HCV infection. Liver Int. 2013;33 Suppl 1:3-13. [PubMed] [DOI] |
| 22. | I-Shyan S, Chun-Yen L, Wen-Juei J, Chang-Wen H, Chien-Hao H, Ji-Yih C. Patients younger than forty years old with hepatitis C virus genotype-1 chronic infection had treatment responses similar to genotype-2 infection and not relatedto interleukin-28B polymorphism. Ann Hepatol. 2013;12:62-69. [PubMed] |
| 23. | Roman F, Hawotte K, Struck D, Ternes AM, Servais JY, Arendt V, Hoffman P, Hemmer R, Staub T, Seguin-Devaux C. Hepatitis C virus genotypes distribution and transmission risk factors in Luxembourg from 1991 to 2006. World J Gastroenterol. 2008;14:1237-1243. [PubMed] [DOI] |
| 24. | Ali A, Nisar M, Ahmad H, Saif N, Idrees M, Bajwa MA. Determination of HCV genotypes and viral loads in chronic HCV infected patients of Hazara Pakistan. Virol J. 2011;8:466. [PubMed] [DOI] |
| 25. | Rong X, Lu L, Wang J, Xiong H, Huang J, Chen J, Huang K, Xu R, Wang M, Zhang X. Correlation of Viral Loads with HCV Genotypes: Higher Levels of Virus Were Revealed among Blood Donors Infected with 6a Strains. PLoS One. 2012;7:e52467. [PubMed] [DOI] |
| 26. | Zhang GY, Pu SB, Yu XY, Yang J, Shi JF, Xu AL, Guo ZH, Qiao MG, Zhang J, Jing YZ. [Study on hepatitis C virus genotypes in Yantai district, Shandong province]. Zhonghua Shiyan He Linchuang Bingduxue Zazhi. 2012;26:259-262. [PubMed] |
| 27. | Cai Q, Zhao Z, Liu Y, Shao X, Gao Z. Comparison of three different HCV genotyping methods: Core, NS5B sequence analysis and line probe assay. Int J Mol Med. 2013;31:347-352. [PubMed] [DOI] |
| 28. | Nucara S, Caroleo B, Guadagnino V, Perrotti N, Trapasso F. Natural history and clinical response: "it's the virus, stupid, or is it the host?". BMC Infect Dis. 2012;12 Suppl 2:S6. [PubMed] [DOI] |