修回日期: 2013-09-12
接受日期: 2013-09-17
在线出版日期: 2013-10-18
目的: 研究10q21基因rs10761659多态性与广西壮族人群炎症性肠病(inflammatory bowel disease, IBD)易感性的相关性.
方法: 采用病例对照研究, 分别收集2009-08/2012-10广西区域无血缘关系的壮族73例和汉族67例IBD患者及广西区域无血缘关系的壮族70例和汉族78例健康对照者的肠黏膜组织. 使用酚-氯仿法提取基因组DNA, PCR扩增目的片段, RFLP筛查rs10761659突变点, 基因测序法再进一步证实突变位点, 最后统计该多态性位点的基因型频率及等位基因频率, 分析其分布与广西壮族人群IBD的相关性.
结果: 广西壮族及汉族IBD患者与正常对照者均发现了10q21基因rs10761659突变型纯合子、突变型杂合子及野生型纯合子, 但其基因型频率及等位基因频率分布在克罗恩病(Chron's disease)患者、溃疡性结肠炎(ulcerative colitis)患者及正常对照者中差异均无统计学意义(P>0.05); 壮族与汉族病例组比较, 其基因型频率分布也无统计学差异(P>0.05).
结论: 10q21基因rs10761659多态性与中国广西壮族人群炎症性肠病无明显相关性.
核心提示: 本研究通过PCR-RFLP法及基因测序法发现与西方国家炎症性肠病(inflammatory bowel disease, IBD)易感性相关的10q21基因rs10761659多态性与广西壮族IBD患者无显著相关性.
引文著录: 龙伟艳, 詹灵凌, 陈兰, 吕小平. 10q21基因rs10761659多态性与广西壮族人群炎症性肠病的相关性. 世界华人消化杂志 2013; 21(29): 3146-3152
Revised: September 12, 2013
Accepted: September 17, 2013
Published online: October 18, 2013
AIM: To analyze the association of 10q21 gene rs10761659 polymorphism with susceptibility to inflammatory bowel disease (IBD) in a Zhuang population from Guangxi, China.
METHODS: A case-control study was conducted from August 2009 to October 2012, which included intestinal tissue samples of 73 Zhuang and 67 Han unrelated IBD patients and 70 Zhuang and 78 Han unrelated healthy volunteers in Guangxi. Genomic DNA was extracted from intestinal tissue using the phenol-chloroform method. The rs10761659 locus of the 10q21 gene was amplified by polymerase chain reaction (PCR), genotyped by restriction fragment length polymorphism (RFLP) and verified by gene sequencing.
RESULTS: The homozygous mutant, heterozygous mutant and wild-type genotypes of the rs10761659 locus were found in both IBD patients and healthy controls, but the distribution of genotypes and allele frequencies was not significantly different among Crohn's disease, ulcerative colitis and control groups (P > 0.05). No significant difference was detected in the 10q21 gene rs10761659 polymorphism between Zhuang and Han patients with Crohn's disease or ulcerative colitis (P > 0.05).
CONCLUSION: The rs10761659 polymorphism of the 10q21 gene is not associated with susceptibility to IBD in the Zhuang population in Guangxi.
- Citation: Long WY, Zhan LL, Chen L, Lv XP. Association of 10q21 gene rs10761659 polymorphism with susceptibility to inflammatory bowel disease in a Guangxi Zhuang population. Shijie Huaren Xiaohua Zazhi 2013; 21(29): 3146-3152
- URL: https://www.wjgnet.com/1009-3079/full/v21/i29/3146.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i29.3146
炎症性肠病(inflammatory bowel disease, IBD)是一种胃肠道的慢性反复发作性炎性疾病, 包括克罗恩病(Chron's disease, CD)及溃疡性结肠炎(ulcerative colitis, UC)两种临床病理类型. 到目前为止, IBD的发病机制仍未明确, 普遍认为是一种由遗传、环境、感染及免疫共同作用的多因素疾病[1,2]. 大量研究证实遗传易感性在IBD的发病及发展过程起重要作用[3-5]. 自2001年由Hugot、Ogura及Hampe等[6-8]报道了人类的第一个CD易感基因之后, 人们通过全基因组关联性研究及对其的Meta分析, 已证实了99个IBD易感性基因位点, 其中UC及CD共享的有28个[9], 而与CD相关的可达71个[10], 近年来许多研究发现了一些新的IBD易感基因位点, 关注较多的如IL-23R-rs11805303, ATG16L1-rs10210302, NOD2-P268S, PTPN2-rs2542151, 10q21-rs10761659等[11-15], 但多数位于基因编码区上, 而rs10761659是位于10q21基因非编码基因间区(和内含子一起统称基因沙漠区)上的IBD多态性位点, 目前越来越多的研究开始关注基因沙漠区的疾病易感基因. 国外已有多项研究证实10q21基因的rs10761659改变可以增加CD的易感性[11,12,16,17], 而对意大利人群的研究显示rs10761659与UC及CD均相关, 并证实其与成人CD的回肠发病相关[18]. 但在我国广东地区却报道了该位点与我国汉族人群UC、CD的发病无显著相关[19], 提示在不同的种族遗传背景中, rs10761659对IBD发病机制的作用存在显著的差异性. 广西是我国壮族人口最大的聚居地, 他的疾病遗传性和易感基因都非常独特. 在广西壮族人群中, 10q21基因变异与IBD的相关性如何目前尚未见报道. 因此, 本研究的目的是探索10q21基因rs10761659多态性与广西壮族人群中IBD的相关性.
收集2009-08/2012-10在广西医科大学第一附属医院消化内科就诊的广西区域无血缘关系的IBD患者140例, 其中UC 98例, CD 42例, 壮族73例, 汉族67例, 男75例, 女65例, 平均年龄40.79岁±13.56岁, IBD的诊断依据2012年中华医学会消化病学分会制定的标准[20], 并排除患有其他自身免疫性疾病者. 并收集广西区域无血缘关系的健康对照者148例, 其中壮族70例, 汉族78例, 男75例, 女73例, 平均年龄41.52岁±14.15岁. IBD组和正常对照组年龄及性别比较差异无统计学意义(P>0.05), 标本采集经医院伦理委员会批准和患者本人或家属知情同意.
1.2.1 基因组DNA的提取: 内镜下钳取25-50 mg的新鲜肠黏膜组织, 立即转移至-80 ℃冰箱. 用生理盐水将解冻的肠黏膜组织洗净, 置EP管中尽量剪碎, 加入50 μL的SDS(10%) 、450 μL TES及5 μL蛋白酶K(20 g/L), 轻柔混匀后放置56 ℃水浴箱中充分消化(7-8 h), 加入等体积的Tris饱和酚, 混匀后在4 ℃、12000 r/min离心机中离心10 min, 提取上清液至另一EP管; 加入等体积的Tris饱和酚、氯仿、异戊醇混合液(25:24:1), 轻柔混匀分层后4 ℃、12000 r/min离心10 min并提取上清液至另一EP管; 再加入等体积的氯仿、异戊醇混合液(24:1), 混匀后同样条件离心10 min, 提取上清液于另一EP管; 然后加入约2.5倍体积的无水乙醇, 用枪头轻轻吹吸至白色絮状物完全析出, 再次离心10 min后弃去上清液, 在白色沉淀中加入适量750 mL/L的乙醇洗涤2次, 相同条件离心5 min后弃去上清液, 放置通风橱自然挥干, 最后加入50-120 μL TE溶解, 置-20 ℃保存备用, 用分光光度仪检测DNA的纯度及浓度.
1.2.2 PCR扩增DNA目的片段: 引物序列设计参考文献[19], 上游: 5'-GCTTAGGAAACAAAGGAATCAC-3'; 下游: 5'-AGTCAAAGAGGAGGGCGTT-3', 由生工生物技术(上海)有限公司合成引物. PCR反应体系包括DNA模板2 μL、引物(10 μmol/L)上下游各1 μL、2×pfuPCR Master Mix(北京天根)10 μL, 最后加ddH2O至20 μL. 反应条件: 预变性94 ℃, 5 min; 变性94 ℃、45 s, 退火60 ℃、45 s, 延伸72 ℃、45 s, 总共35个循环; 72 ℃继续延伸10 min. PCR产物置4 ℃冰箱保存, 取5 μL在含核酸染料的2%琼脂糖凝胶上电泳, 凝胶成像仪上鉴定目的片段.
1.2.3 RFLP筛查突变位点: 根据突变位点(G→A)选择相应限制性内切酶TaaⅠ(HpyCH4Ⅲ)(美国Fermentas公司). 酶切反应体系包括PCR产物5 μL, 10×Buffer Tango缓冲液1 μL, 限制性内切酶0.5 μL, 再加ddH2O至17 μL. 将混匀后的反应体系置于37 ℃水浴15 h, 取10 μL酶切产物加于含核酸染料的3%琼脂糖凝胶上, 在90 V电压下电泳约60 min, 凝胶成像仪上判断酶切产物片段大小.
1.2.4 基因测序进一步确定突变位点: 对RFLP中发现突变者重新用PCR扩增目的片段, 切胶纯化后送往北京三博远志测序部进行基因测序, 测序仪为美国ABI 3730XL, 测序结果在PubMed中的BLAST里进行比对, 用Chromas软件判读基因型.
统计学处理 用SPSS16.0软件包进行分析, 统计rs10761659在壮族与汉族的CD组、UC组及健康对照组中的基因型频率及等位基因频率, 用Hardy-Weinberg进行遗传平衡性检验, 用χ2检验比较各组间基因型及等位基因频率分布的差异, P<0.05为差异有统计学意义.
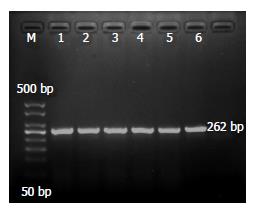
PCR扩增后, 形成了包含rs10761659位点的262 bp目的片段(图1).
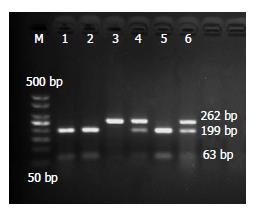
TaaⅠ(HpyCH4Ⅲ)内切酶可识别碱基序列ACNGT, rs10761659的PCR目的片段被其消化后形成了3种基因型的条带, 野生型被切开形成两条条带(199、63 bp), 突变型杂合子被切开形成3条条带(262、199、63 bp), 而突变型纯合子不能被切开, 仍保留262 bp一条条带(图2).
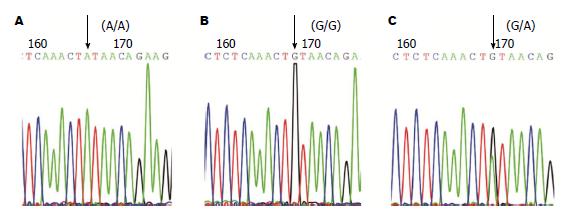
将rs10761659的PCR产物进行测序后, 结果和RFLP的一致, 出现了3种基因型:野生型(G/G)、突变型杂合子(G/A)、突变型纯合子(A/A)(图3).
汉族和壮族的病例组及正常对照组的rs10761659基因型及等位基因频率分布符合Hardy-Weinberg遗传平衡定律(P>0.05), 提示所选样本具有群体代表性. 广西壮族及汉族的CD组、UC组与正常对照组均发现有rs10761659突变型纯合子、突变型杂合子及野生型纯合子, 但其基因型及等位基因频率在CD组、UC组和正常对照组中的分布均无统计学差异(P>0.05); 壮族CD组与汉族CD组比较、壮族UC组与汉族UC组比较, rs10761659基因型频率分布差异也无统计学意义(P>0.05)(表1).
| 民族 | 基因型 | 正常对照组 | CD组 | UC组 |
| 壮族 | GG | 40(57.1) | 9(47.4) | 30(55.6) |
| GA | 26(37.1) | 8(42.1) | 21(38.9) | |
| AA | 4(5.7) | 2(10.5) | 3(5.6) | |
| 等位基因A的频率(%) | 24.2 | 31.5 | 25.0 | |
| 汉族 | GG | 50(64.1) | 12(52.2) | 23(52.3) |
| GA | 24(30.8) | 8(34.8) | 18(40.9) | |
| AA | 4(5.1) | 3(13.0) | 3(6.8) | |
| 等位基因A的频率(%) | 20.5 | 30.4 | 27.2 |
IBD是一种多基因疾病, 近年来, 全基因组关联性研究已成为一种发现复杂疾病(包括IBD)易感基因或位点的有效途径, 使人类对IBD的遗传易感性有了更深的了解. 2008年Barrett等[21]对3个关于欧洲人群CD大样本(包含3230个患者及4829个正常者)的全基因组关联性研究进行了Meta分析及重复研究, 一共定义了30余种CD易感基因, 包括10q21等11个之前所报道的突变体及21个新的CD高危基因如STAT3、JAK2、ICOSLG、CDKAL1、ITLN1等. 随后, 另一个更大规模(包含了6333例患者及15056例正常对照者)的meta分析结果显示在全基因组关联性研究中得到证实的欧洲人CD易感基因已达71个, 包括10q21-rs10761659[10]. 这些研究结果强烈支持了CD是一种多基因相关性疾病, 为将来这些易感基因的致病机制及功能的研究提供了重要线索.
2007年由Rioux等[11]首次报道了10q21-rs10761659与CD的易感性相关, 同年, 由英国病例控制协会(WTCCC)进行的全基因组关联性研究再次证实了10q21-rs10761659与CD显著相关(P = 2.7×10-7)[12]. rs10761659是一个位于染色体10q21上非编码基因间区的SNP位点, 和锌指蛋白基因ZNF365末端的着丝点相距14 kb, 和4型遗蛋白假基因的着丝点相距55 kb[11]. 锌指蛋白基因及假基因的致病机制目前尚未明确, ZNF365基因编码的蛋白又称锌指蛋白, 他作为一种对真核生物的基因转录过程起调控作用的重要转录因子[22], 而假基因可以调节信使RNA的稳定性[23], 当rs10761659发生改变(G突变成A)时, 可能会影响与其相邻的这两个基因的功能, 最终引起IBD的发病.
rs10761659是近几年发现的一个CD易感基因, 但在不同种族及人群中存在着显著差异, 自2007年的一项全基因组关联性研究报道rs10761659与CD的易感性相关之后[11], 在英国[12]、德国[16]、荷兰、比利时[17]及德系犹太人人群[24]中反复得到了证实. 此外, rs10761659除了与意大利人群CD相关外, 还被发现与意大利人群UC相关, 其中与成人CD及UC的易感性均相关, 但仅与儿童UC相关[18]. 一项最近的Meta分析也提示了rs10761659是CD和UC的共享易感位点[9]. 然而对日本人群前期的研究表明位于NKX2-3基因上游区域的rs10761659与日本CD患者弱相关[25], 但之后的另一个研究却报道其与日本人群IBD无关[26], 而我国广东地区也报道了该多态性位点与我国汉族人IBD无关[19], 提示可能是因为研究样本量偏小引起的误差, 或者是该基因多态性不是亚洲人IBD的易感基因位点. 我们此次的研究结果和我国广东汉族人的研究结果一致, 虽然我们发现了10q21-rs10761659在壮族或汉族UC、CD中的突变率均比正常对照者高, 但差异无统计学意义(P>0.05). 这和我国对其他一些IBD易感基因的研究不同的是, 该基因位点在CD患者、UC患者和正常人中的突变率均高, 各组间比较未达统计学差异, 而之前对我国人群的研究显示, NOD2-R702W, G908R, L1007fs[27,28]; TLR4-Asp299Gly, Thr399Ile; TLR2-Arg753Gln, Arg677Trp[29]和OCTN1-C1672T, OCTN2-G-207C[30]等基因位点在IBD患者及正常人中均缺如, NOD2-P268S尽管在我国CD患者中的突变率不高, 但和正常人对比仍然有统计学差异. 综合各项研究数据表明rs10761659对IBD的作用存在明显的种族、地理及人群差异性, 且对IBD的不同病理类型的作用也不同.
很多研究都证明了CD的基因易感性和他的临床特征存在关联性[31,32], 我国广东地区的研究也发现了NOD2/CARD15基因上的P268S多态性改变可能与我国汉族人群CD的发病年龄偏小、回肠病变及合并肠腔狭窄相关[19]. 对于10q21基因rs10761659多态性与CD临床特征的关系研究, 意大利学者仅发现其与成人的回肠型CD相关, 与儿童CD的临床特征并不相关[18], 而其他几个关于rs10761659的研究未见报道其与CD临床特征的相关性[11,12,16,17,19,24], 我们此次的研究并没有发现rs10761659与CD的发病机制相关, 而且壮族与汉族人之间无差别, 因此没有进一步研究他与CD临床特征的关系, 未来对致病基因型及临床表型之间的关系还有待更多的研究去探索.
炎症性肠病(inflammatory bowel disease, IBD)是一种多基因复杂疾病, 自2001年由Hugot等报道了人类的第一个CD易感基因NOD2/CARD15之后, 人们通过全基因组关联性研究已证实99个IBD易感性基因位点, 其中10q21基因rs10761659多态性已被国外多项研究证实与CD的易感性相关, 但在我国汉族人群中未被证实, 尚未见报道其与广西壮族IBD的相关性.
万军, 教授, 中国人民解放军总医院南楼老年消化科.
多数IBD易感基因位点是位于基因的编码区, 而rs10761659位于10q21基因非编码基因间区(和内含子合称沙漠区), 目前对基因沙漠区的疾病易感基因的致病机制及其功能的研究已成为一热点.
rs10761659与CD的相关性已在英国 、德国、荷兰、比利时、意大利等西方国家中反复证实, 意大利还发现rs10761659与UC相关.日本的研究提示rs10761659与日本CD患者弱相关或者无关, 而我国广东未能证实该多态性位点与我国汉族人IBD的易感性相关.
广西是我国壮族人口最大的聚居地, 他的疾病遗传性和易感基因都非常独特. 本研究发现广西壮族IBD患者存在rs10761659突变体, 但未达统计学相关性, 这和西方国家的研究结果相反, 提示IBD的基因易感性存在显著的种族、区域差异性.
10q21基因rs10761659多态性可能不是中国广西壮族人群炎症性肠病的易感基因位点.
本文目的明确, 资料详实, 结果可靠, 有助于深入认识IBD发病机制所涉及的遗传因素中易感基因的种族差异问题.
编辑: 田滢 电编:鲁亚静
| 1. | Xavier RJ, Podolsky DK. Unravelling the pathogenesis of inflammatory bowel disease. Nature. 2007;448:427-434. [PubMed] |
| 2. | Podolsky DK. Inflammatory bowel disease. N Engl J Med. 2002;347:417-429. [PubMed] |
| 5. | Haritunians T, Jones MR, McGovern DP, Shih DQ, Barrett RJ, Derkowski C, Dubinsky MC, Dutridge D, Fleshner PR, Ippoliti A. Variants in ZNF365 isoform D are associated with Crohn's disease. Gut. 2011;60:1060-1067. [PubMed] [DOI] |
| 6. | Hugot JP, Chamaillard M, Zouali H, Lesage S, Cézard JP, Belaiche J, Almer S, Tysk C, O'Morain CA, Gassull M. Association of NOD2 leucine-rich repeat variants with susceptibility to Crohn's disease. Nature. 2001;411:599-603. [PubMed] |
| 7. | Ogura Y, Bonen DK, Inohara N, Nicolae DL, Chen FF, Ramos R, Britton H, Moran T, Karaliuskas R, Duerr RH. A frameshift mutation in NOD2 associated with susceptibility to Crohn's disease. Nature. 2001;411:603-606. [PubMed] |
| 8. | Hampe J, Cuthbert A, Croucher PJ, Mirza MM, Mascheretti S, Fisher S, Frenzel H, King K, Hasselmeyer A, MacPherson AJ. Association between insertion mutation in NOD2 gene and Crohn's disease in German and British populations. Lancet. 2001;357:1925-1928. [PubMed] |
| 9. | Anderson CA, Boucher G, Lees CW, Franke A, D'Amato M, Taylor KD, Lee JC, Goyette P, Imielinski M, Latiano A. Meta-analysis identifies 29 additional ulcerative colitis risk loci, increasing the number of confirmed associations to 47. Nat Genet. 2011;43:246-252. [PubMed] [DOI] |
| 10. | Franke A, McGovern DP, Barrett JC, Wang K, Radford-Smith GL, Ahmad T, Lees CW, Balschun T, Lee J, Roberts R. Genome-wide meta-analysis increases to 71 the number of confirmed Crohn's disease susceptibility loci. Nat Genet. 2010;42:1118-1125. [PubMed] [DOI] |
| 11. | Rioux JD, Xavier RJ, Taylor KD, Silverberg MS, Goyette P, Huett A, Green T, Kuballa P, Barmada MM, Datta LW. Genome-wide association study identifies new susceptibility loci for Crohn disease and implicates autophagy in disease pathogenesis. Nat Genet. 2007;39:596-604. [PubMed] |
| 12. | Wellcome Trust Case Control Consortium. Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls. Nature. 2007;447:661-678. [PubMed] |
| 13. | Chua KH, Hilmi I, Ng CC, Eng TL, Palaniappan S, Lee WS, Goh KL. Identification of NOD2/CARD15 mutations in Malaysian patients with Crohn's disease. J Dig Dis. 2009;10:124-130. [PubMed] [DOI] |
| 14. | Gasche C, Nemeth M, Grundtner P, Willheim-Polli C, Ferenci P, Schwarzenbacher R. Evolution of Crohn's disease-associated Nod2 mutations. Immunogenetics. 2008;60:115-120. [PubMed] [DOI] |
| 15. | Zhao XD, Shen FC, Zhang HJ, Shen XY, Wang YM, Yang XZ, Tu HM, Tai YH, Shi RH. Association of interleukin-23 receptor gene polymorphisms with susceptibility and phenotypes of inflammatory bowel diseases in Jiangsu Han population. Zhonghua Neike Zazhi. 2011;50:935-941. [PubMed] |
| 16. | Franke A, Balschun T, Karlsen TH, Hedderich J, May S, Lu T, Schuldt D, Nikolaus S, Rosenstiel P, Krawczak M. Replication of signals from recent studies of Crohn's disease identifies previously unknown disease loci for ulcerative colitis. Nat Genet. 2008;40:713-715. [PubMed] [DOI] |
| 17. | Weersma RK, Stokkers PC, Cleynen I, Wolfkamp SC, Henckaerts L, Schreiber S, Dijkstra G, Franke A, Nolte IM, Rutgeerts P. Confirmation of multiple Crohn's disease susceptibility loci in a large Dutch-Belgian cohort. Am J Gastroenterol. 2009;104:630-638. [PubMed] [DOI] |
| 18. | Latiano A, Palmieri O, Latiano T, Corritore G, Bossa F, Martino G, Biscaglia G, Scimeca D, Valvano MR, Pastore M. Investigation of multiple susceptibility loci for inflammatory bowel disease in an Italian cohort of patients. PLoS One. 2011;6:e22688. [PubMed] [DOI] |
| 19. | Lv C, Yang X, Zhang Y, Zhao X, Chen Z, Long J, Zhang Y, Zhong C, Zhi J, Yao G. Confirmation of three inflammatory bowel disease susceptibility loci in a Chinese cohort. Int J Colorectal Dis. 2012;27:1465-1472. [PubMed] [DOI] |
| 21. | Barrett JC, Hansoul S, Nicolae DL, Cho JH, Duerr RH, Rioux JD, Brant SR, Silverberg MS, Taylor KD, Barmada MM. Genome-wide association defines more than 30 distinct susceptibility loci for Crohn's disease. Nat Genet. 2008;40:955-962. [PubMed] [DOI] |
| 22. | Wolfe SA, Nekludova L, Pabo CO. DNA recognition by Cys2His2 zinc finger proteins. Annu Rev Biophys Biomol Struct. 2000;29:183-212. [PubMed] |
| 23. | Hirotsune S, Yoshida N, Chen A, Garrett L, Sugiyama F, Takahashi S, Yagami K, Wynshaw-Boris A, Yoshiki A. An expressed pseudogene regulates the messenger-RNA stability of its homologous coding gene. Nature. 2003;423:91-96. [PubMed] |
| 24. | Kenny EE, Pe'er I, Karban A, Ozelius L, Mitchell AA, Ng SM, Erazo M, Ostrer H, Abraham C, Abreu MT. A genome-wide scan of Ashkenazi Jewish Crohn's disease suggests novel susceptibility loci. PLoS Genet. 2012;8:e1002559. [PubMed] [DOI] |
| 25. | Yamazaki K, Takahashi A, Takazoe M, Kubo M, Onouchi Y, Fujino A, Kamatani N, Nakamura Y, Hata A. Positive association of genetic variants in the upstream region of NKX2-3 with Crohn's disease in Japanese patients. Gut. 2009;58:228-232. [PubMed] [DOI] |
| 26. | Nakagome S, Takeyama Y, Mano S, Sakisaka S, Matsui T, Kawamura S, Oota H. Population-specific susceptibility to Crohn's disease and ulcerative colitis; dominant and recessive relative risks in the Japanese population. Ann Hum Genet. 2010;74:126-136. [PubMed] [DOI] |
| 27. | 林 美娇, 吕 小平, 陈 兰, 詹 灵凌. NOD2/CARD15基因R702W、G908R及L1007fs多态性与广西壮族人群炎症性肠病的相关性. 世界华人消化杂志. 2012;20:1210-1215. [DOI] |
| 28. | Gao M, Cao Q, Luo LH, Wu ML, Hu WL, Si JM. [NOD2/CARD15 gene polymorphisms and susceptibility to Crohn's disease in Chinese Han population]. Zhonghua Neike Zazhi. 2005;44:210-212. [PubMed] |
| 29. | Chen L, Lin MJ, Zhan LL, Lv XP. Analysis of TLR4 and TLR2 polymorphisms in inflammatory bowel disease in a Guangxi Zhuang population. World J Gastroenterol. 2012;18:6856-6860. [PubMed] [DOI] |
| 31. | Jung C, Colombel JF, Lemann M, Beaugerie L, Allez M, Cosnes J, Vernier-Massouille G, Gornet JM, Gendre JP, Cezard JP. Genotype/phenotype analyses for 53 Crohn's disease associated genetic polymorphisms. PLoS One. 2012;7:e52223. [PubMed] [DOI] |
| 32. | Cleynen I, González JR, Figueroa C, Franke A, McGovern D, Bortlík M, Crusius BJ, Vecchi M, Artieda M, Szczypiorska M. Genetic factors conferring an increased susceptibility to develop Crohn's disease also influence disease phenotype: results from the IBDchip European Project. Gut. 2012;62:1556-1565. [PubMed] [DOI] |