修回日期: 2012-06-29
接受日期: 2012-08-01
在线出版日期: 2012-08-18
目的: 克隆和分析镇江地区来源于不同疾病的(胃癌、溃疡和胃炎)幽门螺杆菌(Helicobacter pylori, H. pylori)的表皮接触诱导基因iceA1.
方法: 从胃十二指肠疾病患者胃黏膜组织中分离培养获得H. pylori, PCR扩增检测iceA1基因, 并克隆至pMD18-T载体上, 进行测序和序列分析.
结果: 克隆和测序了镇江地区来源于不同疾病的(胃癌、溃疡和胃炎)共12株H. pylori的iceA1基因片段, 并与标准菌株60190比对, 结果显示镇江地区的H. pylori的iceA1基因中存在着3处框内缺失突变热点(780del6、809del5、914del7), 这些缺失突变在溃疡和胃炎中均存在, 但是胃癌株只存在809del5. 对缺失片段周围的序列进行分析, 这些缺失序列的两端基本都与同向重复序列相连, 这可能与复制过程中滑动错配的小片段缺失模型有关.
结论: iceA1序列的变异性有可能作为分析H. pylori群体遗传学的有用工具.
引文著录: 邵长江, 张尤历, 王文兵, 陈慧娟, 孔梅, 陈鑫, 宋永站. 幽门螺杆菌上皮接触诱导基因iceA1的克隆及序列特征. 世界华人消化杂志 2012; 20(23): 2218-2223
Revised: June 29, 2012
Accepted: August 1, 2012
Published online: August 18, 2012
AIM: To clone and sequence the iceA1 gene of Helicobacter pylori (H. pylori) strains isolated from patients with various gastric disorders in Zhenjiang area.
METHODS: H. pylori strains were isolated from the gastric mucosa of patients with chronic gastritis, peptic ulcer or gastric cancer, and cultured on solid agar medium. The iceA1 gene was amplified from H. pylori DNA by PCR, cloned into T vector, sequenced and analyzed using bioinformatics methods.
RESULTS: The iceA1 gene was cloned from 12 H. pylori strains derived from patients with chronic gastritis, peptic ulcer or gastric cancer and sequenced. Sequence analysis led to identification of three deletion hot spots (780del6, 809del5, 914del7) within the iceA1 open reading frame. All these deletion mutations were identified in strains from patients with peptic ulcer or chronic gastritis, but only 809del5 was detected in strains from cancer patients. Local DNA sequence analysis revealed that the endpoints of all there deletions coincided with direct repeats. We also found deletion hot spots that were associated with direct repeats in iceA1 and that favored a small-deletion model of slipped mispairing events during replication.
CONCLUSION: Analysis of iceA1 sequence variations may be a useful tool for analysis of the population genetics of H. pylori.
- Citation: Shao CJ, Zhang YL, Wang WB, Chen HJ, Kong M, Chen X, Song YZ. Cloning and sequence analysis of ithe iceA1 gene of Helicobacter pylori strains isolated from patients with various gastric disorders. Shijie Huaren Xiaohua Zazhi 2012; 20(23): 2218-2223
- URL: https://www.wjgnet.com/1009-3079/full/v20/i23/2218.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v20.i23.2218
幽门螺杆菌(Helicobacter pylori, H. pylori)是一种定植于人胃部的革兰氏阴性杆菌, 是慢性胃炎、消化性溃疡的主要病因, 并与胃癌和胃黏膜相关淋巴组织恶性淋巴瘤等疾病的发生密切相关[1,2], 世界卫生组织已将其列为Ⅰ级致癌因子[3]. iceA基因是H. pylori与胃上皮细胞接触后诱导表达的一种基因, 是1996年美国学者Donahue等[4]在比较1例溃疡和1例胃炎患者的H. pylori菌株的mRNA转录时发现的一新的H. pylori毒力相关基因. 该基因位于cysE(丝氨酸乙酰基转移酶的同源基因)下游, hpyIM上游. iceA基因有两个等位基因型, 分别称为iceA1和iceA2. iceA1基因与胃病之间的联系目前还有争论. 有的研究显示iceA1基因型与胃病之间无明显联系, 而也有一些研究表明, iceA1的基因型和H. pylori感染的临床预后之间有联系[5-7]. 这可能是由于在全球不同地理区域内H. pylori基因型分布不均一引起的, 也反映了H. pylori菌株与其致病的临床表现的地理上差异. 陈慧娟等[8]研究来自镇江地区分离的H. pylori的iceA1基因的阳性率与亚洲其他国家报道的报道相近, iceA1在胃癌中的检出率高于慢性胃炎与消化性溃疡的患者, 但与胃癌、消化性溃疡和慢性胃炎等疾病类型的发生无显著相关性. 陈惠娟等分析了4例来源于镇江地区iceA1基因的结构特征, 发现镇江地区H. pylori菌株的iceA1基因有较独特的结构特征, 镇江地区H. pylori的iceA1基因读码框有3个翻译起始点, 镇江株以第3个起始点翻译为主, 形成较小的蛋白结构. 因此, 我们进一步研究镇江地区iceA1序列特征, 并与标准菌株60190进行序列比对, 找出其核苷酸突变的位点及热点, 以进一步了解iceA1的地域特征.
H. pylori基础培养基、微需氧环境发生袋、选择性抗生素及厌氧培养罐购自德国Merck公司. 改良布氏肉汤培养基由上海腹泻疾病控制中心提供, 无菌羊全血为金坛欣迪公司产品; Escherichia coli DH5α为江苏大学生命科学研究院实验室保存. pMD18-T载体、Taq DNA聚合酶、质粒抽提试剂盒购自TaKaRa大连公司. 酵母提取物、蛋白胨为OXOID公司产品. 其他常规试剂为市售分析纯.
1.2.1 H.pylori菌株的来源: 分离的H. pylori来自2008-05/2008-12因消化系症状来江苏大学附属医院消化科胃镜室检查患者的胃窦黏膜(距幽门5 cm以内)活检组织, 每例取新鲜胃黏膜3块, 其中1块置于输送液中用于分离培养H. pylori, 1块用于快速尿素酶检测, 1块用于病理组织学检查. 疾病的诊断依据胃镜和病理学检查.
1.2.2 H. pylori的培养: 自输送液中取出胃黏膜标本, 将活检新鲜组织用接种环均匀涂于固体琼脂培养基, 在微需氧环境, 37 ℃下培养, 3-5 d后收集细菌. 经菌落形态、涂片染色显微镜下观察以及生化反应(尿素酶、化酶和触酶)证实. 尿素酶试验阳性-细菌使尿素酶试剂变红; 触酶试验阳性-细菌使3%H2O2产生连续的气泡; 氧化酶试验阳性-细菌使浸有氧化酶试剂的滤纸变黑.
1.2.3 H. pylori基因组DNA提取: 刮取培养的菌落, 悬于适量TE缓冲液, 以GES液(EDTA, sarkosyl)-氯仿法提取DNA, 加适量TE液, 4 ℃过夜溶解. 于-20 ℃保存备用.
1.2.4 引物设计与合成: 根据陈惠娟研究的镇江地区H. pylori菌株的iceA1基因读码框有3个翻译起始点, 镇江株以第3个起始点翻译为主, 形成较小的蛋白结构, 因此我们设计目的片段引物的思路是要包含从第1个翻译起始点到第3个翻译起始点这一区域. 根据NCBI数据库中的H. pylori iceA1基因(GenBank登录号: U43917)分别设计iceA1的上下游引物(表1). 引物由上海生工生物工程技术服务有限公司合成.
| 基因 | 引物名称 | 核苷酸序列(5'-3') | PCR产物大小(bp) |
| iceA1 | iceA1F | AATGCGGTGGTGCT- TTCAGAT | 472 |
| iceA1R | TTTGAGTGTTTTCAG- AGTTGCCGTG |
1.2.5 PCR扩增iceA1片段: 以获得的H. pylori基因组DNA为模板, P1和P2为引物, 用Taq DNA聚合酶进行PCR扩增(在20 μL反应体系中, 分别加入dNTP 2.0 μL, 10×PCR buffer 2.0 μL, DNA模板1 μL, P1和P2引物各0.5 μL, Taq DNA聚合酶0.3 μL, ddH2O 13.7 μL, 按照PCR条件扩增, 扩增条件为: 94 ℃预变性5 min后, 按94 ℃ 1 min, 56.2 ℃ 30 s, 72 ℃ 40 s, 38个循环, 最后72 ℃延伸10 min. PCR产物经1%的琼脂糖凝胶电泳检测.
1.2.6 TA克隆: 胶回收试剂盒回收目的片段, 回收 PCR产物与pMD18-T载体在16 ℃连接过夜. 连接产物转化DH5α感受态细胞, 转化后的细菌涂布于含100 μg/mL氨苄青霉素的LB平板上, 37 ℃培养12 h. 挑取菌落转种于含氨苄青霉素100 μg/mL的LB液体培养基, 37 ℃振荡培养12 h后收集细菌, 质粒抽提试剂盒抽提质粒.
1.2.7 重组质粒的酶切鉴定和序列测定: 获得重组质粒pMD18-T-iceA1后用Hind Ⅲ和EcoRⅠ双酶切, 酶切产物经1%的凝胶电泳鉴定. 为了进一步从序列的准确性上得到验证, 将阳性克隆菌株送到上海生物工程有限公司测序.
1.2.8 iceA1序列比对: 应用Expasy在线翻译蛋白; 应用生物软件MEGA 4.0, 将本实验室的12个H. pylori菌株与国际标准菌株60190 iceA1基因(GenBank登录号: U43917)的核苷酸序列进行序列进行比对.
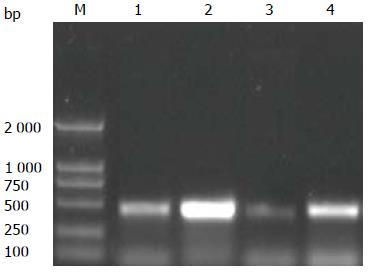
以不同胃十二指肠疾病来源的H. pylori菌株基因组DNA为模板, 扩增出iceA1基因大小为472 bp左右(图1)对于iceA1片段的扩增, PCR产物的大小会有细小的差别, 测序结果表明这些区域内存在缺失突变.
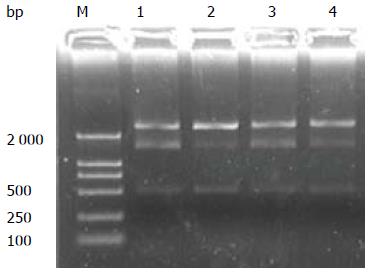
将不同疾病来源菌株的iceA1克隆于pMD18-T载体后用Hind Ⅲ和EcoRⅠ双酶切鉴定, 所获片段大小与预期相符(图2).
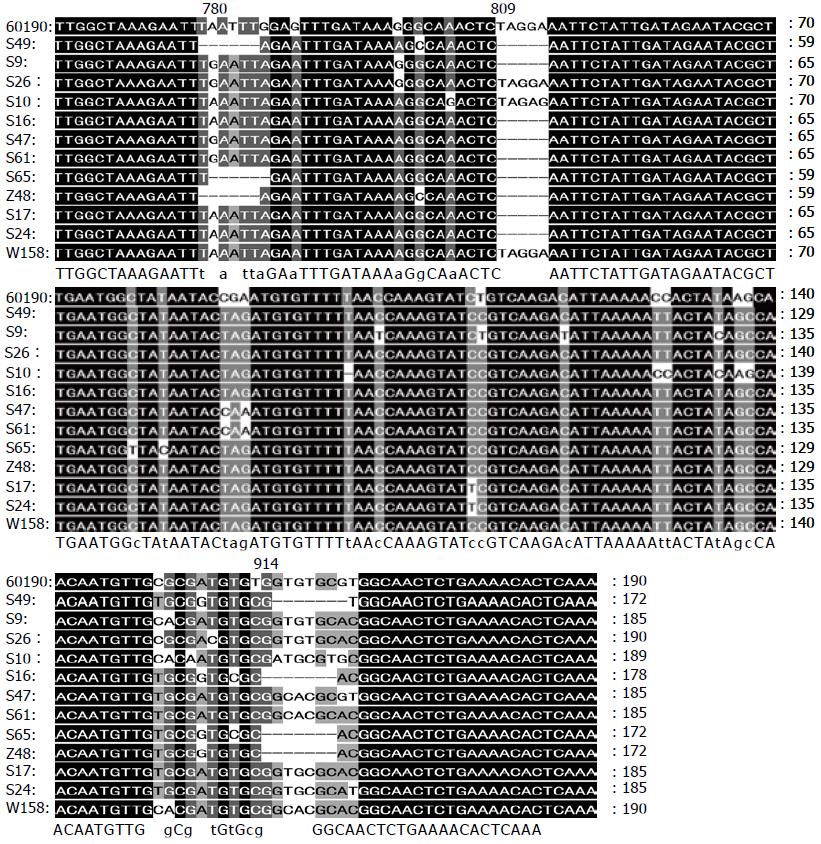
分培养出不同胃十二指肠疾病来源的H. pylori, 挑取iceA1进行TA克隆, 筛出12个阳性克隆(胃癌3株, 溃疡6株, 胃炎3株)的iceA1基因片段, 测序结果大小从428-472 bp不等(图3).
本研究扩增了镇江地区来源于不同疾病的(胃癌、溃疡和胃炎)共12株H. pylori的iceA1基因片段并与标准菌株60190比对(GenBank登录号: U43917), 比对结果显示镇江地区的H. pylori的iceA1基因中存在着框内缺失突变. 本研究发现了对应于标准菌株60190开放读码框第780个核苷酸位置缺失了6个bp(为了方便表述, 记作780del6)核苷酸序列(TAATTT), 第781个核苷酸位置缺失了6 bp核苷酸序列(AATTTG), 这些缺失突变的产生可能是因为同样的缺失机制(位于在同一个同向重复序列中), 其中780del6比781del6产生的频率更高(表2). 我们还发现了另外两个缺失位点, 即60190 iceA1开放读码框的第809个核苷酸位置(809del5)缺失了5 bp的核苷酸序列(TAGGA), 第914个核苷酸位置(914del7)缺失了7 bp的核苷酸序列, 这两个位置的缺失导致了移码突变, 从而导致iceA1转录的提前终止, 从而产生较小的蛋白结构. 这2处缺失突变在镇江地区H. pylori的iceA1中存在频率较高. 其中, 809del5的阳性率为83.3%(10/12), 914del7为33.3%(4/12). 而且有3株H. pylori同时存在3处缺失突变(780del6、809del5和914del7), 3株胃癌H. pylori只含有809del5缺失突变, 而溃疡和胃炎株则含有3个缺失突变. 对缺失片段周围的序列进行分析, 这些缺失序列的两端基本都与5-10 bp核苷酸的同向重复序列相连(表3).
| 菌株标号 | 疾病来源 | 框内缺失 | ||
| 780del6 | 809del5 | 914del7 | ||
| S49 | CSG | + | + | + |
| S9 | CAG | _ | + | _ |
| S26 | CAG | _ | _ | _ |
| S10 | DU | _ | _ | _ |
| S16 | DU | _ | + | + |
| S47 | DU | _ | + | _ |
| S61 | DU | _ | + | _ |
| S65 | DU | + | + | + |
| Z48 | DU | + | + | + |
| S17 | GC | _ | + | _ |
| S24 | GC | _ | + | _ |
| W158 | GC | _ | + | _ |
| 60190 | _ | _ | _ | |
| 缺失位置b | 菌株 | 核苷酸序列a | 缺失突变两端的重复序列 | 阳性缺失株数量(%) |
| 780del6 | 60190 | AGAATTTAATTTGGAGTTT | AGAATTT或AATTT | 3/12(25.0) |
| S26 | AGAATTTGAATTAGAATTT | |||
| S49 | AGAATT----------- AGAATTT | |||
| 809del5 | 60190 | AAACTCTAGGAAATTCTA | AAAYTCTA | 10/12(83.3) |
| S26 | AAACTCTAGGAAATTCTA | |||
| S49 | AAACTC----------AATTCTA | |||
| 914del7 | 60190 | GTGTGGTGTGCGTGGC | GTGCR或GTGTGS | 4/12(33.3) |
| S26 | GTGCGGTGTGCACGGC | |||
| S49 | GTGCG---------------TGGC |
DNA序列分析表明iceA1与nla ⅢR在结构上十分相似, 后者有一个690 bp的开放读码框架(open reading frame, ORF), 能编码230个氨基酸的蛋白产物nla ⅢR. Xu等[9]在对HpCH4株的研究中发现, 其iceA1有一全长为684 bp的ORF, 经IPTG诱导重组大肠杆菌后可成功表达, 其基因产物为228个氨基酸, 非常类似于nla ⅢR, 并且证实该基因产物具有nla ⅢR样限制性内切酶的功能, 表达该产物的H. pylori与临床疾病发生的严重程度关系密切[10-12], 但各家报道并不完全一致[5-7]. 并非所有iceA1皆可编nla ⅢR样蛋白, 其原因为不同地域的iceA1基因其ORF有着菌群特异性框移突变和无义突变, 故不能翻译出完整的nla ⅢR样蛋白. Donhaue等[4]认为, iceA1在大部分H. pylori菌株中的功能并非编码功能蛋白, 而是在转录水平调节其下游hpy IM基因的表达, 通过DNA甲基化水平的改变来调控相关毒性基因的表达, 从而间接参与H. pylori的致病作用.
iceA1在不同地区的及与疾病的关系并不相同. Kidd等[13]在对南非患者H. pylori的研究中发现, iceA1存在情况为: 胃炎24%, 胃溃疡40%, 胃癌67%, iceA1与胃癌有关. 在日本iceA1在胃癌中的比例61%略高于其他消化性溃疡(53%), 未见统计学差异, 但发现iceA1菌株与消化性溃疡及胃粘膜IL-8水平有显著相关性. 北美及荷兰研究的显示, iceA1比iceA2更普遍存在, 且iceA1阳性的H. pylori能刺激胃黏膜产生高水平的IL-8, 与iceA2相比, iceA1与十二指肠溃疡关系更为密切[14]. 东亚以iceA1基因型为主[15], 美国中东部地区则以iceA2为优势基因[16]. Wu等[17]研究台湾东部地区的iceA1与慢性胃炎有关. iceA基因的分布存在地域性差异, 在亚洲、非洲主要表达为iceA1基因, 在美洲大陆则以iceA2为主导, 而欧洲大陆的iceA基因则表现出与亚洲、美洲菌株的相似性, 在大多数地区的研究中未发现iceA1毒力菌株感染与临床疾病发生的因果联系[18], 因此与H. pylori的其他毒力基因相似, iceA1基因对于疾病的意义在不同地区是不一样的.
H. pylori可通过点突变、等位基因交换、基因重排及序列插入等使基因呈现出多样性, 从而导致H. pylori不同基因组间的差异极大[19]. 不同地区流行的H. pylori基因组结构明显不同, 而基因组多样性则影响细菌毒力因子的功能及抗原形成, 即在细菌进化过程中毒力因素也存在生物-地理变异[14]. 我们对镇江地区来源于不同疾病的(胃癌、溃疡和胃炎)共12株H. pylori的iceA1基因片段与标准菌株60190比对, 我们发现一个有趣的现象, 即与标准菌株60190相比, 镇江地区的H. pylori的iceA1基因中存在着3个框内缺失突变热点, 而且这些缺失突变存在的频率很高. 包含重复序列(同向重复序列或者反向重复序列)的局部DNA序列在复制或者重组的过程中可能由于错配的原因而导致序列缺失, 在本项研究中, 我们发现缺失序列末端都与同向重复序列相邻. 因此, 很有可能是同向重复序列导致了镇江地区的H. pylori的iceA1基因内部的这些缺失突变的产生. 我们还发现了3株同时存在3个缺失突变的H. pylori, 而且镇江地区的有高度同源的cagA, vacA和ureI等基因[20,21]. 因此, 我们推测"建立者效应"[5]可能也是导致镇江地区框内突变热点产生的原因之一.研究表明iceA1不同的等位基因型说明了[20,21]在不同地区基因库中的进化多样性,对于菌株型的追踪有一定帮助.iceA1序列的变异性可以作为不同地域菌株多样性的一个分子标记, 有可能成为分析[20,21]群体遗传学的有用工具.
iceA1基因是幽门螺杆菌(H. pylori)与胃上皮细胞接触后诱导表达的一种基因, 是一新的H. pylori毒力相关基因. iceA1基因与胃病之间的联系目前还有争议. 这可能是由于在全球不同地理区域内H. pylori基因型分布不均一引起的, 也反映了H. pylori菌株与其致病的临床表现的地理上差异.
崔莲花, 副教授, 青岛大学医学院公共卫生系
有的研究显示iceA1基因型与胃病之间无明显联系, 而也有一些研究表明, iceA1的基因型和H. pylori感染的临床预后之间有联系.
镇江地区H. pylori菌株的iceA1基因有较独特的结构特征, 存在3个框内缺失突变热点.我们发现缺失序列末端都与同向重复序列相邻, 有可能是同向重复序列导致了镇江地区的H. pylori iceA1基因内部缺失突变的产生.
iceA1序列的变异性可以作为不同地域菌株多样性的一个分子标记, 有可能成为分析H. pylori群体遗传学的有用工具.
建立者效应: 一些异常基因频率在小隔离群体中特别高, 可能是由于该群体中少数始祖所具有的基因, 由于遗传漂变而逐渐达到较高水平.
本研究选题科学, 创新性较好, 实用性较强, 具有一定的学术价值.
编辑: 李军亮 电编:鲁亚静
| 1. | Arfaoui D, Elloumi H, Ben Abdelaziz A. [Helicobacter pylori and gastric adenocarcinoma]. Tunis Med. 2009;87:231-236. [PubMed] |
| 2. | Peter S, Beglinger C. Helicobacter pylori and gastric cancer: the causal relationship. Digestion. 2007;75:25-35. [PubMed] [DOI] |
| 3. | Herrera V, Parsonnet J. Helicobacter pylori and gastric adenocarcinoma. Clin Microbiol Infect. 2009;15:971-976. [PubMed] [DOI] |
| 4. | Donahue JP, Peek RM, Van Doorn LJ, Thompson SA, Xu Q, Blaser MJ, Miller GG. Analysis of iceA1 transcription in Helicobacter pylori. Helicobacter. 2000;5:1-12. [PubMed] [DOI] |
| 5. | Ito Y, Azuma T, Ito S, Suto H, Miyaji H, Yamazaki Y, Kato T, Kohli Y, Keida Y, Kuriyama M. Sequence analysis and clinical significance of the iceA gene from Helicobacter pylori strains in Japan. J Clin Microbiol. 2000;38:483-488. [PubMed] |
| 6. | Ciftci IH, Uslan I, Dilek FH, Aşık G, Ozgür MA, Dilek ON. [Investigation of Helicobacter pylori iceA1 and iceA2 genes in patients with chronic gastritis and gastric cancer]. Mikrobiyol Bul. 2011;45:228-233. [PubMed] |
| 7. | Amjad N, Osman HA, Razak NA, Kassian J, Din J, bin Abdullah N. Clinical significance of Helicobacter pylori cagA and iceA genotype status. World J Gastroenterol. 2010;16:4443-4447. [PubMed] [DOI] |
| 8. | 陈 慧娟, 张 尤历, 唐 炜. 幽门螺杆菌iceA1, babA2基因型与临床消化疾病的关系. 江苏大学学报(医学版). 2009;19:173-175. |
| 9. | Xu Q, Morgan RD, Roberts RJ, Xu SY, van Doorn LJ, Donahue JP, Miller GG, Blaser MJ. Functional analysis of iceA1, a CATG-recognizing restriction endonuclease gene in Helicobacter pylori. Nucleic Acids Res. 2002;30:3839-3847. [PubMed] [DOI] |
| 10. | Peek RM, Thompson SA, Donahue JP, Tham KT, Atherton JC, Blaser MJ, Miller GG. Adherence to gastric epithelial cells induces expression of a Helicobacter pylori gene, iceA, that is associated with clinical outcome. Proc Assoc Am Physicians. 1998;110:531-544. [PubMed] |
| 11. | van Doorn LJ, Figueiredo C, Sanna R, Plaisier A, Schneeberger P, de Boer W, Quint W. Clinical relevance of the cagA, vacA, and iceA status of Helicobacter pylori. Gastroenterology. 1998;115:58-66. [PubMed] [DOI] |
| 12. | Sheu SM, Sheu BS, Yang HB, Li C, Chu TC, Wu JJ. Presence of iceA1 but not cagA, cagC, cagE, cagF, cagN, cagT, or orf13 genes of Helicobacter pylori is associated with more severe gastric inflammation in Taiwanese. J Formos Med Assoc. 2002;101:18-23. [PubMed] |
| 13. | Kidd M, Peek RM, Lastovica AJ, Israel DA, Kummer AF, Louw JA. Analysis of iceA genotypes in South African Helicobacter pylori strains and relationship to clinically significant disease. Gut. 2001;49:629-635. [PubMed] [DOI] |
| 14. | Kauser F, Hussain MA, Ahmed I, Srinivas S, Devi SM, Majeed AA, Rao KR, Khan AA, Sechi LA, Ahmed N. Comparative genomics of Helicobacter pylori isolates recovered from ulcer disease patients in England. BMC Microbiol. 2005;5:32. [PubMed] [DOI] |
| 15. | Yamaoka Y, Kodama T, Gutierrez O, Kim JG, Kashima K, Graham DY. Relationship between Helicobacter pylori iceA, cagA, and vacA status and clinical outcome: studies in four different countries. J Clin Microbiol. 1999;37:2274-2279. [PubMed] |
| 16. | Wong BC, Yin Y, Berg DE, Xia HH, Zhang JZ, Wang WH, Wong WM, Huang XR, Tang VS, Lam SK. Distribution of distinct vacA, cagA and iceA alleles in Helicobacter pylori in Hong Kong. Helicobacter. 2001;6:317-324. [PubMed] [DOI] |
| 17. | Wu CC, Chou PY, Hu CT, Liu ZC, Lin CY, Tseng YH, Lin NT. Clinical Relevance of the vacA, iceA, cagA, and flaA genes of Helicobacter pylori strains isolated in Eastern Taiwan. J Clin Microbiol. 2005;43:2913-2915. [PubMed] [DOI] |
| 19. | Falush D, Kraft C, Taylor NS, Correa P, Fox JG, Achtman M, Suerbaum S. Recombination and mutation during long-term gastric colonization by Helicobacter pylori: estimates of clock rates, recombination size, and minimal age. Proc Natl Acad Sci U S A. 2001;98:15056-15061. [PubMed] [DOI] |